Aminoácidos, péptidos e
proteínas
Algumas funções de proteínas:
Luciferina e
luciferase
Hemoglobina
Queratina
Funções das proteínas
1.Catálise enzimática
2.Transporte e armazenamento
3.Movimento coordenado
4.Suporte mecânico
5.Protecção imune
6.Geração e transmissão de impulsos nervosos
7.Controlo do crescimento e diferenciação
Estrutura geral de um
aminoácido como ião
dipolar
Convenção:
Código de 3 letras : as três primeiras letras do nome
Código com uma letra: Margaret Dayhoff (1925 –
1983) diminuir o tamanho dos ficheiros.
CHIMSV – 6 aa – a 1ª letra do nome (é única)
ex. cisteína, histidina
AGLPT – 5 aa – a 1ª letra (não é única) dos aa mais
comuns . Ex.leucina é mais comum que lisina.
RFYW – 4 aa – sugerido fonéticamente
Ex. aRginine, tYrosine
DNEQ -4 aa – letras sugeridas pelos nomes
Ex. asparDic, asparagiNe
K – lisina - sobravam poucas letras e era a letra mais
perto do L
As proteínas são
constituídas apenas
por L-aminoácidos
Relação estérea dos estereoisómeros da alanina com a
configuração absoluta do L- e D- gliceraldeído
Os aminoácidos podem ser classificados com base nas
propriedades do grupo R
Grupos R alifáticos, não polares
Glicina
Alanina
Prolina
Valina
Leucina
Isoleucina
Metionina
Grupos R aromáticos
Fenilalanina
Tirosina
Triptofano
Grupos R sem carga, polares
Serina
Treonina
Cisteína
Asparagina
Glutamina
Cisteína
Cistina
Cisteína
Grupos R carregados positivamente
Lisina
Arginina
Histidina
Grupos R carregados negativamente
Aspartato
Glutamato
Aminoácidos não comuns também têm papéis
importantes
Paredes celulares
de plantas
Colagéneo
4 - Hidroxiprolina
5 - Hidroxilisina
Colagéneo
6 – N - Metillisina
Miosina – proteína contráctil do músculo
γ - Carboxiglutamato
Protrombina – proteína envolvida na
coagulação do sangue
Outras proteínas que se ligam ao Ca
Alguns resíduos de aa podem ser modificados
transientemente (adição de grupos fosforil, metil,
acetil, etc) – modificações regulatórias
Ornitina e citrulina – aa envolvidos na
biossíntese de arginina e no ciclo da ureia.
Forma não iónica
Forma
zwiteriónica
O estado de ionização de um aminoácido depende do
pH
Os aminoácidos podem funcionar como ácidos ou
bases fracos
Titulação de
um
aminoácido
Efeito do ambiente químico sobre o pka
Curva de titulação do glutamato
Curva de titulação da histidina
Sumário:
•Os 20 aa têm um grupo α-carboxílico e um grupo
α-amino. O C α é assimétrico. Nas proteínas apenas
há L- aa.
• Outros aa menos comuns, existem quer nas
proteínas (após modificação) ou como metabolitos
livres
•Os aa são classificados em 5 grupos com base na
sua polaridade e carga (a pH 7) do seu grupo R.
•Os aa variam nas suas propriedades ácido-base e
têm curvas de titulação características.
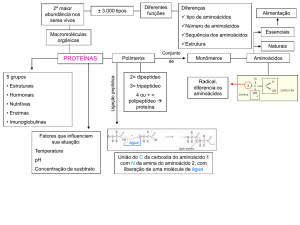
Péptidos e Proteínas
A ligação peptídica
Ligação peptídica
dipéptido
Ligação peptídica
dipéptido
tripéptido
tetrapéptido
pentapéptido
...
oligopéptido...polipéptido
MM‹10 000 Da
Um pentapéptido
Terminal N ou amina
Terminal C ou
carboxilo
Ser–Gly–Tyr–Ala–Leu serilgliciltirosilalanileucine
SGYAL
Estrutura
predominante do
tetrapéptido
alanilglutamilglicillisina
a pH7
Carga global 0
pI = 7
Ponto isoelétrico de
uma proteína – pH ao
qual a carga global é
zero
Péptidos com importância
biológica
adoçante
L-aspartil-L-fenilalanina metiléster
hormona
Piroglutamato
Prolilamida
Histidina
piroGlu-His-Pro-NH2
TRH - hormona de libertação da
tirotropina
hormona
Piroglutamato
Prolilamida
Histidina
piroGlu-His-Pro-NH2
TRH - hormona de libertação da
tirotropina
forma-se no hipotálamo a partir de
Glu-His-Pro
Outras hormonas que são péptidos
Oxitocina (9 res) – pituitária; estimula as
contracções uterinas
Bradicinina (9 res) – inibição da inflamação
de tecidos
Vasopressina – aumento a tensão arterial
Insulina (2 cadeias com 30 res) – reduz a
glucose no sangue promovendo a sua
entrada nas células.
Venenos
Amanitina
Amanitin is
the most
powerful
poison ...
Antibióticos
gramicidinas, polimixinas, bacitracinas,
glicopéptidos, etc
Neuropéptidos:
encefalinas, endorfinas...
Massa Molecular de algumas proteínas
MM
nº res
nºcadeias
Nº aproximado de aa de uma proteína = MM/110
Proteínas constituídas por
uma cadeia polipeptídica
Proteínas multiméricas – dois
ou mais polipéptidos iguais ou
diferentes, associados não
covalentemente.
As subunidades idênticas
chamam-se protómeros.
Ex. hemoglobina
Algumas proteínas têm duas
cadeias polipeptídicas ligadas
covalentemente
Ex.insulina bovina
Composição em
aminoácidos de
duas proteínas
Cálculo do nº de aminoácidos de uma proteína
Massa molecular média dos aminoácidos = 138
Média ponderada dos aminoácidos mais frequentes = 128
Eliminação de uma molécula de água por ligação peptídica
128 – 18 = 110
Massa molecular média de um resíduo de aa numa proteína
110
Massa molecular da proteína / 110 = nº de aa de uma
proteína
“Simples”
Proteínas
Conjugadas
Grupo prostético
Proteína conjugada
g.prostético
exemplo
Lipoproteína
Lípido
Β1-lipoproteína sangue
Glicoproteína
Fosfoproteína
Hemoproteína
glúcido
Imunoglobulina G
Caseína do leite
Hemoglobina
fosfato
Hemo (Fe
porfirinico)
Metaloproteína
Fe
Ferritina
“
“
“
Zn
Alcooldesidrogenase
Ca
Calmodulina
Mo
Dinitrogenase
Flavoproteína
FAD
Succinato desidrogenase
Proteínas em solução
Interacções electrostáticas
entre macroiões
Variação da solubilidade de proteínas com o pH e força
iónica
I = ½ ∑ MiZi 2
Salting in – Aumentando a concentração de sal, a
solubilidade de uma proteína aumenta.
Salting out – A concentrações salinas muito elevadas, a
solubilidade das proteínas diminui.
Células e tecidos usados em estudos bioquímicos
E.coli, S.cerevisiae, chlamydomonas, folha de espinafre, fígado de
rato, músculo esquelético
Fonte homogénea e grande quantidade: culturas de
microrganismos, tecidos, mutantes genéticos.
Alguns materiais são ricos num componente específico:
Músculo esquelético – actina e miosina
Células do pâncreas – RER
Células espermáticas – DNA
Células do fígado – enzimas das vias biosintéticas
Folhas de espinafre, eritrócitos, vírus, …
As proteínas podem ser separadas e purificadas
Tecido ou células
↓ lise
Extracto bruto
↓Fraccionamento subcelular
Extracto
↓
Fraccionamento de proteínas
↓
Proteína em estudo
As proteínas podem ser separadas e purificadas
Solubilidade – “Salting out”
Tamanho – Diálise, cromatografia de exclusão molecular
Carga – Cromatografia de troca iónica
Afinidade – Cromatografia de afinidade
Diálise
Cromatografia em coluna
reservatório
Fase móvel (amostra
de proteínas)
Fase estacionária
(matriz porosa)
suporte
poroso
efluente
proteínas
Carga + +
Carga +
CargaCarga- -
Partículas poliméricas
com grupos funcionais
negativos
As proteínas movem-se através da
coluna a velocidades dependentes da
carga e do pH usado
Cromatografia de troca
iónica (catiónica)
Explora
diferenças de
sinal e
intensidade
de carga
eléctrica de
proteínas a
um
determinado
pH
Questão:
Um biólogo pretende separar dois péptidos por
cromatografia de troca iónica. Ao pH da fase
móvel a usar na coluna, um péptido (A) tem uma
carga global de -3, devido à presença de mais
resíduos Glu e Asp do que Arg, Lys e His. O
péptido B tem uma carga global de +1.
• Qual dos péptidos seria primeiro eluído de uma
resina de troca catiónica?
• Qual seria eluído primeiro de uma resina de
troca aniónica?
Cromatografia por
exclusão ou filtração
gel
Partículas
poliméricas
porosas
As moléculas de
proteína separam-se por
tamanho; as maiores
saiem (eluem) primeiro
Cromatografia de
afinidade
Proteína X
ligando
Amostra inicial
(mistura de
proteínas)
Solução
do ligando
Mistura de proteínas é adicionada
à coluna contendo um polímero
com um ligando específico para a
proteína X
Proteínas diferentes de X são
retiradas por lavagem da
coluna
Proteina X é eluida
por solução de
ligando
HPLC- high performance liquid cromatography
Exemplo de tabela de purificação do enzima X
Passo
Vol.
fracção(ml)
Prot.
total(mg)
Activ.
Activ.
espec.
1 400
10 000
100 000
10
2. prec. c/ sulf. am.
280
3000
96 000
32
3. crom. troca ion.
90
400
80 000
200
4. filtração gel
80
100
60 000
600
5. crom. afinidade
6
3
45 000
15 000
1. extracto cru
Actividade vs actividade
específica
Os dois copos contêm a mesma actividade da proteína. O segundo
tem uma actividade específica mais elevada
electroforese
Amostras
diferentes
em cada
poço
poço
direcção
da
migração
Gel de
poliacrilamida
Purificação de RNA polimerase
de E.coli
1 2 3 4 5 6
Determinação do
peso molecular de
uma proteína
Isoelectrofocagem: separa proteínas
conforme o pI
Pontos isoeléctricos de algumas
proteínas
Electroforese a duas
dimensões:
combinação de
isoelectrofocagem e
electroforese em
acrilamida
Níveis de estrutura na proteína
primário
quaternário
secundário
terciário
Estrutura covalente das proteínas
A purificação de uma proteína é o principio…
A função de uma proteína depende da sua
sequência de aminoácidos
• Proteínas com diferentes funções têm diferentes
composições em aa.
• Doenças genéticas humanas envolvendo
alterações em aa, têm na origem proteínas com
funções alteradas.
• Proteínas com funções semelhantes em espécies
diferentes, têm sequências de aa semelhantes (ex.
a ubiquitina). Polimorfismo.
A sequência de aminoácidos de uma proteína
é invariante?
20-30% das proteínas no homem são
polimórficas (variantes)
Como determinar a sequência ?
Sequência de aminoácidos
da insulina bovina – a 1ª
proteína a ser sequenciada
Determinação da sequência em aminoácidos de
uma proteína
(estrutura primária)
1.Determinação da composição em aminoácidos:
Proteína completamente hidrolisada (110 0 C, 24h
em HCl 6N).
Aa separados e quantificados por cromatografia de
troca iónica
2. Identificação do aminoácido N – terminal
Método de Sanger (FDNB)
Degradação de Edman (fenilisotiocianato)
polipéptido
Reagente
de Sanger
Mistura dos restantes aa
(inutil para prosseguir)
2,4dinitrofenil
deriv. do
polipép.
2,4-dinitrofenil
deriv. do res N
terminal
Sequenciação de um
polipéptido
polipéptido
Reagente
de Sanger
Mistura dos restantes aa
(inutil para prosseguir)
2,4dinitrofenil
deriv. do
polipép.
2,4-dinitrofenil
deriv. do res N
terminal
Apenas permite identificar aa do terminal
N
Sequenciação de um
polipéptido
polipéptido
Reagente
de Sanger
Mistura dos restantes aa
(inutil para prosseguir)
2,4dinitrofenil
deriv. do
polipép.
Reagente
de Edman
2,4-dinitrofenil
deriv. do res N
terminal
PTIC
feniltio
isocia
nato
Identificação do aa
N terminal
aducto
PTC
Polipéptido com n-1
resíduos que reage
de novo com PTIC
cloreto de dansilo
cloreto de dabsilo
3. Cisão das pontes dissulfureto
As ligações dissulfureto interferem com o procedimento
de sequenciação.
Oxidação pelo ácido perfórmico
Redução pelo ditiotreitol(DTT) ou β-mercaptoetanol
lig.persulfureto
Ditiotreitol
(DTT)
oxidação ac.
perfórmico
2 res. ácido
cisteico
Redução por
DTT
acetilação por
iodoacetato
cisteínas
acetiladas
4. Fragmentação dos péptidos
A cadeia polipéptica é fragmentada em
segmentos mais pequenos que são depois
separados por métodos cromatográficos ou
electroforéticos
Especificidade de alguns métodos comuns de fragmentação de cadeias
polipeptídicas
reagente (fonte)
ponto de clivagem
Tripsina (pâncreas bovino)
Submaxillarus protease (glândula submaxilar)
Quimotripsina (pâncreas bovino)
Protease V8 (Staphylococcus aureus)
Asp-N- protease (Psedomonas fragi)
Pepsina (estômago de porco)
Endoprotease Lys C (Lysobacter enzymogenes)
CNBr
5. Sequenciação dos péptidos
Cada péptido é sequenciado pelo procedimento
de Edman.
6. Ordenamento dos fragmentos do polipéptido
inicial
Utilizam-se outros enzimas ou químicos para
reconstruir a sequência da proteína original
Sequenciação de uma proteína
7. Localização das ligações dissulfureto
A proteína é hidrolisada com um enzima (ou químico)
mas sem antes se terem cindido as ligações
dissulfureto.
Os péptidos são separados por electroforese e
comparados com o conjunto de péptidos gerados
pelo mesmo enzima (químico).
Por cada ligação dissulfureto não aparecerão dois
péptidos e será visível um péptido maior.
A sequência de aminoácidos pode ser deduzida por
outros métodos
A sequência de resíduos de aminoácidos de
uma proteína fornece informações sobre:
• Estrutura tridimensional
•Função
•Localização celular
•Evolução
Níveis de estrutura na proteína
primário
quaternário
secundário
terciário