64º Congresso Nacional de Botânica
Belo Horizonte, 10-15 de Novembro de 2013
ESTUDO DO “COMPLEXO Kielmeyera coriacea” (CALOPHYLLACEAE)
ATRAVÉS DE MARCADORES MICROSSATÉLITES
1*
1
2
1
3
Rafaela J. Trad , Maria Beatriz de S. Cortez , Danilo A. Sforça , Anete P. Souza , Maria I. Zucchi , Volker
1
Bittrich & Maria do Carmo E. Amaral
1
2
3
Departamento de Biologia Vegetal – IB/Unicamp; Departamento de Genética – IB/Unicamp; Departamento de
Genética –ESALQ/USP; *[email protected]
Introdução
Tradicionalmente, a circunscrição de espécies é baseada
em caracteres morfológicos, porém isto pode resultar em
táxons que não refletem as espécies reais. Há algum
tempo, sugere-se o uso de marcadores moleculares para
testar a delimitação de espécies, assim como para
estudar processos de hibridização. A descoberta de locos
amplamente variáveis como os microssatélites (SSRs)
permitiu que fosse alcançado o poder estatístico
necessário à realização desse tipo de estudo, o que é,
particularmente, importante no caso de complexos de
espécies. No caso do “complexo K. coriacea”, estudos
apontam a formação de híbridos entre as espécies [1, 2].
Kielmeyera Mart. & Zucc., com cerca de 50 espécies,
possui três conhecidos complexos de espécies, entre
eles: o “complexo K. coriacea”, composto pelas espécies
K. coriacea Mart. & Zucc. (com duas subespécies e sete
variedades) e K. grandiflora (Wawra) Saddi [3].
Através do uso de marcadores microssatélites, o presente
estudo testou a validade das categorias taxonômicas do
“complexo K. coriacea”.
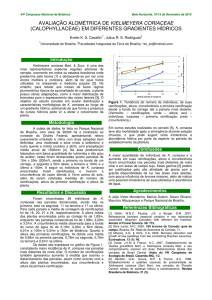
Conforme a Figura 1 é possível identificar dois “pools”
gênicos, representados em verde e vermelho.
Considerando os resultados, é possível notar que: a
ocorrência de híbridos só é indicada para o estado de
Goiás (geograficamente central); a análise não foi capaz
de identificar as espécies do complexo, nem de indicar
uma estruturação genética relacionada às espécies. A
análise de variância molecular indicou que uma
porcentagem muito baixa da diversidade está entre as
espécies, sugerindo que a classificação atual não
apresenta boa circunscrição de táxons.
Figura 1. Agrupamento realizado a partir dos resultados
do Structure para k=2 dividido por estados e populações.
Cada coluna representa um indivíduo e cada cor um
“pool” gênico. BA: 1-4, DF: 5-8, GO: 9-13, MG: 14-18, T:
19-22, SP: 23 e TO: 24-27.
Metodologia
Conclusões
Foram coletadas folhas de 27 populações com 20-30
indivíduos nos estados: BA, GO, MG, MT, SP, TO e no
DF de modo que K. coriacea subsp. coriacea, K. coriacea
subsp. tomentosa e K. grandiflora tivessem, ao menos,
cinco populações amostradas.
As folhas foram liofilizadas e moídas. O DNA foi extraído,
quantificado em gel de agarose 1% e amplificado para 12
locos de microssatélites. Os produtos de PCR foram
quantificados em gel de agarose 3% e visualizados em
geis de poliacrilamida 6% corados com prata [4].
Os dados foram analisados nos softwares Structure
versão 2.3.3 e Arlequin.
Com base no presente trabalho, é possível concluir que a
circunscrição das espécies pertencentes ao “complexo K.
coriacea” deve ser reavaliada, assim como as categorias
infraespecíficas.
Resultados e Discussão
A análise genética foi feita através da genotipagem onde
foram geradas duas planilhas de dados: uma somente
com presença/ausência, utilizada no programa Arlequin
para realizar a analise de variância molecular (AMOVA), e
uma com os tamanhos das marcas encontradas
(codominante) utilizada no programa Structure para
realização de testes de atribuição de indivíduos a grupos
pela estatística bayesiana. A partir dos resultados
gerados, o agrupamento que melhor explica os dados foi
obtido através do calculo do delta K [5], cujo valor foi 2.
Agradecimentos
O presente projeto foi apoiado pela CAPES, pelo CNPq e
pelo FAEPEX/UNICAMP.
Referências Bibliográficas
[1] Trad, R.J. 2012. Estudos taxonômicos e biossistemáticos
no “complexo Kielmeyera coriacea” Mart. & Zucc.
(Calophyllaceae). Dissertação de Mestrado, Universidade
Estadual de Campinas.
[2] Caddah, M.K., Mayer, J.L.S., Amaral, M.C.E. & Bittrich, V.
2012. Species limits in the Kielmeyera coriacea complex
(Calophyllaceae) – a multidisciplinary approach. Botanical
Journal of the Linnean Society 168: 101-115.
[3] Saddi, N. 1982. A taxonomic revision of the genus
Kielmeyera Mart. (Guttiferae). PhD thesis, University of
Reading.
[4] Creste, S., Tulmann Neto, A. & Figueira, A. 2001. Detection of
single sequence repeat polymorphisms in denaturating
polyacrylamide sequencing gels by silver staining. Plant
Molecular Biology Reporter 19: 299–306.
[5] Evanno, G., Regnaut, S. & Goudet, J. 2005. Detecting the
number of clusters of individuals using the software
STRUCTURE: a simulation Study. Molecular Ecology 14: 26112620.