LIGAÇÕES QUÍMICAS
REVISÃO
Ligações químicas e forças atrativas
1- Ligações covalentes – Ligação química entre dois átomos na qual
elétrons são compartilhados
Ligação Forte
Energia de ligação é a energia necessária para romper a ligação
(expressa em kcal/mol ou KJ/mol). 100 – 200 kcal/mol
2- Ligações não covalentes (ligações fracas):
Tipos:
2.1. Ligações iônicas
2.2. Ligações ou Pontes de Hidrogênio
2.3. Forças de Van Der Waals
2.4. Atrações hidrofóbicas
Muito importantes na manutenção de estruturas biológicas
2.1. Ligações iônicas – formam-se quando há uma
transferência completa de elétrons de um átomo para outro,
formando-se dois íons, um com carga positiva e o outro com
carga negativa.
Ocorre atração eletrostática entre os íons.
Ex: Na+Cl-;
Nas proteínas: grupos carboxila (COO-) e grupos amino (NH3+)
do grupo R dos aminoácidos.
Energia de ligação 4 -7 kcal/mol
2.2. Ligações ou Pontes de Hidrogênio - atração eletrostática entre
um átomo eletronegativo (O ou N) e um átomo de hidrogênio que está
ligado covalentemente a um outro átomo eletronegativo.
H2O
Aminoácidos na proteína
Energia de ligação 1-7 kcal/mol
2.3. Forças de van der Waals – atração eletrostática entre átomos ou
moléculas neutras. Surgem por flutuações na distribuição de cargas.
Um lado tem carga mais negativa e o outro lado mais positivo
Energia de ligação
0.5 -1 kcal/mol
2.4. Atrações Hidrofóbicas – Associação entre grupos apolares.
Surgem quando dois grupos não polares se aproximam, reduzindo a
superfície exposta à água.
Aminoácidos na proteína
Energia de ligação variável
Ligações químicas (Resumo)
Energia de ligação (expressa em kcal/mol ou KJ/mol*) é a energia
necessária para romper a ligação.
Esta energia é igual à energia liberada quando a ligação se forma.
Ligação
Energia de Ligação (Kcal/mol)
Covalente
100 – 200
Iônica
4 -7
de Hidrogênio
1 -7
Van der Waals
0.5 -1
Atrações hidrofóbicas
variável
* 1 Caloria = 4,184 Joules
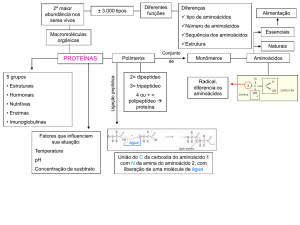
PROTEÍNAS
Definição
Proteínas são polímeros de aminoácidos
Os aminoácidos estão unidos entre si por ligações
peptídicas
Proteínas naturais são polímeros de L-aminoácidos
Nomenclatura
Peptídio – poucos aminoácidos (2 a 30) (resíduos)
Polipeptídio - de 30 a 50 aminoácidos
Proteínas - > 50 aminoácidos
Ligação peptídica
É uma ligação carbono-nitrogênio que se forma pela reação entre o
grupo carboxila (COO--) de um aminoácido e o grupo amino (NH3 +)
do aminoácido seguinte.
Nota: grupos carboxila e amino ligados ao Carbono alfa.
O comprimento de uma ligação peptídica é 1,33 Å
Formação da ligação peptídica
A reação acima JAMAIS ocorre espontaneamente num ser vivo. A ligação
peptídica é feita no aparato de síntese proteica e ocorre nos ribossomos.
Nesta reação os aminoácidos sempre estão ligados a um tRNA. A energia
para a formação da ligação peptídica provem da hidrólise do ATP e GTP.
(ver nas aulas do módulo de Biologia Molecular).
Quebra da ligação peptídica
Hidrólise química: Aquecimento prolongado em presença de ácido forte.
Ex. 6M HCl a 110 oC, por 18 a 24 hs, no vácuo.
Hidrólise enzimática: Incubação com enzimas proteolíticas. Ex: pepsina,
tripsina, quimotripsina. Neste caso pode ser a 37 oC por tempo curto.
Características da ligação peptídica
(ligação amida, covalente)
AA1
O
C
Os 4 átomos ficam no
mesmo plano
AA2
N
H
CARBOXILA
AMINA
Cadeia polipeptídica
a1
a2
a3
ax
ay
az
Amino terminal
1. Sentido da Cadeia Polipeptídica: grupo amino-terminal livre do aminoácido
1 e grupo carboxi-terminal livre do aminoácido z.
2. Grupos R dos aminoácidos estão livres.
Carboxi
terminal
Estrutura Primária (Estrutura linear)
Estrutura Primária = sequência de aminoácidos da proteína. Esta
estrutura é mantida pela ligação peptídica (covalente) entre os resíduos
de aminoácidos. A estrutura é determinada pelo código genético no DNA.
Anotação: sempre da extremidade Amino terminal para a Carboxi terminal
Ala – Val – Lys
diferente de
Lys – Val – Ala
NOTA:
A estrutura primária da proteína determina sua estrutura
tridimensional. A estrutura tridimensional determina sua FUNÇÃO
As proteínas podem ser classificadas quanto à sua forma
em:
•proteínas globulares: a cadeia polipeptídica “enrola-se” sobre si mesma
criando uma estrutura compacta mais ou menos esférica
•proteínas fibrosas: as cadeias se enrolam entre si criando uma estrutura
tipo fibra.
proteínas globulares
proteínas fibrosas
Representação da estrutura de algumas Proteínas Globulares
Níveis de organização estrutural de uma proteína
Toda a proteína possui uma:
Estrutura primária
Regiões de estrutura secundária
Estrutura Terciária
Algumas proteínas possuem estrutura quaternária
Níveis de organização estrutural de uma
proteína
Nota: nem todas
as proteínas
Estrutura Secundária
Mantida por ligações fracas (Pontes de hidrogênio) entre átomos da
cadeia principal (C e N da ligação peptídica)
Estrutura Secundária
Dois tipos principais:
- alfa-hélice
- folha beta pregueada
Presente em segmentos da proteína
Folha beta pregueada
Alfa-hélice
Alfa-hélice
O
C
N
H
R
O
C
C
H
O
C
N
H
N
H
R
O
C
C
H
N
H
Pontes de H = 1 a 7 kcal/Mol
Estrutura Secundária
Proteína com elevado conteúdo de folhas beta
pregueadas
Regiões sem estrutura definida
Estrutura terciária
Conformação tridimensional
final da proteína
Dobramento final da cadeia
polipeptídica por interações com
regiões com estrutura definida
(alfa-hélice e folha beta
pregueada) e regiões sem
estrutura definida.
Estrutura terciária mantida por:
Interações fracas e fortes (pontes S-S) entre átomos da cadeia
lateral (grupo R)
Estrutura terciária
Interações fracas (não covalentes) entre átomos da cadeia lateral (grupo R)
de um aminoácido com átomos da cadeia lateral de outro aminoácido.
Lembrar
Grupos R
Interações na estrutura terciária
Pontes de H – entre AA polares com e sem
carga
Interações hidrofóbicas entre AA apolares
Interações iônicas (eletrostáticas) entre AA
polares
Ponte dissulfeto (entre cisteínas) –
Ligação covalente
Ligações covalentes
Pontes dissulfeto (S-S) formam-se entre os grupos SH de duas cisteínas
O
Cisteína
O
C
+H
C
3N
CH2
C
S
H
CH2
2
H
C
C
N
C
O
SH
Cistina
O
C
H
3N
S
+H
H
+H
O
O
3
Estrutura Quaternária
Muitas proteínas são constituídas por mais de uma cadeia
polipeptídica. A estrutura quaternária descreve a forma com que
as diferentes subunidades se agrupam e se ajustam para formar a
estrutura total da proteína.
Forças que mantém a estrutura quaternária: Interações fracas e pontes S-S
Subunidade A
Subunidade B
Estrutura
quaternária da
Proteína
Exemplo: HEMOGLOBINA
é composta por quatro subunidades
2 subunidades alfa e 2 subunidades beta
Ligação do Oxigênio ao grupo Heme da Hemoglobina
RESUMO
Níveis de organização estrutural das proteínas e
tipo de ligações
Estrutura primária
Ligações covalentes (= ligação peptídica) entre os resíduos de
aminoácidos da proteína
Estrutura secundária
Ligações fracas (Pontes de hidrogênio) entre átomos da cadeia principal,
C=O e H-N
Estrutura terciária
Interações fracas (ligações iônicas, pontes de Hidrogênio, ligações de
van der Waals, atrações hidrofóbicas) entre átomos da cadeia lateral
(grupo R) e interações fortes (covalentes) entre os grupos SH de cisteínas
(pontes dissulfeto)
Estrutura quaternária
Apenas em proteínas que contém mais de uma subunidade.
Nota: Cada subunidade (ou monômero) é uma proteína.
As subunidades interagem por ligações fracas (ligações iônicas,
pontes de Hidrogênio, ligações de van der Waals, atrações
hidrofóbicas) entre átomos da cadeia lateral (grupo R) de cada
subunidade e interações fortes (covalentes) entre os grupos SH de
cisteínas (pontes dissulfeto) das subunidades.
Executar o software de estrutura de proteinas
http://www.iq.usp.br/bayardo/softwares/proteina/menu/index.html
Resumo dos níveis de organização
estrutural de uma proteína
Nota: nem todas
as proteínas
Exemplo: Proteína Actina do músculo
Proteína globular
374 aminoácidos
AA N-terminal – Ac. Aspártico (D)
AA C- terminal – Fenilalanina (F)
Estrutura Primária - sequência linear dos aminoácidos
DDDIAALVVDNGSGMCKAGFAGDDAPRAVFPSIVGRPRHQGVMVGMGQKD
SYVGDEAQSKRGILTLKYPIEHGIVTNWDDMEKIWHHTFYNELRVAPEEHP
VLLTEAPLNPKANREKMTQIMFETFNTPAMYVAIQAVLSLYASGRTTGIVM
DSGDGVTHTVPIYEGYALPHAILRLDLAGRDLTDYLMKILTERGYSFTTTA
EREIVRDIKEKLCYVALDFEQEMATAASSSSLEKSYELPDGQVITIGNERF
RCPEALFQPSFLGMESCGIHETTFNSIMKCDVDIRKDLYANTVLSGGTTMY
PGIADRMQKEITALAPSTMKIKIIAPPERKYSVWIGGSILASLSTFQQMWI
SKQEYDESGPSIVHRKCF
Estrutura Tridimensional da Actina
ATP
ATP
Região com Alfa-hélice
As moléculas de Actina se polimerizam em forma
helicoidal formando um filamento chamado de Actina F
Ao filamento de Actina associam-se outras proteínas: Tropomiosina e
Troponina
Mutações no gene da actina
Patologias: nemaline myopathy, myotubular myopathy, central core myopathy,
congenital fiber type disproportion, and multicore myopathy
DESNATURAÇÃO DE PROTEÍNAS
Desnaturação é a perda da estrutura tridimensional (secundária, terciária
e quaternária) das proteínas. Com a desnaturação as proteínas perdem
sua função. Não há perda da estrutura primária
DESNATURAÇÃO DE PROTEÍNAS
AGENTES
Ocorre em presença de temperaturas elevadas, uréia, ácidos fracos,
álcalis, sais concentrados e álcool
Esses agentes rompem as ligações fracas que mantém a estrutura
tridimensional (secundária, terciária e quaternária).
DESNATURAÇÃO DE PROTEÍNAS (cont.)
A estrutura primária da proteína não é rompida com estes agentes pois é
mantida por ligações covalentes que são ligações fortes.
A estrutura primária da proteína é rompida por situações drásticas (HCl 6N,
100o C) ou pela ação de proteases.
Christian ANFINSEN – Prêmio Nobel em 1972
A sequência dos aminoácidos de uma
proteína determina a conformação da mesma.
EXPERIMENTO:
A Ribonuclease (RNase) é uma proteína que tem 124
aminoácidos.
Apresenta quatro pontes S-S entre 8 Cisteínas
Denaturação da RNase
1. Redução das pontes S-S com beta mercaptoetanol
2. Desnaturação da proteína com 8 M Uréia
Enzima inativa
ANFINSEN
- Remoção da uréia e Oxidação das cisteínas para formar pontes S-S
A Enzima readquiriu sua conformação nativa e recuperou mais de 90%
de sua atividade
Nota: Se as 8 cisteínas se combinassem aleatoriamente 2 a 2, haveria 28 = 256
possibilidades de formar diferentes pontes dissulfeto, mas apenas o arranjo original
de pontes S-S se forma.
Conclusão: A sequência dos aminoácidos de uma proteína (sequência
primária) determina a conformação da mesma.
PROTEÍNAS FIBROSAS
Possuem forma alongada.
A maioria das proteínas fibrosas têm um papel estrutural em células animais
e nos tecidos.
Tropomiosina – Presente na célula muscular
Colágeno – Proteína mais abundante em vertebrados (matriz do osso;
material dos tendões)
Queratina – Cabelo, unhas e pele
Elastina – Fibra elástica em ligamentos e vasos sanguíneos arteriais
Fibroína – Feita pelo bicho da seda e aranhas
ESTRUTURA DO COLÁGENO
O colágeno é um material extracelular
fabricado pelos fibroblastos.
É uma proteína fibrosa que resulta
relativamente insolúvel em água.
O colágeno é formado por três cadeias
polipeptídicas entrelaçadas, formando uma
tripla hélice.