Universidade Federal de Santa Catarina - UFSC
Departamento de Bioquímica - CCB
Disciplina: BQA5125 - Bioquímica para Engenharia Sanitária e Ambiental
Bolsistas REUNI: Priscila G. A. Martins e Tiago Bortolotto
Lista de Exercícios de Bioquímica
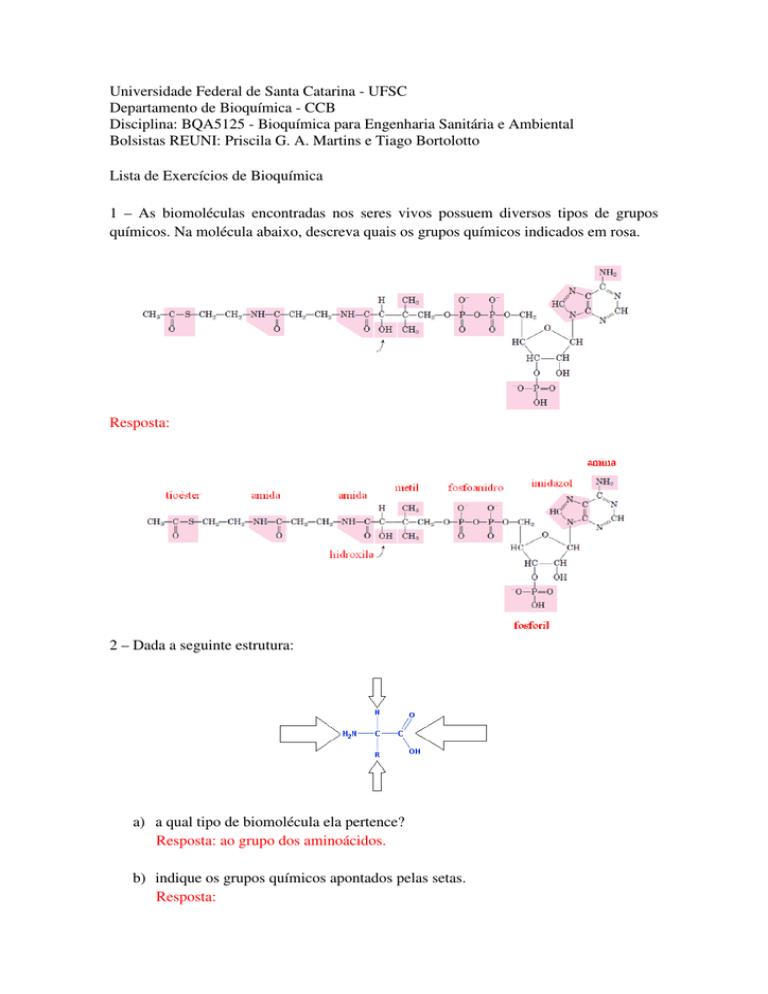
1 – As biomoléculas encontradas nos seres vivos possuem diversos tipos de grupos
químicos. Na molécula abaixo, descreva quais os grupos químicos indicados em rosa.
Resposta:
2 – Dada a seguinte estrutura:
a) a qual tipo de biomolécula ela pertence?
Resposta: ao grupo dos aminoácidos.
b) indique os grupos químicos apontados pelas setas.
Resposta:
c) há pelo menos dois grupos químicos que são ionizáveis em solução. Quais são
eles e quais os respectivos valores de pKa?
Resposta: são os grupos amina e carboxila. O pKa do grupo carboxila varia entre
1,8 e 2,4 enquanto o pKa do grupo amina varia entre 8,8 a 10,9.
d) levando em consideração os valores de pKa desses dois grupos, como estes
devem estar ionizados em pH 7?
Resposta: em pH 7, a maior parte dos aminoácidos deve estar com o grupo
amina protonado (NH3+) pois seu pKa é maior que o pH do meio. Já o grupo
carboxila tende a estar desprotonado (COO-), pois seu pKa é muito menor que o
pH do meio.
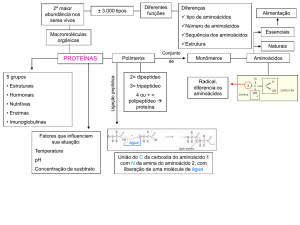
3 – Os aminoácidos são considerados moléculas dipolares ou zwitteriônicas. Qual é a
principal característica deste grupo de moléculas? O que faz dos aminoácidos moléculas
dipolares?
Resposta: moléculas dipolares ou zwitterriônicas, são moléculas que possuem dois
grupos polares em sua estrutura. Em solução, os grupos amina e carboxila tendem a
ficar ionizados e com isso tornam-se polarizados (amina carregada positivamente e
carboxila carregada negativamente), sendo esta a origem do caráter zwitteriônico dos
aminoácidos.
4 – Todos os aminoácidos compartilham de grupos químicos comuns, com exceção de
um. Quais são os grupos comuns a todos os aminoácidos e qual o grupo varia para cada
aminoácido?
Resposta: os grupos em comum a todos os aminoácidos são: amina, carboxila e o
hidrogênio. O grupo variável é a cadeia lateral.
5 – Os 20 aminoácidos que são encontrados na maioria das proteínas (eventualmente
chamados de “aminoácidos comuns”) diferenciam-se entre si pelas diferentes estruturas
da cadeia lateral. As cadeias laterais conferem aos aminoácidos diferentes propriedades
químicas que são utilizadas para separar essas moléculas em grupos (ou classes). Quais
são esses grupos e quais os aminoácidos que os compõem.
Resposta:
6 – Veja a figura abaixo:
a) na figura acima vemos a oxidação (perda de elétrons) de duas moléculas de um
aminoácido formando um “dímero”. Qual é o aminoácido envolvido nessa
reação e qual o nome dado ao “dímero” desse aminoácido?
Resposta: o aminoácido oxidado é a cisteína e o dímero é chamado de cistina.
b) como se chama a ligação covalente (em amarelo) da molécula dimerizada?
Resposta: ligação dissulfeto.
c) esse dímero pode ser novamente convertido ao aminoácido original. Com qual
reação química isso é possível?
Resposta: sim, por meio de uma reação de redução.
7 – Além dos 20 aminoácidos comuns, há outros “aminoácidos não-comuns” que são
encontrados nas proteínas. Com base nisso, descreva:
a) como esses aminoácidos são incorporados nas proteínas;
Resposta: não são incorporados pela via da síntese protéica comum, nos
ribossomos e sim são modificados após já estarem presentes nas proteínas.
b) cite um exemplo;
Resposta: dehidro-alanina
c) a falta de um aminoácido não-comum no colágeno gera uma grave doença
humana. Que aminoácido não-comum é esse e qual é a doença?
Resposta: o aminoácido é a hidroxi-prolina e a doença é o escorbuto.
8 – A ligação entre dois aminoácidos é chamada de ligação peptídica. Faça um esquema
de como ocorre a ligação peptídica entre os aminoácidos glicina e serina e dê o nome do
dipeptídeo gerado.
Resposta:
9 – O dipeptídeo seril-histidina é exatamente igual o dipeptídeo histidil-serina no que
diz respeito a sua estrutura? Prove seu ponto de vista desenhando os dipeptídeos abaixo.
Resposta: as estruturas são diferentes, pois no dipeptídeo histidil-serina o aminácido
histidina é o N-terminal e a serina é C-terminal. No dipeptídeo, seril-histidina ocorre o
inverso. Portanto, os dipeptídeos são estruturalmente diferentes e não apenas isômeros.
10 – Quais são os quatro níveis de organização estrutural das proteínas e o que
caracteriza cada nível?
Resposta: Os quatro níveis de organização são: primário, secundário, terciário e
quaternário.
A estrutura primária de uma proteína compreende o número, espécie e a
sequência dos aminoácidos unidos por ligações peptídicas e pontes dissulfetos. É
especificada por informação genética.
A estrutura secundária abrange os arranjos regulares e recorrentes da cadeia
polipeptídica (α- hélice e folha β pregueada).
A estrutura terciária descreve o dobramento final da cadeia polipeptídica por
interação de regiões com estrutura regular (α- hélice e folha β pregueada) ou de regiões
sem estrutura definida.
A estrutura quaternária é o arranjo espacial de duas ou mais cadeias
polipeptídicas (ou subunidades protéicas) com a formação de complexos
tridimensionais.
11- Quais são os tipos de interações intramoleculares (dentro da própria proteína)
responsáveis em manter a estrutura terciária de uma proteína?
Resposta: Interações não covalentes: Ligação hidrogênio (antigamente conhecida como
ponte de hidrogênio), interações hidrofóbicas e ligações eletrostáticas ou iônicas.
Interação covalente: ponte dissulfeto.
12 – O que é uma proteína desnaturada? Cite 3 fatores que podem ocasionar a
desnaturação de uma proteína.
Resposta: É uma proteína que teve sua estrutura espacial afetada a ponto de ocasionar a
perda de sua função biológica. A proteína dita desnaturada teve sua conformação nativa
destruída devido à quebra de ligações não covalentes e o resultado é uma cadeia
polipeptídica distendida. Os fatores que podem ocasionar a desnaturação de uma
proteína são: elevação de temperatura, alteração de pH, radiação ultravioleta, exposição
a detergentes, agitação vigorosa até formação de espuma, tratamento químico (solventes
orgânicos como álcool e acetona) e uréia.
13 – O que são os efeitos “salting in” e “salting out” e como eles interferem na
solubilidade de proteínas?
Resposta: O efeito “salting in” é o aumento da solubilidade de proteínas devido ao
acréscimo de baixas concentrações de sais em solução. Os íons salinos interagem com
as cargas iônicas das proteínas aumentando assim o número efetivo de cargas e a
quantidade de moléculas de água fixadas à ionosfera protéica.
O efeito “salting out” é a precipitação de proteína em solução por altas
concentrações de sais. Os sais atraem as moléculas de água do meio, de modo a ficar
menos água disponível para as moléculas protéicas o que acarreta na diminuição da
solubilidade e precipitação.
14 – Como ocorre o processo de diálise?
Resposta: A diálise é um tipo de filtração molecular. A mistura de proteína e moléculas
pequenas (como sais) é colocada dentro de um saco de material semipermeável (como o
celofane). Quando o saco de diálise é imerso em tampão as moléculas protéicas ficam
retidas, enquanto que moléculas pequenas ou íons atravessam a membrana de diálise.
15 – Diferencie os diferentes tipos de cromatografia (troca iônica, afinidade e filtração
em gel).
Resposta: Na cromatografia de exclusão molecular (ou filtração em gel) a mistura de
proteínas passa por uma coluna que é formada por polímeros que possuem pequenas
cavidades. Ao contrário das moléculas grandes, as moléculas pequenas conseguem
entrar e sair destas cavidades ficando assim mais tempo retidas na coluna. As moléculas
grandes são então eluídas primeiro seguida das menores, ocorrendo assim a separação
por tamanho molecular.
Na cromatografia por afinidade uma molécula pela qual a proteína de interesse
tem afinidade é ligada a uma matriz insolúvel. A mistura de proteínas é então passada
por esta matriz, a proteína que possui afinidade pelo ligante fica retida (por adsorção)
enquanto que as outras proteínas passam livremente. A proteína adsorvida pode ser
eluída da coluna por adição de solução concentrada de ligante.
A cromatografia de troca iônica é embasada na adsorção e ligação reversível de
moléculas carregadas a grupos carregados com carga oposta ligados a uma matriz
insolúvel. O valor de pH onde uma biomolécula possui carga nata nula é chamado de
ponto isoelétrico (pI). Quando exposta a pH abaixo do seu pI, a biomolécula apresentará
carga positiva e poderá ser atraída por colunas contendo grupos com cargas negativas
(coluna trocadora de cátion). Quando expostas a pH acima do seu pI, a biomolécula
apresentará carga negativa e poderá ser atraída por colunas contendo grupos com cargas
positivas (coluna trocadora de ânion).
16 – Qual tipo de cromatografia poderia ser utilizada para separar:
a) uma proteína de 90 kDa de uma proteína de 10 kDa.
Resposta: a separação poderia ser feita por uma cromatografia de filtração
em gel.
b) uma proteína com carga positiva em pH 5 de uma proteína cujo seu pI é 5?
Resposta: a separação poderia ser feita por uma cromatografia de troca
iônica usando-se um tampão em pH 5 como eluente e uma coluna dita
trocadora de cátions. A proteína que a presenta pI 5 passará livremente pela
coluna enquanto que a outra (com carga positiva) ficará retida.
c) uma proteína que possui afinidade ao metal Zn de uma proteína que não
afinidade a este metal.
Resposta: a separação poderia ser feita por uma cromatografia de afinidade.
Neste caso usaria-se uma coluna cuja matriz possui o metal Zinco
imobilizado, a proteína que possui afinidade a este metal ficaria retida na
coluna enquanto que a outra, por não possuir afinidade ao metal, seria eluída
com o tampão.
17 – Qual é o principio da eletroforese?
Resposta: Em um mesmo pH, proteínas diferentes apresentarão cargas líquidas
diferentes, o que determinará velocidades de migração diferentes, se as proteínas forem
submetidas a um campo elétrico. Este é o princípio da eletroforese.
18 – Quais são os parâmetros de separação de proteínas em uma eletroforese
bidimensional?
Resposta: Os parâmetros de separação de proteínas em uma eletroforese bidimensional
são: pI (ponto isoelétrico) e massa molecular.