GUIA DE USO
Sobre as informações de entrada
Para obter a conformação dos aminoácidos em um determinado trecho de uma proteína:
1) Obtenha o arquivo .pdb (pode ser obtido no Protein Data Bank <http://www.rcsb.org/>)
que representa a proteína a ser simulada. Este arquivo deverá estar faltando alguns
aminoácidos em sua sequência. A sequência faltando será informada em um arquivo
separado na próxima etapa.
2) Crie um arquivo com a extensão .seg, que representará o trecho de aminoácidos faltando
no arquivo .pdb. Você deve utilizar um editor de texto simples (como o bloco de notas)
para criá-lo. Este arquivo deverá ter a seguinte estrutura:
ALA A 200
1
SEGMENT
ASP A 201
1
SEGMENT
ALA A 202
1
SEGMENT
VAL A 203
1
SEGMENT
GLN A 204
1
SEGMENT
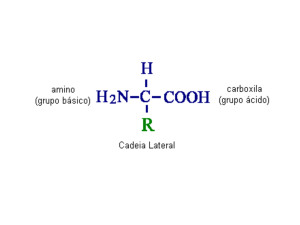
A primeira coluna é a sigla que identifica o aminoácido. A segunda coluna é a
identificação da cadeia*. A terceira coluna é o número que identifica o aminoácido. A
quarta e quinta coluna é padrão para qualquer arquivo de entrada.
*Observe que o arquivo .seg contém a identificação da cadeia (como por exemplo "A" no
modelo apresentado). Esta identificação deve estar presente apenas se ela estiver inclusa
no arquivo .pdb. Caso o arquivo .pdb não contenha esta informação, não inclua esta coluna
no arquivo .seg. A numeração dos aminoácidos no arquivo .seg deve preencher a
numeração faltando no arquivo .pdb. No exemplo apresentado, o arquivo .seg contém os
aminoácidos de 200 a 204.
3) Faça o upload destes arquivos no site. Preencha os campos informando os aminoácidos
iniciais e finais. Neste exemplo: 200 e 204, respectivamente. Informe seu endereço de e-
mail e preencha o reCAPTCHA corretamente para receber os resultados quando forem
concluídos.
Identificação dos componentes da página
Para obter mais informações sobre a metodologia da simulação:
LMProt: An Efficient Algorithm for Monte Carlo Sampling of Protein
Conformational Space
Roosevelt Alves da Silva, Léo Degrève, and Antonio Caliri
Disponível em:
http://www.ncbi.nlm.nih.gov/pmc/articles/PMC1304563/