http://wfdaj.sites.uol.com.br
© 2007 Dr. Walter F. de Azevedo Jr.
Visualização de Aminoácidos
Objetivos: Visualizar a estrutura tridimensional de aminoácidos, usando-se recursos
computacionais. A partir da visualização da estrutura de tridimensional dos aminoácidos
identificar os átomos e os tipos de ligação. Familiarizar-se com a estrutura tridimensional
dos aminoácidos.
Materiais
1. Computador PC;
2. Programa VMD (Visual Molecular Dynamics) para visualização de macromoléculas
biológicas;
3. Arquivos com coordenadas atômicas de aminoácidos;
Procedimento
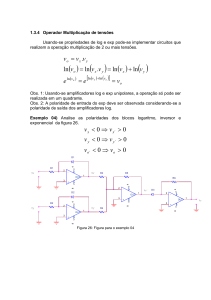
1) Visualização do ala.pdb. Carregar as coordenadas atômicas do aminoácido alanina
ala.pdb, da seguinte forma: No VMD Main clique em File e New Molecule. No menu New
Molecule clique em Browse e selecione o arquivo ala.pdb do diretório proteins/aa. Clique
em Abrir e Load. Feche o Menu Molecule File Browser. Clique na tela gráfica. Você terá o
aminoácido na tela gráfica. As representações indicam linhas para cada ligação covalente
presente nos átomos. Azul para uma ligação covalente saindo de um átomo de nitrogênio,
vermelho para uma ligação covalente saindo de um átomo de oxigênio, branco para uma
ligação covalente saindo de um átomo de hidrogênio e azul claro para uma ligação
covalente saindo de um átomo de carbono. Gire o aminoácido com o botão do mouse da
esquerda, e gire a molécula em torno de um eixo perpendicular à tela usando-se a tecla
da direita do mouse. Clique VMD Main→Mouse→Label→Atoms. Com esta configuração o
VMD identifica o átomo ao clicarmos com o mouse sobre o átomo, use esta opção para
identificar os átomos que formam o aminoácido alanina. Identifique o carbono alfa, o
nitrogênio, o carbono da carbonila, o oxigênio da carbonila e o carbono beta (carbono da
cadeia lateral)
Questão: Desenhe a fórmula molecular da alanina na tabela da próxima página.
2) Visualização do aminoácido usando-se outros esquemas gráficos.. No menu
Graphical Representations, na opção Drawing Method escolha a opção VDW (raio de
Van der Waals).
Questão:
Quais
as
principais
características
da
representação
VDW?__________________________________________________________________
________________________________________________________________________
_________________
No menu Graphical Representations, na opção Drawing Method escolha a opção CPK.
Questão:
Quais
as
principais
características
da
representação
CPK?___________________________________________________________________
________________________________________________________________________
________________
3) Visualização dos outros aminoácidos. Carregar as coordenadas atômicas dos outros
aminoácidos, usando-se o mesmo procedimento descrito no item 1. Feche o Menu
Molecule File Browser. Clique na tela gráfica. Você terá o aminoácido na tela gráfica. As
representações indicam linhas para cada ligação covalente presente nos átomos. Azul
1
http://wfdaj.sites.uol.com.br
© 2007 Dr. Walter F. de Azevedo Jr.
para uma ligação covalente saindo de um átomo de nitrogênio, vermelho para uma
ligação covalente saindo de um átomo de oxigênio, branco para uma ligação covalente
saindo de um átomo de hidrogênio e azul claro para uma ligação covalente saindo de um
átomo de carbono. Gire o aminoácido com o botão do mouse da esquerda, e gire a
molécula em torno de um eixo perpendicular à tela usando-se a tecla da direita.
Nome do
aminoácido
Alanina
Código de
três letras
Ala
Nome do arquivo
pdb
ala.pdb
Arginina
Arg
arg.pdb
Asparagina
Asn
asn.pdb
Aspartato
Asp
asp.pdb
Cisteína
Cys
cys.pdb
Glutamina
Gln
gln.pdb
Glutamato
Glu
glu.pdb
Glicina
Gly
gly.pdb
Histidina
His
his.pdb
Isoleucina
Ile
ile.pdb
Leucina
Leu
leu.pdb
Fórmula molecular
2
http://wfdaj.sites.uol.com.br
© 2007 Dr. Walter F. de Azevedo Jr.
Lisina
Lys
lys.pdb
Metionina
Met
met.pdb
Fenilalanina
Phe
phe.pdb
Prolina
Pro
pro.pdb
Serina
Ser
ser.pdb
Treonina
Thr
thr.pdb
Triptofano
Trp
trp.pdb
Tirosina
Tyr
tyr.pdb
Valina
Val
val.pdb
4) Figuras dos aminoácidos. Carregar as coordenadas atômicas dos aminoácidos (um
de cada vez), usando-se o mesmo procedimento descrito no item 1. Feche o Menu
Molecule File Browser. Clique na tela gráfica. Você terá o aminoácido na tela gráfica. As
representações indicam linhas para cada ligação covalente presente nos átomos. Mude
para a representação gráfica CPK, usando o procedimento já descrito. Gere as figuras no
formato bmp, como segue: Clique em VMD main, e siga as opções File→ Render, então
use a opção render using: snapshot. Use o browse para selecionar o diretório e o nome
do arquivo onde será salva a imagem da estrutura mostrada na tela, depois clique em
start rendering, o programa gerará um arquivo bmp da figura mostrada na tela no diretório
selecionado. Cole a figura gerada para cada aminoácido no local referente ao aminoácido
na tabela a seguir.
3
http://wfdaj.sites.uol.com.br
© 2007 Dr. Walter F. de Azevedo Jr.
Nome do
aminoácido
Alanina
Código de
três letras
Ala
Nome do arquivo
pdb
ala.pdb
Arginina
Arg
arg.pdb
Asparagina
Asn
asn.pdb
Aspartato
Asp
asp.pdb
Cisteína
Cys
cys.pdb
Glutamina
Gln
gln.pdb
Glutamato
Glu
glu.pdb
Glicina
Gly
gly.pdb
Histidina
His
his.pdb
Isoleucina
Ile
ile.pdb
Leucina
Leu
leu.pdb
Lisina
Lys
lys.pdb
Figura do aminoácido
4
http://wfdaj.sites.uol.com.br
© 2007 Dr. Walter F. de Azevedo Jr.
Metionina
Met
met.pdb
Fenilalanina
Phe
phe.pdb
Prolina
Pro
pro.pdb
Serina
Ser
ser.pdb
Treonina
Thr
thr.pdb
Triptofano
Trp
trp.pdb
Tirosina
Tyr
tyr.pdb
Valina
Val
val.pdb
Referências:
ALBERTS, B. et al. Biologia Molecular da Célula. 4a edição. Artmed editora, Porto Alegre,
2004 (Capítulo 3).
LESK, A. M. Introduction to Protein Architecture. Oxford University Press, New York, 2001.
BRANDEN, c. & TOOSE, J. Introduction to Protein Structure. 2nd Ed. Garland Publishing,
Inc. New York, 1999.
http://www.ks.uiuc.edu/Research/vmd/
http://www.fi.muni.cz/usr/mejzlik/mirrors/www.nyu.edu/pages/mathmol/software.html
http://www.rcsb.org/pdb/
5