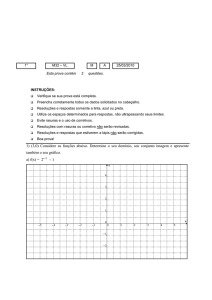
Problema :
Seleção de modelos lineares .
Procedimentos para seleção de modelos buscam identificar o modelo , dentro
de uma classe de candidatos , que melhor explica os dados em questão. Isto
torna-se complicado, uma vez que temos uma vasta quantidade de candidatos.
Este problema é usualmente resolvido utilizando “Eliminação para trás”
(Backward elimination) ou “Inclusão para frente” (Forward Inclusion); aqui é
apresentada um nova proposta.
Proposta :
Otimização do problema através de Algoritmos genéticos , utilizando como
método de comparação dos modelos o AIC1 e uma outra função de ajuste dada
por :
- 2 * log máxima verossimilhança + #de parâmetros * log #de parâmetros
que chamaremos de .
1
AIC = - 2 * log máxima verossimilhança + 2 * #de parâmetros
Dados :
1o – Foi simulado um conjunto de 20 regressores Xi ~ U (-10,10) , i= 1, ... ,20 ,
cada um contendo 100 observações , e dentre estes , 8 verdadeiros , tendo assim o
modelo :
Yi = β1 x1i + β2 x2i + β3 x3i + β4 x4i + β5 x5i + β6 x6i + β7 x7i + β8 x8i + ε
onde ε ~ N ( 0 , 1 ).
2o – Foi simulado , da mesma forma do item anterior , 100 regressores , cada um
com 200 observações , e dentre estes 20 são verdadeiros.
Algoritmo Genético :
O algoritmo genético opera em uma população de candidatos para otimizar o
problema. Através de uma população inicial cria-se uma nova população,
baseado em mecanismos genéticos naturais.
A iteração deste processo nos leva à sucessivas populações de indivíduos que,
esperamos, neste caso, explicarem melhor o nosso modelo. A adaptação dos
indivíduos é feita através de uma função de ajuste,que aqui, serão o AIC e ,
já mencionados anteriormente.
O algoritmo genético começa com uma população gerada aleatoriamente, é
feito o ajuste de cada indivíduo da população e então , novos indivíduos são
selecionados com probabilidade dependendo do valor do ajuste.
Com uma probabilidade fixa são feitos cruzamentos de pares de indivíduos, e
mutações.E então a população é substituída pela nova população e o processo é
repetido até convergência ou um número fixado de gerações.
Implementação :
# Dados simulados #
1o conjunto de dados
x<-matrix(runif(20*100,-10,10),nrow=100, ncol=20)
beta <- c(1,-1,1,-1,1,-1,1,-1,0,0,0,0,0,0,0,0,0,0,0,0)
y<-x%*%beta + rnorm(100,0,1)
2o conjunto de dados
x<-matrix(runif(200*100,-10,10),nrow=200, ncol=100)
beta <- c(1,-1,1,-1,1,-1,1,-1,1,-1,1,-1,1,-1,1,-1,1,-1,1,-1,rep(0,80))
y<-x%*%beta + rnorm(200,0,1)
# Variáveis fixas #
extras<- NULL
faltam<- NULL
verdadeiro<- c(rep(T,20),rep(F,80))
a<- matrix(ncol=80,nrow=1)
b<-matrix(ncol=100,nrow=80)
População
G<-200 Número de gerações
prob.cross<-0.2 Probabilidade de haver cruzamento
prob.mut<-0.05 Probabilidade de haver mutação
pop<- 80
# Ajuste do modelo verdadeiro #
testeverdadeiro<-lm(y~ x[,1:20])
verdade<-AIC(testeverdadeiro,k=log(20))
# População inicial e ajuste da população #
for(i in 1:pop){
csi<- sample(c(T,F), 100,replace=TRUE, prob=c(0.5,0.5))
b[i,]<-csi
teste<-lm(y~x[,b[i,]])
a[i]<-1/(AIC(teste,k=log(sum(b[i,]))))
}
Para as G gerações faremos :
for(g in 1:G){
p<-matrix(a/sum(a),ncol=pop,nrow=1)
ele.ótimo<-(1:pop)[p==max(p)]
( Escolhemos os elementos para próxima fase de acordo com o valor do seu
ajuste )
# Reprodução #
( Escolhemos pop – 10 elementos porque estamos guardando o elemento com
melhor ajuste 10 vezes (elitismo) )
elemento<-sample(1:pop,pop-10,replace=T,prob=p)
b<-b[c(rep(ele.ótimo[1],10),elemento),]
# Cruzamento #
for(j in 1:40){
Número de cruzamentos
elemento1<-sample(1:pop,2)
csi1<-b[elemento1[1],]
csi2<-b[elemento1[2],]
for (i in 1:100){
if(runif(1)<prob.cross){
cross<-csi1[i]
csi1[i]<-csi2[i]
csi2[i]<-cross
}
b[elemento1[1],]<-csi1
b[elemento1[2],]<-csi2
}}
# Mutação
for(j in 1:30){
Número de mutações
elemento2<-sample(1:pop,1)
for(i in 1:100){
if(runif(1)<prob.mut){
b[elemento2,i]<- !b[elemento2,i]
}
}}
# Ajuste da nova população #
for(i in 1:pop){
teste<-lm(y~x[,b[i,]])
a[i]<-1/(AIC(teste,k=log(sum(b[i,]))))
}
}
# Resultados #
result<-(1:pop)[a==max(a)]
Dentre os indivíduos da população
selecionamos aquele que tem o melhor ajuste
modeloselecionado<-b[result[1],]
teste<-lm(y~x[,modeloselecionado])
AIC(teste,k=log(sum(modeloselecionado)))
extras<- sum((modeloselecionado-verdadeiro)>0)
além das do modelo verdadeiro , foram incluídas.
Conta quantas covariáveis
faltam<-sum((modeloselecionado-verdadeiro)<0)
Conta quantas covariáveis
estão no modelo verdadeiro e não estão no modelo selecionado.
Observação : Os valores fixados anteriormente são para o 2o conjunto de
dados. Para o 1o conjunto de dados, foi usada uma população inicial de 50
indivíduos, com número de cruzamentos 15 e mutações 20. Além disso foi
usado um elitismo de 5 indivíduos. O número de gerações para o 1o
conjunto de dados foi 60, pois a convergência era bem mais rápida.
Resultados :
Com ambos os conjuntos de dados, o algoritmo foi rodado 100
vezes e em 100% delas convergiu para o modelo original ou para
um modelo com função de ajuste menor ; sempre incluindo todas
as variáveis verdadeiras, mostrando assim, eficiência.
Para o 1o conjunto de dados o tempo demorado foi mais ou menos
de 1 minuto , enquanto para o 2o, 15 minutos.
Gráficos de convergência para o 2o conjunto de dados :
Usando :
Usando AIC :
Gráficos de convergência para o 1o conjunto de dados :
Usando :
Usando AIC :
Comparando e AIC :
Através dos contadores extras e faltam , definidos no algoritmo, foi possível
comparar as performances do AIC e .
O algoritmo foi rodado 100 vezes para cada função de ajuste e foi obtido o
seguinte resultado :
1o conjunto de dados :
Média de extras:
AIC
0.975
2.4
2o conjunto de dados :
Média de extras:
AIC
5.37
22.55
Como já citado anteriormente o algoritmo nunca deixa de incluir as covariáveis
verdadeiras(faltam = 0 em todos os casos acima)
O resultados mostram que o tem um desempenho bem
melhor que o do AIC, já que a média de variáveis extras é bem
menor. (Pelo princípio da parcimônia devemos escolher o modelo
com menor número de covariáveis)
Poderíamos ter usado como critério de ajuste o BIC2 (Bayesian
criterion information), que penaliza os modelos “maiores” ainda
mais que .
2
BIC = -2 * log máx verossimilhança + # de parâmetros * log # observações
Projeto
Estatística
Computacional
Renata Leite Estrella
Bibliografia :
Irene Poli, Alberto Roverato
“A Genetic Algorithm for Classical Model Selection”