UNIVERSIDADE MACKENZIE
Faculdade de Ciências Biológicas Exatas e Experimentais
Departamento de Biologia
Características do código genético
para tradução de proteínas
Seminário de Biologia Molecular
Síntese de proteínas in vitro
Uma magnífica confirmação das linhas gerais de síntese de proteínas foi
dada por Von Ehrenstein e Lipmann, que conseguiram sintetizar a hemoglobina
em um sistema livre de células. Tal sistema incluía:
1. Ribossomos, na forma de polissomos de reticulócitos (glóbulos vermelhos
imaturos, onde a hemoglobina é quase a única proteína sintetizada) de
coelhos, que incluíam mRNA para hemoglobina de coelho.
2. Um trifosfato doador de energia.
3. tRNA da bactéria E.coli, previamente carregados com aminoácidos,
usando enzimas de E.coli.
O aspecto mais excitante desse trabalho é que Von Ehrenstein e Lipmann
conseguiram sintetizar uma proteína de uma espécie usando seu mRNA e tRNA
de um organismo muito diferente. Não só isto sugere fortemente a universalidade
do mecanismo geral de síntese de proteínas como também parece irrefutável o
“parentesco” fundamental de todos os organismos vivos através de seu DNA. É
muito claro que o DNA é o material genético de todos os organismos vivos (e
muito vírus, também) e que é um vocabulário de informação de quatro “letras”
(adenina, timina, citosina e guanina). As diferenças entre as espécies residem
portanto nas seqüências das “letras” nucleotídicas que formam certos códigos de
“palavras” (aminoácidos), que serão combinadas seqüencialmente em “frases”
protéicas e “parágrafos” fenotípicos.
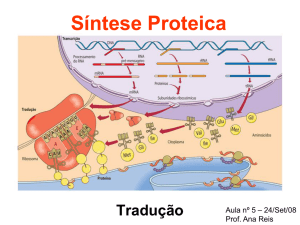
Tradução
É o momento da informação entre genes e proteínas mudar de linguagem
usada. Ao ir do DNA para o RNA, a linguagem (seqüência de nucleotídios)
permaneceu a mesma. Ao ir do RNA para a proteína, a linguagem é mudada de
uma seqüência de nucleotídios para uma seqüência de aminoácidos. Como no
processo de tradução de uma língua para outra, este processo de uso de
informação no RNA para fazer uma proteína é chamado Tradução. Os
aminoácidos isolados não vêm ao ribossomo para ser incorporados na proteína.
Em vez disso, são trazidos ao ribossomo por seu tRNA adequado. A primeira
etapa na incorporação de um aminoácido em uma proteína envolve a ligação do
aminoácido a seu tRNA correto. Este é um processo em duas etapas que se dá da
seguinte maneira:
Ativação do aminoácido.
Cada um dos vinte tipos de aminoácidos tem de ser ativado antes de se ligar
a seu tRNA. Na ativação, uma enzima (aminoacil-tRNA-sintetase) catalisa a
reação de um aminoácido específico com adenosina-trifosfato (ATP), formando
aminoacil-adenosina-monofosfato (aminoacil adenilato) e pirofosfato:
Aminoacil
AA + ATP
sintetase
AA ~ AMP +
Pirofosfato
O aminoacil adenilato (AA-AMP) é referido como um aminoácido ativado, que
é ligado à adenosina por uma ligação éster-fosfato e foi agora levado a um nível
de energia no qual pode reagir com seu tRNA.
Por sua arquitetura, cada tipo de molécula da enzima precisa “reconhecer” e
ser capaz de se ligar a um aminoácido em particular. Cada uma das várias
“espécies” de aminoacil-tRNA-sintetase possui dois sítios de ligação. Cada um
destes “reconhece” um, e só um, aminoácido dos 20. Até 20 diferentes tipos de
aminoácidos podem estar envolvidos; portanto, cada célula tem de ter pelo menos
20 tipos diferentes de aminoacil-sintetase.
O aminoácido ativado, no entanto, fica fortemente ligado a sua enzima e não
pode, nesta condição, ser ligado a outro para começar a formação de uma cadeia
polipeptídica. Este processo requer transferência do aminoácido ativado para o
tRNA.
Transferência do aminoácido ativado para o tRNA.
A mesma aminoacil-sintetase q catalisou a ativação do aminoácido liga-se
em seguida a um local receptor no ribonucleotídio de adenina de uma determinada
molécula de tRNA quando os dois entrarem em contato como resultado de uma
colisão casual. Diz-se então que o tRNA está carregado:
AA ~ AMP + tRNA
AA ~ tRNA + AMP
O aminoacil adenilato liga-se à extremidade 3’ -OH livre da ribose do ácido
adelínico 3’ terminal do tRNA, formando uma ligação acila com o grupamento alfacarboxílico do aminoácido. Note que as enzimas precisam ser capazes de ligar-se
especificamente tanto a um determinado aminoácido quanto a uma determinada
molécula de tRNA. Portanto, além do mínimo de 20 diferentes de sintetase, cada
célula tem de Ter pelo menos 20 espécies diferentes de tRNA. De fato, há boa
evidência experimental para a existência de duas ou mais espécies diferentes de
tRNA com o mesmo grau de especificidade para o mesmo aminoácido.
A especificidade da ligação da sintetase a seu aminoácido e a transferência
ao tRNA correto dependem, pelo menos em parte, da estrutura do grupamento R
do aminoácido e de regiões específicas, provavelmente na alça T ou na D, da
molécula do tRNA. Como as diferenças entre os grupamentos R de alguns
aminoácidos são pequenas (Fig. 1.1), erros ocasionais são feitos nesse processo,
com o aminoácido errado dando ligado ao tRNA. Evidências recentes sugerem
que a aminoacil-sintetase confere a ligação correta antes de liberar o tRNA-AA. Se
foi feito um erro, o aminoácido errado é removido e o correto é ligado.
Montagem de polipeptídios
Após a ativação do aminoácido e sua ligação ao RNA de transferência, o
tRNA difunde-se para o ribossomo, onde ocorre a real montagem das cadeias
polipeptídicas. A descrição a seguir da formação da ligação peptídica aplica-se à
E.coli em particular e a bactérias em geral, organismos nos quais o processo é
mais bem conhecido. Nos eucariotos, aparentemente ela só difere em pequenos
detalhes da série de eventos a seguir.
Quando o mRNA se liga à subunidade menor (isto é, 30s) do ribossomo
bacteriano, ele expõe lá pelo menos dois grupos de três ribonucleotídios cada, que
constituem locais capazes de aceitar moléculas de tRNA carregadas. Esses locais
são designados sítio aminoacil (A) e sítio peptidil (P), respectivamente. Na
extremidade 5’da molécula associada de mRNA, ou perto dela, tem de estar
presente um grupo de três ribonucleotídios iniciadores de cadeia. O primeiro
ribossomo liga-se a este local iniciador de três bases, muitas vezes quando o
mRNA ainda está sendo transcrito (fig.1. 2). Na E.coli, o primeiro aminoácido a ser
incorporado é a N-formilmetionina. Esta é a metionina com um grupamento formil,
O
I
H–Cligado ao grupamento amina:
O H H O
II I
I I
H – C – N – C – C – OH
I
CH2
I
CH2
I
S
I
CH3
A metionina é formalina enzimaticamente após a ligação ao seu tRNA. Nem
todos os complexos metionina-tRNA (met-tRNA) podem ser formilados e há dois
tipos de met-tRNA, e deles só um permite a formilação. O grupamento formil
bloqueia a formação de uma ligação peptídica com o grupamento carboxila de
outro aminoácido. Dependendo do polipeptídeo em particular sendo sintetizado, a
N-formil metionina pode ou não ser mais tarde removida enzimaticamente antes
que a cadeia polipeptídica se torne funcional, mas na maioria dos casos ela é
removida.
O processo de incorporação seqüencial de aminoácidos em proteínas por
formação de ligação peptídica pode ser dividido em três estágios: Início,
Alongamento e Término ( veja também a figura 1.2 ).
Início
1. A subunidade 30s do primeiro ribossomo liga-se ao mRNA em um sítio
iniciador, a uma distância fixa antes do códon de início, que é a
seqüência nucleotídica AGGAGGU e chamada seqüência ShineDelgarno, nome de seus descobridores.
2. O tRNA-fmet , carregado com o primeiro aminoácido (N-formil-metionina)
liga-se à subunidade 30s no sítio P, junto a três ribonucleotídios. São
necessários três fatores protéicos de início (IF) mais guanosina-trifosfato.
3. Imediatamente após Ter sido completada a etapa 2, a subunidade maior,
50s, liga-se à subunidade 30s e completa a montagem do primeiro
ribossomo, que ainda transposta o tRNA carregado com N-formilmetionina no sítio P.
Alongamento
4. O segundo tRNA carregado liga-se a esse primeiro ribossomo no sítio A
deste, com ajuda da proteína fator de alongamento, de modo que
ambos os sítios estão ocupados, isto é, o sítio P com o primeiro tRNA, o
sítio A com o segundo.
5. Forma-se enzimaticamente (pela peptidil-transferase, que é uma proteína
componente da subunidade 50s) uma ligação peptídica entre o
grupamento amino do segundo aminoácido e o grupamento carboxila do
primeiro aminoácido.
6. Ocorre então a translocação, que consiste de três etapas:
a) ejeção do tRNA-fmet do sítio P
b) movimento do tRNA-dipeptídio do sítio A para o P
c) movimento do mRNA de tal modo que o efeito é o movimento
aparente do ribossomo em direção à extremidade 3’do mRNA (de três
nucleotídios).
7. A etapa 6 traz o que antes estava no sítio A (com seu dipeptídio) à
localização no sítio P com a ajuda do fator G de alongamento.
8. Um terceiro tRNA carregado move-se para a posição no novo sítio A com
a ajuda do mesmo fator de alongamento anteriormente usado na Etapa 4.
9. O processo delineado nas Etapas 4 a 8 é então repetido de novo e de
novo, com novos ribossomos sucessivamente juntando-se no local de
início e movendo-se ou sendo movidos ao longo do mRNA na direção 5’
3’. Os detalhes da fase de alongamento estão ilustrados na Fig.1.3.
Término
10. Essas etapas continuam até que o primeiro e então os sucessivos
ribossomos atingem um grupo de término de três nucleotídios, e em tal
ponto os ribossomos (cada um portando um polipeptídio completo) são
sucessivamente ejetados do polissomo com a ajuda de três fatores
protéicos de liberação. As subunidades dissociam-se e são capazes de
repetir todo o processo.
Extensão e velocidade da tradução
Como descrito anteriormente, a vida de qualquer molécula de mRNA é finita.
Ela continua a funcionar de modo descrito enquanto houver ribossomos ligando-se
à sua extremidade 5’, mas esta extremidade é vulnerável a ribonucleases. Estas
são enzimas que hidrolisam o RNA e o degradam a seus nucleotídios
constituintes, que podem então ser usados para construir integralmente novos e
diferentes mRNA. A meia-vida de muitos mRNA bacterianos parece ser da ordem
de um a três minutos, mas é mais longa em eucariotos – de algumas horas em
fígado de camundongo a vários ou muitos dias em reticulócitos. Há relativamente
poucos dados quantitativos disponíveis sobre a estabilidade de mRNA eucariótico.
A tradução ocorre com considerável velocidade. Na produção de
hemoglobina, por exemplo, uma cadeia polipeptídica de perto de 150 aminoácidos
requer cerca de 80 segundos para a síntese. Isto significa adição de um
aminoácido em pouco mais de 0,5 segundo.
Bibliografia
1. Burns, G. W., e Bottino, P. J., 1991. Genética. 9:197-170.
Guanabara Koogan – RJ
2. De Robertis, E. D. P., e De Robertis Jr, E. M. F., 1993. Bases
da Biologia Celular e Molecular. Guanabara Koogan – RJ
3. Zaha A., 1996. Biologia Molecular Básica. Mercado Aberto RS