51º Congresso Brasileiro de Genética
Resumos do 51º Congresso Brasileiro de Genética • 7 a 10 de setembro de 2005
Hotel Monte Real • Águas de Lindóia • São Paulo • Brasil
www.sbg.org.br - ISBN 85-89109-05-4
[email protected]
Palavra-chave: apomixia, reprodução vegetal, forrageiras
Guimarães, LA1,2; Silveira, ED1; Silva, FR; Carneiro, VTC1
1
Embrapa Recursos Genéticos e Biotecnologia; 2Universidade de Brasília-UnB.
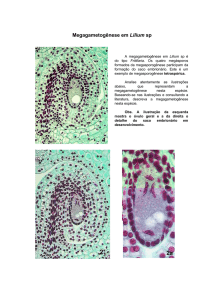
Análise de bibliotecas de cDNA de ovários
de B. brizantha apomítica e sexual
em megasporogênese e megagametogênese
(Analysis of cDNA libraries of ovaries from
apomictic and sexual Brachiaria brizantha in
megasporogenesis and megagametogenesis)
A apomixia é o modo de reprodução assexuada de plantas por meio de sementes. Ela é encontrada em
mais de 300 espécies de plantas, entre elas, Brachiaria brizantha. Essa espécie de forrageira é amplamente
utilizada como pastagem no Brasil e apresenta tanto o modo de reprodução apomítico, quanto o modo
de reprodução sexual. O padrão de expressão gênica observado no desenvolvimento do saco embrionário
pode estar relacionado com a apomixia, pois nas plantas apomíticas este apresenta diferenças em relação
a uma planta sexual. O saco embrionário é formado de uma célula não reduzida e o desenvolvimento
do embrião ocorre sem fertilização. Este trabalho teve como objetivo analisar e comparar a expressão de
genes durante o desenvolvimento de ovários de B. brizantha apomítica e sexual, em megasporogênese e
megagametogênese. RNA total de ovários de B. brizantha foi extraído utilizando TRIZOL® e mRNA foi
isolado com o kit Dynabeads (Dynal®). Foram construídas quatro bibliotecas de cDNA com o kit SMART
cDNA Library Construction Kit (Clontech). Os cDNAs foram clonados em pTriplex2 e transformados em
células de Escherichia coli XL1Blue. As bibliotecas geradas foram seqüenciadas em aparelho automático
ABI 3700. Os cromatogramas foram analisados com o software PHRED e, após o processamento das
seqüências (trimming), estas foram agrupadas com o programa CAP3, e comparadas por BLAST contra
o banco não redundante, para verificar similaridade dos EST’s gerados. Em seguida, as seqüências foram
anotadas utilizando dados fornecidos no Interpro e Gene Ontology. As bibliotecas mostraram um elevado
grau de representatividade. Dos 1536 EST’s seqüenciados até o momento, 82% apresentaram qualidade
mínima para análise. Dentre elas, 76% não foram agrupadas (singlets) e as demais geraram contigs compostos
por 1 a 5 reads, apresentando, assim, baixa redundância. Apenas 18% das seqüências foram descartadas
devido à qualidade do seqüenciamento e a contaminações, sendo menos de 0,1% com seqüências de RNA
ribossomal de planta. Os resultados gerados apresentaram qualidade e representatividade, além de terem
sido identificados EST´s que apresentaram similaridade com seqüências importantes para o desenvolvimento
do saco embrionário. O envolvimento dessas seqüências na expressão da apomixia é discutido neste trabalho
e o seqüenciamento de um número maior de clones dessas bibliotecas poderá colaborar na identificação de
cDNA´s relacionados ao modo de reprodução apomítico em B. brizantha.
Apoio financeiro: Embrapa Recursos Genéticos e Biotecnologia e CNPQ.
596