UNIVERSIDADE FEDERAL DE GOIÁS
PROGRAMA DE PÓS-GRADUAÇÃO EM MEDICINA TROPICAL
E SAÚDE PÚBLICA
SUELLEN ROCHA ARAÚJO CASTILHO
Caracterização fenotípica e molecular de Acinetobacter
baumannii isolados de pacientes de UTIs de Goiânia, com
relação à capacidade de formar biofilmes e resistência aos
carbapenêmicos
Goiânia
2013
Termo de Ciência e de Autorização para Disponibilizar as Teses e Dissertações
Eletrônicas (TEDE) na Biblioteca Digital da UFG
Na qualidade de titular dos direitos de autor, autorizo a Universidade Federal de
Goiás–UFG a disponibilizar gratuitamente através da Biblioteca Digital de Teses e
Dissertações – BDTD/UFG, sem ressarcimento dos direitos autorais, de acordo com a Lei
nº 9610/98, o documento conforme permissões assinaladas abaixo, para fins de leitura,
impressão e/ou download, a título de divulgação da produção científica brasileira, a
partir desta data.
1. Identificação do material bibliográfico:
[ X ] Dissertação
[ ] Tese
2. Identificação da Tese ou Dissertação
Autor(a): SUELLEN ROCHA ARAÚJO CASTILHO
CPF:
007.904.911-76
E-mail:
[email protected]
Seu e-mail pode ser disponibilizado na página?
[ X ]Sim
[ ] Não
Vínculo Empregatício do autor:
Professora da Rede Municipal de Educação
Agência de fomento:
Coordenação de Aperfeiçoamento de Pessoal de Nível Sigla:
CAPES
Superior
País:
Brasil
UF:
GO
CNPJ:
Título:
Caracterização fenotípica e molecular de Acinetobacter baumannii isolados de pacientes
de UTIs de Goiânia, com relação à capacidade de formar biofilmes e resistência aos
carbapenêmicos
Palavras-chave:
Acinetobacter baumannii / formação de biofilme / blaPER-1 / resistência /
carbapenêmicos / OXA
Título em outra língua:
Phenotypic and molecular characterization of Acinetobacter baumannii
isolated from ICU patients in Goiânia, with respect to the ability to form
biofilms and carbapenem resistance
Palavras-chave em outra língua:
Acinetobacter baumannii / biofilm formation / blaPER-1 /
resistance / carbapenem / OXA
Área de concentração:
Microbiologia
Data defesa: (dd/mm/aaaa)
21/02/2013
Programa de Pós-Graduação:
Medicina Tropical e Saúde Pública
Orientador(a):
Dr. André Kipnis
CPF:
075.965.498-02
E-mail:
[email protected]
3. Informações de acesso ao documento:
Liberação para disponibilização?1
[ X ] total
[ ] parcial
Em caso de disponibilização parcial, assinale as permissões:
[ ] Outras restrições: ________________________________________________
Havendo concordância com a disponibilização eletrônica, torna-se imprescindível
o envio do(s) arquivo(s) em formato digital PDF ou DOC da tese ou dissertação.
O Sistema da Biblioteca Digital de Teses e Dissertações garante aos autores, que os
arquivos contendo eletronicamente as teses e ou dissertações, antes de sua
disponibilização, receberão procedimentos de segurança, criptografia (para não permitir
cópia e extração de conteúdo, permitindo apenas impressão fraca) usando o padrão do
Acrobat.
SUELLEN ROCHA ARAÚJO CASTILHO
Assinatura do(a) autor(a)
1
Data: _17_ / _04_ / _2013_
Em caso de restrição, esta poderá ser mantida por até um ano a partir da data de defesa. A extensão deste prazo
suscita justificativa junto à coordenação do curso. Todo resumo e metadados ficarão sempre disponibilizados.
SUELLEN ROCHA ARAÚJO CASTILHO
Caracterização fenotípica e molecular de Acinetobacter
baumannii isolados de pacientes de UTIs de Goiânia, com
relação à capacidade de formar biofilmes e resistência aos
carbapenêmicos
Dissertação de Mestrado apresentada
ao Programa de Pós-Graduação em
Medicina Tropical e Saúde Pública da
Universidade Federal de Goiás para
obtenção do Título de Mestre em
Medicina Tropical e Saúde Pública.
Orientador: Prof. Dr. André Kipnis
Goiânia
2013
ii
Programa de Pós-Graduação em Medicina Tropical e Saúde Pública
da Universidade Federal de Goiás
BANCA EXAMINADORA DA DISSERTAÇÃO DE MESTRADO
Aluno (a): SUELLEN ROCHA ARAÚJO CASTILHO
Orientador (a): DR. ANDRÉ KIPNIS
Membros:
1. Dr. André Kipnis
2. Dr. João Alves de Araújo Filho
3. Dra. Maria Cláudia Dantas P. B. André
Data: 21/02/2013
iii
DEDICATÓRIA
Ao meu esposo Alexandre, pelo carinho, paciência, compreensão e apoio.
Aos meus pais, Péricles e Suelma, por todo amor a mim dedicado.
Amo vocês!
iv
AGRADECIMENTOS
A Deus, por me capacitar diante de todas as dificuldades enfrentadas e por me
fortalecer a cada manhã. Sei que Ele esteve atento às minhas orações e a Sua resposta se
concretiza com esta grande vitória! “Espera no Senhor, anima-te, e Ele fortalecerá o teu
coração; espera, pois, no Senhor.”
Ao meu esposo Alexandre, que em todo o tempo me apoiou, compreendeu e me
aconselhou, mesmo quando era o principal alvo, e muitas vezes o único, dos meus
surtos e crises de nervosismo e desespero, de modo que por vezes acabou por renunciar
a si mesmo. Não há como mensurar o meu amor, respeito e carinho por você!
Aos meus pais, Péricles e Suelma, que sempre acreditaram em mim. Por vezes vi
as lutas que enfrentaram para garantir minha educação e ajudar na formação da pessoa
que me tornei. Devo minha vida a vocês!
À minha querida irmã Suelenne e meu cunhado Uadson, pelos momentos de
descontração que me proporcionaram e por me deixarem fazer parte da vida de uma
princesa chamada Alice. Amo vocês!
À minha sogra Joasira, por fazer de pequenos instantes em sua companhia
momentos de muita alegria. É um prazer fazer parte de sua família!
A todos os meus familiares, que direta ou indiretamente, contribuíram para eu
alcançar esta vitória. Em especial, minha vovó Eledyr e minhas cunhadas Rosângela e
Solange.
Ao meu orientador, Dr. André Kipnis, pela oportunidade de estágio e vaga na
seleção de Mestrado e por todo apoio e confiança a mim dispensados. Seus
conhecimentos e a dedicação em nos tornar cientistas me fizeram ver além das minhas
limitações! Muito obrigada!
Aos professores, Dra. Carla Afonso S. Bitencourt Braga, Dra. Maria Cláudia
Dantas P. B. André e Dr. Plínio Lázaro Faleiro Naves, que participaram da Banca de
Qualificação e moldaram a minha Dissertação com sugestões, críticas, elogios e
conhecimentos compartilhados.
À professora Dra. Ana Paula Junqueira-Kipnis, pela dedicação em corrigir meu
artigo e me ajudar a vencer os desafios para uma escrita perfeita. Não poderia deixar de
registrar a sua ajuda e apoio! Obrigada!
v
Ao autor MSc. Arto Baghdayan, da Universidade de Oklahoma (EUA), que
respondeu com carinho aos meus e-mails, dedicando-me um pouquinho de seu tempo e
instruindo-me na realização da técnica de formação de biofilme. I have no words to
thank such attention! Your help was essential for the conclusion of this work! Thanks a
lot!
Às minhas amigas Sanair Santana e Alyne Pereira, por todo o companheirismo e
por estarem sempre atentas ao meu desespero com conselhos de motivação. Espero
contar sempre com vocês!
A todos os meus colegas de laboratório, em especial Déllys Leonora, Bruna
Daniella, Lázaro Moreira, Renato Beilner e Jacqueline Souza, por toda ajuda a mim
prestada para que fosse possível a conclusão deste trabalho, e também aos colegas
Viviane Lopes, Maysa Paula, Fabiana Metzker, Fábio Muniz, Rogério Coutinho e
Aureliano Guedes, por tornarem nossos dias no laboratório mais leves e descontraídos!
É muito bom ter todos vocês ao meu lado!
Aos meus colegas de trabalho da Escola Municipal Senador Darcy Ribeiro, em
especial à Diretora Valéria Alves, por compreender minhas muitas faltas, e também às
colegas Lucilene Camilo, Rosane Vera, Fernanda Padilha, Louise Gonçalves, Isabella
Barbosa, Valéria de Almeida e Jussimária Almeida, por fazerem meus dias na escola
bem mais divertidos e menos estressantes.
À CAPES, pelo apoio financeiro.
vi
SUMÁRIO
TABELAS, FIGURAS E ANEXOS
ix
SÍMBOLOS, SIGLAS E ABREVIATURAS
xi
RESUMO
xv
ABSTRACT
xvi
1 INTRODUÇÃO / REVISÃO DA LITERATURA
1
1.1 TAXONOMIA DO Acinetobacter baumannii
1
1.2 Acinetobacter baumannii E PATOGÊNESE
2
1.3 IDENTIFICAÇÃO LABORATORIAL DE Acinetobacter baumannii
4
1.3.1
Identificação fenotípica
4
1.3.2
Identificação genotípica
5
1.4 PERFIL DE RESISTÊNCIA
8
1.4.1
β-lactamases de classe D
12
1.4.1.1 β-lacatamase OXA-23
14
1.4.1.2 β-lactamase OXA-40
14
1.4.1.3 β-lactamase OXA-51
15
1.4.1.4 β-lactamase OXA-58
15
1.4.2
Sequência de inserção ISAba1
16
1.4.3
blaPER-1
17
1.5 FORMAÇÃO DE BIOFILMES
17
1.5.1
Biofilme
20
1.5.2
Formação e maturação do biofilme
20
1.5.3
Sistema quorum-sensing
22
1.5.4
Resistência e Formação de Biofilme
23
1.6 EPIDEMIOLOGIA
26
2 JUSTIFICATIVA
32
3 OBJETIVOS
34
3.1 GERAL
34
3.2 ESPECÍFICOS
34
4 MATERIAL E MÉTODOS
35
4.1 ASPECTOS ÉTICOS
35
vii
4.2 RECRUTAMENTO DOS CASOS E COLETAS DE AMOSTRAS
CLÍNICAS
4.3 ISOLAMENTO,
IDENTIFICAÇÃO,
SUSCEPTIBILIDADE
MICROBIANA
35
36
4.4 TESTE DE VIABILIDADE DE Acinetobacter baumannii
37
4.5 EXTRAÇÃO DE DNA CROMOSSOMAL
37
4.6 EXTRAÇÃO DE DNA PLASMIDIAL
38
4.7 TESTE DE FORMAÇÃO DE BIOFILME EM POLIESTIRENO
38
4.8 IDENTIFICAÇÃO GENOTÍPICA
40
4.8.1
Identificação do gene blaPER-1
41
4.8.2
Identificação dos genes OXA e ISAba1
41
5 RESULTADOS
43
5.1 FORMAÇÃO DE BIOFILME
43
5.2 GENE blaPER-1
43
5.3 RELAÇÃO DO GENE blaPER-1 COM A FORMAÇÃO DE BIOFILME
43
5.4 AMPLIFICAÇÃO DOS GENES OXA E ISAba1
44
5.5 RESISTÊNCIA E FORMAÇÃO DE BIOFILME
48
5.6 RELAÇÃO ENTRE A RESISTÊNCIA E O GENE blaPER-1
49
6 DISCUSSÃO
51
7 CONCLUSÕES
56
REFERÊNCIAS
57
ANEXOS
76
Anexo 1. Aprovação pelo Comitê de Ética em Pesquisa
76
Anexo 2. Manuscrito
77
viii
TABELAS, FIGURAS E ANEXOS
Tabela 1.
Provas de identificação
Acinetobacter.
Tabela 2.
Sistema de classificação para bactérias produtoras de β-lactamases
de acordo com o substrato.
11
Tabela 3.
Oligonucleotídeos usados para detecção de blaPER-1, βlactamases do tipo OXA e ISAba1 em Acinetobacter baumannii.
41
Tabela 4.
Relação entre presença do gene blaPER-1 com a formação de
biofilme bacteriano.
44
Tabela 5.
Relação entre a intensidade da formação de biofilme e a presença
do gene blaPER-1.
44
Tabela 6.
Distribuição de genes β-lactamases do tipo OXA em isolados
Acinetobacter baumannii com perfil de resistência aos
antimicrobianos relacionados.
46
Tabela 7.
Relação entre a resistência de Acinetobacter baumannii na
presença de genes β-lactamases do tipo OXA.
47
Tabela 8.
Relação entre a resistência de Acinetobacter baumannii na
presença exclusiva de um dos genes β-lactamases do tipo OXA.
47
Tabela 9.
Resultados do teste de susceptibilidade aos antimicrobianos de
isolados de Acinetobacter baumannii, em porcentagem (%).
48
Tabela 10.
Padrão de resistência multidrogas de formadores e não-formadores
de biofilme.
49
Tabela 11.
Relação entre a presença do gene blaPER-1 e a resistência à
cefepima.
50
Figura 1.
Métodos genotípicos para identificação de espécies Acinetobacter.
7
Figura 2.
Mecanismos potenciais
Acinetobacter.
9
Figura 3.
Divisão das carbapenemases em Classe A, B e D, de acordo com a
classificação de Ambler.
10
Figura 4.
Dendograma obtido para 73 β-lactamases de classe D de acordo
Phillip (clustalW).
13
de
bioquímica
resistência
para
espécies
antimicrobiana
de
4
em
ix
Figura 5.
Desenho esquemático mostrando três exemplos de possíveis
pontos de entrada no corpo para biofilmes infecciosos.
19
Figura 6.
Conceituação de desenvolvimento de biofilme e comportamento
dinâmico em cada etapa da formação.
21
Figura 7.
Representação esquemática das várias etapas de desenvolvimento
de um biofilme de acordo com o modelo aceito para Pseudomonas
aeruginosa.
22
Figura 8.
Três hipóteses para os mecanismos de resistência a antibióticos em
biofilme.
25
Figura 9.
Países que relataram surtos por Acinetobacter baumannii
resistentes aos carbapenêmicos.
26
Figura 10.
Esquema representando a distribuição dos isolados e a disposição
dos controles positivo e negativo e do BRANCO.
39
x
SÍMBOLOS, SIGLAS E ABREVIATURAS
µg
Micrograma
µL
Microlitro
µM
microMolar
AFLP
Polimorfismo de comprimento de fragmentos amplificados (do inglês,
Amplified Fragment Lenght Polymorphism)
AI
Autoindutores
AMI
Amicacina
AMP/SUL
Ampicilina/sulbactam
AmpC
Enzima cefalosporinase cromossomal β-lactamase de classe C
ARDRA
Análise de Restrição do DNA ribossomal Amplificado (do inglês,
Amplified Ribosomal DNA Restriction Analysis)
ARI-1
Acinetobacter resistente ao imipenem (do inglês, Acinetobacter resistant
imipenem)
ATCC
Coleção americana de tipos de cultura (do inglês, American Type
Culture Collection)
blaPER-1
Enzima β-lactamase de espectro estendido que hidrolisa cefalosporinas
CAZ
Ceftazidima
CcrA
Metallo- β-lactamase de Bacteroides fragilis
CFPM
Cefepima
CHDL
β-lactamases da classe D que hidrolisam carbapenêmicos (do inglês,
Carbapenem-Hydrolyzing Class D β-Lactamase)
CLSI
Instituto de Padronização Laboratorial Clínica (do inglês, Clinical
Laboratory Standards Institute)
CPX
Ciprofloxacina
CRAB
Acinetobacter baumannii resistente aos carbapenêmicos (do inglês,
Carbapenem Resistant Acinetobacter baumannii)
CTAB
Brometo de cetiltrimetilamônio (do inglês, cetyl trimethylammonium
bromide)
xi
DNA
Ácido Desoxirribonucléico (do inglês, Deoxyribonucleic Acid)
dp
Desvio padrão
EDTA
Ácido etilenodiamino tetra-acético (do inglês,
ethylenediaminetetraacetic acid)
ELISA
Ensaio Imunoadsorvente Ligado à Enzima (do inglês, Enzyme-Linked
ImmunoSorbent Assay)
EPS
Exopolissacarídeo
ESBL
β-Lactamase de Espectro Estendido (do inglês, Extended-Spectrum βLactamase)
g
Aceleração de gravidade
GEN
Gentamicina
GES
Enzima de espectro estendido descrita em Guiana (do inglês, Guiana
Extended Spectrum)
GIM
Imipenemase descrita na Alemanha (do inglês, German imipenemase)
HCl
Ácido clorídrico
HDT
Hospital de Doenças Tropicais Anuar Auad
IMI
Enzima β-lactamase que hidrolisa o imipenem (do inglês, Imipenemhidrolyzing β-lactamase)
IMP
Impenemase
IPM
Imipenem
IS
Sequência de Inserção (do inglês, Insertion Sequence)
ISAba1
Sequência de Inserção Acinetobacter baumannii (do inglês, Insertion
Sequence Acinetobacter baumannii)
K1
Enzima β-lactamase descrita em Klebsiella
KPC
Klebsiella pneumoniae carbapenemase
LACEN
Laboratório Central de Saúde Pública Dr. Giovanni Cysneiros
LB
Lúria Bertani
LVX
Levofloxacina
MDR
Multidroga resistente
xii
MER
Meropenem
mg
Miligrama
MIR
Enzima β-lactamase mediada por plasmídeo
mL
Mililitro
mM
miliMolar
MYSTIC
Programa de coleção de informações do teste anual de susceptibilidade
ao imipenem (do inglês, Meropenem Yearly Susceptibility Test
Information Collection)
NaCl
Cloreto de Sódio
NaOH
Hidróxido de Sódio
NMC-A
Enzima β-lactamase que hidrolisa carbapenemases da classe A de
Ambler (do inglês, Not Metalloenzyme Carbapenemase)
OD
Densidade óptica (do inglês, Optic Density)
OMP
Proteína de Membrana Externa (do inglês, Outer Membrane Protein)
OXA
Enzima oxacilinase
pb
Pares de base
PBP
Proteínas que se ligam à penicilina (do inglês, Penicillin Bilding Protein)
PBS
Tampão fosfato-salino (do inglês, Phosphate Buffered Saline)
PCR
Reação em Cadeia da Polimerase (do inglês, Polymerase Chain
Reaction)
PDR
Pandroga resistente
pH
Potencial hidrogeniônico
PIP/TAZ
Piperacilina/tazobactam
POL
Polimixina B/colistina
PSE
Enzima específica de Pseudomonas (do inglês, Pseudomonas specífic
enzyme)
rDNA
DNA ribossomal (do inglês, ribosomal DNA)
RNA
Ácido Ribonucleico (do inglês, RiboNucleic Acid)
RNAse
Ribonuclease
xiii
rpoB
Subunidade β da RNA polimerase (do inglês, RNA polymerase betasubunit)
SDS
Dodecil Sulfato de Sódio (do inglês, sodium dodecyl sulfate)
SENTRY
Programa Internacional de Vigilância em Microbiologia (do inglês,
Worldwide Antimicrobial Surveillance Program)
SHV
Cefalosporinase da família sulfidril variável (do inglês, Sulfhydryl
variable)
SIM
Imipenase descrita em Seoul (do inglês, Imipenase Seoul)
SME
Enzima específica de Serratia marcescens (do inglês, Serratia
marcescens enzyme)
SPM
Metallo-β-lactamase descrita em São Paulo (do inglês, São Paulo
metallo-β-lactamase)
TCLE
Termo de Consentimento Livre e Esclarecido
TE
Tris EDTA
TEM
Enzima β-lactamase pertencente à família Temonieira
TET
Tetraciclina
TGC
Tigeciclina
TSA
Ágar de soja tríptica (do inglês, Triptic Soy Agar)
TSB
Caldo de soja tríptica (do inglês, Triptic Soy Broth)
UTI
Unidade de Terapia Intensiva
VIM
Imipenemase descrita em Verona (do inglês, Verona imipenemase)
xiv
RESUMO
A espécie Acinetobacter baumannii é a representante mais importante do gênero, sendo
considerado o patógeno de maior relevância, dentro do complexo A. baumannii – A.
calcoaceticus, para as instituições de saúde mundialmente. Sua importância clínica tem
aumentado nos últimos anos em parte devido a sua notável capacidade de aderência a
superfícies devido a presença de estruturas como pili, produção de EPS
(exopolissacarídeo) e presença de genes que favorecem a formação de biofilme,
proporcionando maior adesão entre as células e consequente resistência aos
antimicrobianos. Outro destaque é sua capacidade de regular ou adquirir resistência, em
particular por meio das oxacilinases, codificadas pelo gene OXA, que apresentam
atividade hidrolítica potente contra penicilinas resistentes às penicilinases. Até o
momento, 250 tipos de OXA já foram descritas. A expressão de genes OXA pode ser
aumentada na presença do elemento de inserção ISAba1, localizado à montante do gene,
conferindo uma vantagem seletiva para o isolado quando este depende da expressão
eficiente de uma variedade de genes de resistência a antibióticos. Na sua ausência, a
resistência geralmente é expressa na presença de dois ou mais genes OXA. A
capacidade de formar biofilme e a presença dos genes blaPER-1, OXA-23, OXA-40,
OXA-51, OXA-58 e ISAba1 foram investigados em 84 isolados provenientes de 64
pacientes de UTIs de Goiânia. Observou-se que 78,5% dos isolados formaram biofilme,
dos quais 23% apresentaram o gene blaPER-1. A presença do gene OXA-51 foi
detectada em todos os isolados, enquanto que 64,3% dos isolados apresentavam o gene
OXA-23, e em 24% dos isolados foi encontrado o gene OXA-58. O gene ISAba1 foi
detectado à montante dos genes OXA-51, OXA-23 e OXA-58, em 33,3% (28/84),
92,6% (50/54) e 10% (2/20) dos isolados, respectivamente. Nenhum isolado estudado
apresentou o gene OXA-40. Dos 62 isolados resistentes ao imipenem e meropenem,
76% dos isolados possuíam o gene OXA-23 enquanto que 24,2% possuíam o gene
OXA-58. Dentre estes isolados, 79% (49/62) apresentaram a sequência de inserção
ISAba1. No presente estudo, a presença do gene blaPER-1 não foi associada com a
formação de biofilmes, enquanto que a presença de genes OXA estava relacionada à
resistência em Acinetobacter baumannii.
xv
ABSTRACT
The species Acinetobacter baumannii is the most important representative of the genus,
being globally considered the pathogen of larger relevance, within the complex A.
baumannii – A. calcoaceticus, to healthcare institutions worldwide. Its clinical
significance has increased in recent years partly due to its notable capacity of adherence
to surfaces due to presence of structures such as pili, production of EPS
(exopolysaccharides) and presence of genes that favor biofilm formation, providing
larger adhesion between the cells and consequent resistance to the antimicrobials.
Another important characteristic is its capacity to regulate or to acquire antibiotic
resistance, specifically through oxacilinases, encoded by the gene OXA, that presents
potent hydrolytic activity against penicilinase-resistant penicillins. Until the moment,
250 types of OXA were already described. The expression of OXA genes can be
increased in the presence of the insertion element ISAba1, which when located upstream
of the gene, confer a selective advantage for the isolate when this depends on the
efficient expression of a variety of resistance genes. In its absence, the resistance is
usually expressed in the presence of two or more OXA genes. The capacity of biofilm
formation and the presence of the genes blaPER-1, OXA-23, OXA-40, OXA-51, OXA58 and ISAba1 were investigated among 84 isolates from 64 patients of the Intensive
Care Units of Goiânia. It was observed that 78.5% of the isolates formed biofilm, of
which 23% presented the gene blaPER-1. The presence of OXA-51 gene was detected
in all isolates, whereas 64.3% of the isolates had the OXA-23 gene and 24.0% of the
isolates had the OXA-58 gene. The ISAba1 gene was detected upstream of the OXA-51,
OXA-23 and OXA-58 genes in 33.3% (28/84), 92.6% (50/54) and 10.0% (2/20) of the
isolates, respectively. None of the isolates studied presented the gene OXA-40. Of the
62 isolates resistant to imipenem and meropenem, 76.0% possessed the OXA-23 gene
while 24.2% of the isolates possessed the OXA-58 gene. Of these isolates, 79.0%
(49/62) presented the gene ISAba1. In the present study, the blaPER-1 gene presence
was not associated with the biofilm formation, while the presence of OXA genes was
related to resistance in Acinetobacter baumannii.
xvi
1 INTRODUÇÃO / REVISÃO DA LITERATURA
1.1 TAXONOMIA do Acinetobacter baumannii
A história da descrição do gênero Acinetobacter remonta ao início do século 20, em
1911, quando Beijerinck, um microbiologista holandês, descreveu um organismo
chamado Micrococcus calcoaceticus que foi isolado do solo por enriquecimento em um
meio contendo acetato de cálcio. Nas décadas seguintes, organismos semelhantes foram
descritos e atribuídos a pelo menos 15 gêneros e espécies diferentes, incluindo
Diplococcus mucosus, Micrococcus calcoaceticus, Alcaligenes haemolysans, Mima
polymorpha, Moraxella lwoffii, Herellea vaginocola, Bacterium anitratum, Neisseria
winogradskyi, Achromobacter anitratus e Achromobacter mucosus (Peleg et al. 2008).
A designação atual do gênero, Acinetobacter (do grego akineto, ou seja, sem
motilidade), foi inicialmente proposta por Brisou e Prévot em 1954 para separar microorganismos móveis de não-móveis dentro do gênero Achromobacter. Antes mesmo de
1968, esta designação do gênero tornou-se mais amplamente aceita. Baumann et al.
(1968) publicou uma pesquisa abrangente e concluiu que as diferentes espécies listadas
acima pertenciam ao mesmo gênero, para o qual o nome de Acinetobacter havia sido
proposto e que a subclassificação entre espécies diferentes com base em características
fenotípicas ainda não era possível. Estas descobertas resultaram no reconhecimento
oficial do gênero Acinetobacter pela Subcomissão Sobre a Taxonomia de Bactéria
Moraxella e afins em 1971 (Lessel 1971 apud Peleg et al. 2008). Na edição de 1974 do
Bergey’s Manual of Systematic Bacteriology (Lautrop 1974 apud Peleg et al. 2008) o
gênero Acinetobacer foi listado com a descrição de uma única espécie, Acinetobacter
calcoaceticus, no qual foi recomendado que o gênero compreendesse apenas isolados
oxidase-negativos. Estudos posteriores incluíram A. lwoffi no gênero, com base na
observação de que algumas cepas eram capazes de acidificar a glicose e outras não
(Skerman et al.1980 apud Peleg et al. 2008).
Com base em dados taxonômicos mais recentes, foi proposto que os membros do
gênero Acinetobacter, como ficou definido, deveria ser retirado da família
Neisseriaceae e classificados na nova família Moraxellaceae (Bergogne-Berezin &
Towner 1996; Giamarellou et al. 2008), a qual inclui Moraxella, Acinetobacter,
Psychrobacter e organismos relacionados (Rossau et al. 1991). Um grande avanço na
1
longa e complicada história do gênero foi realizado em 1986 por Bouvet e Grimont que,
baseado na hibridização do DNA, foram delineados 12 grupos de DNA (genoespécies)
de Acinetobacter, dos quais quatro novas espécies foram propostas, A. baumannii, A.
haemolyticus, A. johnsonii e A. junii e a descrição das espécies já conhecidas, A.
calcoaceticus e A. lwoffii, foram feitas (Bouvet & Grimont 1986).
Desde a década de 1980, em paralelo com a descrição de Acinetobacter como
patógeno nosocomial, a taxonomia do gênero tem sido refinada (Dijkshoorn et al.
2007). Até o ano de 2010, o gênero abrangia 19 espécies com nomes validamente
publicados e uma série de espécies com denominações provisórias (Nemec et al. 2010).
Com a recente descrição de Acinetobacter bereziniae (genoespécie 10), A. guillouiae
(genoespécie 11), o gênero compreende 23 espécies nomeadas e 11 espécies com
denominações provisórias (de Breij et al. 2010; Nemec et al. 2010).
Diversos autores relatam pequenas diferenças genômicas entre as espécies do
gênero Acinetobacter, no entanto as espécies 1 (A. calcoaceticus), 2 (A.baumannii),
genoespécie 3 e genoespécie 13TU apresentam uma relação muito estreita, recebendo a
denominação de complexo A. calcoaceticus – A. baumannii devido à dificuldade na
separação destas espécies a partir de testes convencionais (Bouvet & Grimont 1986;
Gerner-Smidt et al. 1991; Bergogne-Berezin & Towner 1996). Este complexo contém
isolados que são, em sua maioria, acidificantes da glicose (Bergogne-Berezin& Towner
1996) e são responsáveis pela maioria das infecções adquiridas quer na comunidade,
quer no hospital (Peleg et al. 2008). A. baumannii é a espécie de maior importância na
medicina humana dentro do complexo A. baumannii – A. calcoaceticus (Tomaras et al.
2003).
1.2 Acinetobacter baumannii E PATOGÊNESE
Em 1986, um sistema de identificação fenotípica de espécies de Acinetobacter foi
descrito por Bouvet e Grimont, possibilitando a identificação de várias espécies do
gênero, em particular o complexo A. calcoaceticus – A. baumannii (Bouvet & Grimont
1986; Dijkshoorn et al. 2007).
A espécie Acinetobacter baumannii é o representante mais importante, sendo
considerado o patógeno mais relevante para as instituições de saúde mundialmente
(Peleg et al. 2008). São persistentes no ambiente hospitalar e causam uma variedade de
infecções nosocomiais oportunistas (Loehfelm et al. 2008), incluindo infecções no trato
2
respiratório ou urinário, meningite, endocardite, bacteremia, especialmente em pacientes
internados em Unidades de Terapia Intensiva (UTIs), unidades neonatais e enfermarias
de neurocirurgia (Tomaras et al. 2003; Lee et al. 2008; Rao et al. 2008; Nucleo et al.
2009; Tiwari et al. 2012). Frequentemente causam infecções associadas a dispositivos
médicos como, por exemplo, catéteres vasculares, derivações liquóricas, catéteres de
Foley bem como pneumonia associada à ventilação mecânica (Rodriguez-Bano et al.
2008). Sua significância clínica tem sido justificada por sua alta prevalência em
epidemias e situações endêmicas, por sua notável capacidade de regular ou adquirir
resistência aos antibióticos, à dessecação e sua capacidade de formar biofilmes em
dispositivos médicos, contribuindo para a sua persistência no cenário hospitalar
(Roberts et al. 2001; Peleg et al. 2008; de Breij et al. 2010).
O uso extensivo de terapias antimicrobianas em hospitais tem contribuído para o
surgimento e aumento no número de isolados resistentes a uma ampla gama de
antimicrobianos, tornando-se um organismo que ameaça a eficácia dos mesmos (Zarrilli
et al. 2004; Peleg et al. 2008; de Breij et al. 2010).
A. baumannii é responsável por surtos de infecções hospitalares repentinos e de
difícil controle. As circunstâncias locais das unidades de saúde e seu ambiente
determinam o tipo de infecção e, consequentemente, o risco de contaminação, podendo
contribuir para o surgimento de um surto de infecções hospitalares. Esta bactéria é
considerada um agente patogênico de baixa virulência, podendo habitar sobre ou dentro
do corpo humano sem causar doença. Assim, a disseminação por meio das mãos da
equipe de saúde frequentemente não é detectada. Pacientes com sintomas de infecção
por Acinetobacter representam a ponta do iceberg da colonização e disseminação.
Quando as infecções tornam-se aparentes, é provável que o número de pacientes
colonizados já esteja elevado, de modo que se tornam tardias as precauções para evitar
um surto. Uma vez que um surto está estabelecido, todas as superfícies inanimadas no
ambiente podem ser reservatórios de Acinetobacter (Joly-Guillou 2005).
3
1.3 IDENTIFICAÇÃO LABORATORIAL DE Acinetobacter baumannii
Acinetobacter baumannii compreende bactérias em forma cocobacilos Gramnegativas, ubíquas, aeróbias estritas, catalase-positiva e oxidase-negativa, imóveis
(Bergogne-Berezin & Towner 1996), não fastidiosas, não fermentadoras de glicose
(Peleg et al. 2008), que podem ser recuperadas a partir de diversas fontes, tais como o
solo, água, produtos alimentares e ambientes médicos (Tomaras et al. 2003).
Crescem bem em ágar sangue onde formam colônias branco-acinzentadas ou em
ágar MacConkey com colônias levemente rosadas normalmente cremosas, mas que
podem eventualmente ter aspecto mucóide (Peleg et al. 2008). A. baumannii tem
requisitos de crescimento simples explorando uma variedade de fontes de alimentação e
é adaptável a diferentes temperatura, teores de pH, salinidade e níveis de umidade
(Iacono et al. 2008). Suas colônias assemelham-se às de Enterobacteriaceae, com um
diâmetro de 1,5 a 3 cm após 18 horas de crescimento em meios sólidos, enquanto que a
maioria das outras espécies de Acinetobacter produz colônias menores e mais
translúcidas (Peleg et al. 2008).
1.3.1 Identificação Fenotípica
A identificação bioquímica de A. baumannii compreende provas bioquímicas
básicas, como a fermentação de glicose, redução de nitrato a nitrito, detecção de
hemólise em ágar sangue de carneiro, utilização de citrato e outras fontes de carbono, e
principalmente crescimento em caldo de infusão de cérebro e coração a 42°C (tabela 1)
(Bouvet & Grimont 1986).
Tabela 1. Provas bioquímicas de identificação das espécies de Acinetobacter.
OXI
MOT
OFG
Cresc.
a 42°C
CIT
A. baumannii
N
N
O
P
P
P
N
N
A. calcoaceticus
N
N
O
N
P
P
N
N
A. haemolyticus
N
N
I/O
N
P
N
P
P
A. lwoffii
N
N
I
N
N
N
N
N
Acinetobacter sp
N
N
I/O
N
P
V
V
V
Micro-organismos
MAL GEL HEM
OXI: oxidase; MOT: motilidade;
CIT: citrato;
MAL: malonato;
GEL: gelatina
OFG: meio de oxidação fermentação da glicose de Leifson; HEM: hemólise em ágar sangue;
N: negativo; P: positivo;
O: oxidativo;
I: inerte;
I/O: inerte/oxidativo;
V: variável
Fonte: Adaptado de ANVISA (2004a).
4
Em particular, as espécies estreitamente relacionadas e clinicamente mais
relevantes A. baumannii e Acinetobacter genoespécie 13TU não podem ser distinguidas
por identificação fenotípica, enquanto que A. calcoaceticus e Acinetobacter genoespécie
3 só podem ser separadas por suas propriedades de crescimento à diferentes
temperaturas. Isto se deve à similaridade genética e fenotípica das espécies (Peleg et al.
2008).
1.3.2 Identificação Genotípica
O método de hibridização de DNA proposto inicialmente por Bouvet e Grimont,
1986, para identificação e separação do gênero Acinetobacter em espécies não tem sido
apropriado, uma vez que o complexo A. calcoaceticus – A. baumannii é altamente
semelhante, combinando três das espécies clínicas mais relevantes (A. baumannii,
genoespécie 3 e genoespécie 13TU) com uma espécie ambiental (A. calcoaceticus)
(Bouvet & Grimont 1986; Dijkshoorn et al. 2007).
A dificuldade em se distinguir o complexo A. calcoaceticus – A. baumannii têm
levado ao desenvolvimento de métodos genotípicos para a identificação de espécies de
Acinetobacter (Dijkshoorn et al. 2007). Entre eles estão a técnica de Reação em Cadeia
da
Polimerase
(PCR
–
Polimerase
Chain
Reaction)
utilizando
diferentes
oligonucleotídeos iniciadores; a análise do perfil de restrição do produto amplificado do
16S rDNA (ARDRA); a análise fingerprint de alta resolução por Amplified Fragment
Lenght Polymorphism (AFLP) e o sequenciamento da região intergênica do gene 16S23R e ou sequenciamento do gene que codifica a subunidade β da RNA polimerase
(rpoB) (Turton et al. 2006b; Dijkshoorn et al. 2007; Peleg et al. 2008).
A identificação molecular de espécies de Acinetobacter pode ser determinada pelo
sequenciamento de todo o genoma, análise de restrição de uma sequência particular de
DNA e da determinação da sequência de DNA. A figura 1 mostra dois métodos
genotípicos para a identificação de espécies de Acinetobacter: AFLP e ARDRA. AFLP
(figura 1a) compreende os seguintes passos: digestão do DNA celular com duas
enzimas de restrição; ligação dos adaptadores aos fragmentos de restrição; amplificação
seletiva dos fragmentos; eletroforese de fragmentos em um sequenciador; e visualização
dos perfis de restrição. Os perfis são comparados por análise de clusters àqueles de uma
biblioteca de referência de fingerprints de AFLP usando software específico. Este
método usado para a identificação de espécies é útil também para a identificação de
estirpes (tipagem) e clones. ARDRA (figura 1b) compreende a amplificação de
5
sequências de 16S rDNA, seguido de restrição dos fragmentos amplificados com cinco
enzimas de restrição e eletroforese. Os perfis resultantes são comparados com uma
biblioteca de perfis de restrição. Padrões de restrição ARDRA podem ser comparados
entre laboratórios. A análise da sequência de DNA, em particular da sequência 16S
rDNA, é uma abordagem amplamente utilizada para avaliar as relações entre os microorganismos, e os dados são universalmente comparáveis. No entanto, algumas espécies
Acinetobacter não relacionadas tem alta similaridade na sequência 16S rDNA, embora
algumas espécies estreitamente relacionadas são encontradas em diferentes ramos da
árvore filogenética 16S rDNA. Mais estudos são necessários para avaliar os níveis de
cut-off das semelhanças intra e interespécies (Dijkshoorn et al. 2007).
A identificação da espécie A. baumannii também pode ser confirmada pela
detecção da presença do gene OXA-51. Este gene foi identificado a partir de uma ampla
coleção de isolados A. baumannii coletados de diversas áreas geográficas. O gene
ocorre naturalmente no cromossomo de A. baumannii, sendo caracterizado como um
gene intrínseco à espécie (Brown et al. 2005; Heritier et al. 2005; Turton et al. 2006a;
Turton et al. 2006b; Feizabadi et al. 2008).
6
Figura 1. Métodos genotípicos para identificação de espécies de Acinetobacter: AFLP (a) e
ARDRA (b).
Fonte: Adaptado de Dijkshoorn et al. (2007)
7
1.4 PERFIL DE RESISTÊNCIA
Inicialmente, A. baumannii foi considerado um patógeno nosocomial de baixa
virulência, no entanto, estudos recentes têm mostrado que este micro-organismo é mais
virulento do que previamente relatado (Smani et al. 2012). O ambiente hospitalar pode
representar um importante reservatório para A. baumannii durante infecções
nosocomiais; em particular pacientes que permanecem em unidades de cuidado por
períodos prolongados, podem ser colonizados por esta bactéria e carregá-la por longos
períodos sem sintomas visíveis. A sua habilidade à persistência está relacionada à
resistência multidrogas, permitindo que a mesma sobreviva a terapias antimicrobianas
prolongadas em pacientes hospitalizados (Nucleo et al. 2009).
Os isolados clínicos de A. baumannii geralmente são multidroga resistentes às
tetraciclinas, fluoroquinolonas, aminoglicosídeos, maioria dos β-lactâmicos e alguns
carbapenêmicos (Dijkshoorn et al. 2007; Shahcheraghi et al. 2011). A resistência é
mediada por uma variedade de mecanismos (figura 2), como modificações do local
alvo, bombas de efluxo, inativação enzimática de antimicrobianos (Nucleo et al. 2009) e
presença de elementos como plasmídeos, integrons e transposons que são adquiridos,
sugestivamente, por transferência horizontal (Dijkshoorn et al. 2007; D'Arezzo et al.
2009).
A resistência aos aminoglicosídeos tem sido atribuída a pelo menos, nove distintas
enzimas modificadoras, que podem ser encontradas em diferentes combinações em
algumas estirpes. Mutações pontuais específicas nos genes que codificam a DNA-girase
e topoisomerase IV, tem sido correlacionadas com a resistência às fluoroquinolonas e a
resistência às tetraciclinas tem sido associada com genes que codificam as bombas de
efluxo tetraciclinas-específicas (Dijkshoorn et al. 2007). A produção de enzimas βlactamases tem sido relatada como um importante mecanismo de resistência aos
antibióticos β-lactâmicos, hidrolisando o anel β-lactâmico pela quebra da ligação amida,
perdendo assim, a capacidade de inibir a síntese da parede celular bacteriana (Williams
1999), neutralizando o efeito de antibióticos como o imipenem, meropenem e
penicilina. Estas enzimas são produzidas por bactérias Gram-negativas como um
importante meio de autodefesa contra antimicrobianos β-lactâmicos e A. baumannii não
é exceção (Tiwari et al. 2012).
Os carbapenêmicos são as drogas de escolha para o tratamento de infecções
causadas por bacilos Gram-negativos multirresistentes (Quale et al. 2003), mas a
8
resistência de A. baumannii aos mesmos tem aumentado em todo o mundo na última
década (Sohrabi et al. 2012).
Figura 2. Mecanismos potenciais de resistência antimicrobiana em Acinetobacter
O Acinetobacter, como outra bactéria Gram-negativa, tem uma membrana externa e uma
membrana citoplasmática, entre as quais (o espaço periplasmático) residem as β-lactamases
(carbapenemases, AmpC β-lactamases e β-lactamases de espectro estendido). As penicillinbinding proteins (PBPs), localizadas na membrana citoplasmática, constituem o alvo final dos
β-lactâmicos. Uma vez no espaço periplasmático, os β-lactâmicos se ligam às PBPs ou são
ativamente expelidos da estrutura bacteriana através de bombas de efluxo. Acinetobacter pode
abrigar integrons e transposons, elementos genéticos no cromossomo bacteriano ou em
plasmídeos, que podem carregar múltiplos genes de resistência que codificam a síntese de
enzimas (ex: β-lactamases de espetro estendido e metalo β-lactamases).
Fonte: Adaptado de Munoz-Price & Weinstein (2008)
A resistência aos carbapenêmicos em espécies de Acinetobacter ocorre devido à
combinação de vários mecanismos de resistência, incluindo a produção de β-lactamases
9
que pode estar associada com a perda de porinas de membrana externa e/ou superexpressão da bomba de efluxo ou, mais raramente, com a alteração das proteínas que se
ligam às penicilinas (PBPs – penicillin-binding protein) (Gur et al. 2008). A
combinação de diferentes mecanismos são classificados como enzimáticos e nãoenzimáticos, porém o mecanismo mais importante de resistência a esta classe de
antibióticos é a hidrólise enzimática, mediada por enzimas chamadas carbapenemases.
Estas enzimas pertencem a uma das três classes moleculares, de acordo com a
classificação de Ambler (figura 3) (Opazo et al. 2012). Vários estudos têm relacionado a
presença de enzimas metalo- β-lactamases com a resistência de A. baumannii aos
carbapenêmicos, mas as carbapenemases mais prevalentes nesta espécie são as βlactamases de classe D que hidrolisam carbapenêmicos (CHDLs, - carbapenemhidrolizing class D β-lactamases) (Mugnier et al. 2008; Opazo et al. 2012).
Figura 3. Divisão das carbapenemases em Classe A, B e D, de acordo com a classificação de
Ambler. As carbapenemases de classe D são representadas pelas Oxacilinases (OXA).
Fonte: Adaptado de Opazo et al. (2012)
As oxacilinases, também conhecidas como β-lactamase de classe D, são compostas
por enzimas com serina no sítio ativo assim como as β-lactamases de classes A e C de
Ambler, que diferem na estrutura de aminoácidos; enquanto as β-lactamases de classe B
são compostas por enzimas com zinco em seu sítio ativo. A maioria das enzimas de
classe D pertence ao grupo 2d do esquema de classificação funcional de β-lactamases de
Bush et al. (1995) (tabela 2). Entre as quatro classes moleculares, a classe D apresenta
maior diversidade de enzimas, a qual é observada tanto nos níveis genético como
10
bioquímico, com enzimas que possuem hidrólise quer de espectro estreito ou estendido.
Além disso, várias β-lactamases de classe D possuem atividade de espectro estendido,
resultante de mutações pontuais (Poirel et al. 2010).
Tabela 2. Sistema de classificação para bactérias produtoras de β-lactamases de acordo
com o substrato alvo.
Grupo Bush,
Jacoby e
Medeiros
Classe
Molecular
Ambler
1
C
Cefalosporinas
-
-
Enzimas AmpC de bactérias
Gram-negativas; MIR-1
2a
A
Penicilinas
+
-
Penicilinases de bactérias
Gram-positivas
2b
A
Penicilinas e Cefalosporinas
+
-
TEM-1, TEM-2, SHV-1
2be
A
Penicilinas, Cefalosporinas de
espectro reduzido, Cefalosporinas
de amplo-espectro e
Monobactâmicos
2br
A
Penicilinas
2c
A
Penicilinas e Carbenicilinas
2d
D
Penicilinas, Cloxacilina e
Carbapenêmicos
2e
A
Cefalosporinas
2f
A
3
B
4
Inibida por
Substrato alvo
Exemplo
ACa EDTA
TEM-3 a TEM-26, SHV-2 a
SHV-6, K1 K. oxytoca
+
+/-
-
TEM-30 a TEM-36, TRC-1
+
-
PSE-1, PSE-3, PSE-4
+/-
-
OXA-1 a OXA-11, PSE-2
(OXA-10)
+
-
Cefalosporinases induzíveis
de Proteus vulgaris
Penicilinas, Cefalosporinas e
Carbapenêmicos
+
-
NMC-A de E. cloacae, Sme1 de Serratia marcescens
Maioria β-lactâmicos, incluindo
Carbapenêmicos
-
+
L1 de Stenotrophomonas
maltophilia, CcrA de
Bacterioides fragilis
-
?
Penicilinases de B. cepacia
Não determinada Penicilinas
a
AC. Ácido Clavulânico
Fonte: Adaptado de Bush et al. (1995)
Há
diferentes
mecanismos
envolvidos
na
resistência
aos
antibióticos
carbapenêmicos, dentre eles a produção de diferentes β-lactamases do tipo OXA é a
mais prevalente (Sohrabi et al. 2012). A classe D (OXA) tem emergido como a principal
carbapenemase no mundo (Shahcheraghi et al. 2011).
O primeiro surto nosocomial com A. baumannii resistente aos carbapenêmicos
(CRAB – Carbapenem-Resistant A. baumannii) foi relatado nos Estados Unidos em
1991. Desde então, infecções por CRAB e surtos hospitalares têm sido relatados
11
mundialmente (Shahcheraghi et al. 2011). Surtos nosocomiais com isolados de
Acinetobacter que produzem enzimas de resistência OXA tem sido relatados no Brasil,
Polinésia Francesa, Espanha, Sudeste Europeu, Coréia e Argentina (Feizabadi et al.
2008).
1.4.1 β-lactamases de classe D
Originalmente, a denominação oxacilinase (OXA), deveu-se à capacidade deste
grupo de carbapenemases de hidrolisar mais rapidamente a oxacilina, cloxacilina e a
meticilina do que as penicilinas clássicas e ao fato de não serem inibidas pelo ácido
clavulânico e EDTA (Heritier et al. 2005; Opazo et al. 2012). Hoje essa definição não é
válida, uma vez que tem sido descrito, recentemente, que as enzimas inativam
fracamente a cloxacilina e a oxacilina (Opazo et al. 2012) e o grupo é fracamente
inibido pelo ácido clavulânico, tazobactam e sulbactam, enquanto que a sua atividade
pode ser inibida por NaCl in vitro (Heritier et al. 2005; Poirel et al. 2010). Embora
identificados primeiramente em plasmídeos, tem sido demonstrado que muitas espécies
Gram-negativas possuem naturalmente em seus genomas os genes que codificam as
OXA sendo, portanto, consideradas enzimas residentes (Poirel et al. 2010).
Baseado na identidade de suas sequências de aminoácidos, as carbapenemases
foram divididas em grupos filogenéticos (figura 4), dos quais OXA-23, OXA-40, OXA51 e OXA-58 têm sido identificados em A. baumannii.
12
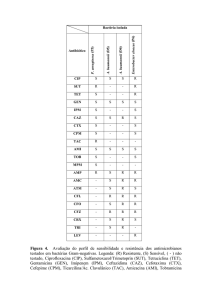
Figura 4. Dendrograma obtido para 73 β-lactamases de classe D de acordo com Phillip
(clustalW). Os comprimentos de ramificação à escala são proporcionais ao número de alterações
no aminoácido. A distância ao longo do eixo vertical não tem qualquer significado. Os
diferentes clusters identificados, permitiram a identificação de nove grupos principais,
considerando que as proteínas de um mesmo grupo têm uma identidade maior que 80% na
sequência de aminoácidos.
Fonte: Adaptado de Poirel et al. (2010)
Até
o
momento,
já
foram
descritas
250
tipos
de
OXA
(http://www.lahey.org/Studies/other.asp#table1) as quais apresentam grande divergência em
termos de propriedades bioquímicas. Enquanto algumas enzimas do tipo OXA possuem
espectro hidrolítico restrito às penicilinas e cefalosporinas de primeira e segunda
geração (oxacilinases de espectro limitado), outras são capazes de hidrolisar
13
cefalosporinas de terceira e quarta gerações (ESBL do tipo OXA), e certas variantes
ainda apresentam atividade hidrolítica contra o imipenem (carbapenemases do tipo
OXA) (Picão 2009).
1.4.1.1 β-lactamase OXA-23
A primeira descrição de OXA em A. baumannii foi em 1985, em um hospital
escocês. Inicialmente recebeu o nome de ARI-1 (Acinetobacter Resistent to Imipenem);
mais tarde seu gene foi sequenciado e designado como OXA-23 (Opazo et al. 2012;
Sohrabi et al. 2012). Desde então, essa enzima tem sido relatada em diferentes partes do
mundo (Feizabadi et al. 2008; Sohrabi et al. 2012), porém somente em Acinetobacter
sp., com uma única exceção, uma vez que este gene também foi identificado no
cromossomo de um Proteus mirabilis resistente a carbapenêmicos isolado na França
(Poirel et al. 2010). A enzima apresenta resistência ao ácido clavulânico (Nordmann &
Poirel 2002) e o gene pode estar localizado no cromossomo ou no plasmídeo de A.
baumannii (Mugnier et al. 2008; Mugnier et al. 2010). De acordo com os resultados
apresentados por Poirel e Nordmann, as medições hidrolíticas mostraram baixa
atividade de OXA-23 contra o imipenem; no entanto, em comparação com outras βlactamases de classe D, esta hidrólise foi significativa e permitiu definir o novo
subgrupo de OXAs, atualmente chamado CHDL (Poirel & Nordmann 2006a). Brown
detectou em seus experimentos uma lenta hidrólise do imipenem pelo OXA-23, mas não
detectou hidrólise do meropenem. O mesmo foi observado com OXA-40 e OXA-51
(Brown et al. 2005).
1.4.1.2 β-lactamase OXA-40
O grupo OXA-40 (inicialmente denominado OXA-24 devido a um erro de
sequenciamento
inicial,
ver
http://www.lahey.org/Studies/)
foi
originalmente
identificado no cromossomo de um isolado de A. baumannii resistente a
carbapenêmicos na Espanha. Em seguida o gene foi identificado em diferentes áreas,
especialmente em Portugal, na Espanha e também nos Estados Unidos. O gene tem sido
identificado no cromossomo ou no plasmídeo (Brown et al. 2005; Poirel et al. 2010). O
perfil de atividade hidrolítica é restrito, inclui principalmente as penicilinas. A hidrólise
do imipenem é de baixo nível, e a do meropenem não foi detectada (Joly-Guillou 2005).
O OXA-40 apresenta 60% de similaridade com a sequência de aminoácido do OXA-23
e não é inibido por NaCl (Poirel et al. 2010; Sohrabi et al. 2012).
14
1.4.1.3 β-lactamase OXA-51
Recentes relatos de diferentes países têm mostrado que o gene OXA-51 é intrínseco
à espécie A. baumannii, sendo encontrado no cromossomo desta espécie (Turton et al.
2006a; Turton et al. 2006b; Feizabadi et al. 2008; Gur et al. 2008). Esta enzima não
contribui significativamente para o padrão de resistência natural observada em A.
baumannii, apresentando fraca atividade carbapenemases (Hu et al. 2007; Poirel et al.
2010). Takagi et al. (2009) relataram a combinação entre a expressão deste gene como
os mecanismos responsáveis pela resistência ao imipenem. Nenhuma das cefalosporinas
é hidrolisada por OXA-51, com exceção da cefaloridina. A hidrólise do imipenem é
lenta e meropenem não é hidrolisado por esta enzima (Brown et al. 2005). Os genes que
codificam OXA-51 e derivativos, foram identificados a partir de uma grande coleção de
isolados de A. baumannii recuperados de amplas áreas geográficas. Provavelmente, a
maioria das variantes de OXA-51, se não todas, partilham propriedades bioquímicas
idênticas (Poirel et al. 2010).
1.4.1.4 β-lactamase OXA-58
A ocorrência de OXA-58 em Acinetobacter sp. é generalizada e consistentemente
associada com a resistência não somente aos carbapenêmicos, mas também a muitos
outros antimicrobianos, assim com β-lactâmicos, fluoroquinolonas e aminoglicosídeos
(Figueiredo et al. 2011). Esta β-lactamase hidrolisa penicilinas e carbapenêmicos em um
nível baixo. Foi identificada pela primeira vez na França, em um isolado A. baumannii
multidroga resistente (Poirel et al. 2010; Sohrabi et al. 2012) e frequentemente é
mediada por plasmídeo, apresenta similaridade superior a 50% com as sequências de
aminoácidos dos outros três grupos (Sohrabi et al. 2012). Turton et al. (2006a)
destacaram em seus resultados a resistência de OXA-58 ao imipenem e meropenem.
Feizabadi et al. (2008) concluíram em seus estudos que a resistência aos
carbapenêmicos está relacionada com a presença de, no mínimo, dois genes do tipo
OXA.
15
1.4.2 Sequência de inserção ISAba1
As sequências de inserção (IS – Insertion Sequence) são os menores e mais
abundantes elementos transponíveis. Estes elementos podem conter fortes promotores
que desempenham um papel importante na expressão dos genes de resistência a
antibióticos, os quais estão localizados próximos ao local de inserção destes elementos.
Recentemente uma sequência de inserção foi identificada em A. baumannii, denominada
ISAba1 ( Insertion Sequence Acinetobacter baumannii) (Sohrabi et al. 2012).
Diferentes IS têm sido descritas em Acinetobacter, cada uma em associação com
genes específicos, desempenhando importante papel no desenvolvimento da resistência
aos carbapenêmicos (Poirel & Nordmann 2006a, 2006b; Turton et al. 2006a; Feizabadi
et al. 2008; Mugnier et al. 2008; Sohrabi et al. 2012). As mais conhecidas são ISAba1,
ISAba2, ISAba3 e ISAba4, onde ISAba1 parece estar relacionada unicamente com a
espécie A. baumannii (Peleg et al. 2008).
A introdução da sequência de inserção ISAba1 pode agir como um forte promotor
aumentando a expressão dos genes (Poirel & Nordmann 2006a; Turton et al. 2006a;
Sohrabi et al. 2012;). Os grupos OXA-23, OXA-51 e OXA-58 possuem com frequência
associação com esses IS. Dentre eles, o OXA-23 é o mais comum e frequentemente
associado aos ISAba1 e ISAba4 (Poirel & Nordmann 2006a; Turton et al. 2006a;
Bogaerts et al. 2008; Mugnier et al. 2010;; Sohrabi et al. 2012). Tem sido relatado a
associação do grupo OXA-51 com os elementos de inserção ISAba1 e ISAba9,
proporcionando sequências promotoras para a expressão de alto nível desta enzima
(Turton et al. 2006a; Figueiredo et al. 2009; Sohrabi et al. 2012). As sequências
promotoras que desempenham importante papel na expressão de OXA-58 foram
descritas como ISAba1, ISAba2 e ISAba3 (Poirel & Nordmann 2006a, 2006b; Turton et
al. 2006a; Bertini et al. 2007; Gur et al. 2008; Sohrabi et al. 2012). A diminuição da
susceptibilidade a carbapenêmicos pode não estar relacionada à presença única de alelos
intrínsecos do tipo OXA-51, sendo uma explicação plausível a regulação mediada por
sequências de inserção ISAba1 (Turton et al. 2006a). No entanto, a detecção de alelos
genéticos que codificam enzimas do tipo OXA-23 e OXA-58 estão associados com, no
mínimo, a diminuição da susceptibilidade aos carbapenêmicos (Woodford et al. 2006).
De acordo com Segal et al. (2005), a abundância de ISAba1 em Acinetobacter sp.
reflete a motilidade do elemento e indica que os eventos de transposição ocorram com
frequência. Sugerem ainda plasticidade do genoma do Acinetobacter como transposição
da sequência de inserção, podendo causar uma variedade de rearranjos genômicos.
16
1.4.3 blaPER-1
O gene blaPER-1 foi identificado pela primeira vez em 1993 na Turquia, em
isolados de Pseudomonas aeruginosa e mais tarde em Salmonella enterica sorovar
Typhimurium e isolados de Acinetobacter (Paterson & Bonomo 2005; Poirel et al.
2005). Um estudo conduzido na Turquia em 1996 revelou uma alta prevalência do gene
blaPER-1 entre isolados nosocomiais de Acinetobacter spp. (46%) e P. aeruginosa
(11%) (Vahaboglu et al. 1997).
Durante anos, pensou-se que a presença de genes que codificam β-lactamases PER
fosse significativa somente na Turquia (PER-1) e na Argentina (PER-2) (Danel et al.
1995; Bauernfeind et al. 1996). No entanto, a identificação do gene blaPER-1 tem sido
relatada recentemente em vários países da Europa (Miro et al. 2005; Hou et al. 2007;
Naas et al. 2007b), Coréia do Sul (Yong et al. 2003; Jeong et al. 2005), China (Hou et
al. 2007) e Japão (Yamano et al. 2006).
Este gene codifica uma enzima que está classificada na classe A de Ambler
(Nordmann & Naas 1994) e confere resistência às penicilinas, cefotaxima, aztreonam
monobactâmico, porém é sensível aos carbapenemas e cefamicinas. A sua atividade é
inibida pelo ácido clavulânico. É um gene comum entre as espécies de Acinetobacter
(Paterson & Bonomo 2005; Poirel et al. 2005).
Estudos recentes têm mostrado uma correlação positiva entre a formação de
biofilme e a presença da β-lactamase de espectro estendido (ESBL – extended spectrum
β-lactamase) codificada pelo gene blaPER-1 entre os isolados de A. baumannii (Sechi et
al. 2004; Lee et al. 2008).
1.5 FORMAÇÃO DE BIOFILMES
Os isolados de A. baumannii têm sido avaliados quanto à capacidade de sobreviver
longos períodos de tempo em superfícies abióticas com habilidade de tolerar a
dessecação (Roberts et al. 2001; Gaddy & Actis 2009), devido a fatores de virulência
como a habilidade em captar o ferro do meio ambiente, sobrevivendo em condições de
déficit de ferro, produção de cápsula polissacarídica, capacidade de aderência a diversas
superfícies e às células do epitélio respiratório por meio de estruturas como pili,
fímbrias, proteínas de membrana externa (OMP) e presença de genes relacionados à
formação de biofilme (Tomaras et al. 2003; Wroblewska et al. 2008; Smani et al. 2012).
17
A potencial habilidade de A. baumannii para formar biofilmes pode explicar sua
excelente resistência a antibióticos e propriedades de sobrevivência (Tomaras et al.
2003; Rao et al. 2008). Gaddy & Actis (2009) e Lee et al. (2008) relataram a formação
de biofilme de A. baumannii em poliestireno. Vidal et al. (1997) destacam que poucos
estudos têm mostrado a capacidade destes isolados clínicos em formar estruturas de
biofilme em superfícies de vidro. Biofilmes bacterianos, arranjo em que as células são
morfológica, metabólica e fisiologicamente diferentes de suas contrapartes planctônicas,
foram observados associados a dispositivos médicos pela primeira vez em 1980 quando,
por microscopia eletrônica, foram visualizadas bactérias depositadas sobre a superfície
de dispositivos de longa permanência, como catéteres intravenosos e marca-passos. A
formação de biofilme também tem sido observada em tubos de incubação, catéteres,
válvulas cardíacas artificiais, catéteres de diálise peritoneal, linhas de água e
instrumentos (figura 5) (Tomaras et al. 2003; Hall-Stoodley et al. 2004).
Os biofilmes são notoriamente difíceis de erradicar, sendo fonte de muitas
infecções crônicas. Um biofilme maduro pode tolerar uma concentração de antibióticos
na ordem de 10 a 100 vezes maior do que é necessário para matar as bactérias
planctônicas. Bactérias em biofilmes são resistentes à fagocitose, tornando
extremamente difícil a erradicação de biofilmes em hospedeiros (Mohamed & Huang
2007). Pouco se sabe sobre a formação de biofilme em A. baumannii (Rao et al. 2008;
Rodriguez-Bano et al. 2008).
18
Figura 5. Desenho esquemático mostrando três exemplos de possíveis pontos de entrada no
corpo para biofilmes infecciosos. O biofilme pode ser disseminado em todo o corpo, quer por
células individuais ou aglomerados de êmbolos protegidos, os quais podem ganhar a corrente
sanguínea próxima aos pontos de entrada (setas). Desprendimentos esporádicos podem levar a
ciclos de bacteremia.
Fonte: Adaptado de Hall-Stoodley et al. (2004).
19
1.5.1 Biofilme
Biofilme é uma comunidade microbiana séssil altamente estruturada (Tomaras et al.
2003; Lee et al. 2008), que é caracterizado por células que são irreversivelmente ligadas
a superfícies bióticas ou abióticas e incorporadas em uma matriz hidratada de
substâncias exopoliméricas, proteínas, polissacarídeos e ácidos nucleicos (Mohamed &
Huang 2007; Lee et al. 2008).
A formação de biofilme é um processo complexo que envolve o desenvolvimento
de fixação e imobilização sobre uma superfície, a interação célula-a-célula, a formação
de microcolônias, a formação de um biofilme confluente e o desenvolvimento de uma
estrutura tridimensional de biofilme. As bactérias em um biofilme se comportam de
maneira diferente de sua forma livre. A regulação da expressão do gene bacteriano em
resposta à densidade de população celular, chamado quorum-sensing, é realizado
através da produção de moléculas de sinais extracelulares, chamados autoindutores
(Mohamed & Huang 2007; de Breij et al. 2010).
Vários fatores ambientais regulam a formação do biofilme bacteriano, tais como
variações na disponibilidade de nutrientes, de oxigênio e de pH e a concentração de
metabólitos bacterianos, o que provoca diferenças entre os biofilmes, de modo que um
biofilme formado numa corrente difere de um formado sobre tecido biológico (Singh et
al. 2006).
No biofilme as bactérias são protegidas contra as defesas do hospedeiro e contra a
ação de antimicrobianos (Shannon et al. 2010).
1.5.2 Formação e maturação do biofilme
Biofilmes não são simplesmente um conjunto de células passivas que estão presas à
superfície, mas são sistemas biológicos complexos que possuem estrutura e são
dinâmicos (figura 6) (Hall-Stoodley et al. 2004). A formação do biofilme é geralmente
descrita como uma série de fases discretas em um ciclo de vida (figura 7), que se inicia
quando as células planctônicas aderem a uma superfície (Nadell et al. 2009). Após a
fixação inicial ocorre divisão binária, resultando na agregação das células aderidas
(Melchior et al. 2006). A aderência inicial geralmente é reversível, de modo que as
células podem se afastar da superfície se as condições alteram. Algumas bactérias
podem posicionar-se usando pili, permitindo a movimentação entre as bactérias e ao
longo do substrato sólido (Nadell et al. 2009).
20
Estes agregados de células primárias produzem exopolissacarídeos para facilitar a
agregação. Assim a fase inicial da formação de biofilme envolve duas etapas: a primeira
fase compreende a aderência das células a uma superfície, facilitada pelas adesinas
associadas à parede celular, que são produtos de diferentes genes. Nesta etapa ocorre a
liberação de autoindutores do quorum-sensing. A aderência às superfícies poliméricas
nativas é aumentada na presença de proteínas de matriz, incluindo fibronectina e
fibrinogênio. A segunda etapa é caracterizada por multiplicação celular e formação de
uma estrutura madura que consiste em muitas camadas de células, ligadas entre si por
polissacarídeos extracelulares. Finalmente no processo de maturação, os componentes
extracelulares interagem com as moléculas orgânicas e inorgânicas do meio
favorecendo a produção de glicocálice, ocorrendo ligações iônicas e protegendo as
células do biofilme (Melchior et al. 2006).
Figura 6. Conceituação de desenvolvimento de biofilme e comportamento dinâmico em cada
etapa da formação.
Fonte: Adaptado de Hall-Stoodley et al. (2004).
21
Figura 7. Representação esquemática das várias etapas de desenvolvimento de um biofilme de
acordo com o modelo aceito para Pseudomonas aeruginosa.
Fonte: Adaptado de Ghigo (2003)
1.5.3 Sistema quorum-sensing
Quorum-sensing é um mecanismo de sinalização célula-célula que se refere à
capacidade das bactérias para responder a moléculas químicas, chamadas autoindutores
(AI), no seu ambiente de uma forma dependente da dose. Os AI podem ser produzidos a
partir de bactérias de mesma espécie ou de gêneros diferentes (Kaper & Sperandio
2005).
Nos diferentes organismos que realizam o quorum-sensing, o processo segue
essencialmente as mesmas etapas. Quando a concentração de AI atinge um limiar
crítico, as bactérias detectam e respondem a este sinal, alterando a expressão do seu
gene alvo. Este fenômeno permite que as bactérias ajam como uma unidade coletiva
(Kaper & Sperandio 2005). O nível de especificidade e de controle é intrigante, dadas as
complexidades associadas com as comunidades microbianas: os biofilmes bacterianos
geralmente são formados por múltiplas espécies, onde cada uma das espécies pode
produzir múltiplos sinais, e o circuito de resposta utilizado por esses sinais também está
interligado (Jayaraman & Wood 2008).
A primeira caracterização de quorum-sensing foi na bactéria marinha Vibrio
fischeri. Esta espécie vive em associação com diferentes hospedeiros animais marinhos
e pode colonizar o órgão de luz do hospedeiro, na qual cresce a altas densidades
emitindo luminescência. Quando as bactérias encontram-se livres na água do mar, a
luminescência não é observada. Ou seja, V. fischeri produz um complexo enzima
22
luciferase codificado pelos genes luxCDABEGH que é responsável pela produção de
luz. No entanto, a transcrição do operon lux e subsequente produção de luz ocorrem
apenas em altas densidades de V. fischeri, e são reprimidos em baixas densidades
(Kaper & Sperandio 2005: Jayaraman & Wood 2008).
O quorum-sensing é mediado de forma semelhante ao de V. fischeri na maioria das
bactérias Gram-negativas. Esse sistema codifica uma gama de funções, incluindo a
produção de sideróforo, a divisão celular, a síntese de polissacarídeo e motilidade
(Jayaraman & Wood 2008; Christiaen et al. 2011).
1.5.4 Resistência e Formação de Biofilme
Os mecanismos de resistência a antibióticos conhecidos, tais como bombas de
efluxo, enzimas modificadoras e mutações alvo (Walsh 2000), não parecem ser
responsáveis pela proteção de bactérias em um biofilme. Mesmo bactérias sensíveis que
não têm uma base genética conhecida para resistência podem ter a sensibilidade
profundamente reduzida quando elas formam biofilme (Stewart & Costerton 2001).
Quando as bactérias estão dispersas de um biofilme, geralmente tornam-se rapidamente
suscetíveis a antibióticos (Anwar et al. 1989; Williams et al. 1997), o que sugere que a
resistência das bactérias em biofilme não é adquirida através de mutações ou elementos
genéticos móveis. Os mecanismos de resistência a antibióticos convencionais não são
suficientes para explicar a maioria dos casos de infecções de biofilme resistentes a
antibióticos, porém este fato não exclui a possibilidade de que os mecanismos de
resistência convencionais sejam expressos em biofilmes contribuindo para a resistência
a antibióticos. Resistência a antibióticos convencionais pode ser desenvolvida em
biofilmes tratados repetidamente, ou por um longo período de tempo, contribuindo para
a persistência de infecções com biofilmes (Bagge et al. 2000).
Os mecanismos de resistência aos antibióticos no biofilme bacteriano estão
começando a ser elucidados (Mah & O'Toole 2001); a figura 8 mostra três principais
hipóteses. A primeira hipótese é a possibilidade de lenta ou incompleta penetração do
antibiótico no biofilme. A mensuração da penetração de antibióticos em biofilmes in
vitro tem mostrado que alguns antibióticos permeiam facilmente o biofilme bacteriano
(Stewart 1996). Não existe nenhuma barreira genérica para a difusão de solutos, do
tamanho de antibióticos, através da matriz do biofilme, que é principalmente água
(Stewart 1998). No entanto, se o antibiótico é desativado no biofilme, a penetração pode
ser retardada. No biofilme tipo-selvagem, o antibiótico é desativado nas camadas
23
superficiais mais rapidamente do que se difunde. Antibióticos que adsorvem na matriz
do biofilme também podem ter uma penetração retardada, o que pode explicar a
penetração lenta de antibióticos aminoglicosídeos (Kumon et al. 1994; Shigeta et al.
1997). Esses agentes carregados positivamente se ligam a polímeros carregados
negativamente na matriz do biofilme (Gordon et al. 1988; Nichols et al. 1988).
A segunda hipótese depende de um microambiente químico alterado dentro do
biofilme. Microescala de gradientes em concentrações de nutrientes são uma
característica bem conhecida de biofilmes. Resultados de estudos com eletrodos
miniaturizados têm mostrado que o oxigênio pode ser totalmente consumido nas
camadas superficiais de biofilme, levando a nichos anaeróbios em camadas profundas
do biofilme (de Beer et al. 1994). Os aminoglicosídeos são menos eficazes contra o
mesmo organismo em condições anaeróbias do que em condições aeróbias (Tack &
Sabath 1985). Alternativamente, a depleção de substratos ou acúmulo de um produto
inibidor residual podem levar à inibição da expansão de algumas bactérias, protegendoas da morte. As penicilinas, que têm como alvo a síntese de parede celular, inibe apenas
o crescimento bacteriano (Tuomanen et al. 1986). Esta segunda hipótese é reforçada
pela visualização experimental direta de zonas metabolicamente inativas dentro de
biofilmes alimentados continuamente (Xu et al. 2000). Além disso, o ambiente
osmótico dentro de um biofilme pode ser alterado, levando à indução de uma resposta
de estresse osmótico (Prigent-Combaret et al. 1999). Tal resposta pode contribuir para
resistência aos antibióticos, alterando as proporções relativas das porinas de uma
maneira que reduz a permeabilidade do envelope celular aos antibióticos (Stewart &
Costerton 2001).
Um terceiro mecanismo da resistência aos antibióticos é que uma subpopulação
de micro-organismos num biofilme forma um único, e altamente protegido, estado
fenotípico – uma diferenciação celular semelhante à formação de esporos. Esta hipótese
está apoiada nos resultados que mostram que há resistência em biofilmes recémformados, mesmo que ainda sejam muito finos para representar uma barreira para a
penetração de um agente antimicrobiano ou substratos metabólicos (Das et al. 1998;
Cochran et al. 2000). Além disso, as bactérias no biofilme, mas não todas, são
rapidamente mortas por antibióticos (Goto et al. 1999; Brooun et al. 2000). As bactérias
sobreviventes, que podem consistir de 1% ou menos da população original, persistem
apesar da exposição continuada ao antibiótico. A hipótese de um estado letárgico
semelhante a esporos, aceita para algumas bactérias do biofilme, proporciona uma
24
poderosa e genérica explicação para a redução da susceptibilidade de biofilmes a
antibióticos e desinfetantes (Stewart & Costerton 2001).
Figura 8. Três hipóteses para os mecanismos de resistência a antibióticos em biofilmes. A
superfície de fixação é mostrada na parte inferior e a fase aquosa contendo o antibiótico, na
parte superior.
Fonte: Adaptado de Stewart & Costerton (2001)
25
1.6 EPIDEMIOLOGIA
As infecções por A. baumannii têm sido uma questão clínica substancial em muitas
partes da Europa (figura 9) (Van Looveren & Goossens 2004). Desde o início de 1980,
os surtos de infecções hospitalares por este patógeno na Europa, principalmente na
Inglaterra, França, Alemanha, Itália, Espanha e Holanda (Bergogne-Berezin & Towner
1996; Villegas & Hartstein 2003; Fournier & Richet 2006), foram investigados usando
métodos epidemiológicos de tipagem molecular. Na maioria dos casos, um ou dois
isolados epidêmicos foram detectados num dado contexto epidemiológico. A
transmissão de tais isolados tem sido observada entre hospitais, muito provavelmente
através de transferência de pacientes colonizados (Turton et al. 2004; van den Broek et
al. 2006; Da Silva et al. 2007).
Figura 9. Países que relataram surtos por Acinetobacter baumannii resistentes aos
carbapenêmicos. Em vermelho os surtos relatados antes de 2006, e em amarelo os surtos
relatados após 2006.
Fonte: Adaptado de Peleg et al. (2008)
A propagação de A. baumannii multidrogas resistentes (MDR) não se limita aos
hospitais dentro de uma cidade, mas também ocorre em escala nacional. Exemplos são a
propagação do clone denominado Sudeste e o OXA-23 clones 1 e 2 no Sudeste da
Inglaterra (Coelho et al. 2006; Turton et al. 2004), a disseminação de um clone A.
baumannii mutidroga resistente em Portugal (Da Silva et al. 2007), a propagação interhospitalar do clone A. baumannii produtora de VEB-1 de espectro estendido de um total
de 55 centros médicos no norte e sudeste da França (Naas et al. 2006) e da propagação
26
de um clone A. baumannii resistente à amicacina observada em nove hospitais em várias
regiões na Espanha (Vila et al. 1999). A transferência internacional de pacientes
colonizados tem levado à introdução e disseminação da epidemia subsequente de
isolados A. baumannii MDR do sul em países do norte da Europa, como a Bélgica e a
Alemanha (Schulte et al. 2005; Bogaerts et al. 2006). A propagação intercontinental
também foi descrita entre a Europa e outros países, como consequências de viagem de
avião (Peleg et al. 2006a; Naas et al. 2007a). Estes eventos destacam a importância do
rastreio adequado e possível isolamento de pacientes com altas taxas de organismos
resistentes às drogas.
Na América do Norte há um destaque para os Estados Unidos, onde no período de
1991 e 1992 foram observados surtos de CRAB em um hospital de Nova York (Griffith
et al. 2006). Os organismos neste surto foram MDR, mantendo a susceptibilidade
somente para polimixinas e ampicilina/sulbactam (Griffith et al. 2006). Outros hospitais
de Nova York tiveram surtos clonais de A. baumannii MDR ou pan-drogas resistentes
(PDR) (Durand et al. 1993; Jones et al. 2004; Griffith et al. 2006; Landman et al. 2007),
e surtos semelhantes têm sido frequentemente relatados em muitas outras regiões dos
Estados Unidos (Jones et al. 2004; Maslow et al. 2005; Lolans et al. 2006). Uma
importante contribuição para a epidemiologia das infecções com A. baumannii nos
Estados Unidos foi o retorno de militares que lutaram no Iraque ou no Afeganistão
(Davis et al. 2005; Murray et al. 2006; Scott et al. 2007). Um aumento nas infecções por
A. baumannii foi descrita pela primeira vez em militares americanos em março de 2003,
logo após que as operações de combate começaram no Iraque. Os militares mais
acometidos foram primeiro tratados em hospitais de campanha, antes de serem
encaminhados para o Centro Médico Regional Landstuhl (Alemanha) ou Walter Reed
Army Medical Center (Estados Unidos) (Scott et al. 2007). A maioria destas infecções
foi detectada no momento ou logo após a admissão a essas instituições (Murray et al.
2006; Scott et al. 2007).
Os dados sobre o grau de resistência de A. baumannii na África são em grande parte
limitados à África do Sul, embora hajam relatos esparsos de outros países (Iregbu et al.
2002; Nejjari et al. 2003). Brink et al. (2007) mostraram que cerca de 30% de isolados
de A. baumannii de corrente sanguínea de pacientes na África do Sul são resistentes aos
carbapenêmicos, mais de 40% são resistentes à cefepima e piperacilina/tazobactam, e
mais de 30% são resistentes à ciprofloxacina e levofloxacina. Esses isolados resistentes
27
são endêmicos em algumas unidades, por exemplo queimaduras e UTIs, e foram
disseminados inter institucionalmente (Marais et al. 2004)
Numerosos surtos têm sido documentados em hospitais na Ásia e Oriente Médio
(figura 9), e uma variedade de carbapenemases também tem sido descritas (Afzal-Shah
et al. 2001; Abbo et al. 2005; Ko et al. 2007). Taxas de resistência em isolados
SENTRY (2001 – 2004) na região Ásia-Pacífico, foram superiores a 25% para
imipenem
e
meropenem,
40%
para
cefepima
e
ceftazidima,
40%
para
ampicilina/sulbactam, 35% para amicacina e 45% para ciprofloxacina (Gales et al.
2006). A resistência para a tigeciclina (Navon-Venezia et al. 2007) e polimixina B
(Gales et al. 2006; Ko et al. 2007) já foi descrita para essa região.
O primeiro surto australiano descrito de A. baumannii adquirido em hospital foi na
Austrália Ocidental (Riley et al. 1996). Estes isolados foram resistentes à gentamicina,
cefalosporinas e ticarcilinas, com alguns isolados também resistentes à ciprofloxacina.
A análise epidemiológica molecular identificou que 11% das amostras provenientes das
mãos de funcionários foram positivas para o mesmo isolado de A. baumannii que
causou a infecção dos pacientes (Riley et al. 1996). Mais recentemente, surtos de A.
baumannii têm afetado outras grandes cidades ao longo da costa leste da Austrália,
incluindo Brisbane, Sydney e Melbourne (Peleg et al. 2006b; Playford et al. 2007;
Valenzuela et al. 2007). Estes surtos têm envolvido isolados CRAB, com OXA-23
contribuindo para este fenótipo (Valenzuela et al. 2007). Surtos de A. baumannii
resistentes a carbapenêmicos também ocorreram na Polinésia Francesa (Naas et al.
2005).
Segundo Turton et al. (2006b), as taxas de resistência ao imipenem, meropenem,
ceftazidima, piperacilina/tazobactam, ciprofloxacina e gentamicina na América Latina,
parecem estar entre as mais altas do mundo. Em estudo de vigilância envolvendo
Argentina, Brasil, Chile e Colômbia, de 1997 a 2001, as taxas de resistência foram mais
elevadas na Argentina, mas em todos os países foram isolados micro-organismos
multirreristentes (Tognim et al. 2004).
Outra questão é a detecção do gene blaPER-1, que desde 1995 tem se disseminado
na Itália (Docquier et al. 2001) e, mais recentemente, na Bélgica (Claeys et al. 2000),
França (De Champs et al. 2004), Espanha (Miro et al. 2005), Romênia (Naas et al.
2007b), Coréia (Jeong et al. 2005), Japão (Yamano et al. 2006) e China (Hou et al.
2007), sendo mais comumente encontrado em espécies de Pseudomonas aeruginosa. A
presença do gene blaPER-1 em espécies de Acinetobacter, tem sido relatada na Coréia,
28
codificando resistência à cefepima e ceftazidima (Yong et al. 2003) e na Turquia
conferindo resistência à ceftazidima, meropenem e imipenem (Kolayli et al. 2005).
No Brasil não existem muitos dados sobre infecções nosocomiais por
Acinetobacter. De acordo com Turrini & Santo (2002), mesmo com a legislação vigente
no país, os dados sobre infecção hospitalar são pouco divulgados pelo fato de não serem
consolidados por muitos hospitais, o que tem dificultado a aquisição de um diagnóstico
que retrate a dimensão real do problema no país. A Organização Mundial de Saúde
considera um índice médio aceitável de infecção hospitalar entre 9% a 20% por espécies
bacterianas. Segundo o Ministério da Saúde e uma pesquisa realizada e publicada em
1995 no Brasil, a taxa média de infecção hospitalar é cerca de 15%, porém este índice
varia significativamente, já que está diretamente relacionado com o grau de atendimento
e complexidade de cada hospital (ANVISA 2004b).
Em hospitais brasileiros, A. baumannii MDR constituem uma causa grave de
infecção hospitalar, compreendendo 8,8% do total de isolados bacterianos nosocomiais
que causam infecções em pacientes de UTI, de acordo com o Programa Brasil MYSTIC
(Meropenem Yearly Susceptibility Test Information Collection) (Kiffer et al. 2005). A
este respeito, os carbapenêmicos permanecem como a opção terapêutica de amplo
espectro para o tratamento de tais infecções. Contudo a resistência a esses agentes
antimicrobianos tem aumentado, resultando na utilização de agentes potencialmente
mais tóxicos, tais como as polimixinas (Fernandez-Cuenca et al. 2003). Embora altas
taxas de resistência aos carbapenêmicos entre isolados de Acinetobacter sp. tenham sido
relatadas, muito pouco se sabe sobre os mecanismos de resistência. De acordo com os
relatórios do Programa Brasil MYSTIC, A. baumannii foi o quarto patógeno mais
prevalente isolado de pacientes hospitalizados em 20 centros brasileiros e apresentou
altas taxas de resistência a todos os antimicrobianos testados (Kiffer et al. 2005).
Dalla-Costa et al. (2003) relataram um surto por CRAB produtores da enzima
OXA-23 em dois hospitais de Curitiba (PR), assim como Schimith Bier et al. (2010)
que detectou OXA-23 nos 172 isolados de CRAB , os quais apresentaram sensibilidade
somente à tigeciclina e polimixina B. Em estudo publicado por Carvalho et al. (2009),
foi comum a ocorrência de isolados A. baumannii MDR produtoras de OXA-23
disseminados em hospitais públicos e privados do Rio de Janeiro (RJ), as quais
apresentaram resistência ao meropenem, ciprofloxacina, amicacina, cefepima,
ceftazidima, piperacilina/tazobactam e ampicilina/sulbactam. A detecção desta enzima
em isolados de CRAB também foi relatada por Martins et al. (2009), em estudo
29
realizado na cidade de Porto Alegre (RS), onde todos apresentaram resistência ao
imipenem, meropenem, piperacilina/tazobactam, gentamicina e ciprofloxacina. Além de
detectar OXA-23 em todos os isolados, Werneck et al. (2011b) em estudo realizado em
oito cidades de quatro diferentes regiões geográficas brasileiras, também detectou a
sequência de inserção ISAba1 à montante de OXA-23 em todos os isolados.
A
resistência aos carbapenêmicos foi relatada para 83,5% dos isolados que possuíam
OXA-23, porém o autor não destaca quantos, dentre estes, possuíam ISAba1.
Até o momento, o único relato que se tem de OXA-40 no Brasil foi publicado por
Werneck et al. (2011a), o qual foi detectado em um isolado de A. baumannii, sendo este
o único isolado do estudo. O mesmo apresentou susceptibilidade à polimixina B e
colistina, redução de susceptibilidade para ampicilina/sulbactam e resistência para
ceftazidime, cefepime, imipenem, meropenem, ciprofloxacina e amicacina.
A detecção do gene OXA-58 no Brasil, Rio de Janeiro, foi relatada por Figueiredo
et al. (2011), apresentando resistência à amicacina, ceftazidima, ciprofloxacina,
cefepima, gentamicina, imipenem, meropenem e piperacilina/tazobactam e por Gusatti
et al. (2012), apresentando ISAba1 à montante do gene e expressando resistência ao
imipenem.
O gene OXA-51 foi detectado em todos os isolados de A. baumannii mencionados
nos estudos brasileiros supracitados.
Em Goiás foi detectado um surto de infecção hospitalar em fevereiro de 2009 pela
Superintendência de Vigilância Sanitária e Ambiental (Svisa), no Hospital de Urgências
de Aparecida de Goiânia (HUAPA). Foram 83 mortes no período de janeiro de 2009 a
janeiro de 2010. A taxa de infecção hospitalar chegou a 88,23% em abril de 2009. De
acordo com a Sociedade Brasileira de Terapia Intensiva (SOBRATI) a taxa de infecção
em todas as alas de um hospital deve chegar, no máximo, a 2% e nas UTI entre 10 e
15%. A contaminação se deve a vários tipos de bactérias, porém a mais comum, letal e
mais persistente nas UTI são as A. baumannii MDR (SVS 2010).
Um relatório técnico da Fundação Oswaldo Cruz (FIOCRUZ) caracterizou, através
de tipagem molecular, os isolados bacterianos de sua rede de monitoramento de
resistência e incluiu 540 amostras enviadas pelos Laboratórios Centrais do Brasil, entre
agosto/2009 e julho/2010. Das 540 amostras recebidas, 165 amostras eram isolados de
A. baumannii. Dos 165 isolados de A. baumannii, 89 amostras foram encaminhadas
pelo LACEN/GO: 13 amostras (de três hospitais de Goiânia) tinham 11 isolados
positivos para OXA-23, e 76 amostras (consideradas de um surto) tinham 66 isolados
30
positivos para OXA-23. Todas as 89 amostras enviadas pelo LACEN/GO pertenciam a
três hospitais públicos goianos e confirmam a presença de padrão genômico de
resistência aos carbapenêmicos no estado de Goiás (FIOCRUZ-Rio de Janeiro, 2010
apud Godoy 2012).
Não foram encontrados relatos sobre a detecção dos genes OXA-40, OXA-58 e
ISAba1 no Estado de Goiás, e do gene blaPER-1 no Brasil.
31
2 JUSTIFICATIVA
Acinetobacter baumannii é um importante patógeno adquirido em hospitais que
causa uma variedade de infecções em seres humanos, especialmente em pacientes
imunocomprometidos (Dalla-Costa et al. 2003; Dijkshoorn et al. 2007). Em hospitais
brasileiros, A. baumannii MDR constituem uma causa significativa de infecção
hospitalar (Kiffer et al. 2005). A resistência a múltiplas drogas mediadas por enzimas do
tipo OXA e a sua capacidade de formar biofilme sob condições de estresse, contribuem
para sua persistência no cenário hospitalar e representam importantes fatores de
virulência (Poirel & Nordmann 2006a, 2006b; Perez et al. 2007; Feizabadi et al. 2008).
Embora seja relevante, poucos estudos tem relacionado a presença do gene blaPER1 com a formação de biofilme, cujos resultados já apresentados têm sido conflitantes.
Sechi et al. (2004) relaciona a produção de blaPER-1 com a aderência, enquanto Lee et
al. (2008) sugere que a presença do gene seja altamente significativa para a aderência e
formação de biofilme. Em contrapartida, a ocorrência de CHDLs tem sido relatada entre
isolados clínicos de A. baumannii em todo o mundo. Os primeiros relatos de OXA-23,
OXA-40 e OXA-58 em CRAB no Brasil foram em 2003, 2010 e 2012, respectivamente
(Dalla-Costa et al. 2003; Werneck et al. 2011a; Gusatti et al. 2012).
Na tentativa de justificar a multirresistência dos isolados investigados, este estudo
analisou a capacidade de formação de biofilme em poliestireno a partir de 84 isolados
clínicos, provenientes de pacientes de cinco UTIs de quatro instituições de referência (3
públicas e 1 filantrópica), que adquiriram A. baumannii através de infecção hospitalar.
Através da técnica de biologia molecular de reação em cadeia da polimerase (PCR) foi
investigada a presença do gene blaPER-1, a qual foi correlacionada com a formação de
biofilme em poliestireno, e dos genes OXA-23, OXA-40, 0XA-51, OXA-58 e ISAba1,
cujas presenças foram relacionadas com a resistência aos antibióticos carbapenêmicos,
tais
como
imipenem,
piperacilina/tazobactam,
meropenem,
ceftazidima,
e
também
amicacina,
à
ampicilina/sulbactam,
gentamicina,
ciprofloxacina,
levofloxacina, polimixina B/colistina, tetraciclina, tigeciclina e cefepima.
Considerando que não foram encontrados relatos da identificação de alguns
destes genes em A. baumannii no Estado de Goiás, nem mesmo no Brasil para o gene
blaPER-1, e prevendo possível relevância destes achados, este estudo tem como
objetivo contribuir para um melhor entendimento da real prevalência das β-lactamases
32
nos hospitais goianos e, consequentemente, auxiliar nas ações de controle dos isolados e
na redução das infecções por A. baumannii.
Cabe ressaltar a importância deste estudo, de grande relevância para o sistema de
saúde, uma vez que os resultados apresentados possibilitam uma análise qualitativa,
comparativa e complementar a outras abordagens científicas do tema, auxiliando na
implementação de estratégias visando a redução dos surtos hospitalares, bem como na
conscientização dos profissionais de saúde quanto à necessidade de maior rigor na
prescrição de antimicrobianos, contribuindo assim para o bem estar da população.
33
3 OBJETIVOS
3.1 GERAL
Analisar a capacidade de formação de biofilme em poliestireno de isolados de
Acinetobacter baumannii e caracterizar, por biologia molecular, a presença dos
genes blaPER-1, blaOXA-23, blaOXA-40, blaOXA-51, blaOXA-58, ISAba1.
3.2 ESPECÍFICOS
Analisar a capacidade dos isolados formarem biofilme em poliestireno;
Investigar a presença do gene blaPER-1 nos isolados;
Avaliar a relação entre a formação (ou não) de biofilme e a presença do gene
blaPER-1;
Avaliar a relação entre a formação (ou não) de biofilme e a resistência aos
antibióticos;
Investigar a prevalência dos genes β-lactamases OXA-23, OXA-40, OXA-51 e
OXA-58 e a presença de ISAba1 à montante dos genes OXA;
Correlacionar a presença de ISAba1 com os níveis de resistência aos
carbapenêmicos.
34
4 MATERIAL E MÉTODOS
4.1 ASPECTOS ÉTICOS
As coletas das amostras usadas neste estudo foram realizadas de acordo com as
normas de conduta da ética médica, após assinatura do Termo de Consentimento Livre e
Esclarecido (TCLE), aprovado pelos Comitês de Ética em Pesquisa das instituições
envolvidas (Protocolos: CEPACCG N° 005/10; HC/UFG N° 070/2010 e HDT N°
002/2010), pelos pacientes e/ou seus responsáveis, nos quais estavam claros os
procedimentos e objetivos do projeto (Anexo I).
4.2 RECRUTAMENTO DOS CASOS E COLETAS DE AMOSTRAS CLÍNICAS
Trata-se de um estudo transversal de caracterização fenotípica e molecular de
isolados clínicos de Acinetobacter baumannii. Para este estudo foram usados os
isolados clínicos provenientes de um estudo anterior (Infecções por Acinetobacter
baumannii em adultos admitidos em Unidades de Terapia Intensiva (UTIs) de Goiânia
e Aparecida de Goiânia), onde foram coletadas 84 amostras de Acinetobacter
baumannii de 64 pacientes adultos no período de julho a dezembro de 2010 nos
seguintes locais:
UTI de atendimento clínico (UTI-1) e UTI de atendimento cirúrgico (UTI-2), com
oito leitos cada, pertencentes a um hospital universitário com 300 leitos;
UTI (UTI-3) de atendimento clínico com nove leitos, pertencente a um hospital
estadual de referência no tratamento de doenças infecciosas e parasitárias com 126
leitos;
UTI (UTI-4) de atendimento cirúrgico com nove leitos, pertencente a um hospital
filantrópico de referência em câncer no Estado de Goiás, com 211 leitos;
UTI (UTI-5) de atendimento clínico com dez leitos, pertencente a um hospital
estadual de referência em urgências com 75 leitos, em Aparecida de Goiânia.
35
4.3 ISOLAMENTO, IDENTIFICAÇÃO E SUSCEPTIBILIDADE MICROBIANA
A coleta dos espécimes biológicos ocorreu conforme a indicação da equipe
médica assistente, sendo que todas as UTIs possuíam assistência de médico
infectologista. As amostras foram encaminhadas e identificadas no laboratório de
referência local segundo a rotina de cada unidade hospitalar, por método automatizado
e/ou manual.
As amostras da UTI-3 e UTI-5 foram encaminhadas ao Laboratório Central de
Saúde Pública (LACEN) e as da UTI-4 para o laboratório do próprio hospital, onde as
culturas foram processadas pelo método manual, com isolamento da bactéria em ágar
MacConkey e identificação através da bacterioscopia com coloração pelo método de
Gram e por provas bioquímicas: motilidade (IAL), crescimento a 42°C, utilização do
citrato, OF-glicose, produção de oxidase, urease, hidrólise da bile-esculina, testes de
descarboxilação (lisina, ornitina e arginina) e OF-lactose a 10%. A avaliação da
susceptibilidade do A. baumannii foi realizada por disco-difusão e pelo E-test®
(polimixina com MIC ≥ 4 µg/ml para resistência) (ANVISA 2004a; CLSI 2009 apud
Godoy 2012).
As amostras dos pacientes das UTIs 1 e 2 foram enviadas para o laboratório
próprio do hospital, que utilizou o equipamento Vitek II (sistema automatizado) para
identificação da bactéria pela provas: DP-300, p-cumarico, controle de crescimento,
acetamida, esculina, indican vegetal, manitol, xilose, rafinose, sorbitol, H 2S, sacarose,
inositol,
adonitol,
raminose,
L-arabinose,
fermentação
de
ONPG,
glicose
(fermentativa), testes de descarboxilação (lisina, ornitina e arginina).
A determinação da susceptibilidade do A. baumannii foi realizada para os
seguintes
antimicrobianos:
piperacilina/tazobactam,
imipenem,
ceftazidima,
meropenem,
amicacina,
ampicilina/sulbactam,
gentamicina,
ciprofloxacina,
levofloxacina, polimixina B/colistina, tetraciclina, tigeciclina e cefepima. Todos os
laboratórios seguiram os padrões de susceptibilidade recomendados pelo Clinical and
Laboratorial Standards Institute 2009 (CLSI 2009 apud Godoy 2012).
Após os testes, confirmação e isolamento, as amostras de A. baumannii foram
encaminhadas para o Laboratório de Bacteriologia Molecular do Instituto de Patologia
Tropical e Saúde Pública (IPTSP)-UFG e armazenadas em freezer -80°C.
36
4.4 TESTE DE VIABILIDADE DE Acinetobacter baumannii
As amostras mantidas no freezer -80°C foram reativadas em meio de cultura
sólido TSA (Tryptic Soy Agar) e incubadas por um período aproximado de 16 horas a
uma temperatura de 37°C. Após incubação, o crescimento foi analisado
microscopicamente pela coloração de Gram, onde observou-se cocobacilos Gramnegativos, como esperado para confirmar a pureza das culturas de A. baumannii.
Com a confirmação, uma colônia isolada da placa com TSA foi repicada em meio
caldo TSB (Tryptic Soy Broth) e incubada nas mesmas condições supracitadas. Após
crescimento foram realizadas duas etapas, onde parte da cultura foi transferida para um
criotubo contendo glicerol 20%, na concentração de 1:1 (500 µL de cultura em 500 µL
de glicerol 20%) e armazenada em freezer -20°C. Na segunda etapa, foi realizada a
extração de DNA.
4.5 EXTRAÇÃO DE DNA CROMOSSOMAL
O DNA cromossomal foi extraído segundo a técnica de van Soolingen et al. (1994)
com pequenas adaptações. Primeiramente, transferiu-se 1,5 mL de cultura bacteriana
crescida para cada tubo de 1,5 mL tipo Eppendorf previamente identificado com a
respectiva amostra, sendo o mesmo centrifugado a 5000 g por 5 minutos, a 14°C.
Posteriormente, o sobrenadante foi descartado e o pellet ressuspendido no vortex.
Nos mesmos tubos, foram adicionados 400µL de tampão TE, seguido de 6 µL de
lisozima (20mg/mL) e 1µL de enzima RNAse (10mg/mL) em cada. A solução foi
levada ao banho-maria a 37°C por 1,5 horas. Transcorrido este tempo, foi acrescentado
70µL de dodecil sulfato de sódio (10%) e 12 µL de proteinase K (20mg/mL). O sistema
foi então incubado por 10 minutos a 56°C. Posteriormente, adicionou-se 100µL de NaCl
5,0 M e 80µL de solução CTAB/NaCl (10% CTAB, 0,73 M NaCl). A solução foi
agitada lentamente até ficar com aspecto leitoso, sendo incubada por 10 minutos a 65°C.
Posteriormente, acrescentou-se 650 µL de clorofil (clorofórmio/álcool isoamílico, 25:1,
v/v), seguido de leve agitação por 30 segundos. A solução foi centrifugada por 12000 g
por 5 minutos a 10°C. O sobrenadante foi transferido para outro tubo identificado, e
adicionou-se 400µL de isopropanol gelado e então o sistema foi homogeneizado e
incubado a -20°C por 24 horas.
37
Após o tempo necessário, as amostras foram centrifugadas a 18.000g, a 4°C por 20
minutos. Realizou-se a lavagem com 1mL de álcool etílico a 70% gelado e deixou-se
secar à temperatura ambiente por 30 minutos. O DNA extraído foi ressuspendido em 50
µL de água ultrapura MiliQ e estocado a -20°C.
4.6 EXTRAÇÃO DE DNA PLASMIDIAL
Para extração do DNA plasmidial utilizou-se o método de lise alcalina de
Birnboimand Doly (1979), com pequenas adaptações. Foi transferido 1,5 mL de cultura
bacteriana crescida para tubos tipo Eppendorf, previamente identificados com as
respectivas amostras, sendo os mesmos centrifugados a 5000 g por 10 minutos a 10°C.
O sobrenadante foi descartado e o sedimento foi ressuspendido com 210 µL da solução
I (150 mM TrisHCl, 10mM EDTA pH 8.0), posteriormente foi adicionando ao mix da
solução 1 µL de RNAse por amostra e em seguida foi homogeneizado em um vortex.
Em seguida foi adicionado 210 µL da solução II (200 mM NaOH, 1% SDS), misturados
por inversão dos tubos e incubados a temperatura ambiente por 5 minutos. Transcorrido
o tempo, foi adicionado 280 µL da solução III (acetato de sódio 3M, ácido Acético 2M,
pH 4,8-5,0), misturados imediatamente 10 vezes por inversão dos tubos e centrifugados
a 12000 g por 10 minutos a 10°C.
O sobrenadante foi transferido para um novo tubo previamente identificado ao qual
adicionou-se 560 µL de isopropanol gelado, o qual foi homogeneizado por inversão dos
tubos e incubados por 10 minutos a temperatura ambiente. Em seguida, os tubos foram
centrifugados a 12000 g por 15 minutos a 10°C. O sobrenadante foi descartado e o
sedimento foi lavado com 500 µL de etanol a 70% gelado. O DNA extraído foi
ressuspendido em 30 µL água ultrapura MiliQ e estocado a -20°C.
4.7 TESTE DE FORMAÇÃO DE BIOFILME EM POLIESTIRENO
A formação de biofilme foi analisada de acordo com o método de Tendolkar et al.
(2004) com pequenas alterações.
Os isolados armazenados em freezer-20°C foram reativados em caldo TSB e
incubados a 36°C sob agitação por 16 horas. As cepas A. baumannii ATCC 19606 e
Escherichia coli HB 101 foram usadas como controles positivo e negativo,
respectivamente, sendo cultivadas sob as mesmas condições.
38
Após crescimento, a densidade das culturas bacterianas foi ajustada com a escala
0,5 de McFarland, o que corresponde a um número aproximado de 1,5 x 10 8 bactérias.
Em uma placa de poliestireno de 96 poços de fundo chato foram pipetados 270 µL
de meio LB (Luria Bertani) diluído em água destilada para ¼ mais glicose a 0,2%
(LB¼-Gli) nos poços das linhas horizontais de A-G. Nos poços da linha horizontal H
foram pipetados 300 µL de LB¼-Gli, os quais foram usados para a leitura do
BRANCO. Foram pipetados 30 µL da cultura ajustada de cada isolado, em réplicas de
4, obtendo uma diluição da cultura bacteriana em 1:10. Nas fileiras horizontais F e G
foram pipetados 30 µL das culturas ajustadas controles positivo e negativo para a
formação de biofilme, respectivamente. A placa foi acondicionada em câmara úmida e
incubada a 29°C por 22 horas. A figura 10 ilustra a distribuição dos isolados em placa
de microtitulação.
Figura 10. Esquema representando a distribuição dos isolados e a disposição dos controles
positivo e negativo e do branco.
Compreendido o período da incubação, foi determinado o crescimento bacteriano
através da leitura da placa em leitor de ELISA em OD 405nm. Em seguida, a placa foi
lavada cuidadosamente por 3 vezes com PBS 1X estéril usando pipeta multicanal,
invertida sobre papel toalha e deixada secar por 3 horas a temperatura ambiente.
Posteriormente foram adicionados 300µL de corante cristal violeta a 0,2% em água a
cada poço e a placa foi incubada por 15 minutos a temperatura ambiente que, depois
39
de aspirado o CV, cada poço foi lavado cuidadosamente 3 vezes com PBS 1X. Após
aspirar todo o PBS, foi adicionado 300µL de etanol-acetona (80:20, v/v) e feita leitura
em OD595nm.
As leituras obtidas nas OD 405 e 595 foram subtraídas pelo branco de cada
ensaio. A formação de biofilme determinada pela leitura a 595nm foi corrigida pelo
crescimento da cultura observada a 405nm, calculando-se a razão dos valores obtidos
(OD595-BRANCO / OD405-BRANCO). Os valores obtidos foram usados para se determinar a
média e o desvio padrão (dp) de cada amostra. A comparação estatística entre os
valores encontrados foi feita pelo teste t de student, e foram considerados valores
significativamente diferentes quando p<0,05.
Cada experimento foi repetido três vezes independentemente.
Para classificar os isolados como formadores de biofilme foram aceitos os
resultados em que o isolado apresentou uma média superior ao do controle negativo,
onde os valores do dp de ambos não se encontravam. Com base nestes resultados, foi
possível afirmar se o isolado formava maior, menor ou a mesma quantidade de biofilme
que o controle positivo, ao comparar a média do isolado em relação ao controle
positivo. Os isolados não-formadores de biofilme receberam esta classificação quando,
ao comparar as médias, os isolados apresentaram valores inferiores ao do controle
negativo ou tiveram o encontro entre os valores do dp. O experimento de cada placa foi
validado quando, pela comparação estatística do teste t do controle positivo em relação
ao controle negativo, o resultado foi inferior a 0,05. Os formadores de biofilme foram
classificados de acordo com a intensidade da formação do biofilme em forte, moderado
e fraco, utilizando a média obtida do quociente das leituras em leitor de ELISA (OD595BRANCO/OD405-BRANCO)
nos três plaqueamentos e aplicadas à estatística descritiva
QUARTIS. Os valores a seguir foram atribuídos para a determinação de biofilme:
MÉDIA dos quocientes das ODs < 0,96 = FRACO
0,96 < MÉDIA dos quocientes das ODs < 1,92 = MODERADO
1,92 < MÉDIA dos quocientes das ODs = FORTE
4.8 IDENTIFICAÇÃO GENOTÍPICA
A presença dos DNAs extraídos foi verificada em gel de agarose a 1% contendo
brometo de etídeo (0,5 µg/mL), visualizadas em fotodocumentador. Foram realizadas
PCRs usando oligonucleotídeos iniciadores para blaPER-1 (Poirel et al. 2005),
40
blaOXA-23, blaOXA-40, blaOXA-51, blaOXA-58 e ISAba1 (Sohrabi et al. 2012), cujas
sequências estão apresentadas na tabela 3.
Os sistemas de PCR foram preparados para um volume final de 20µL, onde em
tubos de 0,2mL foram adicionados 20 ηg de DNA, Tampão Green GoTaq (Promega),
0,47 µM de cada oligonucleotídeo iniciador específico, 250 µM de cada dNTP, e 1U de
Taq DNA Polimerase (Invitrogen).
Tabela 3. Oligonucleotídeos usados para detecção de blaPER-1, β-lactamases do tipo
OXA e ISAba1 em Acinetobacter baumannii.
Primers
blaPER-1 F
blaPER-1 R
OXA-23 F
OXA-23 R
OXA-40 F
OXA-40 R
OXA-51 F
OXA-51 R
OXA-58 F
OXA-58 R
ISAba1 F
ISAba1 R
Tamanho do
produto (pb)
924
1.057
825
641
453
451
Sequência (5’ - 3’)
ATGAATGTCATTATAAAAGC
AATTTGGGCTTAGGGCAGAA
GATGTGTCATAGTATTCGTCGT
TCACAACAACTAAAAGCACTGT
ATGAAAAAATTTATACTTCCTATATTCAGC
TTAAATGATTCCAAGATTTTCTAGC
AACAAGCGCTATTTTTATTTCAG
CCCATCCCCAACCACTTTT
AGTATTGGGGCTTGTGCT
AACTTCCGTGCCTATTTG
CATTGGCATTAAACTGAGGAGAAA
TTGGAAATGGGGAAAACGAA
Temperatura de
anelamento (°C)
55
50
50
50
49
50
4.8.1 Identificação do gene blaPER-1
O termociclador modelo MJ96G (Biocycler) foi programado de acordo com
Vahaboglu et al. (1995), com algumas alterações, para uma desnaturação inicial de 95ºC
por 4 min, seguido por 30 ciclos de desnaturação (90ºC, 30 seg), anelamento (55ºC, 40
seg) e extensão (72ºC, 1 min), com extensão final de 72ºC por 5 min. Os produtos das
PCRs foram analisados em gel de agarose 1,5% contendo brometo de etídeo (0,5μg/mL)
e visualizados em fotodocumentador. O marcador utilizado foi 1Kb plus DNA Ladder
(Invitrogen).
Para avaliar a correlação entre formação de biofilme bacteriano e presença do gene
blaPER-1, utilizou-se o teste de correlação e o teste exato de Fisher.
4.8.2 Identificação dos genes OXA e ISAba1
O termociclador modelo MJ96G (Biocycler) foi programado para uma desnaturação
inicial de 94°C por 4 min, seguido por 35 ciclos de desnaturação (92°C, 1 min),
anelamento (temperatura de acordo com o oligonucleotídeo usado, ver tabela 3, 1 min) e
extensão (72°C, 1 min), com extensão final de 72°C por 5 min. Isolados que
41
apresentaram presença de qualquer um dos genes OXA pesquisados, foram reanalisados quanto a associação entre ISAba1 e OXA-23, OXA-51 e OXA-58 em A.
baumannii. Para tanto os pares de oligonucleotídeos usados, foram uma combinação do
ISAba1 forward com um dos oligonucleotídeos reverse para OXA-23, OXA-51 e OXA58. Os produtos das PCRs foram analisados em gel de agarose 1,5%, contendo brometo
de etídeo (0,5μg/mL) e visualizados em fotodocumentador. Os marcadores utilizados
foram 1Kb plus DNA Ladder (Invitrogen), 1Kb Ladder DNA marker (Axygen) e 100bp
Ladder DNA marker (Axygen).
42
5 RESULTADOS
Este estudo é complementar ao trabalho de Godoy (2012), onde no período de
junho a dezembro de 2010 foram coletados espécimes biológicos em pacientes
hospitalizados em cinco UTIs de quatro hospitais. Dentre as amostras analisadas, 71
culturas coletadas de 57 pacientes foram positivas para Acinetobacter baumannii,
totalizando 10 pacientes com mais de uma amostra positiva.
5.1 FORMAÇÃO DE BIOFILME
Os isolados foram submetidos ao teste de formação de biofilme em placa de
poliestireno de 96 poços onde, dos 71 isolados clínicos, 57 foram classificados como
formadores de biofilme e 14 foram classificados como não-formadores de biofilme.
Entre os formadores de biofilme, 10% (6/57) foram classificados como fortes (>1,9),
10% (6/57) como moderados (1,9>0,95) e 79% (45/57) como fracos (<0,95).
5.2 GENE blaPER-1
Foram consideradas amostras positivas para o gene blaPER-1 aquelas onde seus
produtos obtidos por PCR apresentaram tamanho esperado de 924 pares de bases (pb).
Dentre os 71 isolados clínicos, o gene blaPER-1 foi detectado em 21 isolados.
5.3 RELAÇÃO DO GENE blaPER-1 COM A FORMAÇÃO DE BIOFILME
A relação entre a presença do gene blaPER-1 com a formação de biofilme pode ser
observada na tabela 4. Nesta, percebe-se que 45 amostras capazes de formarem biofilme
não contêm o gene blaPER-1.
43
Tabela 4. Relação entre presença do gene blaPER-1 com a formação de biofilme
bacteriano.
Não Formação de
Formação de Biofilme
Presença do gene
blaPER-1
Ausência do gene
blaPER-1
Total
Total
Biofilme
12
09
21
45
05
50
57
14
71
Relacionando a presença do gene blaPER-1 com a intensidade de formação de
biofilme, o gene blaPER-1 foi detectado em 33% (2/6) dos isolados que formaram
biofilmes moderados e 22% (10/45) dos isolados que formaram biofilmes fracos. O
gene não foi detectado nos isolados classificados como fortes formadores de biofilme
(tabela 5).
Tabela 5. Relação entre a intensidade da formação de biofilme e a presença do gene
blaPER-1.
BIOFILME
FORTE
MODERADO
FRACO
Nº de Isolados
6
6
45
Presença de blaPER-1
0
2
10
Considerando os resultados obtidos na tabela 4, foram aplicados os teste de
correlação obtendo valor de r igual a -0,34 e o teste de Fisher, obtendo valor de p igual
a 0,0025.
5.4 AMPLIFICAÇÃO DOS GENES OXA E ISAba1
Ao investigar a presença dos genes β-lactamases do tipo OXA, o gene OXA-51 foi
encontrado em todas as amostras. Das 71 amostras, 43 (61%) possuíam o OXA-23, e 19
(27%) possuíam o OXA-58. A coexistência de 2 e 3 genes OXA diferentes foi
observada 41% e 20% do isolados analisados, respectivamente. Nenhum isolado
apresentou amplificação para o gene OXA-40. O elemento de inserção ISAba1 foi
detectado em 72% (51/71) dos isolados.
44
Ao comparar os resultados com os testes de sensibilidade realizados em trabalho
precursor (Godoy 2012), foi observado que 96% (68/71) dos isolados expressaram
resistência a algum dos antibióticos testados e 76% (53/70) dos isolados foram
resistentes ao imipenem. O teste de sensibilidade para o imipenem não foi realizado em
01 isolado. Os resultados dos testes mostraram que a menor taxa de resistência foi
percebida contra polimixina B/colistina (10%) e tigeciclina (8%).
Dos isolados sensíveis ao imipenem, 41% (7/17) apresentaram somente o OXA-51
e em 53% (9/17) foi detectado o gene ISAba1 à montante do gene OXA, onde dois
isolados apresentaram os genes OXA-51 + ISAba1/OXA-23 + ISAba1/OXA-58 e
ISAba1/OXA-51 + ISAba1/OXA-23 + OXA-58, os quais estão classificados como fraco
e moderado formadores de biofilme, respectivamente. Em contrapartida, dos 53
isolados que expressaram resistência ao imipenem, 24% (13) possuíam somente OXA51 + ISAba1/OXA-23, 23% (12) possuíam somente ISAba1/OXA-51 + ISAba1/OXA23 e 19% (10) possuíam somente OXA-51 + ISAba1/OXA-23 + OXA-58.
A tabela 6 mostra a distribuição de genes β-lactamases do tipo OXA em isolados de
A. baumannii na resistência aos antibióticos, onde 756% (53/70) apresentaram
resistência ao imipenem e 60% (42/70) dos isolados se destacaram na resistência
multidrogas ao imipenem, meropenem, cefepima, ceftazidima, ciprofloxacina e
piperacilina/tazobactam, dentre os demais carbapenêmicos testados. Nenhum isolado
resistente aos antibióticos supracitados apresentou ISAba1/OXA-51 + ISAba1/OXA-58
ou ISAba1/OXA-51 + ISAba1/OXA-23 + ISAba1/OXA-58.
45
Tabela 6. Distribuição de genes β-lactamases do tipo OXA em isolados Acinetobacter
baumannii com perfil de resistência aos antimicrobianos relacionados.
Resistência ao
Resistência ao
IPM
IPM, MER, CPX,
CAZ, CFPM,
PIP/TAZ
Sensibilidade
ao IPM
10 (19%)
9 (21%)
7 (41%)
Somente ISAba1/OXA-51
3 (6%)
3 (7%)
3 (18%)
OXA-51 + OXA-23
1 (2%)
0
0
OXA51 + OXA-58
1 (2%)
0
1 (6%)
ISAba1/OXA-51 + OXA-58
1 (2%)
0
0
ISAba1/OXA-51 + OXA-23
2 (4%)
2 (5%)
0
OXA-51 + OXA-58 + ISAba1/OXA23
10 (19%)
10 (24%)
1 (6%)
OXA-51 + ISAba1/OXA-23
13 (24%)
8 (19%)
1 (6%)
OXA-51 + ISAba1/OXA-58
0
0
1 (6%)
OXA-51 + ISAba1/OXA-23 +
ISAba1/OXA-58
0
0
2 (12%)
12 (23%)
10 (24%)
0
0
0
1 (6%)
53 (100%)
42 (100%)
17 (100%)
Alelos OXA
Somente OXA-51
ISAba1/OXA-51 + ISAba1/OXA-23
ISAba1/OXA-51 + ISAba1/OXA-23 +
OXA-58
TOTAL
Dos 51 isolados em que o gene ISAba1 foi detectado, 78% (40/51) estavam
associados ao gene OXA-23, 45% (23/51) ao gene OXA-51 e 8% (4/51) ao gene OXA58 (tabela 7). A resistência à cefepima, ceftazidima, ciprofloxacina, imipenem e
meropenem foi observada em 82% (33/40) dos isolados com ISAba1/OXA-23 e em
74% (17/23) dos isolados com ISAba1/OXA-51. Já os isolados com ISAba1/OXA-58
apresentaram sensibilidade ao imipenem e meropenem.
Três isolados expressaram resistência a todos os antimicrobianos testados, onde os
isolados ABs09 amplificou somente para ISAba1/OXA-51, ABs10 amplificou somente
para OXA-51 e o ABs15 amplificou somente para OXA-51 e ISAba1/OXA-23. Em
contrapartida, um isolado foi sensível a todos os antibióticos testados, amplificando
somente para OXA-51.
As tabelas 7 e 8 mostram o perfil de resistência dos isolados a diferentes
antimicrobianos e presença de genes β-lactamases do tipo OXA. A tabela 8 relaciona na
46
exclusividade dos genes OXA-51 e ISAba1/OXA-51. Nos isolados em que o gene
OXA-58 foi detectado, houve a presença de um ou mais genes OXA.
Tabela 7. Relação entre a resistência de Acinetobacter baumannii na presença de genes
β-lactamases do tipo OXA.
OXA-51
(n=71)*
ISAba1/
OXA-51
(n=23)*
OXA-23
(n=43)*
ISAba1/
OXA-23
(n=40)*
OXA-58
(n=19)*
ISAba1/
OXA-58
(n=4)*
AMI
19 (27%)
3 (13%)
10 (23%)
10 (25%)
9 (47%)
3 (75%)
AMP/SUL
44 (62%)
17 (74%)
31 (72%)
29 (72%)
11 (58%)
0
CFPM
68 (96%)
23 (100%)
42 (98%)
39 (98%)
13 (68%)
4 (100%)
CAZ
66 (93%)
23 (100%)
42 (98%)
39 (98%)
13 (68%)
4 (100%)
CPX
65 (91%)
22 (96%)
40 (93%)
37 (92%)
17 (90%)
4 (100%)
GEN
39 (55%)
13 (56%)
23 (54%)
22 (55%)
9 (47%)
2 (50%)
IPM
53 (75%)
18 (78%)
38 (88%)
35 (88%)
12 (63%)
0
LVX
31 (44%)
10 (43%)
19 (44%)
18 (45%)
11 (58%)
4 (100%)
MER
53 (75%)
18 (78%)
38 (88%)
35 (88%)
12 (63%)
0
PIP/TAZ
50 (70%)
20 (87%)
33 (77%)
31 (78%)
11 (58%)
1 (25%)
POL
7 (9%)
3 (13%)
4 (9%)
4 (10%)
0
0
TET
34 (48%)
7 (30%)
22 (51%)
21 (52%)
12 (63%)
3 (75%)
TGC
6 (8%)
2 (9%)
4 (9%)
4 (10%)
0
0
*n representa o número de isolados positivos para o gene OXA ou ISAba1/OXA
Tabela 8. Relação entre a resistência de Acinetobacter baumannii aos β-lactâmicos na
presença ou não do elemento ISAba1 à frente do gene OXA-51.
OXA-51
(n=17)
ISAba1/
OXA-51
(n=6)
Cefepima
15 (88%)
6 (100%)
Ceftazidima
14 (82%)
6 (100%)
Imipenem
10 (59%)
3 (50%)
Meropenem
10 (59%)
3 (50%)
Antibióticos
47
5.5 RESISTÊNCIA E FORMAÇÃO DE BIOFILME
Com base no teste de sensibilidade realizado com os 71 isolados, 68 apresentaram
resistência a algum dos antibióticos testados. Dos 57 isolados formadores de biofilme,
55 apresentaram resistência a algum dos antibióticos e 2 foram sensíveis a todos os
antibióticos. Dos isolados formadores de biofilme que apresentaram resistência, 100%
(55/55) expressaram resistência à cefepima, seguido de 96% (53/55) para ceftazidima e
ciprofloxacina, e 76% (42/55) para imipenem e meropenem.
Observa-se, na tabela 9, que os isolados formadores de biofilme apresentaram
maior resistência contra a cefepima, ceftazidima e ciprofloxacina, seguido do imipenem
e meropenem. A menor resistência (9%) foi observada para tigeciclina, seguido da
polimixina B/colistina (10%).
Tabela 9. Perfil de susceptibilidade aos antimicrobianos de isolados de Acinetobacter
baumannii, em porcentagem (%).
RESISTÊNCIA
ANTIBIÓTICOS
Isolados
formadores de
biofilme
(n=57)
Isolados não- Resistência de
formadores de
todos os
biofilme
isolados
(n=14)
(n=71)
Amicacina
30
14
27
Ampicilina/Sulbactam
66
43
62
Cefepima
96
93
96
Ceftazidima
93
93
93
Ciprofloxacina
93
86
91
Gentamicina
58
43
55
Imipenem
74
78
75
Levofloxacina
47
28
44
Meropenem
74
78
75
Piperacilina/Tazobactam
74
57
70
Polimixina B/Colistina
10
7
10
Tetraciclina
51
36
48
Tigeciclina
9
7
8
48
A tabela 10 mostra o padrão de resistência multidroga dos isolados formadores de
biofilme, onde 5% apresentaram resistência a todos os antibióticos testados. Dos 57
isolados formadores de biofilme, 74% (42) expressaram resistência contra o imipenem e
meropenem.
Tabela 10. Padrão de resistência multidrogas de formadores e não formadores de
biofilme.
Formadores de biofilme
que apresentaram
resistência (n=55)
Não formadores de
biofilme que
apresentaram
resistência (n=13)
Todos antibióticos testados
3 (5%)
0
AMI, AMP/SUL, CFPM, CAZ, CPX, GEN,
IPM, LVX, MER, PIP/TAZ, POL, TET
5 (9%)
0
AMI, AMP/SUL, CFPM, CAZ, CPX, GEN,
IPM, LVX, MER, PIP/TAZ, TET
8 (14%)
1 (8%)
AMP/SUL, CFPM, CAZ, CPX, GEN, IPM,
MER, PIP/TAZ
21 (38%)
4 (31%)
AMP/SUL, CFPM, CAZ, CPX, IPM, MER,
PIP/TAZ
34 (62%)
6 (46%)
CFPM, CAZ, CPX, IPM, MER, PIP/TAZ
36 (65%)
7 (54%)
CFPM, CAZ, CPX, IPM, MER
41 (74%)
10 (77%)
Perfil de Resistência
Dados clínicos indicam atividade microbiana altamente eficaz das polimixinas e
tigeciclinas contra isolados A. baumannii. Dentre os isolados classificados como
formadores de biofilme, 10% (6/57) e 9% (5/57) expressaram resistência às polimixinas
e tigeciclinas, respectivamente. Dentre os não-formadores de biofilme, somente um
isolado expressou resistência aos antimicrobianos mencionados.
5.6 RELAÇÃO ENTRE A RESISTÊNCIA E O GENE blaPER-1
Todos os 21 isolados que possuíam o gene blaPER-1 (tabela 4) expressaram
resistência à cefepima (tabela 11). A resistência à ceftazidima e imipenem também foi
comparada, onde 95% (20/21) e 90% (19/21) dos isolados com o gene expressaram
resistência, respectivamente. A resistência concomitante à cefepima, ceftazidima e
imipenem foi observada em 19 isolados (90%) que possuíam o gene blaPER-1. Dois
isolados foram sensíveis ao imipenem, sendo 01 sensível também à ceftazidima. A
49
sensibilidade concomitante aos três antibióticos mencionados foi observada em apenas
3 isolados, e nenhum possuía o gene blaPER-1.
Tabela 11. Relação entre a presença do gene blaPER-1 e a resistência à cefepima.
Resistência à
Sensibilidade à
cefepima
cefepima
Presença do gene blaPER-1
21
0
21
Ausência do gene blaPER-1
47
03
50
Total
68
03
71
Total
Vale destacar que não houve relação entre a presença do gene blaPER-1 e os óbitos
dos pacientes, uma vez que dentre os 21 pacientes infectados com A. baumannii
carregando o gene blaPER-1, 76% (16/21) foram a óbito, enquanto que o índice de
óbitos entre os pacientes infectados por A. baumannii não-produtora de blaPER-1 foi de
60% (30/50).
50
6 DISCUSSÃO
As infecções por A. baumannii representam um desafio médico global. São
patógenos oportunistas e são, particularmente, bem sucedidos em colonizar e persistir
no ambiente hospitalar. São capazes de resistir à dessecação e sobreviver em superfícies
inanimadas por anos (Jain & Danziger 2004; Jeong et al. 2006; Dijkshoorn et al. 2007).
O interesse por este organismo tem crescido rapidamente devido ao aparecimento
de isolados MDR, alguns dos quais são PDR aos agentes antimicrobianos (Prashanth &
Badrinath 2004; Van Looveren & Goossens 2004; Dijkshoorn et al. 2007;). Ele também
está entre as causas mais comuns de infecção nosocomial relacionada a dispositivos
médicos, sendo resultado da capacidade do organismo de resistir à desinfecção química
e física, muitas vezes formando biofilme. A formação de biofilme tem sido uma
característica chave na patogenicidade, especialmente em relação às infecções na linha
intravascular e pneumonia associada à ventilação mecânica. Geralmente duas
propriedades são frequentemente associadas com a produção de biofilme: o aumento da
síntese de exopolissacarídeo (EPS) e o desenvolvimento de resistência aos antibióticos
(Costerton et al. 1999). Assim, as infecções devidas às bactérias que formam biofilme
são um problema clínico tenaz.
Optamos por investigar a capacidade de formação de biofilme em poliestireno por
isolados clínicos de A. baumannii e a resistência a múltiplas drogas entre elas, assim
como a presença de genes relacionados com a formação de biofilme e resistência a
antimicrobianos, a saber: blaPER-1, OXA-23, OXA-40, OXA-51, OXA-58 e a
sequência de inserção ISAba1.
Para a identificação do produto do gene blaPER-1, das 71 amostras analisadas, 21
mostraram positivas para o gene blaPER-1. Destas, 12 apresentaram formação de
biofilme.
No presente estudo, o método quantitativo para formação de biofilme mostrou 6
(7,5%) isolados como fortes formadores de biofilme, porém na ausência do gene
blaPER-1. O resultado de Fisher revela que há diferença estatística entre os grupos,
admitindo-se pelo teste de correlação que existe uma correlação negativa entre a
presença do gene blaPER-1 e a formação de biofilme, sugerindo que o gene tem efeito
inibidor na formação de biofilme, sendo aceita a possibilidade de que outros fatores
estão mais fortemente associados com a formação de biofilme dos mesmos, tais como a
51
presença de pili, estruturas semelhantes à fímbrias, OmpA (proteínas de membrana
externa) e proteínas que se ligam à fibronectina, conhecidas como FBPs (fibronectinbinding proteins), as quais facilitam a adesão e invasão bacteriana em células
hospedeiras (Gohl et al. 2006; Lee et al. 2006; Gaddy et al. 2009; Henderson et al.
2011). Sechi et al. (2004) e Rao et al. (2008), relacionam a produção de blaPER-1 com a
aderência bacteriana, mas não com a formação do biofilme. No entanto, Lee et al.
(2008) mostraram em seus resultados que os isolados que possuem esse gene,
apresentam capacidade altamente significativa para adesão e formação de biofilme,
quando comparado aos isolados que não carregavam o gene. Em relação à resistência,
todos os isolados que amplificaram para o gene blaPER-1 apresentaram resistência
contra a cefepima, compartilhando de resultados semelhantes apresentados por Yong et
al. (2003). Observa-se, portanto, que a presença do gene blaPER-1 estava associada a
multirresistência às drogas testadas neste trabalho, uma vez que a sensibilidade
concomitante à cefepima, ceftazidima e imipenem foi observada somente na ausência
do gene blaPER-1 (Tabela 11).
Os isolados formadores de biofilme expressaram resistência à cefepima,
ceftazidima, ciprofloxacina, imipenem e meropenem. Em relação aos não-formadores
de biofilme, nosso estudo detectou resistência altamente significativa aos mesmos
antibióticos. O aumento da resistência em isolados não-formadores de biofilme também
foi relatado por Rao et al. (2008). Esta característica pode estar associada à presença de
genes que promovem a resistência, bem como a ação de mecanismos não-enzimáticos
como super-expressão de bombas de efluxo, alteração de porinas e impermeabilidade da
membrana (Gur et al. 2008).
A seleção apropriada do antibiótico para o tratamento de infecções associadas ao
biofilme, assim como a simples presença de A. baumannii, é extremamente importante.
Os carbapenêmicos têm sido as drogas de escolha para o tratamento de infecções
causadas por este organismo (Quale et al. 2003), mas a resistência aos mesmos tem se
tornado comum, diminuindo as opções terapêuticas existentes (Sohrabi et al. 2012).
Neste estudo, 75% (53/71) dos isolados foram resistentes ao imipenem e meropenem,
sendo estes os antibióticos mais comumente prescritos nos hospitais. Além disso, a alta
porcentagem de resistência também foi observada à ciprofloxacina (91%).
Em
concordância com outros estudos, foi observado que dos seis isolados classificados
como fortes formadores de biofilme, 67% (4/6) apresentaram resistência a quase 60%
dos antibióticos testados (Siroy et al. 2006; Rodriguez-Bano et al. 2008). Assim,
52
adquirir a capacidade de formar biofilme torna-se uma boa estratégia para aumentar a
sobrevivência e persistência sob condições de estresse, por exemplo, durante a invasão
hospedeiro ou após o tratamento antibiótico (Rao et al. 2008). Como consequência do
desenvolvimento de biofilme, afirma-se que a capacidade de Acinetobacter para a
transferência de genes é horizontal, facilitando a disseminação de resistência aos
antibióticos nessas micro-comunidades (Lee et al. 2008). No entanto, ainda não se sabe
muito sobre o mecanismo que desencadeia o desenvolvimento de biofilme em A.
baumannii (Lee et al. 2008).
O gene OXA-51 foi detectado nos 71 isolados, corroborando com estudos que
sugerem a presença do gene em todos os isolados de A. baumannii, uma vez que o gene
OXA-51 é intrínseco à espécie (Heritier et al. 2005; Turton et al. 2006b; Sohrabi et al.
2012).
Surtos por isolados de CRAB produtores de OXA-23 têm sido relatado em diversos
países, sendo encontrado disseminado em todo o mundo (Mugnier et al. 2010). Nos
estudos conduzidos por Mak et al. (2009), Zhou et al. (2007) e Figueiredo et al. (2011),
87,5%, 94% e 75% dos isolados resistentes ao imipenem tinham o gene OXA-23,
respectivamente. Neste estudo, 72% (38/53) dos isolados resistentes ao imipenem
possuíam o gene em questão.
Não foi detectada a presença do gene OXA-40 dentre os isolados analisados,
concordando com os resultados apresentados por Sohrabi et al. (2012) realizado no
noroeste do Irã, onde apenas em 1 em cada 100 isolados foi detectado o gene. A alta
prevalência deste gene tem sido relatada em países da Europa (Quinteira et al. 2007;
Ruiz et al. 2007). O único achado deste gene no Brasil foi relatado em um estudo
conduzido por Werneck et al. (2011a), onde OXA-40 foi detectado no único isolado de
A. baumannii avaliado no estudo.
Dos 71 isolados investigados, 27% apresentaram o gene OXA-58. Estes achados
apresentam maior porcentagem de isolados com o gene do que os resultados
apresentados por Sohrabi et al. (2012), Feizabadi et al. (2008) e Taherikalani et al.
(2009), com 3,6%, 9% e 9%, respectivamente. Os isolados avaliados naqueles estudos
foram provenientes do Irã e, portanto, pode ser que exista uma relação geográfica. No
entanto, é equivalente aos resultados apresentados por Ruiz et al. (2007) na Espanha e
Vahaboglu et al. (2006) na Turquia, com 19% e 17% dos isolados contendo o gene
OXA-58, respectivamente. Figueiredo et al. (2011) detectou OXA-58 em 1 isolado, de
um total de 20 isolados estudados no Rio de Janeiro.
53
Recentemente uma sequência de inserção chamada ISAba1 foi identificada em A.
baumannii (Corvec et al. 2003) e associada com a super-expressão de genes de
resistência como AmpC, OXA-23 e OXA-51, que conferem resistência aos
carbapenêmicos (Corvec et al. 2003; Corvec et al. 2007; Turton et al. 2006a). A
amplificação por PCR neste estudo detectou a presença de ISAba1 em 72% dos
isolados. Este resultado está de acordo com outros estudos em que ISAba1 foi
encontrado na maioria do isolados, como por exemplo, Ruiz et al. (2007), Turton et al.
(2006a) e Sohrabi et al. (2012) que detectaram ISAba1 em 74,7%, 84% e 90% dos
isolados provenientes da Espanha, Reino Unido e Irã, respectivamente. Neste estudo, os
resultados revelaram ainda que a maioria, ou seja, 57% (39/68) dos isolados de A.
baumannii resistentes a algum dos antibióticos testados tinham ISAba1 à montante do
gene OXA-23.
Dos isolados resistentes ao imipenem, 77% (41/53) possuíam o gene ISAba1 à
montante do gene OXA, cujo resultado é similar aos estudos conduzidos por Mak et al.
(2009), Turton et al. (2006a) e Zhou et al. (2007), onde 52,6%, 86% e 94% de isolados
A. baumannii resistentes ao imipenem tinham ISAba1, respectivamente. Ainda em
relação aos isolados resistentes ao imipenem, 66% (35/53) tiveram o gene ISAba1 à
montante do gene OXA-23, similar ao resultado apresentado por Sohabi et al. (2012),
com 74,2%. Estes resultados indicam que ISAba1 quando associado ao gene OXA-23
pode estar envolvido na resistência ao imipenem. Vale destacar que a associação entre
ISAba1 e o gene OXA-58 não resultou em resistência ao imipenem.
Dos 43 isolados que continham o gene OXA-23, 93% (40/43) possuíam o gene
ISAba1 à montante deste gene. Zhou et al. (2007) e Turton et al. (2006a) relataram que
97,5% e 100% dos isolados com OXA-23 continham o gene ISAba1 à montante do
OXA-23.
Turton et al.(2006a) relataram que dentre os 20 isolados que possuíam somente
OXA-51, 8 apresentaram ISAba1 associado ao gene e expressando resistência ao
imipenem ou meropenem, concluindo que a expressão do gene OXA-51 é baixa na
maioria dos casos. Sohrabi et al. (2012) relatou que enquanto 3 isolados que foram
resistentes ao imipenem tinham ISAba1 à montante de OXA-51, somente 1 isolado com
apenas OXA-51 expressou resistência ao imipenem, sugerindo que a presença do gene
OXA-51 sozinho não é suficiente para expressar resistência contra os antibióticos
carbapenêmicos. Neste estudo, a presença isolada do gene OXA-51, ou associada ao
ISAba1 (6), foi identificada em 23 isolados. Dentre eles, 56% (13/23) dos isolados
54
foram resistentes ao imipenem e meropenem, onde 23% (3/13) apresentaram a
sequência de inserção ISAba1 à montante do gene. Percebe-se, portanto, que estes
achados indicam uma discordância com os resultados apresentados por Turton et al.
(2006a) e Sohrabi et al. (2012), pois a resistência ao imipenem e meropenem foi
expressa em 10 isolados que possuíam somente o gene OXA-51. Este resultado sugere a
presença de outros genes β-lactamases do tipo OXA que não foram pesquisados neste
estudo, explicando a resistência ao imipenem dos isolados contendo somente o gene
OXA-51.
Este estudo detectou, através da amplificação por PCR, a presença de ISAba1 à
montante do OXA-58 em 4 isolados, porém em 3 isolados o elemento de inserção
estava presente também à montante do gene OXA-23 ou OXA-51. Em um foi detectado
ISAba1/OXA-51 e no outro foi detectado OXA-51 e ISAba1/OXA-23. Todos os quatro
isolados foram sensíveis ao imipenem. Sohrabi et al. (2012) relatam não ter encontrado
ISAba1 à montante do gene OXA-58 nos dois isolados onde OXA-58 foi detectado. Em
estudo realizado no sul do Brasil, Gusatti et al. (2012) relatou encontrar a sequência de
inserção ISAba1 localizada à montante do gene OXA-58 (Gusatti et al. 2012).
Observou-se que a resistência em isolados contendo ISAba1 associado a algum dos
genes OXA foi significativa, sugerindo que ISAba1 é importante na expressão destes
genes. A facilidade em superexpressar genes com este elemento móvel pode ser de
grande vantagem, proporcionando um mecanismo para expressar os genes somente
quando existe uma vantagem seletiva para o fazer. A presença de ISAba1 reflete em A.
baumannii, de modo que o organismo se ligue rapidamente ao DNA, dando-lhe uma
maleabilidade que pode contribuir muito para o seu sucesso (Turton et al. 2006a).
55
7 CONCLUSÕES
Este estudo revelou, pela primeira vez, a ocorrência do gene blaPER-1 em isolados
de A. baumannii no Brasil e o primeiro relato do gene OXA-58 e da sequência de
inserção ISAba1 em isolados de Acinetobacter baumannii no Estado de Goiás.
A maioria dos isolados clínicos de A. baumannii analisados possuiam capacidade
para formação de biofilme, porém, neste estudo, a correlação entre a presença do gene
blaPER-1 e a capacidade de formar biofilme foi negativa, uma vez que a detecção do
gene foi maior nos isolados que apresentaram fraca formação de biofilme, sendo
também detectado na maioria dos isolados não-formadores de biofilme.
Foi observada resistência à cefepima, ceftazidima, ciprofloxacina, imipenem e
meropenem em isolados formadores de biofilme. Porém, o mesmo foi observado em
isolados não-formadores de biofilme, sugerindo que essa resistência esteja relacionada à
presença do gene blaPER-1 nesses isolados.
Os genes OXA-51, OXA-23, OXA-58 e ISAba1 foram detectados em alguns
isolados de Acinetobacter baumannii, sendo maior a prevalência do OXA-23, uma vez
que o OXA-51 é um gene intrínseco à espécie. A sequência de inserção ISAba1 foi
detectada na maioria dos isolados que apresentaram algum dos genes OXA e a
resistência concomitante à cefepima, ceftazidima, imipenem e meropenem foi mais
prevalente entre os isolados que possuíam o gene ISAba1.
56
REFERÊNCIAS
Abbo A, Navon-Venezia S, Hammer-Muntz O, Krichali T, Siegman-Igra Y, Carmeli Y
2005. Multidrug-resistant Acinetobacter baumannii. Emerging infectious diseases,
11, 22-29.
Afzal-Shah M, Woodford N, Livermore DM 2001. Characterization of OXA-25, OXA26, and OXA-27, molecular class D beta-lactamases associated with carbapenem
resistance in clinical isolates of Acinetobacter baumannii. Antimicrobial agents
and chemotherapy, 45, 583-588.
ANVISA. Detecção e Identificação de Bactérias de Importância Médica. Manual de
Microbiologia Clínica para o Controle de Infecção em Serviços de Saúde [serial
on the Internet], 2004a 04 de setembro de 2012 [cited:[1-93 p.]. Available from:
http://www.anvisa.gov.br/servicosaude/manuais/microbiologia.asp.
ANVISA. Manual de Microbiologia Clínica para o Controle de Infecção em Serviços de
Saúde. Edição Comemorativa para o IX Congresso Brasileiro de Controle de
Infecção e Epidemiologia Hospitalar. Agência Nacional de Vigilância Sanitária
[serial on the Internet], 2004b:[381 p.].
Anwar H, van Biesen T, Dasgupta M, Lam K, Costerton JW 1989. Interaction of
biofilm bacteria with antibiotics in a novel in vitro chemostat system.
Antimicrobial agents and chemotherapy, 33, 1824-1826.
Bagge N, Ciofu O, Skovgaard LT, Hoiby N 2000. Rapid development in vitro and in
vivo of resistance to ceftazidime in biofilm-growing Pseudomonas aeruginosa due
to chromosomal beta-lactamase. APMIS : acta pathologica, microbiologica, et
immunologica Scandinavica, 108, 589-600.
Bauernfeind A, Stemplinger I, Jungwirth R, Mangold P, Amann S, Akalin E, Ang O,
Bal C, Casellas JM 1996. Characterization of beta-lactamase gene blaPER-2,
which encodes an extended-spectrum class A beta-lactamase. Antimicrobial
agents and chemotherapy, 40, 616-620.
Baumann P, Doudoroff M, Stanier RY 1968. A study of the Moraxella group. II.
Oxidative-negative species (genus Acinetobacter). Journal of bacteriology, 95,
1520-1541.
57
Bergogne-Berezin E, Towner KJ 1996. Acinetobacter spp. as nosocomial pathogens:
microbiological, clinical, and epidemiological features. Clinical microbiology
reviews, 9, 148-165.
Bertini A, Poirel L, Bernabeu S, Fortini D, Villa L, Nordmann P, Carattoli A 2007.
Multicopy blaOXA-58 gene as a source of high-level resistance to carbapenems in
Acinetobacter baumannii. Antimicrobial agents and chemotherapy, 51, 23242328.
Birnboim HC, Doly J 1979. A rapid alkaline extraction procedure for screening
recombinant plasmid DNA. Nucleic acids research, 7, 1513-1523.
Bogaerts P, Cuzon G, Naas T, Bauraing C, Deplano A, Lissoir B, Nordmann P,
Glupczynski Y 2008. Carbapenem-resistant Acinetobacter baumannii isolates
expressing the blaOXA-23 gene associated with ISAba4 in Belgium.
Antimicrobial agents and chemotherapy, 52, 4205-4206.
Bogaerts P, Naas T, Wybo I, Bauraing C, Soetens O, Pierard D, Nordmann P,
Glupczynski
Y
2006.
Outbreak
of
infection
by
carbapenem-resistant
Acinetobacter baumannii producing the carbapenemase OXA-58 in Belgium.
Journal of clinical microbiology, 44, 4189-4192.
Bouvet PJM, Grimont PAD 1986. Taxonomy of the Genus Acinetobacter with the
Recognition of Acinetobacter baumannii sp. nov. Acinetobacter haemolyticus sp.
nov. Acinetobacter johnsonii sp. nov. and Acinetobacter junii sp. nov. and
Emended Descriptions of Acinetobacter calcoaceticus and Acinetobacter lwofii.
Int J Syst Bacteriol, 36, 228-240.
Brink A, Moolman J, da Silva MC, Botha M 2007. Antimicrobial susceptibility profile
of selected bacteraemic pathogens from private institutions in South Africa. South
African medical journal = Suid-Afrikaanse tydskrif vir geneeskunde, 97, 273-279.
Brisou J, Prevot AR 1954. [Studies on bacterial taxonomy. X. The revision of species
under Acromobacter group]. Annales de l'Institut Pasteur, 86, 722-728.
Brooun A, Liu S, Lewis K 2000. A dose-response study of antibiotic resistance in
Pseudomonas aeruginosa biofilms. Antimicrobial agents and chemotherapy, 44,
640-646.
Brown S, Young HK, Amyes SG 2005. Characterisation of OXA-51, a novel class D
carbapenemase found in genetically unrelated clinical strains of Acinetobacter
baumannii from Argentina. Clinical microbiology and infection : the official
58
publication of the European Society of Clinical Microbiology and Infectious
Diseases, 11, 15-23.
Bush K, Jacoby GA, Medeiros AA 1995. A functional classification scheme for betalactamases and its correlation with molecular structure. Antimicrobial agents and
chemotherapy, 39, 1211-1233.
Carvalho KR, Carvalho-Assef AP, Peirano G, Santos LC, Pereira MJ, Asensi MD 2009.
Dissemination of multidrug-resistant Acinetobacter baumannii genotypes carrying
bla(OXA-23) collected from hospitals in Rio de Janeiro, Brazil. International
journal of antimicrobial agents, 34, 25-28.
Christiaen SE, Brackman G, Nelis HJ, Coenye T 2011. Isolation and identification of
quorum
quenching
bacteria
from
environmental
samples.
Journal
of
microbiological methods, 87, 213-219.
Claeys G, Verschraegen G, de Baere T, Vaneechoutte M 2000. PER-1 beta-lactamaseproducing Pseudomonas aeruginosa in an intensive care unit. The Journal of
antimicrobial chemotherapy, 45, 924-925.
Cochran WL, McFeters GA, Stewart PS 2000. Reduced susceptibility of thin
Pseudomonas aeruginosa biofilms to hydrogen peroxide and monochloramine.
Journal of applied microbiology, 88, 22-30.
Coelho JM, Turton JF, Kaufmann ME, Glover J, Woodford N, Warner M, Palepou MF,
Pike R, Pitt TL, Patel BC, Livermore DM 2006. Occurrence of carbapenemresistant Acinetobacter baumannii clones at multiple hospitals in London and
Southeast England. Journal of clinical microbiology, 44, 3623-3627.
Corvec S, Caroff N, Espaze E, Giraudeau C, Drugeon H, Reynaud A 2003. AmpC
cephalosporinase hyperproduction in Acinetobacter baumannii clinical strains.
The Journal of antimicrobial chemotherapy, 52, 629-635.
Corvec S, Poirel L, Naas T, Drugeon H, Nordmann P 2007. Genetics and expression of
the carbapenem-hydrolyzing oxacillinase gene blaOXA-23 in Acinetobacter
baumannii. Antimicrobial agents and chemotherapy, 51, 1530-1533.
Costerton JW, Stewart PS, Greenberg EP 1999. Bacterial biofilms: a common cause of
persistent infections. Science, 284, 1318-1322.
D'Arezzo S, Capone A, Petrosillo N, Visca P, Ballardini M, Bartolini S, Bordi E, Di
Stefano A, Galie M, Minniti R, Meledandri M, Pacciani L, Parisi G, Prignano G,
Santini C, Valmarin M, Venditti M, Ziantoni S 2009. Epidemic multidrugresistant Acinetobacter baumannii related to European clonal types I and II in
59
Rome (Italy). Clinical microbiology and infection : the official publication of the
European Society of Clinical Microbiology and Infectious Diseases, 15, 347-357.
Da Silva G, Dijkshoorn L, van der Reijden T, van Strijen B, Duarte A 2007.
Identification of widespread, closely related Acinetobacter baumannii isolates in
Portugal as a subgroup of European clone II. Clinical microbiology and infection :
the official publication of the European Society of Clinical Microbiology and
Infectious Diseases, 13, 190-195.
Dalla-Costa LM, Coelho JM, Souza HA, Castro ME, Stier CJ, Bragagnolo KL, ReaNeto A, Penteado-Filho SR, Livermore DM, Woodford N 2003. Outbreak of
carbapenem-resistant Acinetobacter baumannii producing the OXA-23 enzyme in
Curitiba, Brazil. Journal of clinical microbiology, 41, 3403-3406.
Danel F, Hall LM, Gur D, Akalin HE, Livermore DM 1995. Transferable production of
PER-1 beta-lactamase in Pseudomonas aeruginosa. The Journal of antimicrobial
chemotherapy, 35, 281-294.
Das JR, Bhakoo M, Jones MV, Gilbert P 1998. Changes in the biocide susceptibility of
Staphylococcus epidermidis and Escherichia coli cells associated with rapid
attachment to plastic surfaces. Journal of applied microbiology, 84, 852-858.
Davis KA, Moran KA, McAllister CK, Gray PJ 2005. Multidrug-resistant Acinetobacter
extremity infections in soldiers. Emerging infectious diseases, 11, 1218-1224.
de Beer D, Stoodley P, Roe F, Lewandowski Z 1994. Effects of biofilm structures on
oxygen distribution and mass transport. Biotechnology and bioengineering, 43,
1131-1138.
de Breij A, Dijkshoorn L, Lagendijk E, van der Meer J, Koster A, Bloemberg G,
Wolterbeek R, van den Broek P, Nibbering P 2010. Do biofilm formation and
interactions with human cells explain the clinical success of Acinetobacter
baumannii? PloS one, 5, e10732.
De Champs C, Chanal C, Sirot D, Baraduc R, Romaszko JP, Bonnet R, Plaidy A, Boyer
M, Carroy E, Gbadamassi MC, Laluque S, Oules O, Poupart MC, Villemain M,
Sirot J 2004. Frequency and diversity of Class A extended-spectrum betalactamases in hospitals of the Auvergne, France: a 2 year prospective study. The
Journal of antimicrobial chemotherapy, 54, 634-639.
Dijkshoorn L, Nemec A, Seifert H 2007. An increasing threat in hospitals: multidrugresistant Acinetobacter baumannii. Nature reviews. Microbiology, 5, 939-951.
60
Docquier JD, Luzzaro F, Amicosante G, Toniolo A, Rossolini GM 2001. Multidrugresistant Pseudomonas aeruginosa producing PER-1 extended-spectrum serinebeta-lactamase and VIM-2 metallo-beta-lactamase. Emerging infectious diseases,
7, 910-911.
Durand ML, Calderwood SB, Weber DJ, Miller SI, Southwick FS, Caviness VS, Jr.,
Swartz MN 1993. Acute bacterial meningitis in adults. A review of 493 episodes.
The New England journal of medicine, 328, 21-28.
Feizabadi MM, Fathollahzadeh B, Taherikalani M, Rasoolinejad M, Sadeghifard N,
Aligholi M, Soroush S, Mohammadi-Yegane S 2008. Antimicrobial susceptibility
patterns and distribution of blaOXA genes among Acinetobacter spp. Isolated
from patients at Tehran hospitals. Japanese journal of infectious diseases, 61,
274-278.
Fernandez-Cuenca F, Martinez-Martinez L, Conejo MC, Ayala JA, Perea EJ, Pascual A
2003. Relationship between beta-lactamase production, outer membrane protein
and penicillin-binding protein profiles on the activity of carbapenems against
clinical isolates of Acinetobacter baumannii. The Journal of antimicrobial
chemotherapy, 51, 565-574.
Figueiredo DQ, Santos KR, Pereira EM, Schuenck RP, Mendonca-Souza CR, Teixeira
LM, Mondino SS 2011. First report of the bla(OXA-58) gene in a clinical isolate
of Acinetobacter baumannii in Rio de Janeiro, Brazil. Memorias do Instituto
Oswaldo Cruz, 106, 368-370.
Figueiredo S, Poirel L, Papa A, Koulourida V, Nordmann P 2009. Overexpression of
the naturally occurring blaOXA-51 gene in Acinetobacter baumannii mediated by
novel insertion sequence ISAba9. Antimicrobial agents and chemotherapy, 53,
4045-4047.
Fournier PE, Richet H 2006. The epidemiology and control of Acinetobacter baumannii
in health care facilities. Clinical infectious diseases : an official publication of the
Infectious Diseases Society of America, 42, 692-699.
Gaddy JA, Actis LA 2009. Regulation of Acinetobacter baumannii biofilm formation.
Future microbiology, 4, 273-278.
Gaddy JA, Tomaras AP, Actis LA 2009. The Acinetobacter baumannii 19606 OmpA
protein plays a role in biofilm formation on abiotic surfaces and in the interaction
of this pathogen with eukaryotic cells. Infection and immunity, 77, 3150-3160.
61
Gales AC, Jones RN, Sader HS 2006. Global assessment of the antimicrobial activity of
polymyxin B against 54 731 clinical isolates of Gram-negative bacilli: report from
the SENTRY antimicrobial surveillance programme (2001-2004). Clinical
microbiology and infection : the official publication of the European Society of
Clinical Microbiology and Infectious Diseases, 12, 315-321.
Gerner-Smidt P, Tjernberg I, Ursing J 1991. Reliability of phenotypic tests for
identification of Acinetobacter species. Journal of clinical microbiology, 29, 277282.
Ghigo JM 2003. Are there biofilm-specific physiological pathways beyond a reasonable
doubt? Research in microbiology, 154, 1-8.
Giamarellou H, Antoniadou A, Kanellakopoulou K 2008. Acinetobacter baumannii: a
universal threat to public health? International journal of antimicrobial agents,
32, 106-119.
Godoy CSM 2012. Infecções por Acinetobacter baumannii em adultos admitidos em
Unidades
de Terapia
Intensiva
(UTIs) de Goiânia
e Aparecida
de
Goiânia.Dissertação, Universidade Federal de Goiás, Goiânia, 71 pp.
Gohl O, Friedrich A, Hoppert M, Averhoff B 2006. The thin pili of Acinetobacter sp.
strain BD413 mediate adhesion to biotic and abiotic surfaces. Applied and
environmental microbiology, 72, 1394-1401.
Gordon CA, Hodges NA, Marriott C 1988. Antibiotic interaction and diffusion through
alginate and
exopolysaccharide of
cystic
fibrosis-derived
Pseudomonas
aeruginosa. The Journal of antimicrobial chemotherapy, 22, 667-674.
Goto T, Nakame Y, Nishida M, Ohi Y 1999. In vitro bactericidal activities of betalactamases, amikacin, and fluoroquinolones against Pseudomonas aeruginosa
biofilm in artificial urine. Urology, 53, 1058-1062.
Griffith ME, Ceremuga JM, Ellis MW, Guymon CH, Hospenthal DR, Murray CK 2006.
Acinetobacter skin colonization of US Army Soldiers. Infection control and
hospital epidemiology : the official journal of the Society of Hospital
Epidemiologists of America, 27, 659-661.
Gur D, Korten V, Unal S, Deshpande LM, Castanheira M 2008. Increasing carbapenem
resistance due to the clonal dissemination of oxacillinase (OXA-23 and OXA-58)producing Acinetobacter baumannii: report from the Turkish SENTRY Program
sites. Journal of medical microbiology, 57, 1529-1532.
62
Gusatti CS, Bertholdo LM, Otton LM, Marchetti DP, Ferreira AE, Corção G 2012. Fist
occurrence of blaOXA-58 in Acinetobacter baumannii isolated from a clinical
sample in southern Brazil. Braz J Microbiol, 243-246.
Hall-Stoodley L, Costerton JW, Stoodley P 2004. Bacterial biofilms: from the natural
environment to infectious diseases. Nature reviews. Microbiology, 2, 95-108.
Henderson B, Nair S, Pallas J, Williams MA 2011. Fibronectin: a multidomain host
adhesin targeted by bacterial fibronectin-binding proteins. FEMS microbiology
reviews, 35, 147-200.
Heritier C, Poirel L, Fournier PE, Claverie JM, Raoult D, Nordmann P 2005.
Characterization of the naturally occurring oxacillinase of Acinetobacter
baumannii. Antimicrobial agents and chemotherapy, 49, 4174-4179.
Hou TW, Yin XL, Jiang CY, Wang ZH, Chen QK, Chen X, Li W, Bai Y 2007.
[Microbiology and clinical analysis of six cases of hospital-acquired pneumonia
caused by Acinetobacter baumannii]. Zhonghua jie he he hu xi za zhi = Zhonghua
jiehe he huxi zazhi = Chinese journal of tuberculosis and respiratory diseases, 30,
35-39.
Hu WS, Yao SM, Fung CP, Hsieh YP, Liu CP, Lin JF 2007. An OXA-66/OXA-51-like
carbapenemase and possibly an efflux pump are associated with resistance to
imipenem in Acinetobacter baumannii. Antimicrobial agents and chemotherapy,
51, 3844-3852.
Iacono M, Villa L, Fortini D, Bordoni R, Imperi F, Bonnal RJ, Sicheritz-Ponten T, De
Bellis G, Visca P, Cassone A, Carattoli A 2008. Whole-genome pyrosequencing
of an epidemic multidrug-resistant Acinetobacter baumannii strain belonging to
the European clone II group. Antimicrobial agents and chemotherapy, 52, 26162625.
Iregbu KC, Ogunsola FT, Odugbemi TO 2002. Infections caused by Acinetobacter
species and their susceptibility to 14 antibiotics in Lagos University Teaching
Hospital, Lagos. West African journal of medicine, 21, 226-229.
Jain R, Danziger LH 2004. Multidrug-resistant Acinetobacter infections: an emerging
challenge to clinicians. The Annals of pharmacotherapy, 38, 1449-1459.
Jayaraman A, Wood TK 2008. Bacterial quorum sensing: signals, circuits, and
implications for biofilms and disease. Annual review of biomedical engineering,
10, 145-167.
63
Jeong SH, Bae IK, Kwon SB, Lee K, Yong D, Woo GJ, Lee JH, Jung HI, Jang SJ, Sung
KH, Lee SH 2005. Investigation of a nosocomial outbreak of Acinetobacter
baumannii producing PER-1 extended-spectrum beta-lactamase in an intensive
care unit. The Journal of hospital infection, 59, 242-248.
Jeong SH, Bae IK, Park KO, An YJ, Sohn SG, Jang SJ, Sung KH, Yang KS, Lee K,
Young D, Lee SH 2006. Outbreaks of imipenem-resistant Acinetobacter
baumannii producing carbapenemases in Korea. J Microbiol, 44, 423-431.
Joly-Guillou ML 2005. Clinical impact and pathogenicity of Acinetobacter. Clinical
microbiology and infection : the official publication of the European Society of
Clinical Microbiology and Infectious Diseases, 11, 868-873.
Jones RN, Deshpande L, Fritsche TR, Sader HS 2004. Determination of epidemic
clonality among
multidrug-resistant
strains
of
Acinetobacter
spp.
and
Pseudomonas aeruginosa in the MYSTIC Programme (USA, 1999-2003).
Diagnostic microbiology and infectious disease, 49, 211-216.
Kaper JB, Sperandio V 2005. Bacterial cell-to-cell signaling in the gastrointestinal tract.
Infection and immunity, 73, 3197-3209.
Kiffer C, Hsiung A, Oplustil C, Sampaio J, Sakagami E, Turner P, Mendes C 2005.
Antimicrobial susceptibility of Gram-negative bacteria in Brazilian hospitals: the
MYSTIC Program Brazil 2003. The Brazilian journal of infectious diseases : an
official publication of the Brazilian Society of Infectious Diseases, 9, 216-224.
Ko KS, Suh JY, Kwon KT, Jung SI, Park KH, Kang CI, Chung DR, Peck KR, Song JH
2007. High rates of resistance to colistin and polymyxin B in subgroups of
Acinetobacter baumannii isolates from Korea. The Journal of antimicrobial
chemotherapy, 60, 1163-1167.
Kolayli F, Gacar G, Karadenizli A, Sanic A, Vahaboglu H 2005. PER-1 is still
widespread
in
Turkish
hospitals
among
Pseudomonas
aeruginosa
and
Acinetobacter spp. FEMS microbiology letters, 249, 241-245.
Kumon H, Tomochika K, Matunaga T, Ogawa M, Ohmori H 1994. A sandwich cup
method for the penetration assay of antimicrobial agents through Pseudomonas
exopolysaccharides. Microbiology and immunology, 38, 615-619.
Landman D, Bratu S, Kochar S, Panwar M, Trehan M, Doymaz M, Quale J 2007.
Evolution
of
antimicrobial
resistance
among
Pseudomonas
aeruginosa,
Acinetobacter baumannii and Klebsiella pneumoniae in Brooklyn, NY. The
Journal of antimicrobial chemotherapy, 60, 78-82.
64
Lautrop H 1974. Bergey’s manual of determinative bacteriology. Williams & Wilkins
Co., Baltimore, MD.
Lee HW, Koh YM, Kim J, Lee JC, Lee YC, Seol SY, Cho DT 2008. Capacity of
multidrug-resistant clinical isolates of Acinetobacter baumannii to form biofilm
and adhere to epithelial cell surfaces. Clinical microbiology and infection : the
official publication of the European Society of Clinical Microbiology and
Infectious Diseases, 14, 49-54.
Lee JC, Koerten H, van den Broek P, Beekhuizen H, Wolterbeek R, van den Barselaar
M, van der Reijden T, van der Meer J, van de Gevel J, Dijkshoorn L 2006.
Adherence of Acinetobacter baumannii strains to human bronchial epithelial cells.
Research in microbiology, 157, 360-366.
Lessel EF 1971. Subcommittee on nomenclature of Moraxella and allied bacteria. Int J
Syst Bacteriol, 21, 213-214.
Loehfelm TW, Luke NR, Campagnari AA 2008. Identification and characterization of
an Acinetobacter baumannii biofilm-associated protein. Journal of bacteriology,
190, 1036-1044.
Lolans K, Rice TW, Munoz-Price LS, Quinn JP 2006. Multicity outbreak of
carbapenem-resistant
Acinetobacter
baumannii
isolates
producing
the
carbapenemase OXA-40. Antimicrobial agents and chemotherapy, 50, 2941-2945.
Mah TF, O'Toole GA 2001. Mechanisms of biofilm resistance to antimicrobial agents.
Trends in microbiology, 9, 34-39.
Mak JK, Kim MJ, Pham J, Tapsall J, White PA 2009. Antibiotic resistance determinants
in nosocomial strains of multidrug-resistant Acinetobacter baumannii. The
Journal of antimicrobial chemotherapy, 63, 47-54.
Marais E, de Jong G, Ferraz V, Maloba B, Duse AG 2004. Interhospital transfer of panresistant Acinetobacter strains in Johannesburg, South Africa. American journal
of infection control, 32, 278-281.
Martins AF, Kuchenbecker R, Sukiennik T, Boff R, Reiter KC, Lutz L, Machado AB,
Barth AL 2009. Carbapenem-resistant Acinetobacter baumannii producing the
OXA-23 enzyme: dissemination in Southern Brazil. Infection, 37, 474-476.
Maslow JN, Glaze T, Adams P, Lataillade M 2005. Concurrent outbreak of multidrugresistant and susceptible subclones of Acinetobacter baumannii affecting different
wards of a single hospital. Infection control and hospital epidemiology : the
official journal of the Society of Hospital Epidemiologists of America, 26, 69-75.
65
Melchior MB, Vaarkamp H, Fink-Gremmels J 2006. Biofilms: a role in recurrent
mastitis infections? Vet J, 171, 398-407.
Miro E, Mirelis B, Navarro F, Rivera A, Mesa RJ, Roig MC, Gomez L, Coll P 2005.
Surveillance of extended-spectrum beta-lactamases from clinical samples and
faecal carriers in Barcelona, Spain. The Journal of antimicrobial chemotherapy,
56, 1152-1155.
Mohamed JA, Huang DB 2007. Biofilm formation by enterococci. Journal of medical
microbiology, 56, 1581-1588.
Mugnier P, Poirel L, Pitout M, Nordmann P 2008. Carbapenem-resistant and OXA-23producing Acinetobacter baumannii isolates in the United Arab Emirates. Clinical
microbiology and infection : the official publication of the European Society of
Clinical Microbiology and Infectious Diseases, 14, 879-882.
Mugnier PD, Poirel L, Naas T, Nordmann P 2010. Worldwide dissemination of the
blaOXA-23 carbapenemase gene of Acinetobacter baumannii. Emerging
infectious diseases, 16, 35-40.
Munoz-Price LS, Weinstein RA 2008. Acinetobacter infection. The New England
journal of medicine, 358, 1271-1281.
Murray CK, Roop SA, Hospenthal DR, Dooley DP, Wenner K, Hammock J, Taufen N,
Gourdine E 2006. Bacteriology of war wounds at the time of injury. Military
medicine, 171, 826-829.
Naas T, Coignard B, Carbonne A, Blanckaert K, Bajolet O, Bernet C, Verdeil X,
Astagneau P, Desenclos JC, Nordmann P 2006. VEB-1 Extended-spectrum betalactamase-producing Acinetobacter baumannii, France. Emerging infectious
diseases, 12, 1214-1222.
Naas T, Kernbaum S, Allali S, Nordmann P 2007a. Multidrug-resistant Acinetobacter
baumannii, Russia. Emerging infectious diseases, 13, 669-671.
Naas T, Levy M, Hirschauer C, Marchandin H, Nordmann P 2005. Outbreak of
carbapenem-resistant Acinetobacter baumannii producing the carbapenemase
OXA-23 in a tertiary care hospital of Papeete, French Polynesia. Journal of
clinical microbiology, 43, 4826-4829.
Naas T, Nordmann P, Heidt A 2007b. Intercountry transfer of PER-1 extendedspectrum beta-lactamase-producing Acinetobacter baumannii from Romania.
International journal of antimicrobial agents, 29, 226-228.
66
Nadell CD, Xavier JB, Foster KR 2009. The sociobiology of biofilms. FEMS
microbiology reviews, 33, 206-224.
Navon-Venezia S, Leavitt A, Carmeli Y 2007. High tigecycline resistance in multidrugresistant Acinetobacter baumannii. The Journal of antimicrobial chemotherapy,
59, 772-774.
Nejjari N, Zerhouni F, Bouharrou A, Habzi A, Najdi T, Lahbabi M, Benomar S 2003.
[Nosocomial infections caused by Acinetobacter: experience in a neonatal care
unit in Casablanca]. La Tunisie medicale, 81, 121-125.
Nemec A, Musilek M, Sedo O, De Baere T, Maixnerova M, van der Reijden TJ,
Zdrahal Z, Vaneechoutte M, Dijkshoorn L 2010. Acinetobacter bereziniae sp.
nov. and Acinetobacter guillouiae sp. nov., to accommodate Acinetobacter
genomic species 10 and 11, respectively. International journal of systematic and
evolutionary microbiology, 60, 896-903.
Nichols WW, Dorrington SM, Slack MP, Walmsley HL 1988. Inhibition of tobramycin
diffusion by binding to alginate. Antimicrobial agents and chemotherapy, 32, 518523.
Nordmann P, Naas T 1994. Sequence analysis of PER-1 extended-spectrum betalactamase from Pseudomonas aeruginosa and comparison with class A betalactamases. Antimicrobial agents and chemotherapy, 38, 104-114.
Nordmann P, Poirel L 2002. Emerging carbapenemases in Gram-negative aerobes.
Clinical microbiology and infection : the official publication of the European
Society of Clinical Microbiology and Infectious Diseases, 8, 321-331.
Nucleo E, Steffanoni L, Fugazza G, Migliavacca R, Giacobone E, Navarra A, Pagani L,
Landini P 2009. Growth in glucose-based medium and exposure to subinhibitory
concentrations of imipenem induce biofilm formation in a multidrug-resistant
clinical isolate of Acinetobacter baumannii. BMC microbiology, 9, 270.
Opazo A, Dominguez M, Bello H, Amyes SG, Gonzalez-Rocha G 2012. OXA-type
carbapenemases in Acinetobacter baumannii in South America. Journal of
infection in developing countries, 6, 311-316.
Paterson DL, Bonomo RA 2005. Extended-spectrum beta-lactamases: a clinical update.
Clinical microbiology reviews, 18, 657-686.
Peleg AY, Bell JM, Hofmeyr A, Wiese P 2006a. Inter-country transfer of Gramnegative organisms carrying the VIM-4 and OXA-58 carbapenem-hydrolysing
enzymes. The Journal of antimicrobial chemotherapy, 57, 794-795.
67
Peleg AY, Franklin C, Bell JM, Spelman DW 2006b. Emergence of carbapenem
resistance in Acinetobacter baumannii recovered from blood cultures in Australia.
Infection control and hospital epidemiology : the official journal of the Society of
Hospital Epidemiologists of America, 27, 759-761.
Peleg AY, Seifert H, Paterson DL 2008. Acinetobacter baumannii: emergence of a
successful pathogen. Clinical microbiology reviews, 21, 538-582.
Perez F, Hujer AM, Hujer KM, Decker BK, Rather PN, Bonomo RA 2007. Global
challenge of multidrug-resistant Acinetobacter baumannii. Antimicrobial agents
and chemotherapy, 51, 3471-3484.
Picão RC 2009. Estudo das β-lactamases envolvidas na resistência às cefalosporinas de
amplo espectro em isolados clínicos de Pseudomonas aeruginosa Tese,
Universidade Federal de São Paulo, São Paulo 122 pp.
Playford EG, Craig JC, Iredell JR 2007. Carbapenem-resistant Acinetobacter baumannii
in intensive care unit patients: risk factors for acquisition, infection and their
consequences. The Journal of hospital infection, 65, 204-211.
Poirel L, Cabanne L, Vahaboglu H, Nordmann P 2005. Genetic environment and
expression of the extended-spectrum beta-lactamase blaPER-1 gene in gramnegative bacteria. Antimicrobial agents and chemotherapy, 49, 1708-1713.
Poirel L, Naas T, Nordmann P 2010. Diversity, epidemiology, and genetics of class D
beta-lactamases. Antimicrobial agents and chemotherapy, 54, 24-38.
Poirel L, Nordmann P 2006a. Carbapenem resistance in Acinetobacter baumannii:
mechanisms and epidemiology. Clinical microbiology and infection : the official
publication of the European Society of Clinical Microbiology and Infectious
Diseases, 12, 826-836.
Poirel L, Nordmann P 2006b. Genetic structures at the origin of acquisition and
expression of the carbapenem-hydrolyzing oxacillinase gene blaOXA-58 in
Acinetobacter baumannii. Antimicrobial agents and chemotherapy, 50, 14421448.
Prashanth K, Badrinath S 2004. In vitro susceptibility pattern of Acinetobacter species
to commonly used cephalosporins, quinolones, and aminoglycosides. Indian
journal of medical microbiology, 22, 97-103.
Prigent-Combaret C, Vidal O, Dorel C, Lejeune P 1999. Abiotic surface sensing and
biofilm-dependent regulation of gene expression in Escherichia coli. Journal of
bacteriology, 181, 5993-6002.
68
Quale J, Bratu S, Landman D, Heddurshetti R 2003. Molecular epidemiology and
mechanisms of carbapenem resistance in Acinetobacter baumannii endemic in
New York City. Clinical infectious diseases : an official publication of the
Infectious Diseases Society of America, 37, 214-220.
Quinteira S, Grosso F, Ramos H, Peixe L 2007. Molecular epidemiology of imipenemresistant Acinetobacter haemolyticus and Acinetobacter baumannii isolates
carrying plasmid-mediated OXA-40 from a Portuguese hospital. Antimicrobial
agents and chemotherapy, 51, 3465-3466.
Rao RS, Karthika RU, Singh SP, Shashikala P, Kanungo R, Jayachandran S, Prashanth
K 2008. Correlation between biofilm production and multiple drug resistance in
imipenem resistant clinical isolates of Acinetobacter baumannii. Indian journal of
medical microbiology, 26, 333-337.
Riley TV, Webb SA, Cadwallader H, Briggs BD, Christiansen L, Bowman RA 1996.
Outbreak of gentamicin-resistant Acinetobacter baumanii in an intensive care
unit: clinical, epidemiological and microbiological features. Pathology, 28, 359363.
Roberts SA, Findlay R, Lang SD 2001. Investigation of an outbreak of multi-drug
resistant Acinetobacter baumannii in an intensive care burns unit. The Journal of
hospital infection, 48, 228-232.
Rodriguez-Bano J, Marti S, Soto S, Fernandez-Cuenca F, Cisneros JM, Pachon J,
Pascual A, Martinez-Martinez L, McQueary C, Actis LA, Vila J 2008. Biofilm
formation in Acinetobacter baumannii: associated features and clinical
implications. Clinical microbiology and infection : the official publication of the
European Society of Clinical Microbiology and Infectious Diseases, 14, 276-278.
Rossau R, Landschoot AV, Gillis M, Ley JD 1991. Taxonomy of Moraxellaceae fam.
nov., a New Bacterial Family To Accommodate the Genera Moraxella,
Acinetobacter, and Psychrobacter and Related Organisms. Int J Syst Bacteriol, 41,
310-319.
Ruiz M, Marti S, Fernandez-Cuenca F, Pascual A, Vila J 2007. High prevalence of
carbapenem-hydrolysing oxacillinases in epidemiologically related and unrelated
Acinetobacter baumannii clinical isolates in Spain. Clinical microbiology and
infection : the official publication of the European Society of Clinical
Microbiology and Infectious Diseases, 13, 1192-1198.
69
Schimith Bier KE, Luiz SO, Scheffer MC, Gales AC, Paganini MC, Nascimento AJ,
Carignano E, Dalla Costa LM 2010. Temporal evolution of carbapenem-resistant
Acinetobacter baumannii in Curitiba, southern Brazil. American journal of
infection control, 38, 308-314.
Schulte B, Goerke C, Weyrich P, Grobner S, Bahrs C, Wolz C, Autenrieth IB,
Borgmann S 2005. Clonal spread of meropenem-resistant Acinetobacter
baumannii strains in hospitals in the Mediterranean region and transmission to
South-west Germany. The Journal of hospital infection, 61, 356-357.
Scott P, Deye G, Srinivasan A, Murray C, Moran K, Hulten E, Fishbain J, Craft D,
Riddell S, Lindler L, Mancuso J, Milstrey E, Bautista CT, Patel J, Ewell A,
Hamilton T, Gaddy C, Tenney M, Christopher G, Petersen K, Endy T, Petruccelli
B 2007. An outbreak of multidrug-resistant Acinetobacter baumanniicalcoaceticus complex infection in the US military health care system associated
with military operations in Iraq. Clinical infectious diseases : an official
publication of the Infectious Diseases Society of America, 44, 1577-1584.
Sechi LA, Karadenizli A, Deriu A, Zanetti S, Kolayli F, Balikci E, Vahaboglu H 2004.
PER-1 type beta-lactamase production in Acinetobacter baumannii is related to
cell adhesion. Medical science monitor : international medical journal of
experimental and clinical research, 10, BR180-184.
Segal H, Garny S, Elisha BG 2005. Is IS(ABA-1) customized for Acinetobacter? FEMS
microbiology letters, 243, 425-429.
Shahcheraghi F, Abbasalipour M, Feizabadi M, Ebrahimipour G, Akbari N 2011.
Isolation
and
genetic
characterization
of
metallo-beta-lactamase
and
carbapenamase producing strains of Acinetobacter baumannii from patients at
Tehran hospitals. Iranian journal of microbiology, 3, 68-74.
Shannon O, Morgelin M, Rasmussen M 2010. Platelet activation and biofilm formation
by Aerococcus urinae, an endocarditis-causing pathogen. Infection and immunity,
78, 4268-4275.
Shigeta M, Tanaka G, Komatsuzawa H, Sugai M, Suginaka H, Usui T 1997. Permeation
of antimicrobial agents through Pseudomonas aeruginosa biofilms: a simple
method. Chemotherapy, 43, 340-345.
Singh R, Paul D, Jain RK 2006. Biofilms: implications in bioremediation. Trends in
microbiology, 14, 389-397.
70
Siroy A, Cosette P, Seyer D, Lemaitre-Guillier C, Vallenet D, Van Dorsselaer A,
Boyer-Mariotte S, Jouenne T, De E 2006. Global comparison of the membrane
subproteomes between a multidrug-resistant Acinetobacter baumannii strain and a
reference strain. Journal of proteome research, 5, 3385-3398.
Skerman VBD, McGowan V, Sneath PHA 1980. Approved list of bacterial names. Int J
Syst Bacteriol, 30, 225-420.
Smani Y, McConnell MJ, Pachon J 2012. Role of fibronectin in the adhesion of
Acinetobacter baumannii to host cells. PloS one, 7, e33073.
Sohrabi N, Farajnia S, Akhi MT, Nahaei MR, Naghili B, Peymani A, Amiri Z, Rezaee
MA, Saeedi N 2012. Prevalence of OXA-Type beta-Lactamases Among
Acinetobacter baumannii Isolates from Northwest of Iran. Microb Drug Resist,
18, 385-389.
Stewart PS 1996. Theoretical aspects of antibiotic diffusion into microbial biofilms.
Antimicrobial agents and chemotherapy, 40, 2517-2522.
Stewart PS 1998. A review of experimental measurements of effective diffusive
permeabilities and effective diffusion coefficients in biofilms. Biotechnology and
bioengineering, 59, 261-272.
Stewart PS, Costerton JW 2001. Antibiotic resistance of bacteria in biofilms. Lancet,
358, 135-138.
SVS. Secretaria de Vigilância em Saúde. Ministério da Saúde.Clipping 06 a 08 de
março de 2010 2010:[292 p.].
Tack KJ, Sabath LD 1985. Increased minimum inhibitory concentrations with
anaerobiasis for tobramycin, gentamicin, and amikacin, compared to latamoxef,
piperacillin, chloramphenicol, and clindamycin. Chemotherapy, 31, 204-210.
Taherikalani M, Fatolahzadeh B, Emaneini M, Soroush S, Feizabadi MM 2009.
Distribution of different carbapenem resistant clones of Acinetobacter baumannii
in Tehran hospitals. The new microbiologica, 32, 265-271.
Takagi EH, Lincopan N, Cassettari VC, Passadore LF, Mamizuka EM, Martinez MB
2009. Carbapenem-resistant Acinetobacter baumannii outbreak at University
Hospital. Braz J Microbiol, 40, 339-341.
Tendolkar PM, Baghdayan AS, Gilmore MS, Shankar N 2004. Enterococcal surface
protein, Esp, enhances biofilm formation by Enterococcus faecalis. Infection and
immunity, 72, 6032-6039.
71
Tiwari V, Vashistt J, Kapil A, Moganty RR 2012. Comparative Proteomics of Inner
Membrane Fraction from Carbapenem-Resistant Acinetobacter baumannii with a
Reference Strain. PloS one, 7, e39451.
Tognim MC, Andrade SS, Silbert S, Gales AC, Jones RN, Sader HS 2004. Resistance
trends of Acinetobacter spp. in Latin America and characterization of international
dissemination of multi-drug resistant strains: five-year report of the SENTRY
Antimicrobial Surveillance Program. International journal of infectious diseases :
IJID : official publication of the International Society for Infectious Diseases, 8,
284-291.
Tomaras AP, Dorsey CW, Edelmann RE, Actis LA 2003. Attachment to and biofilm
formation on abiotic surfaces by Acinetobacter baumannii: involvement of a novel
chaperone-usher pili assembly system. Microbiology, 149, 3473-3484.
Tuomanen E, Cozens R, Tosch W, Zak O, Tomasz A 1986. The rate of killing of
Escherichia coli by beta-lactam antibiotics is strictly proportional to the rate of
bacterial growth. Journal of general microbiology, 132, 1297-1304.
Turrini RN, Santo AH 2002. [Nosocomial infection and multiple causes of death].
Jornal de pediatria, 78, 485-490.
Turton JF, Kaufmann ME, Warner M, Coelho J, Dijkshoorn L, van der Reijden T, Pitt
TL 2004. A prevalent, multiresistant clone of Acinetobacter baumannii in
Southeast England. The Journal of hospital infection, 58, 170-179.
Turton JF, Ward ME, Woodford N, Kaufmann ME, Pike R, Livermore DM, Pitt TL
2006a. The role of ISAba1 in expression of OXA carbapenemase genes in
Acinetobacter baumannii. FEMS microbiology letters, 258, 72-77.
Turton JF, Woodford N, Glover J, Yarde S, Kaufmann ME, Pitt TL 2006b.
Identification of Acinetobacter baumannii by detection of the blaOXA-51-like
carbapenemase gene intrinsic to this species. Journal of clinical microbiology, 44,
2974-2976.
Vahaboglu H, Budak F, Kasap M, Gacar G, Torol S, Karadenizli A, Kolayli F, Eroglu C
2006. High prevalence of OXA-51-type class D beta-lactamases among
ceftazidime-resistant clinical isolates of Acinetobacter spp.: co-existence with
OXA-58 in multiple centres. The Journal of antimicrobial chemotherapy, 58, 537542.
Vahaboglu H, Hall LM, Mulazimoglu L, Dodanli S, Yildirim I, Livermore DM 1995.
Resistance to extended-spectrum cephalosporins, caused by PER-1 beta72
lactamase, in Salmonella typhimurium from Istanbul, Turkey. Journal of medical
microbiology, 43, 294-299.
Vahaboglu H, Ozturk R, Aygun G, Coskunkan F, Yaman A, Kaygusuz A,
Leblebicioglu H, Balik I, Aydin K, Otkun M 1997. Widespread detection of PER1-type extended-spectrum beta-lactamases among nosocomial Acinetobacter and
Pseudomonas aeruginosa isolates in Turkey: a nationwide multicenter study.
Antimicrobial agents and chemotherapy, 41, 2265-2269.
Valenzuela JK, Thomas L, Partridge SR, van der Reijden T, Dijkshoorn L, Iredell J
2007. Horizontal gene transfer in a polyclonal outbreak of carbapenem-resistant
Acinetobacter baumannii. Journal of clinical microbiology, 45, 453-460.
van den Broek PJ, Arends J, Bernards AT, De Brauwer E, Mascini EM, van der Reijden
TJ, Spanjaard L, Thewessen EA, van der Zee A, van Zeijl JH, Dijkshoorn L 2006.
Epidemiology of multiple Acinetobacter outbreaks in The Netherlands during the
period 1999-2001. Clinical microbiology and infection : the official publication of
the European Society of Clinical Microbiology and Infectious Diseases, 12, 837843.
Van Looveren M, Goossens H 2004. Antimicrobial resistance of Acinetobacter spp. in
Europe. Clinical microbiology and infection : the official publication of the
European Society of Clinical Microbiology and Infectious Diseases, 10, 684-704.
van Soolingen D, de Haas PE, Hermans PW, van Embden JD 1994. DNA fingerprinting
of Mycobacterium tuberculosis. Methods in enzymology, 235, 196-205.
Vidal R, Dominguez M, Urrutia H, Bello H, Garcia A, Gonzalez G, Zemelman R 1997.
Effect of imipenem and sulbactam on sessile cells of Acinetobacter baumannii
growing in biofilm. Microbios, 91, 79-87.
Vila J, Ruiz J, Navia M, Becerril B, Garcia I, Perea S, Lopez-Hernandez I, Alamo I,
Ballester F, Planes AM, Martinez-Beltran J, de Anta TJ 1999. Spread of amikacin
resistance in Acinetobacter baumannii strains isolated in Spain due to an epidemic
strain. Journal of clinical microbiology, 37, 758-761.
Villegas MV, Hartstein AI 2003. Acinetobacter outbreaks, 1977-2000. Infection control
and hospital epidemiology : the official journal of the Society of Hospital
Epidemiologists of America, 24, 284-295.
Walsh C 2000. Molecular mechanisms that confer antibacterial drug resistance. Nature,
406, 775-781.
73
Werneck JS, Picao RC, Carvalhaes CG, Cardoso JP, Gales AC 2011a. OXA-72producing Acinetobacter baumannii in Brazil: a case report. The Journal of
antimicrobial chemotherapy, 66, 452-454.
Werneck JS, Picao RC, Girardello R, Cayo R, Marguti V, Dalla-Costa L, Gales AC,
Antonio CS, Neves PR, Medeiros M, Mamizuka EM, Elmor de Araujo MR,
Lincopan N 2011b. Low prevalence of blaOXA-143 in private hospitals in Brazil.
Antimicrobial agents and chemotherapy, 55, 4494-4495; author reply 4495.
Williams I, Venables WA, Lloyd D, Paul F, Critchley I 1997. The effects of adherence
to silicone surfaces on antibiotic susceptibility in Staphylococcus aureus.
Microbiology, 143 ( Pt 7), 2407-2413.
Williams JD 1999. Beta-lactamases and beta-lactamase inhibitors. International journal
of antimicrobial agents, 12 Suppl 1, S3-7; discussion S26-27.
Woodford N, Ellington MJ, Coelho JM, Turton JF, Ward ME, Brown S, Amyes SG,
Livermore DM 2006. Multiplex PCR for genes encoding prevalent OXA
carbapenemases in Acinetobacter spp. International journal of antimicrobial
agents, 27, 351-353.
Wroblewska MM, Sawicka-Grzelak A, Marchel H, Luczak M, Sivan A 2008. Biofilm
production by clinical strains of Acinetobacter baumannii isolated from patients
hospitalized in two tertiary care hospitals. FEMS immunology and medical
microbiology, 53, 140-144.
Xu KD, McFeters GA, Stewart PS 2000. Biofilm resistance to antimicrobial agents.
Microbiology, 146 ( Pt 3), 547-549.
Yamano Y, Nishikawa T, Fujimura T, Yutsudou T, Tsuji M, Miwa H 2006. Occurrence
of PER-1 producing clinical isolates of Pseudomonas aeruginosa in Japan and
their susceptibility to doripenem. The Journal of antibiotics, 59, 791-796.
Yong D, Shin JH, Kim S, Lim Y, Yum JH, Lee K, Chong Y, Bauernfeind A 2003. High
prevalence of PER-1 extended-spectrum beta-lactamase-producing Acinetobacter
spp. in Korea. Antimicrobial agents and chemotherapy, 47, 1749-1751.
Zarrilli R, Crispino M, Bagattini M, Barretta E, Di Popolo A, Triassi M, Villari P 2004.
Molecular epidemiology of sequential outbreaks of Acinetobacter baumannii in an
intensive care unit shows the emergence of carbapenem resistance. Journal of
clinical microbiology, 42, 946-953.
Zhou H, Pi BR, Yang Q, Yu YS, Chen YG, Li LJ, Zheng SS 2007. Dissemination of
imipenem-resistant Acinetobacter baumannii strains carrying the ISAba1
74
blaOXA-23 genes in a Chinese hospital. Journal of medical microbiology, 56,
1076-1080.
75
ANEXOS
Anexo 1. Aprovação pelo Comitê de Ética em Pesquisa
76
Anexo 2. Manuscrito
Acinetobacter baumannii PRODUTORES DE blaPER-1 ISOLADOS DE
PACIENTES DE UTIS DE GOIÂNIA E SUA CORRELAÇÃO COM A
FORMAÇÃO DE BIOFILME E RESISTÊNCIA MULTIDROGAS NA
PRESENÇA DE GENES TIPO OXA
Autores:
Suellen Rocha Araújo Castilho a
Cássia Silva de Miranda Godoy a, b
Adriana Oliveira Guilarde a
Ana Paula Junqueira-Kipnis a
André Kipnis a
a
b
Instituto de Patologia Tropical e Saúde Pública da Universidade Federal de Goiás
Hospital de Doenças Tropicais / Hospital Anuar Auad
77
Acinetobacter baumannii PRODUTORES DE blaPER-1 ISOLADOS DE
PACIENTES DE UTIS DE GOIÂNIA E SUA CORRELAÇÃO COM A
FORMAÇÃO DE BIOFILME E RESISTÊNCIA MULTIDROGAS NA
PRESENÇA DE GENES TIPO OXA
blaPER01 Producing Acinetobacter baumannii isolates form patients at ICUs of
Goiania and correlation between biofilm formation and multidrug resistance in the
presence of OXA genes.
Abstract
Background: Gram negative bacilli are one of the major causes of nosocomial
infections worldwide. Among them, non-fermenting Gram Negative bacilli play a
considerable part. Acinetobacter baumannii is one of the main representatives of this
group. Purpose: To investigate by a quantitative method the biofilm formation and to
examine the correlation between biofilm and antibiotic resistance among clinical
isolates of A. baumannii. The association between biofilm formation and presence of
extended spectrum β-lactamases blaPER-1, as well as correlation between biofilm and
presence of OXA-type genes to understand the antibiotic resistance among isolates were
also analyzed. Methods: A total of 71 clinical isolates of A. baumannii were subjected
to identification testing by Gram staining and biochemical tests, and drug susceptibility
testing by disc diffusion. The biofilm production was done by quantitative method in
microtitre plate assay. The presence of blaPER-1 and OXA-type genes was detected by
PCR. Results: In our study, 29% of the isolates presented blaPER-1 gene, and all of
them were resistant to cefepime. The concomitant resistance to cefepime, ceftazidime,
ciprofloxacin, and imipenem was observed in 81% of the blaPER-1 positive isolates.
Fifty seven of the 71 isolates were able to form biofilm. Almost 60% of the blaPER-1
positive isolates formed biofilm. Also, most of the isolates (90%) that did not have
blaPER-1 formed biofilm. The majority of the isolates that were capable of forming
biofilm also presented resistance to several classes of antimicrobial drugs. Among the
55 isolates biofilm producers that were resistant to some antibiotics, 20% had only the
OXA-51 gene, 62% had OXA-23 gene, 56% had ISAba1/OXA-23, 40% had
ISAba1/OXA-51, 24% had OXA-58 and 5% had ISAba1/OXA-58. Conclusion: The
blaPER-1 gene promotes resistance to cefepime, but the presence of this gene was not
related to the biofilm formation among the isolates evaluated. This study detected a high
percentage of ISAba1/OXA-23 producers isolates among the biofilm formation,
suggesting that the antibiotic resistance of isolates in question also relates to the
expression of these genes.
Resumo
Bacilos Gram-negativos são uma das principais causas de infecções hospitalares em
todo o mundo. Entre eles, os bacilos Gram-negativos não fermentadores desempenham
um papel considerável. O Acinetobacter baumannii é um dos principais representantes
desse grupo. Objetivo: Investigar por método quantitativo a formação de biofilme e
examinar a correlação entre biofilme e resistência antimicrobiana entre os isolados
clínicos de A. baumannii. Foi verificado também a associação entre biofilme e a
presença da β-lactamase de espectro estendido blaPER-1, bem como a correlação entre
78
biofilme e a presença de genes do tipo OXA a fim de compreender a resistência
multidrogas entre os isolados. Métodos: Um total de 71 isolados clínicos de A.
baumannii foram submetidos a testes de identificação por coloração de Gram e provas
bioquímicas, e susceptibilidade microbiana pelo método de disco difusão. A produção
de biofilme foi feita pelo método quantitativo em placa de microtitulação. A presença de
blaPER-1 e dos genes tipo OXA foram detectados por PCR. Resultados: Em nosso
estudo, 29% dos isolados apresentaram o gene blaPER-1, e todos eles foram resistentes
à cefepima. A resistência concomitante à cepepima, ceftazidima, ciprofloxacina e
imipenem foi observada em 81% dos isolados positivos para blaPER-1. Para formação
de biofilme, 57 dos 71 isolados foram capazes de formar biofilme. Quase 60% dos
isolados blaPER-1 positivos formaram biofilme. No entanto, a maioria dos isolados
(90%) que não tinham blaPER-1, também formaram biofilme. A maioria dos isolados
que foram capazes de formar biofilme também apresentaram resistência à várias classes
de antimicrobianos. Dentre os 55 isolados formadores de biofilme que apresentaram
resistência a algum antibiótico, 20% possuíam apenas o gene OXA-51, 62% possuíam
OXA-23, 56% possuíam ISAba1/OXA-23, 40% possuíam ISAba1/OXA-51, 24%
possuíam OXA-58 e 5% possuíam ISAba1/OXA-58. Conclusão: O gene blaPER-1
promove resistência à cefepima, porém a presença deste gene não está relacionada com
a formação de biofilme dentre os isolados avaliados. Este estudo apresenta uma alta
porcentagem de isolados produtores de ISAba1/OXA-23 entre os formadores de
biofilme, sugerindo que a resistência antimicrobiana dos isolados em questão também
esteja relacionada com a expressão destes genes.
79