20/03/2012
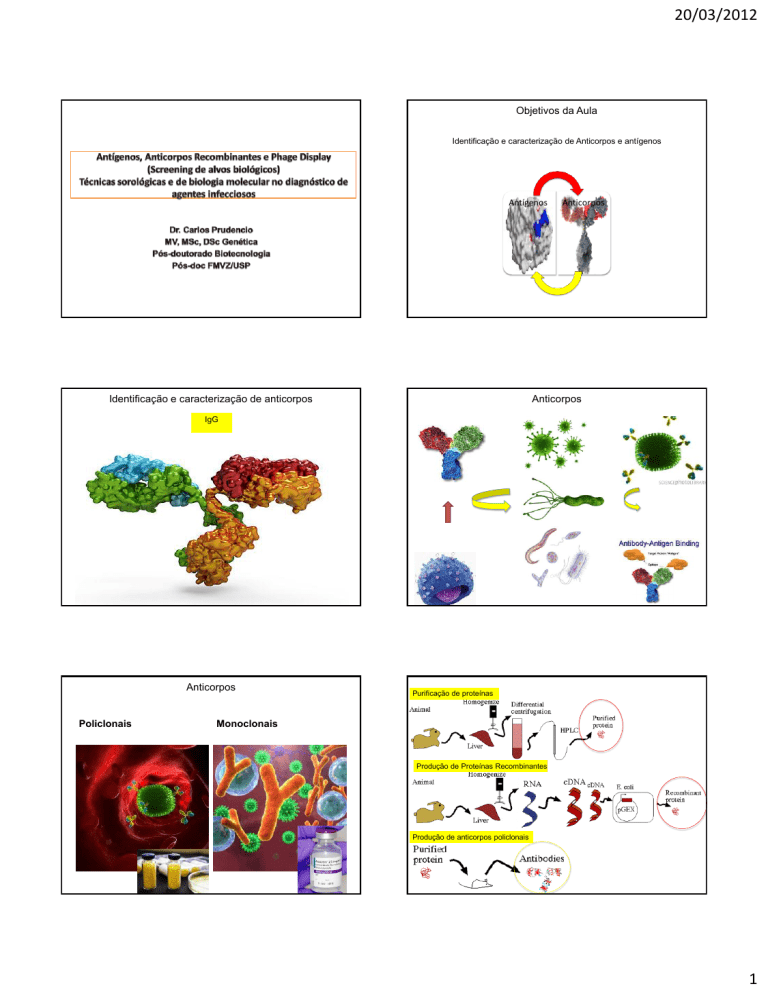
Objetivos da Aula
Identificação e caracterização de Anticorpos e antígenos
Antígenos
Identificação e caracterização de anticorpos
Anticorpos
Anticorpos
IgG
Anticorpos
Policlonais
Purificação de proteínas
Monoclonais
Produção de Proteínas Recombinantes
Produção de anticorpos policlonais
1
20/03/2012
Anticorpos Monoclonais
Anticorpos Monoclonais
kits para diagnósticos
Pesquisa científica
Imunoterapia
(Humanização)
Kits de Diagnóstico
Imunoterapia
Vantagens mAbs
•
•
•
•
•
•
•
•
Poucos animais em teste
Alta especificidade;
Alta afinidade;
Determinação de um só epítopo
de um antigénio;
Alta homogeneidade;
Ausência de anticorpos não
específicos;
Maior facilidade de
caracterização;
Reduzida variabilidade de lote
para lote;
2
20/03/2012
Desvantagens mAbs
Estrutura de anticorpos
• Normalmente são extremamente caros
• Reconhecimento de um só epítopo de um antigénio. Esse epítopo pode
estar destruído (por ex: pela fixação) mesmo que exista a molécula total
do antigénio
• Se o soro não foi produzido contra um epítopo específico do antigénio
que se quer detectar e se existirem outros antigénios com esse epítopo,
a imensa vantagem da alta especificidade será inútil
Paratopo
VH E VL
FRAME WORK
CDRS
REPERTÓRIO IMUNE
3
20/03/2012
Hibridoma
s
Produção
Métodos de identificação de mAbs
Indústria
Sequenciamento de Repertório Imune
Phage Display: O que é?
Phage display is a powerful tool for selecting peptides, proteins, or
antibodies with specific binding properties from a large number of variants.
The technology consists in expressing peptides, proteins or antibody
fragments at the surface of phage particles (Smith, 1985; Winter et al.,
1994; Kay and Hoess,1996). The purpose is to screen for proteins with
specific properties selected from libraries of DNA variants expressed into
populations of protein variants on phages surfaces.
Phage display: Para que serve?
Reagentes para
diagnóstico,
Bionanotecnologia
Antígenos vacinais
Método para
estudo da
interação entre
biomoléculas
Estudos moleculares
na relação patógeno
hospedeiro
Novas drogas
Ativadores/inibidores
enzimáticos
4
20/03/2012
O Que é Phage Display?
Source: Carolos F. Barbas et al. 1991. PNAS 79:78
Gao C et al. PNAS 1999;96:6025-6030
©1999 by The National Academy of Sciences
Estrutura bacteriófagos filamentosos
Ciclo de bacteriófagos filamentosos
M13
Escherichia coli
Source: Carolos F. Barbas et al. 1991. PNAS 79:78
M13
Histórico sobre a metodologia do Phage Display
George P. Smith, Science (1985)
Expressão funcional de polipetídeos de E. coli em bacteriófagos com atividade
funcional de enzimas
5
20/03/2012
Os grandes diferenciais da técnica:
(i) Ligação física entre o
genótipo (DNA) e o fenótipo
(proteína expressa)
Bioseleção (Seleção para enriquecimento)
Amplificação
de fagos em
E. coli
(ii) A triagem envolve
bilhões de peptídeos ou
proteínas (1012)
(iii) flexibilidade de
plataformas de seleções
Relação sobre a Bioseleção e especificidade
Fagos específicos
1012
Biblioteca
de fagos
fagos 108
Seleção
específica de
clones raros
Seleção por
afinidade in
vitro
Fagos inespecíficos
104
Diversidade
0
1
2
3
4
Ciclos de seleção
5
Construção de bibliotecas imunes de anticorpos
Linfócitos B
6
20/03/2012
Molecules 2011, 16, 412-426;
doi:10.3390/molecules16010412
Schematic representation of the selection strategy.
Produção de scFvs neutralizantes de toxinas de
serpentes
Fig.
1
Hof D et al. Mol Cell Proteomics 2006;5:245-255
©2006 by American Society for Biochemistry and Molecular Biology
Genetically Engineered Pea Seeds Protect Against
Parasites
Combinatorial antibody libraries from survivors of the Turkish H5N1 avian influenza outbreak
reveal virus neutralization strategies
Example sequences displaying the immunochemical basis of neutralization found from survivor 5
libraries after H5N1 Vietnam panning
virus neutralization strategies
Zimmermann J et al. (2009) Antibody expressing pea seeds as fodder for prevention
of gastrointestinal parasitic infections in chickens. BMC Biotechnol 9, 79
PNAS April 22, 2008 vol. 105 no. 16 5986-5991
7
20/03/2012
Identificação e Caracterização de Antígenos
Recombinantes
Produtos comerciais
Anti - TNFa
rheumatoid arthritis
Vacinas
Métodos de Diagnóstico
Patógeno Hospedeiro
Enzimas Substratos
Entrega de drogas
Detalhe sobre a construção de bibliotecas genômicas
(DNA)
Appl Microbiol Biotechnol
(2008) 80:447–458
Seleção de polipeptídeos imunogênicos de
microorganismos (Mycoplasma hyopneumoniae )
Identification of immunogenic polypeptides
from a Mycoplasma hyopneumoniae genome
library by phage display
Appl Microbiol Biotechnol (2008) 80:447–
458
8
20/03/2012
Validação para antigenicidade dos clones isolados:
Phage ELISA
Enriquecimento de clones específicos
O Sequenciamento de DNA permite Determinação das
sequencias peptídicas isoladas por afinidade ao ligante
Inhibition Dot Blot assay
The phage particles spotted on nitrocellulose membrane can not be recognized by the purified
IgY when first incubated with the total extract proteins ofR. (B.)microplus.
Sequenciamento de DNA
C
1
2
3
4
5
6
7
8
9
10
11
12
Figura – Phage DNA
DNA traduzido
DNA Sequencing
the selected phage clones could be inhibited in a manner dose response when the extract
proteins was add gradually in the purified IgY solution.
1.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
30.
31.
32.
33.
34.
35.
36.
37.
38.
39.
40.
41.
42.
43.
44.
45.
46.
47.
48.
49.
YNSTASFSTPHL
HTMTSASTSSLL
QYAPSTNSYLLL
INPIHNSTASFT
FVPFASTSSNLA
VYANQASTASRL
HSTEAASTSSFA
FASDMRPSTSSF
FNSTSSLLTSSP
LESRYFPVITPT
FNNTNSTSSFVS
QWNTSSFAVYDP
LEYHQVTLRDVL
ESAAPYSTNSFA
HLASSSTSSSLG
FNRLPYSTSSSL
NDRNSLNTTSFL
ASQMVSTSSFND
HVMSPHPQSTLL
TPVNEAQNTASF
GTWNNTSSLLLN
MHMTHSSTASYL
SALNSTSSFSSH
MRAVSTSSFPAL
KLSLHNTSSVLT
ANLHHPSTSSQL
DQRMASTASFSS
QYSTSSSLGISI
NRYTTSHILSPV
GDKHSTSSFLIT
TSNSTASFSSPA
MDMAQLSTSSNL
NYHHQIAYALLD
TPYSMSSTASRL
GYSFENSTSSPL
KPLLASSTSSTL
WALAPSTASFLS
TLTYAPSTSSPL
MYHNNTSSWLGV
SPPEFSNSTASF
MHISDMSTSSML
YTQNSTASFGPW
SPNTSSFTEADL
FINHASSTSSSL
LAPIGNSTTSFL
EISKAFSTSSFH
APNQFRSTASEL
QNWTSSTSSFSQ
LPTIPYSTASDL
O alinhamento e a busca de similaridade de sequências
se faz necessário para a identificaçao de alvos em
bancos de dados
Epitope mapping and identification of potential
vaccine candidate antigens
Mapeamento clássico de epítopos por phage display em mAbs de B. bigemina
Antigenic validation
A B C A B C A B C A B C
An IgG-binding protein A homolog in
Staphylococcus hyicus
A B C A B C A B C
Blank
Control
phages
35/49
(Critical motif for
this epitope)
)
Figura – Alinhamento linear das seqüências de aminoácidos dos peptídeos selecionados por afinidade com a proteína
MSP1 de Anaplasma marginale realizado pelo programa MATCH.
Veterinary Microbiology 149 (2011) 273–276
9
20/03/2012
Para a validação dos alvos biológicos e necessário a
expressão do gene completo
Bibliotecas de peptídeos naturais (shot-gun) X sintéticas
(NNK)
Libraries
Desenvolvimento de vacinas:
Mimotopos
General Procedure: Biopanning
PIII-protein
MIMOTOPOS
Figuras da dyax
Seleção por adorsorção
10
20/03/2012
Bibliotecas combinatoriais de anticorpos contra
proteínas do R. microplus para a descoberta de
antígenos vacinais
Ticks colection
Tick Protein
extract
overlap
(scFv)
Phagemide ligation
Transformation and add
Helper phage
Sequencing of
phage DNA
Panning with phage
libraries
Selection of tick peptides
epitopes
In silico analyses
•
SDS PAGE
Immunized
Chickens
Total Proteins
Amplification of
the light (VL)
and heavy
chain
fragments (VH)
Phage-ELISA
Panning with peptides
libraries
cDNA
Neutralização/Adesão
Salivary gland
metalloprotease
G-protein coupled receptor
Calreticulin
Glutathione S-transferase
Glyceraldeyde-3-phosphate
dehydrogenase
?
GP80 Precursor
Protective antigen 4D8
TheRhipicephalus
(Boophilus)
microplusproteins
matched by the
phagotopes.
Rev. Bras. Parasitol. Vet., v. 18, n. 1, p. 39-41,
jan.-mar. 2009 ISSN 1984-2961
humoral response in naturally
infested animals
Colected the Splen
Dot Blots,
Anticorpos
Western Blots (2D) and
N-terminal sequencing
scFV Production and
purification
The data indicate that the recombinant phage proteins can be specifically recognized by the
target.
Validation tests (in vitro,
in vivo)
Grouping of the
consensus sequences
among the phagotopes.
Dot Blots tests = Mostly of the 110 different sequences
were recognized by the purified IgY to different degrees.
ELISA Titering
mRNA from Spleen
Sequencing of the
light and heavy
variable chains of
the scFv fragments
Immunoreativity analysis of the phagotopes.
Egg cathepsin
Bm95
GP80 Precursor
Intracellular Cystatin
Bm86 Intestinal Protein
Phospholipids
Salivary gland metalloprotease
Cathepsin-type protein precursor
Paramyosin
Calreticulin
Membrane B antigen
P450 CYP319A1
Heme-binding aspartatic proteinase
Reverse transcriptase-type protein
Glyceraldehyde-3-phosphate
dehydrogenase
we have used those sequences to produce multiple antigen peptide system
(MAPS), which will be used in future field tests.
Artificial Gene A:
Cytochrome oxidase Subunit 1
NADH dehydrogenase subunit 2
Cytochrme P450
Acetylcholinesterase
Egg cathepsin
GP80 Precursor
Glucose 6-phosphate dehydrogenase
isoform
Actin
Salivary gland metalloprotease
Ferritin
Membrane B antigen
Acetylcholinesterase
esterase
Heme-binding aspartatic proteinase
Cytochrome P450
Carboxylesterase-related protein
Cytochrome P450 4W1
Angiotensin converting enzyme
Prudencio, C.R., et al., Recombinant peptides as new immunogens for
the control of thebovine tick, Rhipicephalus (Boophilus) microplus. Vet.
Parasitol. (2010), doi:10.1016/j.vetpar.2010.04.012
Artificial Gene B:
IPTG M
TO
T1H
T2H
T3H
Immunoreactivity
analysis of the
artificial tick
genes.
M
GA
GB
GA
GB
GA
GB GA
GB
Western blot analysis showed that the artificial tick protein expressed by origami cells
transformed with pET-32 reacted with the Chicken pAbs rased against ticks pts.
Mapping protective epitopes in the tick and mosquito
subolesin ortholog proteins
Vaccine. 2010 Jul 26;28(33):5398-406.
Pepscan
A) Subolesin
B) Subolesin Ortholog
11
20/03/2012
Localization of the predicted conformational
discontinuous epitopes
4D12 monoclonal antibody recognizes a new linear epitope from
SAG2A Toxoplasma gondii tachyzoites, identified by phage display
bioselection.
Vaccine. 2010 Jul 26;28(33):5398-406.
J.P. Cunha-Ju ́nior et al. / Immunobiology 215 (2010) 26–37
4D12 monoclonal antibody recognizes a new linear epitope from
SAG2A Toxoplasma gondii tachyzoites, identified by phage display
bioselection. Immunobiology, v. 215, p. 26-37, 2010.
Peptide mimicking antigenic and immunogenic epitope of
neuwiedase from Bothrops neuwiedi snake venom.
Métodos para imunoaglutinação em micro e nanoesferas de poliestireno.
J.P. Cunha-Ju ́nior et al. / Immunobiology 215 (2010) 26–37
Antigenic Fingerprinting of H5N1 Avian Influenza Using
Convalescent Sera and Monoclonal Antibodies Reveals
Potential Vaccine and Diagnostic Targets
Elucidation of antibody repertoires elicited in humans
after vaccination with unadjuvanted and adjuvanted
subunit H5N1 vaccines.
Epitope Profile in H5N1 Survivors
post–second vaccination sera
April 2009 | Volume 6 |
post–third vaccination sera
Science translational medicine 2, no. 15 (January 2010)
12
20/03/2012
Definições das propriedades da resposta imune contra
os epítopos de antígenos vacinais
HIV-C gp160
•
to define properties of immune responses against biomedically important antigens as they occur in
vivo.
(From Persson et al. (2006) J Mol Biol 357, 607-620.)
Dissecting the natural
humoral immune
response in virusinfected persons able
to control the viremia
Estudo de interações entre patógenos e hospedeiros
(Avian anticoccidial activity of a novel membrane-interactive peptide selected
from phage display libraries)
PW2
A. da Silva, Jr et al. / Molecular & Biochemical Parasitology
120 (2002) 53–60
host–pathogen
and in vivo
panning
Investigation
of interactions
host–pathogen
interactions
Entrega de medicamentos e drogas
13
20/03/2012
Entrega de medicamentos e drogas
Entrega de medicamentos e drogas
Interactome discovery pipeline.
Estratégia: Construção de bibliotecas a partir de fragmentos de
DNA genômico de R. rickettsii em vetores do tipo fagomídeos
Di Niro R et al. Nucl. Acids Res. 2010;38:e110-e110
© The Author(s) 2010. Published by Oxford University Press.
Análises de Bioinformática
Expressão e validação de proteínas candidatas
14
20/03/2012
MUITO OBRIGADO!
15