nº 36 (1)
ISSN: 1980-8976
agoa Comprida (Macaé-RJ)
Foto: L. P. Nielsen
Maio de 2007
Foto: Alex Enrich-Prast
Editor
Alex Enrich-Prast
[email protected]
Editores Executivos
Ana Lúcia Santoro
Angela M. Sanseverino
Breno A. Guimarães-Souza
Humberto Marotta
Luana Q. Pinho
Luiz Fernando J. Bento
[email protected]
SBL 2005 - 2007
Presidente
Ricardo Motta Pinto Coelho
[email protected]
Primeira Secretária
Renata Panosso
[email protected]
Primeiro Tesoureiro
Marcos Callisto
[email protected]
Vice Presidente
Antônio Fernando M. Camargo
[email protected]
Segunda Secretária
Andrea Figueiredo
[email protected]
Segundo Tesoureiro
Luís Maurício Bini
[email protected]
Mensagem do presidente ................................. 1
Mensagem do Editor do Boletim ..................... 2
Mensagem do Editor da Acta........................... 3
Métodos e Equipamentos ................... 4
Tópicos Especiais................................12
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
Maio de 2007
ISSN: 1980-8976
Mensagem do Presidente
Prezados Limnólogos
Este é o último trimestre da atual diretoria.
Ainda não deu tempo para que pudéssemos fazer
uma avaliação mais detalhada do que pudemos
ou não fazer nesse período. No entanto, já estamos
disponibilizando para os nossos sócios uma
prestação preliminar de nossas contas que já pode
ser acessada a partir do nosso portal na internet:
http://www.sblimno.org.br/. Através dessa
prestação de contas, o nosso associado poderá
perceber que não foram dois anos muito tranqüilos
e que a SBL enfrentou pesadas turbulências nessa
gestão.
No momento, estamos muito envolvidos nos
preparativos do XI Congresso Brasileiro de
Limnologia que será realizado entre os dias 24 e
28 de agosto em Macaé. Já estivemos no Rio de
Janeiro (UFRJ) em duas ocasiões. Recentemente,
recebemos a vista do Prof. Vinicius Farjalla, um
dos membros do comitê organizador aqui na
UFMG e, logo em seguida, o nosso tesoureiro,
Marcos Callisto, esteve em Macaé onde
encontrou-se com o Prof. Francisco Esteves. A
nossa secretária Andréa Figueiredo tem trabalhado
em conjunto com o comitê organizador atuando
principalmente no delineamento e na captação de
recursos para uma série de eventos centrais que
serão oferecidos no XI CBL. E a nossa secretária,
a Profa. Renata Panosso, apesar da distância (ela
está em Natal, RN), tem dado constante apoio em
diversas questões administrativas. Ela também tem
atuado no sentido de afinar a sintonia entre a
diretoria da sociedade e o comitê organizador.
Apesar da constante troca de e-mails, avaliamos
o quanto foram (e são) importantes esses contatos
pessoais. Organizar um congresso do porte do XI
CBL não é fácil. Ainda mais quando temos que
enfrentar muitas dificuldades sejam elas no âmbito
da falta de infra-estrutura administrativa (ver
abaixo). Os nossos sócios podem até imaginar que
a S.B.L dispõe de muitos recursos, ao consultarem
nº 36 (1)
os nossos extratos na internet. No entanto,
quando comparamos os orçamentos de alguns
congressos de grandes sociedades científicas
brasileiras (exemplo: Associação Brasileira de
Recursos Hídricos – ABRH) é fácil constatar que
a S.B.L ainda tem um longo caminho a percorrer
não só em termos de ter uma capacidade de
alocar maiores investimentos, mas sobretudo em
trabalhar com uma maior antecedência na
organização dos nossos grandes eventos
científicos. Hoje, o ideal seria se a S.B.L. já
tivesse uma comissão organizando o XII C.B.L.
Apesar dos percalços, estamos muito
felizes com os números parciais de inscrições e
envio de resumos ao XI C.B.L. O prazo final
para as inscrições e envio de resumos expira-se
em 30 de maio, mas os nossos números de hoje
(dia 11/5) indicam 1.455 inscrições feitas das
quais 381 delas já estão efetivamente pagas. 266
resumos já foram enviados ao nosso portal. O
total arrecadado é de R$ 51.020,00 (11/5). O
portal da SBL tem se mostrado eficaz para o
gerenciamento das inscrições bem como no
gerenciamento dos resumos recebidos. Temos
tido alguma dificuldade em termos de apoio por
telefone aos usuários do portal, um serviço que
demanda considerável investimento em mão de
obra devidamente treinada para essa finalidade.
Esperamos que esse serviço seja disponibilizado
pela comissão organizadora, em breve. Outro
serviço que será implantado em breve será a
introdução do pagamento das inscrições via
cartão de crédito VISA o que irá facilitar muito
o acesso dos limnólogos estrangeiros ao XI CBL.
Embora existam muitos desafios a serem
vencidos, estamos confiantes com a equipe que
concebeu e vem administrando todo o software
de gerenciamento de inscrições e de resumos.
Ela foi montada integralmente com estudantes
de pós-graduação da UFMG e tem se mostrado
competente para administrar e promover
contínuas melhorias no sistema. Para que nossos
sócios tenham uma idéia do desafio, quase todos
os dias o nosso servidor tem sido alvo de ataques
http://www.sblimno.org.br
1
de todo tipo de hackers. O sistema desenvolvido
tem se mostrado bastante eficiente. Uma série de
ferrramentas tais como o back up em disco rígido
externo, atualizações de sistema operacional feitas
em freqüência diária, vários tipos de firewall e o
uso de antivírus mais potentes têm garantido uma
grande estabilidade e confiabilidade nos acessos dos
usuários do portal da SBL.
Apesar de estarmos muito focados na
organização do XI C.B.L., um outro aspecto
positivo nos chamou a atenção, recentemente. Tratase da constatação do Editor Chefe da Acta
Limnologia Brasiliensia, Prof. Raoul Henry, do
claro aumento do número de novas submissões que
vem ocorrendo em 2007. Esse fato possivelmente
reflete toda uma série de medidas que vem sendo
tomadas não somente pela diretoria da SBL, mas
também pelo editor chefe da revista que somente
agora começam a aparecer. Dentre essas medidas,
podemos mencionar o suporte dado pelo CNPq à
editoração da Acta e a criação da web site da revista
que culminou com a sua inclusão no portal da
CAPES no início de 2007. Apesar de todos esses
avanços, a diretoria entende que a Acta deve mudar
em muitos aspectos. Nesse sentido, estamos
propondo uma reunião com o editor chefe da revista
em junho próximo, em Belo Horizonte. Essa reunião
pretende gerar um conjunto de sugestões de
melhorias para essa revista que possa ser oferecido
para a devida apreciação em nossa próxima
Assembléia Geral.
Gostaria ainda de lembrar aos nossos sócios
que este é um ano muito importante para a S.B.L
pois ela está completando 25 anos de vida. A SBL
foi fundada em uma reunião da S.B.P.C. em
Campinas, no longínquo ano de 1982. Como seria
de se esperar, a sociedade passou por bons e maus
momentos desde então. Hoje, eu não tenho dúvidas
em afirmar que, apesar do grande crescimento em
termos quantitativos e de termos atingido metas
gloriosas, a S.B.L. ainda necessita de cuidados
especiais por parte dos seus associados. Quando
eu assumi a S.B.L., em agosto de 2005, o expresidente Fábio Roland já alertava para a atual
diretoria sobre a urgência para o estabelecimento
de uma sede fixa, com um corpo administrativo
estável que garantisse a continuidade na gestão da
sociedade. Na atual gestão, não tivemos condições
concretizar essa antiga aspiração da S.B.L.
Esperamos que a próxima diretoria possa ter as
condições para realizar essa importante tarefa. A
criação de uma sede fixa é de fundamental
importância para toda a vida administrativa da
S.B.L. Contamos hoje com um número elevado
de sócios e já estamos décima primeira versão
de nosso congresso. A simples administração
desses dois aspectos já demanda um
considerável esforço em termos de trabalho de
secretaria. Inúmeras pendências são
direcionadas para a diretoria todos os dias.
Hoje, sabemos que as grandes sociedades
científicas brasileiras já organizam seus
congressos até com quatro anos de
antecedência. Fora isso, ainda existe a questão
da administração do portal de internet que a
cada dia fica mais complexo e, em futuro
próximo, ele deverá oferecer toda uma série
de serviços adicionais tais como novas
publicações on line, pesquisas com os sócios,
fóruns de discussão e uma variada gama de
atividades e novos serviços. Sendo assim, está
mais do que na hora da S.B.L. se preparar de
forma adequada para esses novos desafios.
Não gostaria de terminar esse texto sem
antes estimular a todos os limnólogos atuantes
no Brasil a comparecerem ao XI CBL em
Macaé. Tenho a convicção de que esse será um
excelente congresso. Precisamos também do
comparecimento do maior número possível de
associados em nossa Assembléia Geral da
sociedade. A SBL necessita de tomar
importantes decisões que somente podem ser
tomadas com o apoio de todos.
Ricardo Motta Pinto Coelho
[email protected]
Universidade Federal de Minas Gerais
Mensagem do Editor do Boletim
Mais um volume com importantes
contribuições foi finalizado. A nossa principal
novidade é a obtenção do ISSN, o “Número
Internacional Normalizado para Publicações
Seriadas” para o Boletim da Sociedade
Brasileira de Limnologia (BSBL). A obtenção
deste número possibilita uma maior
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
2
visibilidade e reconhecimento ao BSBL. Na
próxima assembléia da Sociedade Brasileira de
Limnologia será discutido se o BSBL continua
sendo publicado apenas eletronicamente, se seus
artigos deveriam ser submetidos à revisores
externos, e quais rumos ele deveria tomar.
A seção “Tópicos Especiais” deste volume
tem como tema a Microbiologia Aquática.
Diversos autores deram excelentes contribuições
nesta área, e mais uma vez agradecemos a todos
eles. Como recebemos poucas submissões
espontâneas sobre outros assuntos, a maioria dos
artigos publicados nesta edição refere-se àquele
tema. Novamente faço um apelo para que
limnólogos colaborem e submetam artigos,
comentários e opiniões. O BSBL é mais do que a
seção “Tópicos Especiais”. Existe espaço para a
publicação de opiniões, comunicações, resumos
de eventos científicos, resenhas de livros, novos
métodos, dentre outros. Há muito que comentar e
discutir sobre Limnologia e temas afins, e todos
nós do corpo editorial gostaríamos de incentivar
o envio de contribuições para a divulgação de
idéias.
A próxima edição do BSBL será
comemorativa aos 25 anos da Sociedade
Brasileira de Limnologia, e o vice-presidente da
SBL, Prof. Antonio Camargo, vem gentilmente
me auxiliando em sua editoração. Vários
limnólogos já estão preparando artigos
relacionados à história da Limnologia no Brasil.
Estamos também fazendo um levantamento dos
congressos, diretorias da SBL e da Acta
Limnologica Brasiliensia, dentre outros assuntos.
Além disso, alguns limnólogos foram convidados
a escrever sobre os desafios e o papel da SBL nos
próximos anos. Gostaríamos de pedir a vocês que
escrevam dando sugestões de limnólogos
brasileiros e estrangeiros que tenham contribuído
para o avanço da Limnologia brasileira, bem como
enviem artigos, fotos e textos com estórias
interessantes para que esta edição comemorativa
enriqueça a história da Limnologia e da nossa
Sociedade.
Mensagem do Editor da Acta
Limnologica Brasiliensia
Alex Enrich-Prast
[email protected]
Universidade Federal do Rio de Janeiro
Raoul Henry
[email protected]
UNESP- Botucatu
Aos colegas, informamos que o apelo feito
no último boletim foi atendido. De fato, o número
de trabalhos submetido para a revista aumentou
substancialmente a partir de 01 de janeiro de
2007. Até a presente data (30 de março 2007),
20 (vinte) manuscritos foram enviados (média
acima de 6 por mês). O auxílio pleiteado junto
ao CNPq (Programa de Apoio às Publicações)
foi aprovado, mas somente receberemos
aproximadamente 1/3 (um terço) do montante
solicitado. Isto não permite ainda que
dispensemos a cobrança-auxílio feita aos autores
quando do envio da “prova”. Na realidade, o
valor da contribuição dos autores corresponde
ao pagamento das separatas. Nesta oportunidade,
gostaria de falar sobre o papel dos assessores. A
revisão e emissão de pareceres garantem à revista
um padrão mínimo de qualidade nas informações
científicas que são divulgadas pela revista. Este
é um trabalho gratuito desenvolvido por
pesquisadores veteranos e jovens com produção
já comprovada, selecionados pelo editor em
função da temática do trabalho apresentado.
Sempre é pedido que se faça a emissão do parecer
em um mês, para que se possa manter a
continuidade e a regularidade da revista. Muitos
atendem a essa solicitação; para alguns, é
necessário um lembrete (cobrança!), o que
implica num atraso na reformulação do artigo e,
por conseqüência, da revista. Uma minoria de
assessores não responde, nem devolve os
trabalhos, quando eventualmente, por sobrecarga
de atividades, doença, férias, viagens ou outro
motivo não pode atender a nossa solicitação. Para
todos os assessores, faço um apelo por uma
agilidade maior e, para a minoria mencionada
anteriormente peço, por favor, uma satisfação:
devolvam imediatamente os trabalhos quando
houver a impossibilidade do atendimento. Tenho
certeza que este apelo será entendido por todos.
http://www.sblimno.org.br
3
Métodos e Equipamentos
Determinação das Taxas Potenciais de
Nitrificação em Ambientes Aquáticos
O processo de nitrificação é realizado por
dois grupos de bactérias aeróbicas obrigatórias e
é constituído por duas reações principais: a
oxidação da amônia (NH4+) a nitrito (NO2-) e a
posterior oxidação deste a nitrato (NO3-). Este
processo conecta as fases reduzidas e oxidadas
do ciclo do nitrogênio e representa a principal
fonte autóctone de substrato para desnitrificação,
processo anaeróbico de redução do nitrato a
nitrogênio atmosférico (N2).
Existem várias metodologias para
obtenção das taxas de nitrificação. Isótopos
estáveis são utilizados para determinação das
taxas reais deste processo, mas esta metodologia
necessita da disponibilidade de um espectrômetro
de massa, um equipamento relativamente caro.
Os espectrômetros de massa quadrupolos, cujo
preço é mais reduzido, também têm sido
utilizados para trabalhos com isótopos estáveis.
Dependendo do objetivo da pesquisa, uma das
metodologias mais simples e bastante utilizada é
o acompanhamento das concentrações de nitrato
ao longo de um período de tempo. Em alguns
procedimentos a oxidação do nitrito a nitrato é
inibida por acetileno ou nitrapirina e a taxa é
obtida através do aumento das concentrações de
nitrito (Belser & M ays 1980. Appl. Environ.
Microb. 39: 505 a 510). No entanto, o uso de um
inibidor ao processo de oxidação do nitrito pode
alterar a atividade das bactérias oxidadoras de
amônia, dependendo das condições de incubação.
O valor potencial representa a taxa
máxima de atividade da comunidade presente,
que ocorre quando todo o substrato necessário é
fornecido em abundância, impedindo qualquer
tipo de limitação. Estas geralmente não são as
condições naturais e as taxas obtidas não
representam a atividade in situ no processo.
Entretanto, bactérias nitrificantes possuem taxas
de duplicação de no mínimo 8 horas e em
incubações curtas não há mudanças na densidade
bacteriana. Deste modo, as taxas mensuradas
representam a atividade potencial das bactérias
presentes no meio e muitas vezes são próximas
às taxas reais. Esta taxa potencial é aplicada
como um indicativo da densidade da comunidade
nitrificante, e pode ser utilizada para demonstrar
a importância ou a influência de determinado
parâmetro ou fator sobre o processo de
nitrificação (Belser 1979. Annu. Rev. Microbiol.
33: 309 a 333; Risgaard-Petersen et al. 2004.
Appl. Environ. Microb. 70: 5528 a 5537). Este
método pode ser aplicado à água, ao sedimento,
ao perifíton ou à rizosfera de macrófitas
aquáticas.
O objetivo deste artigo foi descrever o
procedimento para obtenção das taxas potenciais
de nitrificação no sedimento através da variação
das concentrações de nitrato após um período
curto de incubação. Esta metodologia foi
utilizada para avaliar a influência da densidade
de algas bentônicas sobre as taxas de nitrificação
no sedimento de algumas lagoas do Parque
Nacional da Restinga de Jurubatiba em Macaé
(estado do Rio de Janeiro).
O sedimento pode ser coletado com
testemunhos do tipo “core”, reservando-se o
primeiro centímetro, visto que é a faixa onde
ocorre a maior densidade de bactérias aeróbicas
e, conseqüentemente, onde as bactérias
nitrificantes estão mais ativas em virtude da
disponibilidade de oxigênio (Fig. 1). Sugere-se
que o primeiro centímetro de 5 cores sejam
incubados separadamente, como réplicas
verdadeiras. Deve ser destacado que o sedimento
geralmente é um compartimento extremamente
heterogêneo, e que o desvio padrão entre as
amostras é bastante elevado. Além disso,
ressalta-se a necessidade de coleta de água do
ambiente para a incubação.
Figura 1: Sedimento coletado em um testemunho. O primeiro
centímetro do perfil de sedimento aparece em destaque.
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
4
Em laboratório o primeiro centímetro de
cada amostra deve ser homogeneizado e uma
amostra de volume conhecido (aprox. 2-3ml) é
então transferida para um tubo de plástico (tipo
“falcon”) de 50ml. Neste tubo acrescenta-se de
20 a 30ml de água do próprio ambiente, a fim de
manter as mesmas condições abióticas como
salinidade e pH. Adiciona-se então 1ml de NH4Cl
(20mM) e 1ml de KH2PO4 (4mM) para que não
haja limitação por substrato. Os tubos devem ser
submetidos à incubação por 3 a 5 horas em mesa
agitadora a 100 rpm de maneira que a mistura
água-sedimento mantenha-se oxigenada (Fig. 2).
É importante que, para isso, o tubo não esteja
totalmente cheio permitindo que a rotação
provoque a oxigenação da mistura.
Figura 3: Exemplo de regressão linear das concentrações de nitrato
produzidas ao longo de 4,5h de incubação. O valor de a (35,5) foi
utilizado no calculo da taxa de nitrificação que no caso foi de 177 nmol
NO3- cm-3 h-1, que equivaleria a 65 nmol NO3- cm-2 h-1.
Figura 2: Disposição dos tubos de mistura na mesa agitadora. Várias
amostras podem ser incubadas simultaneamente.
Em intervalos que podem ser de 0,5, 1 ou
até de 2 horas, alíquotas de 6ml devem ser
retiradas e centrifugadas a 5000 rpm (ou mais)
para posterior análise das concentrações de
nitrato (nitrato + nitrito) do sobrenadante. Esta
sub-amostra deve ser rapidamente centrifugada,
evitando que o meio se torne anóxico permitindo
a atuação de bactérias desnitrificantes. No caso
de ambientes húmicos as amostras de NO3- não
devem ser acidificadas ou congeladas para evitar
a floculação das substâncias húmicas, o que pode
eventualmente interferir na determinação das
concentrações de nitrato da amostra e provocar a
subestimativa das taxas potenciais de nitrificação.
O cálculo da taxa potencial é realizado a
partir do ângulo de inclinação (α) da reta obtida
com a regressão linear das concentrações de
nitrato ao longo do tempo (Fig.3), e em função
dos volumes de água e sedimento, perifíton ou
rizosfera utilizados na incubação. A taxa é então
calculada a partir da seguinte fórmula:
A taxa potencial de nitrificação é obtida em
nmol NO3- cm-3 h-1, onde á é expresso em nmol
NO3- L-1 h-1, o volume de água em litros e o
volume de sedimento em cm3. Quando as taxas
forem obtidas em amostras d’água, estas serão
expressas em nmol NO3- L-1 h-1 e quando obtidas
em amostras de perifíton, o volume de sedimento
pode ser substituído pelos valores de material em
suspensão (mg L-1) ou de clorofila-a (µg L-1).
Através do volume de sedimento e da área
do core utilizado é possível converter os valores
e obter a taxa potencial de nitrificação por área
de sedimento. O volume total de sedimento
coletado corresponde ao conteúdo volumétrico
do primeiro centímetro do core de diâmetro
conhecido. Este volume corresponde à área do
cilindro de 1 cm de altura. A partir deste, calculase a área correspondente aos 2 ml de sedimento
utilizados na incubação para se obter o valor
potencial de nitrificação por área.
Ana Lúcia Santoro
[email protected]
Alex Enrich-Prast
[email protected]
Universidade Federal do Rio de Janeiro
http://www.sblimno.org.br
5
Hibridização in situ Fluorescente
(FISH) como Opção de Ferramenta em
Ecologia Microbiana Aquática
“- Como saber que estão lá, se não posso
enxergá-los?”
O conhecimento científico sempre esteve
ligado às descobertas de novas tecnologias e
metodologias. Ciência e tecnologia sempre
caminharam juntas e têm importância recíproca
uma no avanço da outra. Tratando-se de pesquisas
com microrganismos, isto se torna mais evidente.
A constatação da existência destes seres vivos
só se tornou inquestionável com o advento do
microscópio no século XVII. A partir de então, a
presença de bactérias vem sendo registrada nos
mais diversos ambientes, incluindo os aquáticos.
Conseqüentemente à constatação da presença de
microrganismos nos ambientes aquáticos, veio
sua quantificação, que foi intensificada a partir
da contagem direta de bactérias em filtros de
membrana (Hobbie et al.1977. Appl. Environ.
Microbiol. 33: 1225-1228).
Assim, o número e o metabolismo têm
revelado a importância dos microrganismos nos
ciclos biogeoquímicos e na cadeia alimentar
aquática. Durante a quantificação e as medidas
de produção e respiração microbiana, este vasto
mundo invisível é agrupado dentro de uma
“caixa” denominada MICRORGANISMOS ou
somente BACTÉRIAS. Nesta “caixa”, são
colocadas bactérias de diferentes formas e
tamanhos, que podem ou não pertencer ao mesmo
grupo taxonômico. Levando em consideração a
classificação taxonômica e filogenética,
constatamos que a diversidade de
microrganismos é grande também nos ambientes
aquáticos. Entretanto, nem todos os
microrganismos têm a mesma participação na
dinâmica destes sistemas, nem todos são
abundantes e nem estão distribuídos
uniformemente nos mais diversos ambientes
aquáticos (Kirchman 2002. FEMS Microbiol.
Ecol. 39: 91-100). O que nos leva a seguinte
questão: “Como saber quem está lá?”. São várias
as metodologias utilizadas para responder esta
pergunta. Nos últimos 20 anos, técnicas e
métodos de biologia molecular vêm sendo
empregados como ferramentas no nível
filogenético.
Hibridização in situ fluorescente (FISH)
como uma opção.
O FISH, abreviatura do inglês
“Fluorescence In Situ Hybridization”, é uma
destas ferramentas da biologia molecular e vem
sendo empregada em análise da comunidade
microbiana aquática há quase 20 anos
(Giovannoni et al. 1988. J. Bacteriol. 170: 720726). É basicamente uma técnica que utiliza
marcadores fluorescentes e que permite a
identificação filogenética de bactérias de
assembléias misturadas sem a necessidade de
cultivo prévio.
Os marcadores fluorescentes podem ser
delineados para serem altamente específicos e
reconhecerem apenas um gene ou grupos de
espécies. Podem ser construídos a partir de
oligonucleotídeos de RNAr (Amann et al. 1990.
J. Bacteriol. 172: 726-770), de polinucleotídeos
de DNA (DeLong et al. 1999. Appl. Environ.
Microbiol. 65: 4973-5563), de RNAtm de
plasmídeos ou de RNAm (Zwirglmaier 2005.
FEMS Microbiol. Lett. 246: 151-158). Os
primeiros e mais utilizados são os marcadores
construídos a partir de oligonucleotídeos de
RNAr pela propriedade conservativa deste RNA
e por estar presente em todos os organismos. Os
marcadores podem também ser construídos com
diferentes fluorocromos, sendo o Cy3
(carbocianina) o mais utilizado.
Ao longo dos anos, esta técnica vem
sendo aprimorada, inclusive sendo combinada
com outras técnicas. Estas modificações têm
permitido uma melhor visualização dos
resultados e também uma avaliação da atividade
das células identificadas, ligando estrutura e
funcionalidade da comunidade microbiana.
FISH e algumas combinações
1) CARD-FISH (do inglês: “catalysed
reporter deposition”) ou TSA-FISH
(Schonhuber et al. 1997. Appl. Environ.
Microbiol. 63: 3268-3273): é a técnica
que utiliza a deposição de catalizador nos
sítios de hibridização com deposição de
tiramida (TSA do inglês: “tyramid signal
amplification”).
Uma
melhor
visualização das células hibridizadas é
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
6
2)
3)
4)
5)
conseguida mesmo em condições de
baixa atividade celular.
RING-FISH (Zwirglmaier et al. 2004.
Mol. Microbiol. 51: 89-96): técnica que
permite a detecção in situ de genes
individuais ou fragmentos de genes em
plasmídeos e DNA cromossômico em
uma única célula. Nesta metodologia,
utiliza-se uma grande concentração de
marcadores polinucleotídeos. Uma
estrutura secundária é formada, o que
facilita a sintetização de marcadores com
seqüências menores e, portanto, mais
específicos. Um “anel” (em inglês, “ring”
que também é a abreviatura de
“recognition of individual genes”) é
formado pelo sinal fluorescente entorno
da célula marcada.
RAINBOW-FISH (Sunamura &
Maruyama 2005. FEMS Microbiol. Ecol.
55: 159-166): metodologia desenvolvida
para visualização de grupos bacterianos
corados com fluorocromos diferentes em
uma mesma amostra. Após captura e
tratamento de imagens, as bactérias
marcadas resultam em sete cores, como
um arco-íris. A imagem formada se dá
através da coloração, saturação da cor e
intensidade de cada fluorocromo.
FISH-DC (Maruyama & Sunamura 2000.
Appl. Environ. Microbiol. 66: 22112215): técnica baseada na contagem
direta das células hibridizadas
simultaneamente com diferentes
marcadores ligados a diferentes
fluorocromos.
STAR-FISH, FISH-MAR e MICROFISH (respectivamente, Lee et al. 1999.
Appl. Environ. Microbiol. 65: 1289-1297;
Ouverney & Fuhrman 1999. Appl.
Environ. Microbiol. 65: 1746-1752;
Cottrell & Kirchman 2000. Appl. Environ.
Microbiol. 66: 5116-5122): são diferentes
nomes para as técnicas que combinam o
FISH com a microautoradiografia. Estas
técnicas permitem identificar qual célula
está ativa, ou melhor, qual está
absorvendo compostos marcados
radioativamente. Essa combinação das
técnicas permite ainda saber a qual grupo
bacteriano estas células ativas pertencem.
Assim, além da análise da estrutura,
temos uma análise funcional da
comunidade microbiana presente.
6) DVC-FISH (Nishimura et al. 1993. J.
Oceanogr. 49: 51-56): a técnica baseiase na incubação das amostras em
antibióticos e nutrientes. Os antibióticos
utilizados agem na inibição da síntese de
DNA. Assim, evitam a divisão celular,
permitindo apenas o crescimento em
tamanho das células bacterianas. Dessa
forma, a quantidade de RNAr intracelular
aumenta nas amostras, melhorando a
eficiência de detecção das bactérias alvos
pelos marcadores específicos. Uma
limitação desta técnica é a possibilidade
de resistência bacteriana aos antibióticos
utilizados.
Fundamentos Metodológicos – “Como fazer o
“FISH”?
Os protocolos de preparação de FISH
estão constantemente sendo adaptados às rotinas
dos laboratórios, proporcionando diferentes e
importantes avanços. Assim como têm
contribuindo para aumentar de forma exponencial
a disponibilidade comercial de marcadores e de
softwares para a construção dos mesmos.
Os principais itens que devem ser
considerados durante a aplicação da técnica de
FISH são: o tipo fixador, o tamanho e seqüência
do marcador, a composição do tampão de
hibridização e da solução de lavagem, a
temperatura de hibridização e o método de
contagem (Bouvier & del Giorgio 2003. FEMS
Microbiol. Ecol. 44: 3-15).
A especificidade de um marcador para se
ligar a um sítio alvo depende principalmente das
condições de fixação da amostra, hibridização e
lavagem. O fixador está diretamente relacionado
a permeabilização da célula para a entrada do
marcador. O tempo de fixação (1-24 horas) é
variável de acordo com o tipo de fixador usado e
a origem da amostra a ser analisada. Este tempo
é menor para amostras com bactérias ativas.
Depois de fixadas, as amostras podem ser
estocadas por vários meses até o processo de
hibridização.
Durante o processo de hibridização
propriamente dito, marcadores são adicionados
http://www.sblimno.org.br
7
a tampões em concentrações de saturação de
formamida para maximizar a ligação à unidade
alvo. Diferentes concentrações de saturação são
utilizadas para cada marcador. Na hibridização,
as amostras são incubadas em elevadas
temperaturas em frascos fechados com vapor de
tampão de hibridização para evitar alteração na
concentração devido à evaporação. A etapa de
lavagem, também em altas temperaturas, serve
principalmente para retirar o excesso de
moléculas dos marcadores, evitando ligações não
específicas. A concentração de cloreto de sódio
(NaCl) na solução de lavagem varia com o tipo
de marcador.
A visualização dos resultados pode ser
realizada em microscopia de epifluorescência, em
microscopia de varredura ou, ainda, em citometria
de fluxo. Na Fig. 1, podem-se observar bactérias
coradas com DAPI (à esquerda) e bactérias
coradas com um marcador específico (à direita)
em um mesmo campo observado em microscopia
de epifluorescência.
– Marine Microbiology. Academic Press, 207226).
Na revisão de Bouvier & del Giorgio
(2003. op. cit.), a variação de EUB338, o
marcador mais comum para identificar o grupo
das Eubactérias, foi de 1 a 100% em diferentes
trabalhos, com média de 56%. Estes autores
acreditam que existe um componente
metodológico nesta variação, mas também que
existe um componente ambiental forte. O tipo de
ecossistema e o grupo filogenético dominante
influenciam a proporção das eubactérias.
Mesmo assim, melhores instrumentos e
facilidades na aquisição de marcadores tem
tornado o FISH um método atrativo para
ecologistas microbianos, muitas vezes avessos ou
resistentes à biologia molecular. Porém, o mais
desafiante no uso de FISH em amostras
ambientais não está na técnica de corar em si,
mas na construção e aplicação de novos
marcadores dentro de um contexto particular da
comunidade microbiana não conhecida.
Lembrando sempre que, antes da técnica vem a
pergunta a ser respondida. Da mesma forma que
a escolha do nível filogenético apropriado para
cada marcador depende da questão específica de
cada estudo.
Algumas aplicações e contribuições da FISH
Fig. 1. Bacterioplâncton corado com DAPI (à esquerda) e corados com
probes específicos (à direita). Neste caso, o probe utilizado foi CF319a
*
para o grupo Cytophaga-Flavobacter.
Como qualquer técnica, limitações
também existem nos protocolos de FISH. O maior
problema seria a baixa intensidade de sinais. Isto
pode ser atribuído à permeabilidade insuficiente
das células aos marcadores usados (Zwirglmaier
2005. op cit.). As primeiras pesquisas utilizando,
com sucesso, esta metodologia para amostras
ambientais foram em ambientes altamente
eutróficos, onde bactérias crescem rapidamente
e melhores sinais de hibridização podem ser
observados. Como grande parte das bactérias de
ambientes aquáticos são pequenas, com
crescimento lento, ou ainda, inativas, a
intensidade de sinais de células do
bacterioplâncton hibridizadas pode se encontrar
abaixo do limite de detecção ou se perder nos
altos níveis de fluorescência (Pernthaler et al.
2001. Em Paul (eds). Methodos in Microbiology
*
*
*
As informações geradas pelos estudos de
ecologia microbiana, utilizando a técnica
de FISH sob as mais variadas condições
ambientais, têm papel fundamental na
identificação da composição da
comunidade microbiana em diferentes
níveis filogenéticos.
Flutuações espaciais e temporais na
composição
das
comunidades
microbianas têm sido observadas através
da técnica de FISH, mesmo quando a
densidade bacteriana total não é alterada.
O FISH também tem sido utilizado em
estudos que comparam a composição
microbiana entre ambientes.
Resultados obtidos com FISH confirmam
que diferentes grupos de bactérias podem
absorver diferentes quantidades e
qualidade de matérias orgânica e
inorgânica com diferentes taxas de
absorção e, ainda, que pode haver
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
8
*
*
predação
seletiva
entre
os
microrganismos.
O FISH, com o avanço de metodologias
diferenciadas, começa a ser utilizado
também para identificação de infecções
virais, ou de organismos eucariontes
aquáticos.
Indicadores microbiológicos de
qualidade ambiental mais confiável e de
fácil determinação têm sido identificados
utilizando marcadores específicos para
FISH.
-”E o futuro?”
A diversidade de microrganismos está
diretamente relacionada à diversidade funcional
dos microrganismos derivada da complexidade
de rotas de matéria e energia e, das cadeias
tróficas dos ecossistemas aquáticos. Nós estamos
só começando a examinar a ligação entre a
função e a estrutura da comunidade microbiana.
Portanto, avaliar esta diversidade, independente
da metodologia utilizada, torna-se essencial para
um melhor conhecimento do sistema. Passamos
a perguntar: “-Quem está lá e fazendo o quê?”.
Ainda assim, FISH e suas combinações e
adaptações continuarão a ser uma opção de
ferramenta.
ainda por muito tempo. Assim, desde a década
passada, quando se verificou que a grande maioria
dos microrganismos não estava representada nos
experimentos de isolamento, a busca por
ferramentas para estudar essa parcela das
comunidades microbianas se tornou prioridade
para grande parte dos microbiologistas
ambientais. Deste modo, técnicas como DGGE,
TGGE, FISH, T-RFLP, RISA, ARISA e
seqüenciamento de bibliotecas de clone vêm
sendo empregadas para avaliar a diversidade
microbiana nos mais diversos ambientes. Dessas
técnicas, iremos nesse manuscrito detalhar o
seqüenciamento de bibliotecas de clones, dando
ênfase na montagem de uma biblioteca e
seqüenciamento dos clones e ao fim exemplificar
algumas metodologias de análise dos dados
obtidos. Observe esquema que demonstrando a
ordenação metodológica envolvendo a técnica de
seqüenciamento na figura 1.
Alessandro Del´Duca
[email protected]
Dionéia César
[email protected]
Universidade Federal de Juiz de Fora
Métodos Moleculares para Análise de
Comunidades Microbianas em
Ambientes
Aquáticos:
IV.
Seqüenciamento.
Hoje, sabemos que cerca de 99% dos
microrganismos presentes em qualquer ambiente
não são cultiváveis pelas metodologias
convencionais e, como as novas metodologias
de cultivo estão ainda em sua infância, esse
número deve persistir nesta ordem de grandeza
Fig. 1: Ordenação metodológica discutidas envolvendo a técnica de
seqüenciamento.
A primeira etapa de todos os trabalhos
envolvendo ecologia molecular é a obtenção dos
ácidos nucléicos. Podendo ser tanto o DNA
quanto o RNA. A qualidade desse material é
crucial na qualidade do resultado final das
análises, pois inicialmente, um DNA de má
qualidade poderá conter substancias inibitórias
das enzimas utilizadas nas técnicas a serem usadas
em seguida.
Em seguida, existem duas possibilidades
de estratégia de clonagem, uma clonagem guiada
por seqüência, onde a biblioteca gerada conterá
somente incertos de um único gene (amplificado
por PCR). Esse tipo de estratégia é muito utilizada
http://www.sblimno.org.br
9
para descrever-se a diversidade genética de uma
dada comunidade ou comparando comunidades
microbianas. Utilizando-se da PCR pode-se gerar
bibliotecas de uma ampla gama de genes, entre eles
pode-se destacar o rrs que codifica para o rRNA
16S, esse é o gene utilizado na grande maioria dos
trabalhos que se utilizam desta metodologia.
Podemos citar também diversos genes funcionais
ligados a ciclos biogeoquímicos (amoA, nifH,
pamoA, etc.) degradação de xenobiontes (catA,
tmoA, etc.).
A segunda envolve a clonagem aleatória de
fragmentos de DNA de origem ambiental. No
entanto, como a diversidade bacteriana é muito
grande e pouco se conhece ainda sobre o
metabolismo da parcela não cultivada desses
trabalhos envolvem mais trabalho e também maior
custo. Assim, nesse manuscrito só será descrito
trabalhos envolvendo clonagem e sequenciamento
de produtos de PCR.
Com o amplicon obtido por PCR, a etapa
subseqüente é a sua purificação. Isso é necessário
devido a presença de restos de primers na reação.
Como os primers são fragmentos de DNA de
tamanho menor que o amplicon da reação, esses
tem maior facilidade de se ligar ao plasmídeo assim,
ao se realizar a clonagem, esses clones formados
por esses insertos seriam falsos positivos. Nessa
etapa é indicada a utilização de kits comerciais, pois
seu maior rendimento e rapidez facilitam as etapas
subseqüentes. Muitos pesquisadores não realizam
essa etapa e acabam obtendo poucos falso positivos
ao não realizarem essa etapa.
Como muitos kits de clonagem de produtos
de PCR utilizam a característica da Taq polimerase
de acrescentar uma adenina ao terminal 3' ao
amplicon ao fim da amplificação além de utilizar
polimerases que não apresentam essa função (ex.:
Pfu polimerase), é importante realizar uma
adenilação desse terminal. Essa reação consiste em
acrescentar ATP na presença de polimerase com
função de adenilação do 3' dos fragmentos (ex.: Taq
polimerase) a um certo volume de amplicon.
Com os fragmentos devidamente
purificados, é possível prosseguir com a ligação
deste ao plasmídeo com uso de kits comerciais.
Nessa etapa deve-se prestar atenção a concentração
de incerto com relação à concentração de plasmídeo.
Os kits comerciais sempre indicam a concentração
ideal de incerto e a margem na qual se obterá uma
boa quantidade de clones. Ao se montar uma
biblioteca de clones de um determinado gene,
deve-se evitar que a ligação seja um dos fatores
a inserir uma tendência a sua analise, assim é
preferível sempre trabalhar na margem superior
da faixa desta relação de modo a se obter uma
quantidade de clones superior à que será
amostrada. Nas indicações de cada Kit devese observar detalhes sobre a ligação, como
tempo e temperatura e sempre procurar realizar
a ligação de modo a se maximizar a quantidade
de clones obtidos no final.
A etapa a seguir, a clonagem, pode ser
realizada, principalmente, de duas maneiras:
eletroporação e “heat-shock”. O primeiro
utiliza um choque elétrico para abrir poros na
membrana da bactéria por onde o DNA irá
entrar. O segundo, baseia-se no uso de
incubação das células a frio na presença de
cátions divalentes (normalmente cálcio) que
iriam formar poros na membrana por onde
entrará o DNA através de choque térmico
(42°C por 30-60s). A escolha do método vai
depender principalmente da infra-estrutura e
histórico do laboratório. Infelizmente, no Brasil
é muito difícil comprar células competentes de
boa qualidade, principalmente devido ao
transporte ineficiente do material. Desta
maneira, é normalmente necessário realizar a
preparação dessas no laboratório o que requer
pelo menos um dia de trabalho longo e
enfadonho. Essa é a etapa mais complicada e
é indicado que seja realizada com supervisão
de pessoas com experiência na técnica, pois
envolve a organização dos materiais que
otimizem o trabalho e aumentem o rendimento
da reação e da clonagem. Assim como a
preparação de células competentes, durante a
clonagem é importante que se tenha ajuda de
alguém com bastante experiência com o
procedimento. Dessa maneira pode-se
assegurar que todos os detalhes estão sendo
seguidos para aumentar a eficiência da análise.
Posteriormente, o vetor contendo o
gene de interesse pode ser inserido na E. coli
selecionada de acordo com o tipo de
transformação que será realizada (algumas
cepas são mais apropriadas para um tipo de
transformação que outras). As células
transformadas são então inoculadas em meio
sólido, contendo antibiótico, esse é
acrescentado para seleção das células contendo
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
10
o plasmídeo desejado, pois esse contém gene de
resistência ao antibiótico em questão. Isso, porém,
não garante que todas as células conterão o
inserto, assim um segundo marcador existente nos
plasmídeos é examinado, o operon para
catabolismo da galactose. Quando algo é inserido
no plasmídeo esse gene não pode ser expresso
devido ao inserto entrar no meio do gene, sendo
assim incapaz de expressar essa característica.
Para isso se acrescenta ao meio de cultura X-gal,
que é uma molécula de galactose contendo um
radical que confere a colônia capaz de utilizar
galactose uma coloração azul. Assim, apenas as
colônias brancas (cor normal das colonias de E.
coli) contem algo inserido no plasmídeo delas.
Outro detalhe é o número de clones que
seriam necessários para representar um ambiente,
vários autores já calcularam esse número e
obviamente esse valor varia dependendo do
ambiente. Porém, ao se analisar a literatura, podese observar que a maioria dos trabalhos
publicados possuem um número de clones que
varia de 96 a 300 por amostra. Embora esse valor
seja extremamente baixo se comparado com o
número de clones teórico que se consideraria ideal
para uma amostra ambiental (alguns autores
indicam até um milhão de clones para amostras
de solo, que apresentam cerca de 10.000 a 100.000
espécies bacterianas em um grama), já se obtém
informações o suficiente para se comparar
diferentes bibliotecas utilizando-se os métodos
que serão descritos abaixo.
Um jeito prático e rápido de se manipular
os clones é a extração do DNA direto da colônia.
Como as E. coli são bactérias Gram negativas que
possuem uma parede celular bastante frágil é
bastante fácil lisar as células apenas com uma
simples fervura, assim pode-se pegar as colônias
diretamente da placa e extrair seu DNA após
suspende-las em água Milli-Q ou um tampão
contendo EDTA. E utilizar esse material para a
amplificação por PCR, esse material será em
seguida usado para seqüenciamento.
O seqüenciamento, normalmente, é
realizado pelo método de Sanger, método que se
baseia no fato de a polimerase duplicar o DNA
adicionando uma nova base a fita de DNA que
está sendo alongada pelo oxigênio ligado ao
carbono 3' do ultimo nucleotídeo desta. Quando
se coloca um nucleotídeo que não apresenta o
oxigênio no carbono (dideoxinucleotídeo) a
replicação do DNA cessa após sua adição desse.
Os seqüenciadores atuais usam esses
dideoxinucleotídeos ligados a moléculas de
fluorescentes (uma cor pra cada um dos 4
nucleotídeos diferentes) para identificar qual a
molécula que está naquela posição. Assim, ao
se correr as amostras em eletroforese (gel de
acrilamida ou capilar) pode-se separar as
moléculas por seu tamanho e identificar o
nucleotídeo utilizando-se laser e sensores para
captar a fluorescência das moléculas. Assim,
somando-se o tamanho das moléculas e o
nucleotídeo terminal, pode-se saber a seqüência
da molécula em questão. Existem outros métodos
de seqüenciamento menos populares. Um,
porém, tem se mostrado bastante promissor e foi
inclusive empregado no seqüenciamento do
genoma do Neanderthal, o Pyrosequencing.
Nesse
método
uma
reação
de
quimioluminescência enzimática ocorre a cada
vez que um nucleotídeo é acrescentado a uma
fita, e como cada um dos nucleotídeos é
disponibilizado para a polimerase em momento
diferente é possível saber qual está sendo
acrescentado a cada fita a cada momento.
Embora este método gere seqüências curtas, se
comparado com o método de Sanger, pela sua
velocidade e capacidade de seqüenciar um
número muitas vezes maior de amostras, ele é
capaz de seqüenciar o genoma completo de uma
bactéria com apenas uma máquina em menos de
uma semana.
A parte mais importante, e com certeza
onde está a ciência de tudo, é a análise dessa
grande quantidade de dados. Inicialmente, vamos
imaginar que o objetivo do trabalho é apenas
identificar os componentes de uma determinada
comunidade. Para isso o ideal seria construir uma
biblioteca de seqüências do gene para o rRNA
16S. A ferramenta mais simples e completa para
se analisar esses dados é o RDPII (ribossomal
database project; http://rdp.cme.msu.edu/).
Nesse site se encontram ferramentas diversas que
permitem a análise em varias etapas, desde a
qualidade da seqüência obtida até classificação
e montagem de árvores filogenéticas. Porém,
esse site é um tanto incompleto, por sofrer uma
curação cuidadosa dos dados lá disponíveis, e
portanto ainda não possui em seus bancos alguns
grupos de bactérias. Desta maneira é importante
também checar a seqüência no bancos de dados
http://www.sblimno.org.br
11
não específicos como o Genebank do NCBI
(www.ncbi.nlm.nih.org), que embora não curada
apresenta um banco de dados mais completo.
Caso o objetivo do trabalho seja a
comparação das comunidades de dois ou mais
ambientes, estão disponíveis algumas ferramentas
estatísticas específicas para realizar tais
comparações. A primeira e mais completa é o
software DOTUR (Distance based OTU and
richness determination; http://rdp.cme.msu.edu/
). Esse software é bastante útil para, como o nome
já diz determinar quais sequências são a mesma
OTU (operational taxonomic unit). A riqueza de
espécies baseado na determinação das OTUs e
construção de curvas de rarefação, são usadas para
comparar as comunidades. Essa comparação se
baseia no número de seqüências necessárias na
nossa amostragem para se ter uma boa cobertura
da comunidade. Porém, duas comunidade
diferentes, mas apresentando diversidade similar
vão apresentar um comportamento semelhante em
suas curvas de rarefação, sendo assim, necessário
uma outra ferramenta para avaliar se duas
comunidades são iguais ou diferentes.
Uma
ferramenta
desenvolvida,
especificamente, para comparação de duas ou
mais comunidades é o Libshuff (http://
whitman.myweb.uga.edu/libshuff.html). Essa
analisa o quanto uma determinada amostra
representa a outra, ou seja, quanto mais similares
forem duas comunidades melhor elas se
representaram. Deste modo, a comparação de duas
comunidades é feita com base não na diversidade
nela contida, mas sim no conteúdo desta.
Com esses dados na mão, além dos demais
coletados em campo, o pesquisador pode então
explicar os eventos observados tanto na
comunidade como no ambiente. Infelizmente,
ainda é muito difícil analisar por seqüenciamento
um número grande de amostras, devido ao custo.
No entanto, a cada dia que passa o custo de
seqüenciamento é menor, e, um maior número de
trabalhos contendo um número maior de amostras
seqüenciadas e mais clones por amostra é
encontrado na literatura. Por esse motivo, a análise
de bibliotecas de clone de amostras ambientais é
hoje uma metodologia padrão nos estudos de
microbiologia ambiental.
Rodrigo Gôvea Taketani
[email protected]
Natália Oliveira Franco
[email protected]
Alexandre Soares Rosado
[email protected]
Universidade Federal do Rio de Janeiro
Tópicos Especiais
Ecologia Microbiana
Macroecologia de Microorganismos:
Uma Perspectiva Ecológica à
Diversidade Microbiana
A extraordinária diversidade de espécies
é considerada por muitos cientistas como uma
das mais intrigantes características da vida na
Terra, e um dos principais objetivos da ecologia
de comunidades é encontrar explicações para os
padrões de abundância e riqueza destas espécies
observadas no espaço e no tempo. A distribuição
da abundância de indivíduos entre diferentes
espécies, a relação entre a distribuição geográfica
e a abundância, a variação na diversidade entre
diferentes localidades e o aumento da
diversidade com o aumento da área, em
particular, são temas amplamente abordados em
estudos ecológicos. Eles formam parte do campo
da Macroecologia (Brown 1995. Macroecology,
The University of Chicago Press, Chicago, 269p.
) que se volta para a descrição e interpretação de
padrões ecológicos mais amplos. Estes padrões
fornecem importantes pistas sobre os
mecanismos que influenciam a estrutura das
comunidades e são fundamentais para
estabelecer prioridades em conservação.
Relações espécies-área, distribuições de
abundância relativas, padrões espaciais de
ocupação foram extensivamente discutidos
teoricamente e verificados em campo, porém a
maioria do conhecimento gerado deriva de
estudos com plantas e animais. Poucos são os
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
12
estudos que forneçam este tipo de informação a
respeito de comunidades microbianas, embora
microorganismos detenham talvez a maior parcela
da biodiversidade de todo planeta e exerçam um
papel decisivo nos ciclos biogeoquímicos e no
funcionamento de ecossistemas.
É possível que a falta de perspectiva
ecológica da diversidade bacteriana tenha origem
nas restrições técnicas da microbiologia. Quantificar
a diversidade bacteriana representa um grande
desafio. A maioria dos microorganismos
procariontes e eucariontes não podem ser
identificados morfologicamente e o uso de métodos
de cultura revela apenas uma fração da diversidade
real. Recentemente, com o avanço de técnicas
moleculares na identificação dos microorganismos,
alguns pesquisadores iniciaram estudos mais
precisos sobre a abundância e a diversidade
bacteriana (Head et al. 1998. Microbial Ecology
35:1-21). Por exemplo, a partir de técnicas
moleculares, Lambais et al. (2006. Science 312)
quantificaram a riqueza microbiana associada às
folhas de árvores tropicais (filosfera) e, através de
extrapolações estes alcançaram o número de 2 a 13
milhões de novas espécies de bactérias.
Uma das questões mais importantes em
estudos biogeográficos tradicionais é determinar a
importância relativa dos fatores ambientais locais
e dos eventos históricos regionais nos padrões de
distribuição das espécies. Se considerarmos a
distinção entre províncias e habitats, quatro
diferentes hipóteses podem ser traçadas a respeito
da diversidade microbiana (Fig. 1). No entanto,
historicamente foi estabelecido que devido seu
pequeno tamanho e grande abundância
populacional, microorganismos apresentariam altas
taxas de dispersão, o que implicaria na anulação de
eventos evolutivos e ecológicos do passado.
Portanto a biogeografia microbiana seria
influenciada apenas pela variação das características
ambientais contemporâneas, historicamente
conhecida como a hipótese de Baas-Becking “todos
estão em todos os lugares – o ambiente seleciona”.
No entanto, assim como plantas e animais,
estudos recentes mostram que a diversidade
microbiana pode ser influenciada tanto por
processos históricos quanto por fatores ambientais.
Alguns estudos realizados tanto em ambientes
aquáticos quanto terrestres encontraram correlações
entre a composição da comunidade e características
ambientais, como concentração de matéria orgânica,
salinidade e profundidade. Estes estudos
evidenciam que a heterogeneidade espacial
pode ser determinante para os padrões
espaciais da diversidade de microorganismos,
embora pouco se saiba sobre o efeito destas
variáveis no número de taxa da comunidade.
Em contrapartida, o isolamento geográfico
tanto em escala global (entre continentes)
quanto local (entre habitats) foi responsável por
influenciar padrões biogeográficos de
comunidades microbianas que habitam fontes
termais, não apresentando qualquer correlação
com variáveis ambientais, evidenciando desta
forma certo provincianismo. Apesar da maioria
dos estudos se focarem em avaliar estes
mecanismos isoladamente, um crescente
número de trabalhos que avaliam ambos os
processos apontam que tanto a heterogeneidade
ambiental quanto fatores históricos podem
atuar simultaneamente na estruturação de
comunidades microbianas numa escala local.
No entanto, a despeito do aparente avanço na
identificação dos mecanismos que atuam na
estruturação destas comunidades, pouca
atenção foi dada ao papel das interações entre
as espécies neste contexto. Talvez isto se deva
a um legado histórico da ecologia geral, apenas
Fig. 1: Quatro hipóteses sobre a contribuição dos fatores históricos
e ambientais na biogeografia microbiana. Características ambientais
e históricas influenciariam a diversidade microbiana se a similaridade
biótica for correlacionada com a similaridade ambiental ou com a
distância geográfica respectivamente. A palavra província refere-se
a uma região em que a diversidade resulta de conexões passadas e
isolamento com outras regiões. Já a palavra habitat refere-se ao
conjunto de variáveis bióticas e abióticas locais que influenciam a
composição microbiana. (adaptado de Martiny et al. 2006. Nat.
Microbial Rev. 4: 107-112).
http://www.sblimno.org.br
13
em meados da década de 60, após o
desenvolvimento da teoria clássica de
competição, é que as interações entre as espécies
passaram a ser consideradas como um importante
fator estruturador das comunidades. Por
apresentarem curtos tempos de geração e
facilidades metodológicas quanto sua
manipulação, microorganismos podem se tornar
valiosas ferramentas na avaliação do papel das
interações bióticas na comunidade e assim
contribuir de forma decisiva para consolidar, ou
não, a importância das interações na teoria
ecológica.
A relação espécies-área (relação entre a
riqueza de espécies e a área amostrada –REA),
um dos padrões mais estudado em ecologia, nos
fornece evidencias adicionais da biogeografia de
microorganismos. Embora nenhuma relação
espécies-área possa ser generalizada para todos
os habitats, grupos taxonômicos ou escalas
espaciais, uma função potencial (S=cA z) é
freqüentemente empregada. Valores empíricos de
“z” (taxa do aumento do número de espécies com
o aumento da área) para plantas e animais variam
em torno de 0,10 a 0,35 considerando habitats
contínuos e ilhas. Já os valores de “z” observados
para bactérias e outros microorganismos
geralmente encontram-se abaixo de 0,10, embora
valores mais altos (0,26) foram observados em
estudos realizados em corpos aquáticos
descontínuos (Bell et al. 2005. Science 308).
Algumas razões foram apontadas para explicar
os baixos valores de “z” na relação entre a
diversidade microbiana e a área quando
comparados com os obtidos para plantas e
animais. A maioria dos trabalhos com
microorganismos que investigaram a REA a fez
em habitats contínuos com baixa heterogeneidade
onde a colonização de áreas adjacentes
rapidamente homogeneíza a comunidade,
conseqüentemente, diminuindo o valor de “z”.
Outro fator importante é a resolução taxonômica
empregada em trabalhos com microorganismos.
Valores de “z”, em geral, aumentam com o
aumento da resolução taxonômica. A definição
operacional de unidade taxonômica mais
comumente utilizada para bactérias assume 70%
de homologia do genoma. Se o mesmo critério
fosse aplicado para macroorganismos, lêmures,
chimpanzés, gorilas e seres humanos seriam
considerados uma única espécie. Apesar das
semelhanças qualitativas (relação positiva) entre
a REA para micro e macroorganismos, as
diferenças quantitativas observadas ainda não
possuem uma explicação definitiva, o que
encoraja um maior número de estudos que visem
desvendar que características e processos
contribuem para estas diferenças.
O esforço de se compreender a
diversidade microbiana não se limita em apenas
determinar padrões, mas descobrir os processos
que geram tais padrões. Isto implica basicamente
em entender como os processos de dispersão,
especiação e extinção se dão no nível microbiano.
Baseando-se em propriedades alométricas e
excetuando o processo de dispersão ativa,
acredita-se que dado seu tamanho de corpo,
microorganismos apresentem maiores taxas de
dispersão e densidade populacional, além de
apresentarem maior potencial para rápida
diversificação genética e baixas taxas de
extinção. Deste modo, alguns aspectos
biogeográficos podem ser comuns para todas as
formas de vida (plantas, animais e
microorganismos), mas existem determinados
aspectos que são únicos aos microorganismos e
portanto, os processos que influenciam sua
diversidade seriam fundamentalmente diferentes
daqueles observados para organismos de maior
tamanho.
Historicamente a ecologia microbiana
como disciplina desenvolveu-se separada da
ecologia geral, nascendo como uma subdisciplina da microbiologia ambiental e
apresentando suas próprias abordagens e
perspectivas. O resultado final desta separação
é a falta de bases sólidas teóricas a ecologia
microbiana e, por outro lado, a ausência dos
microorganismos na construção do conhecimento
ecológico. A perspectiva ecológica sobre a
diversidade bacteriana se faz crucial para a união
entre estas duas disciplinas. Embora os detalhes
individuais de cada organismo e de sistemas
ecológicos tenham sua relevância, ecólogos em
geral ganhariam mais ao tentar desvendar os
padrões e processos que regem a natureza
(Lawton 1999. Oikos 84: 177–192). Portanto,
Uma visão sintética da ecologia microbiana, de
animais e de plantas não é apenas possível, mas
fundamental para o aprimoramento e
desenvolvimento das bases teóricas do
conhecimento ecológico geral.
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
14
Vinicius Fortes Farjalla
[email protected]
Rafael Dettogni Guariento
[email protected]
Universidade Federal do Rio de Janeiro
Bactérias Magnetotáticas
Bactérias magnetotáticas ou magnéticas
são microrganismos que se orientam e navegam
propelidas pelos seus flagelos ao longo das linhas
de campos magnéticos devido às partículas
denominadas
magnetossomos.
Os
magnetossomos são considerados organelas
formadas por um cristal magnético envolvido por
uma membrana lipo-protéica. Cada cristal é
composto pelo óxido de ferro magnetita (Fe3O4)
ou pelo seu isomorfo magnético de enxofre, a
greigita (Fe3S4). No interior de cada célula, os
magnetossomos formam cadeias, de 10 a 30
partículas, que impõem à célula bacteriana um
momento de dipolo magnético capaz de orientála ao longo das linhas de um campo magnético,
tal como o campo geomagnético ou mesmo um
campo externo aplicado.
Os magnetossomos bacterianos são
caracterizados pela distribuição de tamanho
restrita à faixa dos 50 a 200nm, pela morfologia e
hábitos cristalinos únicos. Eles são espécieespecíficos, ou seja, cada espécie bacteriana
produz invariavelmente o mesmo tipo de
magnetossomo, embora haja exceções. As
bactérias magnetotáticas são capazes de sintetizar
cristais magnéticos de magnetita ou greigita, a
temperatura e pressão ambientes, por processos
desconhecidos, porém muitos menos drásticos do
que aqueles atualmente empregados na síntese
inorgânica de compostos magnéticos. Os
magnetossomos não são encontrados na natureza
oriundos de fontes não-biológicas, além de não
serem reproduzidos por processos inorgânicos de
síntese. A formação de cristais altamente
organizados, com características magnéticas e
cristalográficas altamente definidas, é um
exemplo marcante de um processo de
mineralização biologicamente controlado, que
tem estimulado pesquisas em diversas áreas
desde a sua descoberta. Acredita-se que a
membrana biológica que circunda cada
magnetossomo seja a principal estrutura
envolvida na sua formação e que várias proteínas
presentes na membrana desempenhem papel
fundamental na síntese e na disposição dos
magnetossomos na célula (Bazylinski & Frankel,
2004. Nat. Rev. Microbiol. 2: 217-230).
Os mecanismos de formação dos
magnetossomos podem ser relevantes para a
síntese de biomateriais avançados, com
propriedades definidas. As principais
características que tornam os magnetossomos
atraentes do ponto de vista biotecnológico são:
propriedades magnéticas e cristalográficas
perfeitas; presença de uma membrana
envolvendo cada cristal, o que permite o
acoplamento químico de substâncias de
interesse; alta pureza química; distribuição de
tamanhos estreita e controle biológico de
formação. Aplicações biotecnológicas e
comerciais de magnetossomos já foram
sugeridas, incluindo a manufatura de meios
magnéticos para a indústria, separação celular e
aplicações médicas como agente de contraste em
ressonância magnética (Schüler & Frankel, 1999.
Appl. Microbiol. and Biotech. 52: 464-473).
Portanto, o entendimento da estrutura,
composição e mecanismo de formação dos
magnetossomos é de interesse crucial para o
desenvolvimento de futuras aplicações
tecnológicas.
A formação dos magnetossomos
bacterianos é um bom modelo para o estudo de
biomineralização de materiais magnéticos e
biomagnetismo em outros organismos, uma vez
que cristais de magnetita semelhantes aos
encontrados nos magnetossomos foram
encontrados em uma grande variedade de
organismos superiores, incluindo seres humanos
(Kirschvink et al. 1992. Proc. Nat. Acad. Sci.
USA 89: 7683-7687). Recentemente, partículas
de magnetita ultra-finas de um meteoro de
origem marciana, que lembram os cristais dos
magnetossomos em bactérias recentes, foram
citados como evidência para que a vida tenha
tido uma origem extraterrestre, em Marte
(Thomas-Keprta et al., 2000. Geochim. et
Cosmochim. Acta 64: 4049-4081).
http://www.sblimno.org.br
15
Nos ambientes aquáticos, acredita-se que
as cadeias de magnetossomos são usadas para
navegação na coluna d’água ou entre os grãos do
sedimento, utilizando o fraco campo magnético
da Terra. No hemisfério Norte, o campo magnético
aponta para baixo (Fig. 1). Quando as bactérias
obtêm a informação de que estão em um ambiente
oxidante elas nadam para baixo, no mesmo sentido
Fig. 1: Presume-se que o fenômeno da magnetotaxia seja
fundamentalmente igual nos dois hemisférios: a bactéria utiliza o campo
geomagnético (B geo) como um eixo para encontrar eficientemente
concentrações ideais de compostos redutores e oxidantes, geralmente na
Zona de Transição Óxica-Anóxica (ZTOA) de ambientes estratificados.
A orientação magnética dos magnetossomos no interior de cada bactéria
é oposta em cada hemisfério, como mostrado pelas setas dentro de cada
célula. O sentido do nado é modificado pela mudança no sentido de
rotação flagelar. (Figura modificada de Bazylinski & Frankel, 2004. Nat.
Rev. Microbiol. 2: 217-230).
do campo geomagnético. Se, ao contrário, a
informação é de que elas estão em um ambiente
redutor, a bactéria inverte o sentido de rotação do
flagelo e nada para cima, antiparalelo ao campo
geomagnético. No hemisfério Sul, o campo
magnético da Terra aponta para cima. Como as
bactérias preferem os mesmos ambientes, em
condições oxidantes, elas também nadam para
baixo, mas agora antiparalelo ao campo
geomagnético. Já em condições redutoras, elas
nadam para cima, paralelamente ao campo
magnético da Terra. A propriedade de orientação
e navegação ao longo das linhas de campos
magnéticos exibida por bactérias é conhecida
como magnetotaxia e as bactérias que a
apresentam são denominadas bactérias
magnetotáticas (Fig. 1). De uma maneira geral,
nos dois hemisférios, as bactérias magnetotáticas
tendem a parar em ambientes com pouco ou
nenhum oxigênio. No equador geomagnético
encontram-se os dois tipos em quantidades
aproximadamente iguais.
A detecção de bactérias magnetotáticas
é relativamente simples podendo ser feita com
um ímã comum e um microscópio de luz. Uma
gota contendo sedimento e água do local de
coleta é colocada sobre a lâmina no microscópio
e um ímã é posicionado ao lado da amostra. Após
alguns minutos, as bactérias magnetotáticas
estarão acumuladas na borda da gota mais
próxima ou na oposta ao ímã. Invertendo o ímã,
as bactérias magnetotáticas nadam para o outro
extremo da gota. Quando as bactérias morrem,
elas não se deslocam, mas giram em torno do
próprio eixo, devido ao seu alinhamento passivo
ao campo magnético aplicado. Como as bactérias
magnetotáticas se orientam e nadam conforme
o sentido do campo magnético, podemos isolar
bactérias magnetotáticas de vários ambientes. Na
fig. 2 são apresentadas algumas bactérias
magnetotáticas e suas estruturas.
O cultivo em laboratório de bactérias
magnetotáticas é muito difícil. Até hoje umas
poucas cepas de espécies pertencentes a um
espirilo do gênero Magnetospirillum e alguns
isolados de cocos e víbrios são mantidos em
culturas puras. Fisiologicamente, as bactérias do
gênero Magnetospirillum são desnitrificantes,
utilizando oxigênio e nitrato como aceptores
terminais de elétrons. A fixação do nitrogênio
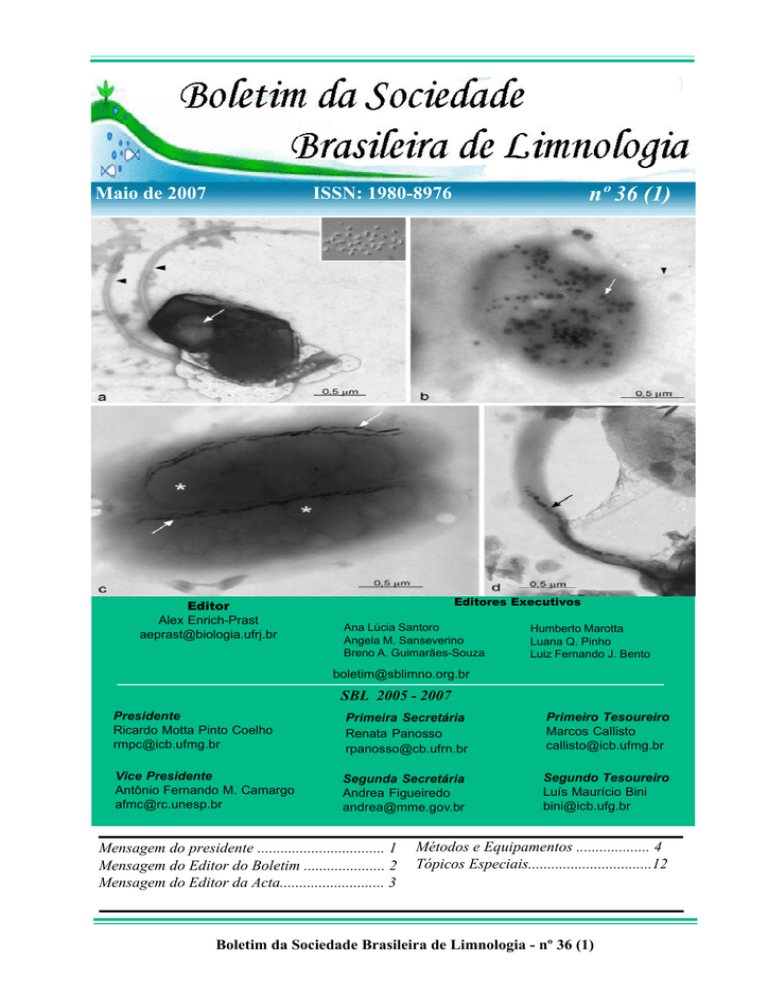
Fig. 2: Bactérias magnetotáticas encontradas nas lagoas Encantada e
do Robalo, no Parque Nacional da Restinga de Jurubatiba, por
microscopia eletrônica de transmissão. (a) Cocos magnetotáticos, vistos
também por microscopia óptica (detalhe); (b) Uma célula que se soltou
de um morfotipo semelhante ao MMP; (c) Bacilos magnetotáticos e (d)
Espirilo magnetotático. As setas apontam os magnetossomos, que podem
ser arredondados, prismáticos ou em forma de ponta de lança. As cabeças
de seta em (a) e (b) mostram os flagelos e os asteriscos em (c) indicam
as inclusões citoplasmáticas.
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
16
pode ser uma capacidade muito útil na zona de
transição óxica-anóxica, onde fontes de nitrogênio
fixado podem ser limitadas e, portanto, alvo de
intensa competição. Algumas bactérias
magnetotáticas crescem autotroficamente; outras
parecem ser heterotróficas anaeróbicas, reduzindo
sulfato ou ferro. No entanto, alguns métodos
independentes de cultivo podem ser utilizados
para estudar a diversidade, ecologia e geoquímica
das comunidades de bactérias magnetotáticas no
ambiente, assuntos hoje pouco conhecidos. Ainda
há dúvidas se a magnetotaxia ou a síntese dos
magnetossomos são subprodutos do metabolismo
energético, mas há indícios que a síntese de
magnetita não possa prover energia suficiente para
o crescimento celular. De qualquer forma, é
possível que a quantidade significativa de energia
investida pelas bactérias para captar grandes
quantidades de ferro e biomineralização dos
magnetossomos possa ter vantagens adicionais à
magnetotaxia.
Recentemente, os aspectos ecológicos das
bactérias magnetotáticas começaram a ser
estudados. O papel das bactérias magnetotáticas
no ciclo do ferro sempre foi cogitado, em função
do grande acúmulo citoplasmático desse elemento
nos magnetossomos. Elas podem ser as maiores
contribuintes não reconhecidas para os fluxos
mais importantes de ferro reduzido no ambiente.
No entanto, tem-se pouca informação nesse
sentido. Talvez a biomassa bacteriana retenha
concentrações nanomolares de ferro. Também se
especula o envolvimento dessas bactérias com os
ciclos de enxofre, nitrogênio e carbono. Sabe-se,
contudo, que as bactérias magnetotáticas habitam
preferencialmente ambientes marinhos
estratificados nas colunas d’água ou sedimento,
e ambientes de água doce, podendo chegar a
números relativamente grandes, 104 células/mL,
na interface óxica-anóxica e em regiões
estritamente anóxicas ou em ambas. Há apenas
um relato de bactérias magnetotáticas em solos
alagados. Em ambientes de água doce encontramse apenas bactérias produtoras de magnetita,
enquanto que em ambientes marinhos já foram
descritas produtoras de magnetita e de greigita.
Acredita-se que a zona de transição óxicaanóxica seja uma região propícia à ocorrência de
reações redox espontâneas ou mediadas por
microrganismos. As bactérias magnetotáticas são
típicas bactérias de gradiente, pois obtém energia
para o crescimento da proximidade entre
compostos redutores e oxidantes na interface
química e ocorrem em finas camadas horizontais,
transversais ao gradiente vertical. Pela sua
relação com o elemento ferro, as bactérias
magnetotáticas podem estar envolvidas pelo
menos em reações químicas com o par FeII/FeIII.
Em estudos com microcosmos, as
bactérias magnetotáticas são mais abundantes
nas condições subóxicas imediatamente abaixo
da interface óxica-anóxica. Em Salt Pond,
Massachusetts, foram realizados os primeiros
experimentos correlacionando a presença de
bactérias magnetotáticas com as condições
abióticas do local (Simmons et al. 2004. Appl.
Environ. Microbiol. 70: 6230-6239). Foi
observado um gradiente decrescente de oxigênio
de acordo com o aumento da profundidade por
causa do consumo de matéria orgânica por
organismos heterotróficos aeróbicos. Uma
quimioclina de extensão variável se desenvolve
onde o oxigênio acaba, produzida pelo fluxo
ascendente de sulfeto continuamente gerado
pelas bactérias redutoras de sulfato na região
anóxica. Um pico de ferro reduzido particulado
está presente no topo da quimioclina, devido ao
fluxo desse ferro em direção às águas com
oxigênio. Os cocos magnetotáticos são as
bactérias magnetotáticas predominantes nessa
profundidade. Já na base da quimioclina, podese observar um pico de ferro reduzido dissolvido,
pois as concentrações de sulfeto são insuficientes
para seqüestrar o ferro em sulfetos sólidos.
Relacionados a esse pico, estão os
microrganismos multicelulares magnetotáticos
(MMM), um conjunto de bactérias
magnetotáticas que apresenta características de
multicelularidade. Entre a zona rica em cocos e
a rica em MMM estão as cadeias de diplococos
(“barbells”) que vivem supostamente na região
de menor concentração de oxigênio e sulfeto.
Um pouco abaixo dos MMM estão os bacilos,
que são as bactérias magnetotáticas que
requerem maiores concentrações de sulfeto. A
concentração mínima de ferro dissolvido para a
presença de bactérias magnetotáticas em
qualquer parte da quimioclina ainda é
desconhecida. A presença de oxidantes e
redutores na quimioclina, assim como o
gradiente físico de densidade entre as águas
mornas da superfície e as águas frias da base,
http://www.sblimno.org.br
17
resultam em um alto número de bactérias e placas
densas de microrganismos fototróficos
anaeróbicos onde há luz disponível. A
classificação das bactérias foi feita com bases
morfológicas porque ainda não foram
estabelecidos genes para a detecção e
identificação das bactérias magnetotáticas a partir
de amostras naturais. Não foi estudado nessa
ocasião o fluxo de ferro para fora da quimioclina,
mas os protistas predadores podem colaborar com
essa exportação. Esse trabalho forneceu
evidências que a composição química do
ambiente pode ser tanto o número total de
bactérias magnetotáticas como a abundância
relativa das diferentes espécies.
Sugeriu-se que magnetossomos
fossilizados das bactérias magnetotáticas
pudessem se tornar indicadores paleoambientais
em registros rochosos ou de sedimentos, visto que
os magnetossomos têm características
morfológicas que os distinguem de minerais
inorgânicos e podem ser responsáveis por parte
considerável do magnetismo remanescente de
registros fósseis. O maior fator limitante ao uso
dos magnetossomos como paleo-indicadores é a
falta de informações quantitativas sobre os
ambientes redox, favorecendo a produção de
greigita ou magnetita pelas bactérias nos dias de
hoje, para que essa informação possa ser
extrapolada para paleoambientes. Pouco ainda se
sabe sobre a biologia das bactérias
magnetotáticas, mas certamente a abordagem
multidisciplinar que esses microrganismos
proporcionam tem contribuído para a elucidação
desse fascinante fenômeno microbiológico.
Thaís Souza Silveira
[email protected]
Ulysses Lins
[email protected]
Universidade Federal do Rio de Janeiro
Cadeia Microbiana Heterotrófica em
Fitotelma
A família Bromeliaceae, muito
representativa em restingas (Araújo 1992. Em
Seeliger (eds.) Coastal plant communities of
Latin America. Academic Press, San Diego,
p.337-347), pode formar microhábitats para
diversos grupos de organismos. Esse fato é
devido à capacidade das bromélias em guardar
umidade na base das folhas e, dependendo da
espécie, formar um reservatório de água no
centro da sua roseta. Bromélias da espécie
Neoregelia cruenta tipicamente formam
fitotelma. Esta espécie apresenta diferentes
morfotipos que se encontram muitas vezes
dispostos em famílias com estágios de vida
distintos, isto é, apresentam indivíduos jovens,
adultos e senescentes em um mesmo estolão.
Muitos
estudos
relacionando
comunidades de macrofauna que habitam
bromélias já foram realizados, porém pouco se
conhece ainda sobre a comunidade microbiana
em fitotelma de bromélias. Brighigna et al.
(2000. Rev. Biol. Trop. 48: 511-517)
determinaram a densidade de bactérias e fungos
associados a folhas de bromélias do gênero
Tillandsia, comparando plantas encontradas em
áreas poluídas e não poluídas da cidade de San
José, Costa Rica.
A importância da alça microbiana para
os ambientes aquáticos faz com que estas
bactérias devam ser avaliadas e quantificadas na
teia alimentar, visto que insere uma nova visão
da atividade microbiana em todos os ambientes.
Segundo a teoria de Biogeografia de Ilhas
de MacArthur & Wilson (1963. Inter. J. Org.
Evol. 54: 970 – 984), o tamanho e a idade de
uma ilha podem determinar a sua abundância e
riqueza de organismos. Diante desta teoria, caso
ela seja aplicada a fitotelmas como ilhas, temos
que: bromélias com maior volume de fitotelma,
tenderiam a ter maior riqueza de espécies e que
em bromélias mais velhas, este padrão se
repetiria. Temos que a teoria de Biogeografia faz
referencia também a distância da ilha em relação
ao continente, considerando este como uma fonte
de espécies. Desta forma, ilhas mais próximas
ao continente, teriam uma maior abundância e
riqueza de espécies que uma ilha mais distante.
Por não haver aparentemente, relação entre esta
afirmação com o presente trabalho, decidimos
não analisá-la.
O objetivo deste estudo foi avaliar a
cadeia microbiana heterotrófica em fitotelma de
Neoregelia cruenta em diferentes estágios de
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
18
vida, além de verificar se a teoria de Biogeografia
de Ilhas também se aplica ao fitotelma.
As coletas foram realizadas em matas de
dominância de Clusia sp. no Parque Nacional da
Restinga de Jurubatiba, município de Macaé, Rio
de Janeiro, em agosto de 2006. Foram escolhidas
céls.105.mL-1. Nas bromélias adultas, a densidade
variou entre 3 e 77 céls.105.mL-1, enquanto as
bromélias jovens apresentaram valores entre 2 e
5 céls.105.mL-1. Foi observada uma correlação
entre a densidade bacteriana e a estágio de
120
Tab. I : Caracterização das bromélias e fitotelma encontrada em N.
cruenta.
110
Família
1
2
3
4
5
S1
A1
J1
S2
A2
J2
S3
A3
J3
S4
A4
J4
S5
A5
J5
85
73
35
54
70
22
65
56
34
59
59
33
61
60
36
4,5
5,5
5,5
3,5
4,0
3,5
4,5
5,0
6,5
3,0
5,0
5,0
2,5
4,0
3,5
19,5
18,5
15,0
21,5
23,0
13,0
25,0
18,5
16,0
18,5
16,0
12,0
18,5
20,0
13,5
24,6
25,3
24,7
28,0
26,5
24,2
25,4
25,8
28,7
29,3
28,8
29,3
29,4
3,3
2,7
4,2
6,4
3,4
4,1
3,6
4,4
3,6
5,3
3,0
2,8
5,4
Volume (mL)
pH
110
250
60
80
100
30
100
70
90
55
180
40
35
100
40
7,2
6,2
5,8
3,7
5,1
4,8
4,1
5,3
5,0
4,0
5,2
5,0
7,5
7,1
4,6
aleatoriamente cinco famílias de bromélias em que
se encontravam pelo menos três indivíduos
distintos quanto ao ciclo de vida (um jovem, um
adulto e um senescente), totalizando 15 bromélias.
A altura e volume de água encontrado foram os
parâmetros utilizados para caracterizar as
bromélias quanto a sua estrutura física. A
caracterização das bromélias e dos fitotelmas
encontra-se na Tab. I.
As amostras retiradas das bromélias foram
acondicionadas em frascos. As alíquotas para a
análise de densidade bacteriana foram fixadas com
formalina 40 % , filtradas e coradas com DAPI.
Então, 20 campos aleatórios foram analisados em
microscópio de epifluorescêcia Olympus IX-71.
Para a análise da comunidade zooplanctônica, as
amostras foram fixadas com formalina 4% e
concentradas em rede de 68 mm. Posteriormente,
foram analisadas quantitativamente através de
uma câmara de Sedgwick-rafter em microscópio
óptico Olympus BX-41.
Testes estatísticos de regressão linear,
ANOVA e Teste de Tukey foram realizados no
programa Statistica 6.0.
A densidade bacteriana nas bromélias
senescentes apresentou valores entre 3 e 120
Densidade Bacteriana (cels.10 5/mL)
100
Altura Diâmetro Perímetro base Temperatura Oxigênio
Bromélia
0
(cm) tanque (cm) (cm)
(mg/L)
( C)
90
80
70
60
50
40
30
20
Mediana
25%-75%
Whiskers
10
0
S
A
J
Fig. 1. Box Plot da densidade bacteriana (cels.105/mL) nos diferentes
estágios de senescência (S = senescente, A = adulto, J = jovem).
senescência das bromélias (p<0,05). O teste de
Tukey mostrou que a densidade bacteriana
apresentou diferença entre jovens e senescentes
(Fig. 1 e Tab. II). A densidade bacteriana não
apresentou diferença entre as cinco famílias
analisadas (p>0,05). A densidade bacteriana foi
dez vezes maior nas senescentes quando
comparada aos indivíduos jovens da mesma
família, o que pode ser explicado pela Teoria de
Biogeografia de Ilhas (MacArthur & Wilson
1963 op. cit.). Entretanto, alguns trabalhos
questionam se realmente existe uma biogeografia
Tab. II: Densidade (ind/mL) e riqueza de táxons de organismos
zooplanctônicos e densidade de bactérias (céls.105/mL) nas bromélias
estudadas.
Densidade
Família Bromélia Nematódeo Tacameba Zooplanctônica
1
2
3
4
5
S1
A1
J1
S2
A2
J2
S3
A3
J3
S4
A4
J4
S5
A5
J5
http://www.sblimno.org.br
42,0
0,0
0,4
0,0
0,8
0,0
0,2
0,0
0,0
0,0
0,0
0,0
0,0
0,1
0,0
11,3
0,0
0,0
0,0
0,0
0,0
1,0
0,0
0,0
0,0
0,0
0,0
0,2
0,0
0,0
5,1
0,6
29,4
0,6
1,0
0,2
0,5
1,4
0,3
0,3
0,3
1,5
2,9
3,8
0,5
Riqueza de
Densidade
táxons do
Bacteriana
Zooplâncton
4
5
4
5
8
4
6
3
4
4
4
1
5
6
4
120
5
3
116
59
5
61
77
2
3
3
4
76
4
4
19
microbiana, já que potencialmente os
microrganismos conseguiriam estar dispersos por
todos os ambientes. As barreiras geográficas
seriam, de certa forma, irrelevantes para
distribuição espacial das bactérias. No entanto,
nem todos os taxas microbianos estariam
distribuídos em mesma proporção pelos
ambientes (Dolan 2005. Aquat. Microb. Ecol. 41:
39-48; Dolan 2006. J. Biogeogr. 33: 199-200).
O padrão da distribuição da densidade
bacteriana não foi observado para a comunidade
zooplanctônica. Os rotíferos foram encontrados
em todas as bromélias (exceto em um indivíduo
jovem). As fases larvais de insetos, organismos
que apresentaram menor densidade, foram
encontradas apenas em indivíduos jovens. As
análises da comunidade zooplanctônica não
demonstraram relação entre o estágio de
senescência das bromélias e a riqueza de espécies,
e nem relação entre as cinco famílias estudadas
e a riqueza de espécies (p>0,05). Mesmo assim,
observou-se que as bromélias adultas e
senescentes apresentaram maior riqueza de
grupos zooplanctônicos do que as jovens. O que
pode ser explicado por serem ambientes
ecologicamente mais maduros, portanto mais
estáveis, logo suportariam uma maior
diversidade. Dolan (2005. op. cit.) afirma, em sua
revisão, que a riqueza taxonômica de
microrganismos cresce com o tamanho do
sistema e a composição da comunidade pode se
relacionar com os corpos de água vizinhos.
Entre as famílias de bromélias analisadas,
encontramos maiores densidade bacteriana e
riqueza de espécies zooplanctônicas na família
2. Em contra partida, a família 4 apresentou os
menores valores para estas mesmas variáveis
(Tab. II).
Através da análise de regressão linear
simples, foi observada correlação positiva entre
densidade bacteriana e densidade de nemátodos
e de tecamebas (p<0,05). Resultados diferentes
foram encontrados por Mayhew & Stephenson
(1997. Environ. Technol. 18: 883-892). Estes
autores, observaram que o crescimento de
protozoários, rotíferos, nematódeos e oligoquetas
reduzem a biomassa bacteriana por predação.
A disponibilidade de nutrientes e a
predação controlam não apenas a densidade
bacteriana total, bem como a estrutura, a atividade
e o crescimento desta comunidade (Gasol et al.
2002. Limnol. Oceanogr. 47: 62-77; Simek et al.
2005. Appl. Environ. Microbiol. 71: 2381-2390).
Também foi observada ausência de correlação
entre as densidades das comunidades bacteriana
e zooplanctônica (p>0,05). Entretanto, a menor
densidade bacteriana foi encontrada na bromélia
onde a densidade de ciliados foi a maior.
Através das relações encontradas no estudo
de fitotelma de N. cruenta, podemos sugerir que
a teoria de Biogeografia de Ilhas também é
aplicável neste microhabitat. Assim como, sugere
que a alça microbiana é importante na
manutenção e sustentação deste ambiente.
Agradecimentos
Agradecemos ao Dr. Carlos Eduardo
Grelle, coordenador do Curso de Campo do
Programa de Pós-Graduação em Ecologia da
Universidade Federal do Rio de Janeiro realizado
em Macaé (RJ) no ano de 2006, e a todos os
professores que colaboraram com essa disciplina.
Agradecemos ao auxílio dos alunos do curso no
trabalho de campo. Agradecemos ao Msc. Marcos
Figueiredo pela ajuda nos métodos estatísticos e
a Priscila Gomes Rosa pela colaboração na
identificação da comunidade zooplanctônica.
Agradecemos ao Dr. Alex Prast e à Dra. Dionéia
Cesar pelas valiosas sugestões para a realização
deste trabalho.
Carlos Henrique Duque-Estrada
[email protected]
Alessandro Del´Duca
[email protected]
Universidade Federal de Juiz de Fora
Aplicações da Citometria de Fluxo na
Ecologia Microbiana
A ecologia microbiana pertence a uma nova
linha que tem se desenvolvido bastante com a
aplicação de tecnologias modernas voltadas para
outras áreas. Uma delas é a citometria de fluxo
(FCM, do inglês Flow Citometry). Originalmente
desenvolvida para o estudo de células de sangue
e câncer (Shapiro 2003. Practical Flow
Cytometry, John Wiley & Sons, 4th ed., 736p.), a
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
20
Fig. 1: (a) Detalhe da formação do fluxo laminar. (b) Esquema da iluminação da amostra por um laser, e o espalhamento frontal, lateral e as
diferentes fluorescências. Figuras modificadas de Purdue.
FCM é uma poderosa técnica que atua através de
análises múltiplas em células individuais. É
importante destacar que dois de seus principais
inventores (Howard Shapiro e Leonard
Heizemberg) jamais protegeram suas descobertas
por patentes, que hoje fornecem muito dinheiro
às principais indústrias de FCM, como a Dako,
Becton, Dickinson & Co., Partec.
A FCM é a técnica de medida, em fluxo, de
processos celulares. O termo citometria de fluxo
muitas vezes é confundido com a sigla FACS
(Fluorescence Activated Cell Sorting), o processo
desenvolvido por Leo Heizemberg, em Stanford,
para separar células, baseado em propriedades
medidas em fluxo, técnica muito usada na
imunologia. Na FCM, a detecção dos eventos ou
células é baseada na fluorescência emitida pelas
células, tanto a auto-fluorescência, como a
fluorescência emitida por marcadores específicos.
Para conhecer mais sobre a FCM, é necessário
entender o que é o processo de fluorescência, a
relação inversa entre comprimento de onda e
energia, o que são comprimentos de onda para a
excitação e emissão.
As bases do processo de FCM são (1) os
fluídos, (2) a ótica, e (3) a eletrônica.
No que diz respeito à fluídica, as células
devem estar suspensas e mantidas em fluxo, em
sentido único na direção da cubeta. Na maioria
dos instrumentos isto é feito injetando um fluído
carreador (sheat fluid, solução salina tipo PBS ou
mesmo água deionizada), que passa sob pressão
por um orifício (entre 70 e 300 µm). Se a
geometria do aparelho for adequada, a amostra
ao ser injetada fluirá por uma área central dentro
do fluxo de carreador, não se misturando com ele.
Isto é denominado fluxo laminar (Fig. 1a). Os
detalhes dependem do fabricante e da construção
do aparelho, em alguns sistemas (Dako) é grande
a pressão diferencial entre o fluxo da amostra e
do carreador, permitindo grandes velocidades de
análise. Em qualquer sistema, a configuração
ideal é quando o fluxo laminar está bem ajustado
(velocidade e volume), de forma que uma célula
passe a cada vez em frente à fonte de iluminação,
recebendo a(s) excitação(ões) e emitindo
fluorescência(s). Os diferentes aparelhos
existentes podem permitir contagens de células/
partículas em velocidades de 100 eventos por
segundo até 50 mil por segundo. Para amostras
ambientais (bactérias), uma boa medida é realizar
contagens em até mil eventos por segundo, o que
torna possível contar 10 mil células em menos
de 1 minuto.
Quanto à ótica, é necessária uma fonte de
luz, que deve ser focalizada no exato ponto da
cubeta onde as células passam enfileiradas. Os
lasers são as melhores opções para esta fonte de
luz na FCM, pois fornecem luz monocromática,
unidirecional e em fase. Após a iluminação das
partículas, elas absorvem a luz e emitem
fluorescências diversas. Todos os aparelhos
coletam ao menos a fluorescência frontal (foward
scatter ou FSC), a lateral (side scatter ou SSC) e
algumas cores (verde, laranja e vermelho). A Fig.
1b ilustra o processo. Cada ponto significa um
evento ou uma célula analisada. O segredo é
treinar o olho e o cérebro junto ao software para
reconhecer esses eventos e separá-los de
qualquer outro que não interesse. As
fluorescências ou emissões das células
analisadas podem ainda ser selecionadas por
filtros específicos, que apenas deixem passar
alguns comprimentos de onda, tornando o
processo ainda mais seletivo.
No que se refere à eletrônica, os sinais são
percebidos por fotodetectoras, e aí transformados
http://www.sblimno.org.br
21
de analógicos para digitais. São necessários bons
recursos computacionais para realizar estas
tarefas seqüenciais em velocidades da ordem de
milhares por segundo. Esta é uma complexa área
e cada indústria tem seu padrão e procedimentos,
sendo grande parte das boas idéias patenteadas.
Existem várias opções de software, alguns poucos
livres (WinMDI e CytoWin) e muitos pagos (FloJo, Summit, Cell Quest – este último para Mac).
O uso de um bom programa pode fazer uma
grande diferença no potencial de produção de
informações a partir de dados de FCM. Os
arquivos gerados não são pequenos, e como
podem ser muitas amostras de muitos usuários e
projetos, necessita-se de espaço para o
armazenamento de dados.
Inicialmente os pesquisadores utilizavam
citômetros grandes, complicados e de alto custo
(mais de US$ 250,000), equipados com lasers
Fig. 2: Número de publicações no período entre 1995 e 2006 contendo
as palavras chave “bacterioplankton” e “flow cytometry” (fonte Web of
Science).
emitindo UV para o uso de fluorocromos como
o DAPI e o Hoechst 33342. Um fato importante
foi o aparecimento, na década de 1990, de
citômetros relativamente mais simples e portáteis,
a
e de menor custo (na faixa de US$ 100,000), com
lasers azuis (488 nm). Em paralelo a isto, houve
o desenvolvimento de corantes excitáveis com
alta eficiência por estes lasers azuis. Desta forma,
os protocolos foram simplificados e uma enorme
quantidade de artigos, de muitos pesquisadores
utilizando citometria em fluxo para contagem de
bactérias, vem sendo publicada (Fig. 2). Pode-se
afirmar que a contagem de células e análise de
populações naturais de bacterioplâncton por
citometria em fluxo é rotina em muitos
laboratórios, e está se tornando uma técnica
essencial em estudos de microbiologia aquática
(Gasol & del Giorgio 2000. Sci. Mar. 64:197224).
Metodologia e cuidados
Para a determinação de bactérias, o
procedimento mais comum é coletar as amostras,
fixar com paraformaldeído 2% (ou em amostras
mais sensíveis, paraformaldeído 1% +
glutaraldeído 0,01%) durante 15 minutos, e
congelar em nitrogênio líquido. Depois, pode-se
manter a -196°C, ou transferir para um freezer 80°C. A transferência para freezer a -20°C pode
ser suficiente, mas descongelamentos acidentais
produzem resultados catastróficos. De qualquer
modo, manter em geladeira ou congelar em
freezer comum não é recomendado na literatura.
No laboratório, pequenas alíquotas (250
µL) são coradas com algum marcador de ácidos
nucléicos (syto13 e sybr green são os mais
usados), adicionadas de microesferas de látex (1,0
ou 1,5 µm), e analisadas em um citômetro de
fluxo. As esferas são importantes marcadores
para: (1) normalizar os sinais de fluorescência
tanto quanto ao local e a intensidade e (2)
quantificar as bactérias. Um parâmetro
comumente utilizado para a separação por
tamanho é o espalhamento frontal. Para bactérias,
o
espalhamento
lateral
(SSC)
é
comprovadamente uma proxi de tamanho até
mais adequada que o FSC. Os ácidos nucléicos
das bactérias vão fluorescer em verde, e a
clorofila vai autofluorescer em vermelho. Usando
Fig. 3: Citogramas típicos com os diferentes painéis e informações obtidas da FCM, ilustrando em verde bactérias HNA, em azul bactérias
LNA, em amarelo as microesferas de látex de 1,5 µm. (a) Espalhamento lateral (SSC) contra a fluorescência verde (FL1). (b) Fluorescência verde
(FL1) contra a fluorescência vermelha (FL4). (c) Histograma da fluorescência verde (FL1).
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
22
estes parâmetros mais o espalhamento lateral,
podem se analisar as diferentes populações de
bactérias com facilidade. Os resultados da análise
são nuvens de pontos em gráficos escolhidos pelo
operador. O mais usual é dispor os dados em (1)
gráfico do espalhamento lateral contra a
fluorescência verde, (2) gráfico da fluorescência
verde contra a fluorescência vermelha e um
histograma da fluorescência verde (Fig. 3).
Além da contagem total de células, uma
informação interessante que pode ser obtida é a
separação de células por “conteúdo aparente de
ácidos nucléicos”. As células de bactérias podem
ser divididas em pelo menos dois grupos, de
acordo com as características do espalhamento
lateral (SSC), da fluorescência (do syto13 ligado
aos ácidos nucléicos) e a auto-fluorescência da
clorofila (quando se retiram os autotróficos da
contagem). Na Fig. 3 podem ser vistos estes
grupos: em verde são as bactérias com alto
conteúdo aparente de ácidos nucléicos (HNA), em
azul as bactérias com baixo conteúdo aparente de
ácidos nucléicos (LNA). Neste mesmo gráfico,
em amarelo, estão as microesferas de látex de 1,5
µm e, em cinza, o ruído. Estes grupos podem
representar bactérias com diferentes estados
metabólicos.
Além da contagem de bactérias, também
existem outras aplicações da FCM na
microbiologia aquática. Empregando a autofluorescência dos pigmentos encontrados nas
células, é possível discriminar os organismos
autotróficos em uma população de bactérias, em
especial para avaliar as populações de
Prochlorococcus e Synecococcus. Ainda
empregando a auto-fluorescência, também se
pode analisar células do fitoplâncton, existindo
até citômetros especiais dedicados a esta
finalidade
(como
a
Citoboia
–
www.cytobuoy.com), capazes de analisar a forma
das células. Outros organismos também
analisados por FCM são as leveduras e protistas.
Todas estas são abordagens diretas de estimativa
de números de organismos, mas também podem
ser investigados inúmeros processos celulares
(atividade, divisão celular, apoptose, etc.), desde
que baseados em marcadores fluorescentes.
Outra importante aplicação da FCM é a
separação celular ou “cell sorting”, que consiste
na separação de células durante o processo de
fluxo. Este tema será objeto de outro artigo. Uma
parte significativa dos citômetros instalados no
mundo está localizada em instituições médicas
ou de áreas afins, visto sua aplicação na área de
saúde. Outra enorme utilização e que compra
quase 1/3 dos citômetros produzidos no mundo,
bem menos conhecida, é na separação de sêmen
X e Y de bois, cavalos, porcos e carneiros. O
aparelho para esta aplicação custa cerca de 1
milhão de dólares, mas agrega muito valor ao
produto, pois permite ao produtor escolher que
tipo de rebanho (p. ex., corte ou leiteiro) deseja
criar.
As técnicas tradicionais de microscopia
não se adaptam a estudos em larga escala, como
operações oceanográficas e monitoramentos em
alta freqüência amostral, que geralmente
produzem mais amostras do que a capacidade
de analisá-las. Então uma das maiores vantagens
da FCM é a capacidade de avaliar um grande
número de células (10 mil) em pouco tempo
(menos de 1 minuto), e ainda possibilitando
ensaios multiparamétricos. Devido à
possibilidade de analisar dezenas de milhares de
células em minutos, a citometria em fluxo pode
realmente reduzir o tempo necessário para a
determinação da abundância bacteriana, o
tamanho das células e sua atividade, oferecendo
informações simultâneas sobre a estrutura
(heterogeneidade) de uma população bacteriana
com uma incomparável vantagem estatística
sobre as contagens ao microscópio.
Apesar das muitas vantagens da FCM
descritas neste texto, nem tudo é assim tão
maravilhoso. Por esta abordagem técnica não é
possível avaliar a forma das células analisadas,
pois elas são pontos de luz. Outra enorme
desvantagem é o custo do equipamento (a partir
de 100 mil dólares), proibitivo para muitas
instituições no mundo. A necessidade de uma
maior especialização do operador também acaba
sendo um impedimento para o avanço desta
aplicação nas ciências ambientais. Mas são
problemas contornáveis e minorados pelas
vantagens da abordagem.
No Brasil:
As atividades de citometria de fluxo
aplicadas a pesquisas ambientais foram iniciadas
no Brasil em 1996, quando começamos a operar
um FacsCalibur (BD) gentilmente cedido pelo
Dr. Alberto Nóbrega (Instituto Microbiologia,
http://www.sblimno.org.br
23
Universidade Federal do Rio de Janeiro). Os
citômetros são dedicados a análises médicas de
imunologia, e em geral seus operadores não
costumam analisar amostras de água. Mas este
nobre colega, por seu espírito colaborador e
gentileza, foi o pilar do desenvolvimento desta
área no país. Em 2003 foi obtido um
financiamento da Finep para a compra de um
citômetro dedicado às pesquisas em meio
ambiente. Um citômetro CyAn (Dako) foi
adquirido e instalado em 2005, e com isto pode
ser criada a Unidade de Citometria Aplicada a
Ecologia Aquática e a Oceanografia – UCEA,
instalada no Departamento de Biologia Marinha
do Instituto de Biologia (Universidade Federal do
Rio de Janeiro).
Nestes 10 anos de citometria de fluxo
aplicada ao meio ambiente no Brasil, foram
analisadas amostras da Baía de Guanabara, da
Lagoa Rodrigo de Freitas, do Oceano Atlântico,
e de muitos rios do Brasil durante o projeto Brasil
das Águas (ver p. ex., Andrade et al. 2003. J.
Microbiol. Meth. 55: 481-490). Nossos objetivos
futuros envolvem ampliar as colaborações da
UCEA, de forma a avançar na técnica de
citometria, pois boa parte do que se faz no mundo
hoje é relativamente simples em termos da técnica
em si. Em um primeiro momento estamos
ampliando o uso para amostras de sedimento e
ar. Portanto, existe um grande espaço para o
desenvolvimento de novas aplicações e
colaborações, assim como projetos interessantes
e de qualidade.
Rodolfo Paranhos
[email protected]
Luciana Andrade
[email protected]
Universidade Federal do Rio de Janeiro
Relações entre Microorganismos e Luz
Solar na Degradação da Matéria
Orgânica Dissolvida em Ecossistemas
Aquáticos
Bactérias
planctônicas
são
microorganismos extremamente importantes para
o ciclo de energia e matéria nos ecossistemas
aquáticos, uma vez que tem papel central na
remineralização de nutrientes orgânicos e porque
formam a base de uma cadeia trófica planctônica
alternativa conhecida como alça microbiana.
Nesse contexto, as bactérias absorvem a matéria
orgânica dissolvida (MOD) do meio,
transformando-a em biomassa (através da
produção secundária), disponível para os níveis
tróficos superiores, e em dióxido de carbono
(CO 2), como produto final do processo de
obtenção de energia (respiração). Muitos estudos
vêm sendo realizados sobre a demanda
energética e capacidade produtiva desses
organismos (Roland & Cole 1999. Aquat.
Microb. Ecol. 20: 31-38), porém os mecanismos
que determinam o balanço do carbono destinado
ao crescimento ou a obtenção de energia ainda
são bastante obscuros. Temperatura,
disponibilidade de nitrogênio e fósforo, assim
como a qualidade do carbono orgânico dissolvido
(COD) são reconhecidos como fatores
importantes e limitantes para o metabolismo
bacteriano (Farjalla et al. 2002. Arch. Hydrobiol.
156: 97-119).
O processo de foto-degradação da MOD
(ou do COD) consiste na interação dos raios
solares com alguns compostos orgânicos
dissolvidos na água. Essa reação pode resultar
na decomposição total ou parcial desses
compostos, produzindo moléculas inorgânicas
(e.g. CO2) e/ou alterando a composição e a
disponibilidade dos mesmos para o crescimento
bacteriano (para revisao ver Suhett et al. 2006.
Oecol. Bras. 10: 186-204). Sendo assim, a fotodegradação e a degradação bacteriana podem ser
estudadas como processos paralelos uma vez que
ambas são consideradas relevantes para a
mineralização da MOD em ambientes aquáticos
(Jonsson et al. 2001. Limnol. Oceanogr. 46:
1691-1700; Amado et al. 2006. FEMS Microbiol.
Ecol. 56: 8-17). Por exemplo, Amado et al. (2006
op. cit.) demonstraram que a importância relativa
destes dois processos pode variar sazonalmente
em um mesmo ecossistema, e estes se
complementam na degradação total da MOD.
Entretanto, não menos importante é o resultado
da integração entre estes dois processos, uma vez
que ambos atuam sobre o mesmo conjunto de
moléculas (Cotner & Biddanda 2002.
Ecosystems 5: 105-121). O objetivo desse artigo
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
24
é mostrar as possíveis relações entre a fotodegradação e a produção e respiração bacterianas
e propor alguns mecanismos reguladores destas
relações.
Alguns estudos mostraram que a fotodegradação da MOD pode resultar na redução das
taxas de produção e respiração bacteriana,
enquanto que outros registraram aumento dessas
taxas (e.g. Anesio et al. 2000. Microb. Ecol. 40:
200-208). Diante desse panorama, poder-se-ia
concluir que as interações entre a degradação
bacteriana e a foto-degradação da MOD são
incertas e ambíguas. Tranvik e Bertilsson (2001.
Ecol. Lett. 4: 458-463), em uma revisão com
inúmeros ambientes que formam um gradiente de
produtividade, concluíram que quando algas
planctônicas são uma importante fonte de MOD
para a coluna d’água (ambientes mais produtivos),
o processo de foto-degradação resulta na redução
do metabolismo bacteriano. Por outro lado,
quando a MOD é fundamentalmente de origem
húmica (substâncias húmicas), o processo de fotodegradação resulta no aumento do metabolismo
bacteriano. Entretanto, os mecanismos que
levaram a esses diferentes resultados não foram
explicados por aqueles autores (Tranvik &
Bertilsson 2001 op. cit.).
A produção bacteriana é um processo
dependente da disponibilidade de COD (energia
e substrato para o crescimento) e de outros
nutrientes, como o nitrogênio (N) e o fósforo (P),
usados principalmente na formação de proteínas,
material genético (DNA, RNA) e armazenamento
de energia (ATP) (Makino et al. 2003. Funct. Ecol.
17: 121-130). A foto-degradação da MOD pode
liberar nutrientes, como N e P, antes imobilizados
no interior de complexas moléculas orgânicas,
tornando-os disponíveis para utilização
microbiana (Cotner & Heath 1990. Limnol.
Oceanogr. 35: 1175-1181; Vahatalo et al. 2003.
Arch. Hydrobiol. 156: 287-314). Dessa forma, o
aumento na produção bacteriana em função da
foto-degradação indica não somente que a fotodegradação forma compostos orgânicos mais
lábeis para o crescimento bacteriano, mas também
que esta possivelmente promove a liberação de
outros nutrientes que estimulem o crescimento das
células com a mesma quantidade de energia
disponível. Por outro lado, a redução das taxas de
produção bacteriana pode ser explicada pela fotodegradação de moléculas lábeis ou pela formação
de compostos mais complexos e mais refratários,
pelo processo de humificação (Kieber et al. 1997.
Limnol. Oceanogr. 42: 1454-1462). Finalmente,
as alterações na respiração bacteriana em função
da foto-degradação parecem estar apenas
relacionadas a alterações na disponibilidade do
COD (Amado et al. 2006 op. cit.).
As bactérias, por sua vez, ao utilizarem
o carbono como fonte de energia e matéria,
transformam o estoque de MOD presente no
ecossistema aquático. De maneira geral, as
bactérias excretam na água compostos ricos em
anéis aromáticos que promovem um
escurecimento do estoque de MOD, aumentando
a foto-reatividade (Nieto-Cid et al. 2006. Limnol.
Oceanogr. 51: 1391-1400). Dessa forma, pode
existir, em certos ambientes, uma cooperação na
degradação da MOD entre os processos de
degradação biológica e fotoquímica, na qual a
luz forma moléculas mais lábeis para o
crescimento bacteriano e as bactérias
transformam o estoque de MOD liberando
moléculas mais reativas à foto-degradação. Por
outro lado, conforme citado anteriormente, há
moléculas específicas que são simultaneamente
lábeis à degradação microbiana e à fotodegradação. Sendo assim, pode existir em alguns
casos uma relação de competição entre os
processos biológico e fotoquímico pela
degradação de compostos específicos que
compõem a MOD (Cotner & Biddanda 2002 op.
cit.; Obernosterer & Benner 2004. Limnol.
Oceanogr. 49: 117-124).
O balanço final das interações entre a
degradação bacteriana e a foto-degradação está
fortemente ligado à identidade e abundância dos
compostos que formam o estoque de MOD. Por
exemplo, estudos recentes sugerem que a
competição tende a ocorrer em ambientes
produtivos (Amado et al. submetido). Esses
ambientes apresentam assinaturas químicas,
como a presença de aminoácidos ricos em
compostos aromáticos, que indicam uma alta
atividade bacteriana. Esses aminoácidos
(triptofano, histidina, metionina, tirosina e
cisteína), por exemplo, além de serem
importantes fontes de nitrogênio para as
bactérias, são extremamente reativos ao oxigênio
singleto (1O2) (Ronsein et al. 2006. Quim. Nova
29: 563-568), um estado excitado de moléculas
de oxigênio (O2), resultado da interação dos raios
http://www.sblimno.org.br
25
solares com a MOD, sobretudo de origem húmica
(Latch & McNeill 2006. Science 311: 1743-1747).
Dessa forma, em ambientes nos quais esses
aminoácidos, ou outros compostos de
comportamento similar, representam uma grande
fração da MOD, a competição, como balanço final
entre a foto e a bio-degradação, e provável de
ocorrer, culminando com a redução das taxas de
degradação de ambos os processos.
Após uma explosão nos estudos sobre a
foto-degradação da MOD em ecossistemas
aquáticos na década passada, principalmente
relacionada à questão do aumento do buraco da
camada de ozônio, hoje observamos um
decréscimo do número destes estudos
possivelmente associado a um comprovado
processo de recuperação desta camada. Porém,
várias questões permanecem sem resposta e vários
padrões encontrados continuam sem explicação.
Recentemente, o ciclo do carbono têm tido
grande destaque na comunidade científica
mundial, devido ao aumento do Efeito Estufa. Tal
aumento está relacionado ao aumento da
concentração atmosférica de gases biogênicos,
como o CH4 e o CO2. O estoque de MOD presente
nos ecossistemas aquáticos é um dos maiores
estoques de carbono no planeta, superando o
estoque de carbono fixado nas plantas terrestres
e nos organismos marinhos combinados.
Processos que atuem diretamente neste estoque,
liberando CO2 na água e aumentando o grau de
heterotrofia dos ecossistemas aquáticos, são de
grande importância para o entendimento do
balanço de carbono no planeta. Assim, em outra
questão climática de larga de escala, a fotodegradação da MOD volta a ganhar destaque na
comunidade científica, agora acompanhada da
respiração microbiana, como dois dos principais
processos mineralizadores da MOD presente nos
ecossistemas aquáticos.
André Megali Amado
[email protected]
Vinicius Fortes Farjalla
[email protected]
Universidade Federal do Rio de Janeiro
O Papel dos Fungos nos Ecossistemas
Aquáticos
Em praticamente todos os ecossistemas
aquáticos, sejam marinhos ou continentais,
lóticos ou lênticos, preservados ou impactados,
existem fungos, que são microrganismos
predominantemente sapróbios possuidores de
características taxonômicas, ecológicas e
fisiológicas complexas, além de elevada
adaptabilidade ao meio ambiente. Atualmente o
grupo dos fungos possui representantes em três
diferentes reinos: Protozoa, Chromista
(Straminipila) e Fungi (Kirk et al. 2001.
Ainsworth & Bisby´s Dictionary of the Fungi,
CAB International, 655p.).
Basicamente podem ser encontrados dois
grupos de fungos nos ecossistemas aquáticos:
os fungos zoospóricos e os fungos “nãozoospóricos”. Os zoospóricos são organismos
que possuem estruturas especializadas para a
mobilidade, as quais são extremamente
importantes para sua dispersão. De maneira
contrária, os “não-zoospóricos” são desprovidos
de estruturas locomotoras, porém apresentam
modificações morfológicas e metabólicas que
conferem adaptabilidade para a vida aquática
(Schoenlein-Crusius & Malosso 2007. Em
Deshmukh & Rai (eds), Fungi – multifaceted
Microbes, Anamaya Publishers, 321p.).
Os fungos zoospóricos, com
representantes nos três reinos, são
microrganismos de origem polifilética, providos
de esporos flagelados denominados zoósporos,
quando assexuados e, planogametas, quando
sexuados. São dependentes da água em pelo
menos algum momento do ciclo de vida, já que
muitos necessitam viver inteiramente em corpos
d’água, e outros podem habitar ambientes
terrestres, nos quais podem ser encontrados sob
a forma de resistência (Moore-Landecker 1996.
Fundamental of the fungi, Prentice-Hall Inc.,
574p.). Esses microrganismos possuem elevada
resistência às condições hidrológicas adversas,
como amplas variações de pH, temperatura,
saturação de oxigênio e eutrofização. Eles
apresentam ampla distribuição geográfica e são
capazes de colonizar substratos celulósicos,
quitinosos e queratinosos presentes nos diversos
ambientes aquáticos, com algumas espécies
parasitas de macrófitas, algas planctônicas e
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
26
bentônicas, peixes, anfíbios, crustáceos, outros
fungos e, até mesmo do homem (Alexopoulos et
al. 1996. Introductory mycology, John Wiley &
Sons Inc., 869p.). A essas potencialidades somamse a tolerância e a capacidade biossortiva de
alguns metais, como o cobre, chumbo e zinco,
evidenciadas em linhagens procedentes de locais
contaminados (Souza 2006. Mucorales de solo
contaminado com metais pesados na região do
pólo cerâmico de Santa Gertrudes, SP:
ocorrência e capacidade de biossorção de
chumbo e zinco, Tese de Doutorado, UNESP, Rio
Claro, 221p.).
A micota não-zoospórica é composta por
representantes de Ascomycota, Basidiomycota,
leveduras, Hyphomycetes aquáticos e fungos de
origem terrestre denominados geofungos, com
diferentes proporções de acordo com as condições
do ambiente aquático (Schoenlein-Crusius et al.
2004. Em Bicudo & Bicudo (eds), Amostragem
em limnologia, Editora RiMa, São Carlos, p.179191).
Os fungos, cuja reprodução é somente
possível com a presença da água, como no caso
dos fungos zoospóricos, dos Hyphomycetes
aquáticos e de algumas espécies de Ascomycota
e Basidiomycota, têm sido considerados
verdadeiramente aquáticos. Entretanto, relatos
destes fungos em substratos terrestres, ou seja,
em condições adversas no que se refere à
disponibilidade de água, dificultam a
circunscrição deste conceito (Schoenlein-Crusius
& Grandi 2003. Braz. J. Microbiol. 34: 183-193).
Os Hyphomycetes aquáticos são fungos
anamórfos, isto é, apresentam reprodução
assexuada, com formação de conídios em
estruturas diferenciadas e semelhantes às
estruturas somáticas, denominadas conidióforos
que ficam livres no micélio (Ingold 1975. An
illustrated guide to aquatic and water-borne
Hyphomycetes (Fungi Imperfecti) with notes on
their biology, Freshwater Biological Association,
96p.). Os conídios apresentam formas
hidrodinâmicas, sigmóides ou tetrarradiadas,
leves, o que permite flutuação e rápida fixação
em novos substratos. As condições hidrológicas,
como teor de oxigênio, temperatura e turbulência,
têm grande influência sobre o desenvolvimento e
distribuição das espécies. Devido às
peculiaridades das formas de seus conídios, os
Hyphomycetes aquáticos são denominados fungos
tetrarradiados e ingoldeanos, em homenagem ao
Prof. Dr. C. T. Ingold, que os estudou
intensivamente após a década de 40 (Bärlocher
1992. The ecology of aquatic Hyphomycetes,
Springer Verlag, 225p.).
A importância dos fungos para os
diversos ecossistemas é amplamente
reconhecida, já que estes são importantes
organismos decompositores de matéria orgânica
e possuem grande capacidade de degradação de
resíduos compostos por carbono e nitrogênio,
como açúcares simples, celulose, hemicelulose,
lignina, pectinas, proteínas (quitina e queratina),
além de xilano, ácidos húmicos, entre outras
substâncias Em um dos estudos pioneiros sobre
a importância dos fungos nos ambientes
aquáticos, Kaushik & Hynes (1968. J. Ecol. 56:
229-543) verificaram acréscimos de proteínas
acima de 11% em folhas colonizadas por fungos,
baixas concentrações em folhas colonizadas
apenas por bactérias e em folhas tratadas com
antifúngicos.
Posteriormente diversos estudos
comprovaram que, da matéria orgânica
particulada total que é incorporada aos ambientes
aquáticos, apenas uma pequena parte é
prontamente utilizada pelos detritívoros, sendo
necessária prévia colonização destas por
microrganismos, principalmente pelos fungos,
que as tornam mais palatáveis e iniciam
transformações de grande quantidade de estoque
de energia em formas mais prontamente aceitas
por esses organismos (Bärlocher 1992 op cit.).
A decomposição de folhedo e a atividade
dos fungos podem afetar as concentrações de
nutrientes na água dos ecossistemas. Também é
fato conhecido que os microrganismos
associados aos substratos submersos em
decomposição podem obter nitrogênio e fósforo
diretamente da água (Suberkropp 1995. Can. J.
Bot. 73: 1361-1369).
Uma vez estabelecido o papel desses
fungos na cadeia trófica, os estudos abrangeram
a investigação da riqueza das espécies, potencial
de produção de unidades reprodutivas e,
sobretudo, a produção de biomassa, como
expressão da intensidade de colonização dos
substratos. Entretanto, os fungos são organismos
de difícil quantificação por conta do tipo de
associação existente entre a biomassa fúngica e
os substratos, no qual o micélio fúngico
http://www.sblimno.org.br
27
encontra-se imerso, dificultando a separação e
quantificação (Newell 1992. Em Carrol &
Wicklow (eds), The fungal community: its
organization and role in the ecosystem, Marcel
Dekker, p.521-561). Para se entender a cadeia de
detritos e a decomposição de serapilheira, tanto
em ambientes aquáticos como terrestres, tornase necessário conhecer os mecanismos
decompositores e as interações microbianas que
resultam na ciclagem de nutrientes, adicionando
informações quanto à importância desses
microrganismos no processamento da matéria
orgânica e na dinâmica trófica dos diferentes
ecossistemas (Gessner & Chauvet 1997. Limnol.
Oceanogr. 42 (3): 496-505).
Suberkropp (1997. Freshwater Biol. 38:
169-178) estudou a contribuição fúngica durante
a decomposição de folhas submersas e encontrou
relação significativa entre a quantidade de
esporos, biomassa (ergosterol) e de folhas
encontradas no ambiente aquático, verificando
decréscimo da diversidade das espécies e da
biomassa fúngica nas folhas durante o verão,
como conseqüência da menor quantidade de
matéria orgânica disponível. Em trabalho
realizado por Gessner & Chauvet (1997 op cit.),
foram verificadas elevações crescentes nas
concentrações de biomassa fúngica durante a
decomposição, semelhante ao ocorrido no lago
hipereutrófico estudado por Moreira (2006.
Avaliação da diversidade e biomassa de Fungos
associados a folhas em decomposição de
Tibouchina pulchra Cogn. submersas em
reservatórios de Parque Estadual das Fontes do
Ipiranga (PEFI), São Paulo, SP. Dissertação de
Mestrado, Instituto de Botânica, São Paulo, 111p.)
e aos resultados obtidos por Kuehn et al (1999.
Microb. Ecol. 38: 50-57) em estudo sobre
decomposição de macrófitas aquáticas).
Nos países de clima temperado, os
Hyphomycetes
aquáticos
apresentam
comportamento sazonal bem definido, ocorrendo
predominantemente no final do verão e outono,
quando a senescência das folhas é maior do que
em outras épocas (Bärlocher 1992 op cit.). Nos
estudos em regiões de clima tropical (Sridhar &
Kaveriappa 1989. Nova Hedwigia 49(3-4): 455467), incluindo os estudos Brasileiros
(Schoenlein-Crusius & Malosso 2007 op cit.), a
ocorrência dos Hyphomycetes aquáticos parece
estar mais associada ao estágio de decomposição
das folhas, em especial aos teores de nutrientes
do que a sazonalidade expressa por variações
climáticas das épocas secas e chuvosas.
Apesar dos Hyphomycetes aquáticos
serem associados a ambientes lóticos bem
aerados e com águas moderadamente turbulentas
(Bärlocher 1992 op cit.), a presença de diversas
espécies têm sido reportada em ambientes
altamente poluídos por efluentes orgânicos (Au
et al. 1992. Can. J. Bot. 70: 1071-1079) e metais
pesados, sendo encontrados também em águas
subterrâneas contaminadas por cobre (Krauss et
al. 2005. Arch. Hydrobiol. 162(3): 417-429).
Nos estudos realizados em sistemas
lóticos de regiões de clima temperado, os fungos
zoospóricos foram considerados colonizadores
primários de persistência reduzida nos
substratos, ao contrário dos geofungos, que,
dependendo da tolerância às condições do
ambiente aquático, poderiam permanecer por
mais tempo. Como a diversidade, atividade e
produção de biomassa dos Hyphomycetes
aquáticos suplantaram a dos outros fungos, a
estes foi atribuído o papel principal na
decomposição dos substratos vegetais submersas
(Bärlocher 1992 op cit.). Deve-se salientar, no
entanto, que a metodologia utilizada nesses
estudos permitiu somente a observação de alguns
representantes de Oomycetes (Achlya,
Saprolegnia, Pythium, entre outros), pois não foi
utilizada a técnica de iscagem múltipla (Milanez
1989. Em Fidalgo & Bononi (eds), Técnicas de
coleta, preservação e herborização de material
botânico. Instituto de Botânica, p. 17-20), a qual
possibilita o isolamento de Chytridiomycetes e
Hyphochytriomycetes.
No entanto, em estudos conduzidos na
Mata Atlântica de Paranapiacaba, SP (PiresZottarelli et al. 1993. Rev. Microbiol. 24 (3): 192197; Schoenlein-Crusius & Milanez 1998. Rev.
Bras. Bot. 21(1): 73-79), verificou-se que a
associação das técnicas de iscagem múltipla,
observação direta e a utilização de meios de
cultura tornou possível a observação e o
isolamento de Hyphomycetes aquáticos,
geofungos e elevada diversidade de fungos
zoospóricos, havendo entre estes o predomínio
de quitridiomicetos, com Polychytrium
aggregatum Ajello ocorrendo abundantemente
diretamente no interior das folhas, confirmando
a importância dos fungos zoospóricos na
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
28
decomposição de substratos submersos. Esta
observação também tem sido corroborada pela
utilização de técnicas moleculares, que revelaram
a existência de elevada diversidade de fungos no
folhedo submerso, entre os quais os zoospóricos
fizeram-se presentes de forma bastante expressiva
(Nikolcheva & Bärlocher 2004. Mycol. Progress
3(1): 41-49).
Outro grande grupo de fungos presentes
nos ambientes aquáticos são as leveduras, que
constituem um grupo polifilético de fungos
ascomicetos e basidiomicetos, caracterizadas por
possuir uma fase de crescimento unicelular e um
estágio sexual que não está relacionado à
produção de corpos de frutificação.
Aproximadamente 100 gêneros e 700 espécies são
conhecidas. Estes fungos são cosmopolitas e
podem ser encontrados em ambientes aquáticos
e terrestres. Muitos são sapróbios, sendo
importantes decompositores de material vegetal
ou animal. Embora a maioria das espécies seja
decompositora, algumas são parasitas para as
plantas, animais e para os homens (Kurtzman &
Fell 2004. Em Muller et al. (eds), Biodiversity of
Fungi: inventory and monitoring methods,
Elsevier Academic Press, p.337-342).
As leveduras aquáticas, também
caracterizadas como fungos nativos do ambiente
aquático, possuem crescimento rápido por
brotamento e elevada produção de enzimas
capazes de degradar variados tipos de substratos.
Algumas são sensíveis às variações ambientais,
como presença de poluentes; outras mais
resistentes, assemelhando-se aos fungos
cosmopolitas terrestres (Schoenlein-Crusius &
Milanez 1996. Em Bicudo & Menezes (eds),
Biodiversity in Brazil, a first approach, p. 31-48).
São classificadas de acordo com o ciclo de
reprodução e por meio de certas características
morfológicas e fisiológicas, como ausência de
produção de esporos formados por reprodução
sexuada, método de brotamento do esporo,
produção de urease, morfologia química e
ultraestrutural da parede celular, assimilação de
carboidratos e fermentação (Alexopoulos et al.
1996 op cit.).
No Brasil, estudos sobre leveduras podem
ser diferenciados como de importância médica
(Paula et al. 1983. Rev. Microbiol. 14(2): 136143) e ambiental. A diversidade de leveduras em
ambientes aquáticos e terrestres, bem como em
frutos, demonstram heterogeneidade na
composição das comunidades quando
comparados os diferentes ecossistemas (Rosa et
al. 1995. Hydrobiologia 308: 103-108). O estudo
de diversidade e distribuição de leveduras em
ambientes aquáticos indica que hábitats
diferentes podem apresentar comunidades de
leveduras características, revelando diversos
biótipos diferentes que provavelmente
representam novas espécies (Hagler et al. 1995.
Oecologia 1: 225-244).
A influência do grau de trofia dos
sistemas aquáticos sobre a diversidade dos
fungos é bastante complexa. Com base nos
resultados citados na literatura, Bärlocher (1992
op cit.) considera que a adição de poluição
orgânica pode modificar a abundância de
espécies fúngicas individuais, retardando a
decomposição de folhas e outros substratos. O
autor afirma, ainda, que para avaliar a extensão
dos efeitos antrópicos, que podem acarretar uma
expressiva redução da quantidade de espécies, a
situação ideal é que se conheça a diversidade da
micota nativa antes da ocorrência do impacto,
podendo-se prever com maior precisão quais as
conseqüências para o ecossistema e quais as
alternativas para recuperá-lo.
Diante do comportamento bastante
plástico dos fungos frente aos fatores de stress,
Niyogi et al. (2002. Ecosystems 5: 554-567)
postularam que a diversidade da micota
geralmente é sensível ao stress baixo a moderado,
enquanto a biomassa da comunidade e as funções
da mesma no ecossistema podem permanecer
estáveis ou até apresentam um enriquecimento,
decrescendo somente quando o nível de stress
for consideravelmente elevado. Entretanto, a
remoção ou a diminuição do stress, podem tornar
a comunidade vulnerável à invasão por outras
espécies, antes limitadas pelas condições do
ambiente. Esta hipótese explica porque a
velocidade de decomposição de folhas submersas
em águas poluídas pode permanecer inalterada
enquanto a diversidade dos fungos apresenta
declínio significativo e porque acréscimos de
fósforo e nitrogênio em águas lóticas podem
elevar a concentração de conídios de
Hyphomycetes aquáticos, redefinindo a
diversidade da micota (Gulis & Suberkropp
2004. Mycologia 96(1): 57-65).
http://www.sblimno.org.br
29
De maneira geral, os fungos aquáticos
apresentam tolerância e elevada adaptabilidade a
condições adversas do ambiente, sendo que a
especificidade de substrato e o comportamento
sazonal são mais flexíveis do que no caso de
outros microrganismos. Entretanto, verifica-se
que cada espécie possui potencialidades inerentes
que se combinam no ambiente de forma
sinergética, como no caso da decomposição de
folhedo submerso, realizada pela ação conjunta
de detritívoros, bactérias e fungos (Hieber &
Gessner 2002. Ecology 83(4): 1026-1038).
Relacionar espécies com os ecossistemas
tem sido um dos maiores desafios para a ecologia
contemporânea (Cardinale et al. 2000. Oikos 91:
175-183). Uma das maneiras de fazê-lo consiste
em analisar os efeitos dos atributos da comunidade
como riqueza ou diversidade sobre algum
processo relevante como produção primária,
decomposição e mineralização de folhedo
(Emerson et al. 2001. Nature 411: 73-77) e a
quantificação do papel individual das espécies ou
da comunidade.
Estima-se que nos trópicos há
aproximadamente 300 espécies de Hyphomycetes
aquáticos observados em águas continentais,
distribuídas em 143 gêneros e 280 espécies (Goh
1997. Em Hyde (ed), Biodiversity of tropical
microfungi, Hong Kong University Press, p.189227). Na América do Sul, os Hyphomycetes
aquáticos são em torno de 90 espécies, com
predomínio no Brasil devido a maior quantidade
de estudos ecológicos e taxonômicos do grupo
(Schoenlein-Crusius & Grandi 2003. Braz. J.
Microbiol. 34: 183-193). No caso dos fungos
zoospóricos, se conhece 328 táxons no Brasil, o
que corresponde a somente 18% dos táxons
atualmente conhecidos por literatura.
As estimativas mostram que ainda há
muitas espécies de fungos aquáticos zoospóricos
ou não a serem descobertas para a Ciência, e que
a compreensão do papel de cada uma nas águas
ainda está longe de ser totalmente elucidada.
Iracema Helena Schoenlein-Crusius
[email protected]
Instituto de Botânica, Secretaria de Estado de
Meio Ambiente
Carolina Gasch Moreira
[email protected]
Universidade Federal de Santo Amaro
Carla Lídia Amorim Pires-Zottarelli
[email protected]
Instituto de Botânica, Secretaria de Estado de
Meio Ambiente
Importância, Desafios e Perspectivas no
Estudo da Ecologia Aquática
Microbiana no Brasil
Introdução
A compreensão do funcionamento de um
ecossistema aquático passa pelas relações e
fluxos de energia entre os produtores,
consumidores e decompositores. Pensando neste
termo e em ecossistemas aquáticos, existem
ecossistemas onde os produtores, consumidores
e decompositores são todos microrganismos, mas
não existe ecossistema terrestre ou aquático sem
a presença destes. Refletindo sobre a afirmativa
de Reynolds (1984, The ecology of freshwater
phytoplankton, Cambridge University Press, 384
pp) que foi salientada pela Profa. Vera Huzsar
(2006, BSBL 35: 91-93), penso que se “a
importância do fitoplâncton esta fora de
questão...”, como definir a importância das
bactérias, vírus e outros microrganismos em
qualquer ecossistema? O objetivo deste texto é
ressaltar a importância do estudo sobre
microrganismos em geral e o papel destes no
funcionamento de ecossistemas aquáticos
continentais, bem como sobre os desafios e as
perspectivas da nova área do conhecimento
Ecologia Aquática Microbiana no Brasil e no
mundo.
História
Os estudos em microbiologia ecológica
vem se intensificando sobremaneira nas últimas
décadas. Este avanço é atribuído principalmente
ao avanço de técnicas que possibilitaram
demonstrar e quantificar a importância dos
microrganismos em ecossistemas aquáticos ou
terrestres. Embora muitos pesquisadores sempre
apontassem para a importância dos
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
30
microrganismos em ambientes aquáticos, a
ausência da disponibilidade de metodologias que
demonstrassem esta importância fazia com que
o estudo desta comunidade fosse negligenciado,
por exemplo, em relação ao fito e zooplâncton.
Pomeroy (1974. Bioscience 24: 499-504) discutiu
a importância dos microrganismos em teias
tróficas marinhas, e Hobbie et al. (1977. Appl.
environ. microbiol. 33: 1225-1228) quantificou
a densidade de bactérias na água. Segundo Duarte
et al. (1997. Aquat.Microb. Ecol. 13: 101-111),
o artigo de Hobbie et al. é o mais citado em
Ecologia Aquática Microbiana. Os resultados
obtidos em estudos posteriores resultaram na
publicação do artigo “The role of water-column
microbes in the sea” (Azam et al. 1983. Mar. Ecol.
Prog. Ser. 10: 257-263), que cunhou o termo
“microbial loop”, um marco no estudo da
Fig 1: Árvore filogenética universal demonstrando os 3 domínios e os
diferentes ramos de cada um. Notar a reduzida representatividade em
termos de diversidade genética dos organismos macroscópicos em
relação aos outros Eucarya. Adaptado de Pace (1997. Science 276:734740).
Ecologia Aquática Microbiana. Após a
publicação deste artigo, o papel dos
microrganismos nos ambientes aquáticos passou
a ser mais estudada, e sua importância relativa
cada vez mais confirmada. Embora este termo
tenha sido cunhado dentro de um contexto
aquático, na verdade este fenômeno também
descreve o fluxo de energia da maioria dos
ecossistemas terrestres, pois a maior fração de
sua produção primária é degradada por
microrganismos.
Outro importante artigo foi o de Carl Woese
(1987. Microbiol. Rev. 51: 221-271), que
modificou não somente a visão da microbiologia,
mas de toda a Biologia. Baseado em nas
seqüências de rRNA, Woese (1987 op. cit.) e
Woese et al. (1990. Proc. Natl. Acad. Sci. 87:
4576-4579) propuseram uma árvore filogenética
com a divisão de 3 domínios: Archaea, Bacteria
e Eucarya. Desde então, esta árvore filogenética
vem sendo melhor estudada, vários trabalhos
sobre relações filogenéticas, com base molecular,
de integrantes destes domínios vem sendo
realizados, e a importância relativa da diversidade
de microrganismos em relação aos organismos
eucariotos (Eucarya) tem ficado cada vez mais
evidente (Pace 1997. Science 276: 734-740; Fig.
1).
Importância geral dos microrganismos
Atualmente reconhece-se o papel chave dos
microrganismos no funcionamento de qualquer
ecossistema. Podemos encontrar ecossistemas
sem a presença de eucariotos, mas nunca sem
procariotos (Archaea e Bacteria). No entanto, a
importância dos microrganismos não se restringe
ao seu atual papel nos ecossistemas.
Em todas as considerações sobre a origem
da vida no nosso planeta, não se questiona o fato
do ancestral comum ter sido um microrganismo.
Durante anos, debateu-se se este ancestral seria
um organismo heterótrofo, fotoautotrófo ou
quimioautotrófo, e hoje, a hipótese mais aceita,
é de que caso a vida tenha surgido neste Planeta,
ela ocorreu quimioautotroficamente.
A evolução dos ciclos biogeoquímicos,
logo após o surgimento da vida no Planeta,
também foi determinada pela ação de
microrganismos. O próprio acúmulo de oxigênio
na atmosfera que se iniciou há cerca de 2 bilhões
de anos é atribuído às cianobactérias.
A diversidade de microrganismos também
é muito superior a de todos os eucariotos juntos
(Fig. 1). Estimativas sugerem que o número de
espécies de insetos, que durante muito tempo foi
considerado o grupo mais diverso deste Planeta,
pode chegar a 30 milhões. A estimativa atual da
diversidade de microrganismos em nosso Planeta
chega a 1 bilhão de espécies!!! E deve-se
considerar que, para a obtenção destas
estimativas, assume-se como espécie organismos
com menos de 97% de homologia de 16S rRNA
(ou 70% de homologia de todo o genoma). Como
foi mencionado anteriormente nesta edição por
Farjalla e Guariento, se o mesmo critério fosse
http://www.sblimno.org.br
31
aplicado para macroorganismos, lêmures,
chimpanzés, gorilas e seres humanos seriam
considerados uma única espécie.
Nos
ambientes
aquáticos,
os
microrganismos participam da produção de
matéria orgânica através das produções foto
(energia solar) e quimiosintetizante (energia
oriunda da oxidação de compostos reduzidos),
além de atuar como consumidores. Embora
organismos superiores tenham participação ativa
na cadeia detritívora, o papel dos microrganismos
no fluxo de energia sobrepõe o destes organismos
superiores em até duas ordens de magnitude. A
degradação de matéria orgânica realizada pelos
microrganismos é essencial para redisponibilização dos nutrientes aos produtores
primários. Também foi demonstrado que a
produção bacteriana é o principal condutor entre
os ambientes terrestre e aquático, pois a matéria
orgânica dissolvida de origem terrestre pode ser
incorporada em biomassa bacteriana e
posteriormente ser transferida à cadeia trófica via
o “microbial loop”.
Desafios na área da Ecologia Microbiana
Um dos principais objetivos da Ecologia
Microbiana é combinar a identificação e atividade
de microrganismos nos ecossistemas. As
perguntas “Que bactérias estão lá?” e “O que elas
esta fazendo?” tem movido ecólogos microbianos
há muitas décadas. Os avanços metodológicos
obtidos nos últimos 20 anos têm auxiliado nas
respostas a estas perguntas, pois possibilitaram
uma maior compreensão do mundo
microbiológico. Na verdade, “mundo
microbiológico” não é nem de longe um termo
apropriado. Para se ter idéia dos desafios da
Ecologia Microbiana, o número de bactérias em
nosso planeta é maior do que o número de estrelas
em nosso universo (Curtis 2007. Environ.
Microbiol. 9(1): 1).
Várias questões básicas, como a
distribuição espacial e temporal dos
microrganismos, sua biogeografia e sua
biodiversidade funcional, ainda estão longe de
terem resposta (Dubilier 2007. Environ.
Microbiol. 9: 2-3). Embora na biologia o conceito
de espécie, às vezes, seja colocado em discussão,
zoólogos e botânicos conseguem diferenciar e
classificar espécies. O conceito de espécie para
microrganismos é ainda mais obscuro em virtude
do percentual de homologia do genoma ser
totalmente arbitrário (99%). Além disso, entre
microrganismos ocorre transferência horizontal
de genes, o que dificulta ainda mais uma
definição clara de espécie.
Apesar
dos
enormes
avanços
metodológicos nos últimos anos, as ferramentas
atualmente utilizadas ainda estão longe de ser as
ideais. Um exemplo é o trabalho realizado por
Sogin et al. (2006. Proc. Natl. Acad. Sci. 103:
12115-12120), que, utilizando uma nova
metodologia, demonstrou que os estudos até
então subestimavam a diversidade de
microrganismos marinhos em até duas ordens de
grandeza.
Novos métodos vêm sendo desenvolvidos
e novos equipamentos são disponibilizados no
mercado, possibilitando avanços ainda maiores
nesta área. Enquanto um seqüenciador
tradicionalmente utilizado nos laboratórios de
microbiologia seqüencia cerca de 70kbp de DNA
por análise, os mais modernos podem produzir
cerca de 30Mbp, e existem perspectivas de
equipamentos que produzam 1Gbp. Uma nova
metodologia
denominada
nanoSIMS
(“Secondary Ion Mass Spectrometry”), que
combina a técnica de FISH (DeLucca e César,
nesta edição) com a espectrometria de massa de
íons secundários, possibilita a determinação da
composição química, radio-isotópica ou de
isótopos estáveis em níveis individuais e
submicrométricos (p. ex. uma única bactéria).
Kuypers e Jorgensen (2007. Environ. Microbiol.
9: 6-7) consideram que, em virtude da elevada
sensibilidade e resolução espacial desta técnica,
será possível, em um futuro próximo, identificar
e definir a função metabólica de indivíduos em
estudos realizados em campo. É opinião de
muitos especialistas que o desenvolvimento de
novas tecnologias possibilitará o obtenção de
respostas em nível celular e no ambiente onde o
microrganismo se desenvolve.
Atualmente, o maior desafio a ser superado
é a proposta de novas teorias. Prevê-se que, nos
próximos 2-3 anos, milhares de genomas serão
seqüenciados, milhares de espécies de
microrganismos serão descritas, mas muitas
vezes sem o embasamento teórico. Jim Prosser e
colaboradores publicaram um excelente artigo
que merece ser lido não somente por limnólogos,
mas por biólogos de todas as áreas (Prosser et al.
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
32
6
artigos.ano
-1
5
4
750
Número publicações
Panorama da Ecologia Aquática Microbiana
no Brasil
O número de trabalhos produzidos na área
de Ecologia Aquática Microbiana no Brasil ainda
é muito reduzido. Thomaz (2006. BSBL 35: 2327) realizou uma pesquisa no na base Thomson
ISI (Thomson Scientific) com as palavras
“macrophyte” e “Brasil or Brazil”, e demonstrou
que o número de publicações no estudo de
macrófitas
aquáticas
aumentou
consideravelmente, e que nos últimos 20 anos o
fatores que justifiquem esta baixa quantidade de
artigos publicados nesta área no Brasil.
Uma comparação entre o número total e o
número de artigos na área de Microbiologia
Aquática, entre os periódicos Limnology &
Oceanography (L&O), Hydrobiologia e Acta
Limnologica Brasiliensia (Acta) nos últimos 20
anos, mostra que o número total de trabalhos
publicados nas duas primeiras revistas é mais
elevado do que na Acta (Figs. 2A e 2C). Este
resultado certamente pode ser atribuído ao fato
da L&O e Hydrobiologia serem revistas
internacionais, que recebem uma maior
quantidade de submissões de pesquisadores de
todo mundo, além é da maior disponibilidade de
recursos financeiros. A L&O, por exemplo,
publica majoritariamente artigos de
pesquisadores americanos, sendo os Estados
Unidos o país que mais investe em Ciência e
A
500
250
0
25
Microrganismos (%)
2007. Nature Rev. Microbiol. 5: 384-392).
Segundo estes autores, a Ecologia Microbiana
sofre uma revolução no momento, com um rápido
acúmulo de dados que, no entanto, estão sendo
obtidos sem a aplicação de teorias que
possibilitem a organização e favoreçam o
aumento do poder preditivo dos mesmos. O
avanço da Ecologia Microbiana, baseada no
desenvolvimento de novas tecnologias e não em
um avanço teórico, levará esta nova área do
conhecimento a um avanço cego, custoso
economicamente e não-generalista. Segundo Jim
Prosser (com. pess.), embora os estudos de
“genomics, transcriptomics, proteomics e
metabolomics” sejam muito importantes, o que
realmente está faltando nesta nova ciência
emergente é a proposta de novas teorias, ou
“theoromics”.
B
20
15
10
5
3
0
2
50
0
1985
1990
1995
2000
2005
2010
Fig 2: Número de trabalhos (total = 23) encontrados na base Thomson
ISI utilizando-se “bacterioplankton*” no campo “topic” e “Brasil or
Brazil” no campo “address”, entre os anos de 1986 e 2006.
número total de artigos publicados foi de 151.
Repeti esta análise com as palavras
“bacterioplankton” e “Brasil or Brazil” e obtive
um padrão relativamente parecido, porém com
um número total bem mais reduzido de 23
trabalhos. O desenvolvimento recente de técnicas
para o estudo de comunidades de microrganismos
em ecossistemas aquáticos pode ser um dos
Microrganismos (n)
1
40
30
C
LO
Hydro
Acta
20
10
0
1985
1990
1995
2000
2005
Ano
Fig 3: Avaliação dos artigos publicados nos periódicos Limnology &
Oceanography (LO); Hydrobiologia (Hydro) e Acta Limnologica
Brasiliensia (Acta) entre 1986 e 2006. Número total de artigos publicados
por ano (A); Percentual de artigos publicados sobre microrganismos
(B) e Número de artigos publicados sobre microrganismos (C).
http://www.sblimno.org.br
33
Tecnologia e que tem a maior produção científica
mundial.
No entanto, uma comparação entre o
percentual de trabalhos publicados na área da
Ecologia Aquática Microbiana entre as 3 revistas
(Fig. 2B) mostra que na L&O houve um aumento
de 4 % no final da década de 80 para 17% no
início deste século. Na Hydrobiologia e na Acta,
este percentual passou de cerca de 1 % do final
da década de 80 para cerca de 4% no início deste
século. Este resultado sugere que, em termos
percentuais, nossa produção na área de Ecologia
Aquática Microbiana está bem próxima a
Hydrobiologia. No entanto, este percentual é
cerca de 4 vezes menor do que publicado pela
L&O.
Uma comparação entre o percentual dos
trabalhos sobre diferentes comunidades
aquáticas, encontrados na Revista Brasileira de
Biologia (RBB), Acta, Hydrobiologia e L&O de
2003 a 2006, e nas apresentações do IX
Congresso Brasileiro de Limnologia (IX CBL,
2003, Juiz de Fora) indica uma dominância das
seguintes comunidades (Fig. 3):
Acta - macroinv. > zooplâncton > peixes;
RBB - peixes >> macróf. aquát. > zooplâncton e
IX CBL - macróf. aquát. > fitoplâncton >
zooplâncton
Este resultado contrasta um pouco com a
dominância dos estudos de fito, zoo e
ictioplâncton observado para Hydrobiologia e
L&O, mas demonstra claramente que o número
40
de pesquisas sobre Ecologia Aquática Microbiana
no Brasil precisa aumentar, para que esta área
tão importante possa enriquecer a Limnologia
Brasileira.
Desafios no Brasil
Antes de iniciar este artigo, reli os trabalhos
de Thomaz (2006. BSBL 35: 23-28), Nessimian
& e Ferreira-JR (2006. BSBL 35(2): 54-56) e
Huzsar (2006. BSBL 35(3): 91-93) e percebi que
os desafios para o estudo das comunidades de
macrófitas aquáticas, de macroinvertebrados
bentônicos e de fitoplânton, em nosso país, não
são pequenos, o que provavelmente ocorre em
muitas outras áreas também. No entanto, percebi
que a situação é ainda mais crítica quanto ao
conhecimento sobre a Ecologia Aquática
Microbiana no Brasil. Embora muito ainda
precise ser feito em todas as áreas, os padrões
limnológicos de várias comunidades como de
fito, zôo e ictioplâncton, de macrófitas aquáticas
e de macroinvertebrados bentônicos nos
ecossistemas aquáticos brasileiros são
relativamente compreendidas. O conhecimento
existente sobre a ecologia de microrganismos
aquáticos é muito incipiente, defasado e muitas
vezes inexistente se comparado com outras
comunidades.
Segundo Thomaz (2006 op. cit.) e vários
pesquisadores brasileiros por ele citados, a
ausência de produção de conhecimento novo
(novas teorias) é um dos principais problemas
49
63
P u b lic a ç õ e s X C o m u n id a d e s
Percentual
30
L&O
H y d ro
A c ta
RBB
IX CBL
20
10
pe
ix
es
m
ac
ró
fit
as
pe
rif
íto
n
m
ac
ro
in
v.
ba
ct
er
io
pl
.
o
fit
zo
o
0
Fig 4: Percentual de artigos publicados nos periódicos Limnology & Oceanography (L&O); Hydrobiologia (Hidrob); Acta Limnologica Brasiliensia
(Acta); Revista Brasileira de Biologia (RBB) entre 2003 e 2006 e resumos do IX Congresso Brasileiro de Limnologia ocorrido em Juiz de Fora
em 2003 (IX CBL). Foram consideradas as comunidades de zooplâncton (zoo); fitoplâncton (fito); bacterioplâncton (bacteriopl.); macroinvertebrados
bentônicos (macroinv.); perifíton; macrófitas aquáticas e peixes. Notar que o valor máximo de cada categoria encontra-se ao lado da barra,
quando o percentual ultrapassa 40%.
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)
34
no estudo, não somente da limnologia, mas
sobretudo da Ecologia Brasileira. Embora
consigamos desenvolver de forma competente
vários estudos realizados em países que lideram
o cenário científico internacional, estes são
muitas vezes uma repetição.
No entanto, como a necessidade do
desenvolvimento de novas teorias nesta área não
é um “privilégio” brasileiro, mas sim um
obstáculo a ser superado por todos os cientistas
que se dedicam ao estudo de microrganismos,
nossa distância dos países desenvolvidos não é
tão grande. Segundo Prosser et al. (2007 op. cit.),
um dos principais obstáculos à elaboração de
novas teorias em ecologia microbiana é a falta
de ferramentas e disciplinas da ecologia teórica,
que deveriam fazer parte da formação da maioria
dos microbiólogos. Considerando que a maioria
dos limnólogos brasileiros tem sua formação
básica em ecologia, acredito que tenhamos as
mesmas condições de desenvolver novas teorias
nesta nova área do conhecimento.
Em minha opinião, há muito o que se fazer
e propor em termos de pesquisas para o futuro
da Ecologia Aquática Microbiana no Brasil. A
maioria dos trabalhos em Ecologia Aquática
Microbiana refere-se à ambientes temperados.
Inicialmente temos que olhar e tentar interpretar
os resultados já disponíveis na literatura, usando
nosso conhecimento para propor teorias à luz de
resultados obtidos em ambientes temperados.
Podemos repetir estudos de outras regiões em
nossos ecossistemas com o objetivo de testar
hipóteses, discutir e comparar os resultados. A
quantidade de informação e de dados na literatura
é cada vez maior, e a necessidade de se extrair
padrões destes dados é premente não somente no
Brasil, mas em todo o mundo. Estudos que
descrevam os padrões ecológicos e
microbiológicos em nossos ecossistemas
aquáticos certamente são necessários. Estas
investigações devem ir além das repetições e
comparações de padrões, elas devem ser
inovadoras e aplicadas ao conhecimento dos
nosso ambientes tropicais. Por último, precisamos
de uma mudança em relação aos equipamentos
já existentes nas instituições de pesquisa. A
dificuldade em se conseguir financiamento de
órgãos públicos e privados para aquisição de
equipamentos é enorme, e a falta de pessoal
técnico especializado para o uso e a manutenção
desses equipamentos é um sério problema em
nossas instituições de pesquisa. Como
conseqüência, os equipamentos conseguidos
arduamente pelos pesquisadores para seus
laboratórios muitas vezes têm seu acesso a outros
pesquisadores dificultado ou mesmo negado.
Como conseqüência, provoca-se uma corrida por
recursos para aquisição de equipamentos muitas
vezes já existentes na mesma instituição, fato que
por princípio deveria ser considerado inaceitável
em um país com tantas carências como o nosso.
Como os equipamentos para se estudar Ecologia
Aquática Microbiana são onerosos e de cara
manutenção, seria fundamental que
pesquisadores unissem esforços não somente para
a obtenção, mas também para o uso conjunto e
colaboração. Esta união de esforços iria facilitar
o desenvolvimento de pesquisas, intercâmbio de
aprendizados e idéias, contribuindo para o avanço
da ciência brasileira.
Finalizo este artigo com as inspiradoras
palavras de E. O. Wilson (1997. O Naturalista,
368p.), muito bem citadas por Farjalla (2003.
Fontes de matéria orgânica dissolvida (MOD) e
seu consumo pelas bactérias planctônicas em
lagoas costeiras e em um lago amazônico, Tese
de doutorado, PPGE, UFRJ, 240p.):
“Se eu pudesse começar tudo de novo e reviver
minha visão no século XXI, seria um ecólogo
microbiano. Dez bilhões de bactérias vivem em
um grama de solo comum, uma simples pitada
mantida entre o indicador e o polegar. Elas
representam milhares de espécies, ignoradas pela
ciência em sua esmagadora maioria. Abriria meu
caminho por florestas clônicas que se estendem
por grãos de areia, viajaria em um submarino
imaginário pelo meio de gotas d’água que
equivalem ao tamanho de lagoas e rastrearia
predadores e presas a fim de descobrir novos
sistemas de vida e estranhas redes alimentares.
Para tudo isso não precisaria aventurar-me a mais
de dez passos do prédio do meu laboratório. As
onças, formigas e orquídeas ainda ocupariam
florestas distantes em todo o seu esplendor, mas
a elas se juntaria agora um mundo vivo
virtualmente sem fim, ainda mais estranho e
muitíssimo mais complexo, com possibilidades
infinitas...”
Alex Enrich-Prast
[email protected]
Universidade Federal do Rio de Janeiro
http://www.sblimno.org.br
35
Boletim da Sociedade Brasileira de Limnologia - nº 36 (1)