VARIABILIDADE GENÉTICA DE ACESSOS E CULTIVARES
DE MELANCIA BASEADA EM MARCADORES RAPD
Ricarte, A. O (1); Albuquerque, L. B.(1); Antonio, R. P.(2); Silveira, L. M.(3);
Carvalho, K. K. A.(1); Silva, M. F. N (1); Filho, A. J. R. S (1).
[email protected]
(1)
Laboratório de Biotecnologia Molecular Vegetal. Universidade Federal Rural do
Semi-Árido - UFERSA, Mossoró – RN;
(2)
Empresa Brasileira de Pesquisa Agropecuária – Embrapa Semi-Árido Mossoró –
RN;
(3)
Laboratório de Recursos Genéticos Vegetal. Universidade Federal Rural do SemiÁrido - UFERSA, Mossoró – RN.
RESUMO
Parte da produção de melancia realizada no Nordeste brasileiro é feita principalmente
por pequenos produtores, os quais cultivam diversos tipos de frutos. Desta forma,
conduziu-se este trabalho com o objetivo de caracterizar a variabilidade genética entre
24 acessos pertencentes à coleção de germoplasma de melancia da UFERSA e duas
cultivares comerciais, por meio de marcadores RAPD. As reações foram feitas com
base em 26 amostras de DNA, utilizando-se 10 primers RAPD. Foi amplificado um
total de 1679 bandas, sendo 706 polimórficas. Com a obtenção do dendrograma, a um
corte de 60%, houve a formação de 10 grupos distintos. Dois acessos (M22 e M24) e
as duas cultivares utilizadas nas análises ficaram englobados em um mesmo grupo. Os
acessos de melancia da coleção pertencente à UFERSA possuem alta diversidade para
ser explorada em programas de melhoramento genético para esta espécie.
Palavras-chave: Diversidade Genética, Marcadores Moleculares, Germoplasma.
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
INTRODUÇÃO
Parte da produção de melancia cultivada no Nordeste brasileiro é feita
basicamente por pequenos produtores que utilizam suas próprias
sementes e fazem a própria seleção de frutos, utilizando as sementes
para o cultivo no ano seguinte. Desta forma, ano após ano uma seleção
continuada destes genótipos é realizada, sendo cultivados diversos tipos
de frutos (Ramos; Queiroz, 2005).
Para impedir que ocorra a erosão genética destes materiais, torna-se
necessário a realização de coletas e a caracterização dos mesmos a fim
de se conhecer o grau de variabilidade genética que estes genótipos
possuem. Isto deve ser realizado com o intuito de se indicar genótipos
mais promissores para os trabalhos de melhoramento e também para
melhorar as condições de cultivo e produção dos agricultores,
permitindo o uso racional destes genótipos na agricultura familiar.
Uma das formas mais eficientes de se caracterizar a diversidade
genética entre diferentes genótipos consiste no uso de marcadores
moleculares, os quais se baseiam no DNA das plantas (Vidal et al.,
2005).
Para caracterização da diversidade, há atualmente vários marcadores
moleculares disponíveis, sendo os que usam a técnica de PCR
2
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
(Polymerase Chain Reaction), como os RAPD (Random Amplification
of Polymorphic), um dos mais utilizados por ser uma técnica simples,
rápida, de baixo custo e aplicável a qualquer tipo de organismo, que se
caracteriza (Grattapaglia, 2007). Além disso, não é necessário
conhecimento prévio sobre o genoma da espécie avaliada e os primers
utilizados são aleatórios com 10 pares de bases de extensão (Vidal et
al., 2005).
Marcadores RAPD têm sido aplicados com sucesso na caracterização da
diversidade genética de acessos de melancia (Silva et al., 2006). No
Brasil, têm sido aplicados na diversidade genética de algumas coleções
de germoplasma (Faleiro et al., 2001; Conte et al., 2006).
No presente trabalho, objetivou-se, caracterizar a variabilidade genética
entre vinte e quatro acessos e duas cultivares de melancia da coleção de
germoplasma da UFERSA, por meio de marcadores RAPD.
MATERIAL E MÉTODOS
Sementes de 26 genótipos de melancia, sendo duas cultivares e 24
acessos oriundos da coleção de germoplasma de melancia da
UFERSA,oriundos de diferentes cidades do estado do Rio Grande do
Norte, foram plantados em casa de vegetação e 15 dias após a
3
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
germinação suas folhas foram coletadas para a extração de DNA,
conforme o protocolo descrito por Doyle e Doyle (1990).
A quantificação foi feita em gel de agarose a 0,8% (p/v) comparando-se
visualmente a intensidade das bandas do DNA dos genótipos extraídos,
com bandas de peso molecular conhecido de 1 kb. Cada um dos
genótipos teve o seu material genético diluído à uma concentração de
10 ηg.µL-1.
Para a realização de reações de amplificação com os primers RAPD foi
usado um coquetel preparado para um volume final de 13 µL, contendo
4,2 µL de água ultrapura, 1,3 µL de Buffer (10x), 2,0 µL de MgCl2
(25mM), 1,3 µL de dNTP (2,5mM), 0,2 µL de Taq DNA Polimerase
(5U.µL-1) (Kit da Fermentas Life Sciences/USA), 1,0 µL de primer (5
µM), e 3,0 µL de DNA (10ηg). As reações foram conduzidas em
termociclador Axygen Axygene, cujo programa de ciclagem foi
padronizado para todos os primers RAPD e apresentou a seguinte
programação: pré-desnaturação à 96 °C por 1 minuto, seguido de 40
ciclos à 92 °C por 1 minuto, 40 ºC por 1 minuto e 72 °C por 2 minutos,
com extensão final de 5 minutos à 72 °C com base em (SILVA et al.,
2006), com posterior manutenção da temperatura à 4 °C, até que fossem
submetidas a eletroforese horizontal.
4
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
Os produtos de amplificação foram separados por eletroforese em gel
de agarose a 1,0% (p/v), usando tampão TBE (Tris - Ácido Bórico EDTA) pH 8,0 1X. Os géis foram corados com quatro µL de brometo
de etídeo (10 mg mL-1) e fotodocumentados em transluminador, através
de luz ultravioleta. O padrão de peso molecular de 1 kb (New England
Biolabs/USA) foi utilizado visando comparação com os fragmentos
amplificados. Uma matriz binária baseada na presença (1) ou ausência
(0) de bandas RAPD, foi gerada e utilizada para estimar a similaridade
genética entre os genótipos. A estimativa de similaridade genética entre
cada par de genótipos foi calculada pelo coeficiente de Sorence-Dice.
Os genótipos foram agrupados pelo método UPGMA (Unweighted
Pair-Group Method), obtendo-se dessa forma, um dendrograma. Todas
as análises foram realizadas com o auxílio do programa Genes versão
2013.5.1 (Cruz, 2006)
RESULTADOS E DISCUSSÃO
Foi realizada uma triagem entre 12 primers RAPD testados
inicialmente, dos quais 10 apresentaram bom perfil de amplificação e
destes, nove detectaram e revelaram polimorfismo entre os 26 genótipos
de melancia.
5
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
De acordo com os dados obtidos pela amplificação, o maior número de
bandas foi gerado pelos primeres OPAA 04 e OPO-10, com 11 bandas
cada e o menor número foi gerado pelo primer OPAA 07 com 6 bandas.
A avaliação realizada utilizando o marcador RAPD mostrou que esta é
uma importante ferramenta para estudos de polimorfismo em acessos e
cultivares de melancia. Do total de 1679 bandas amplificadas com os 10
primers, 973 bandas apresentaram-se monomórficas e 706 apresentaram
polimorfismo. Elevados níveis de polimorfismo também foram
encontrados por (Silva et al., 2006) ao utilizarem seis primers RAPD
em genótipos de melancia, sendo 42 acessos e uma cultivar.
Todos os 10 primers selecionados pela triagem apresentaram bom perfil
de amplificação permitindo a identificação de bandas consistentemente
polimórficas. Devido ao excelente padrão apresentado, apenas as
bandas mais intensas foram analisadas (Figura 1).
6
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
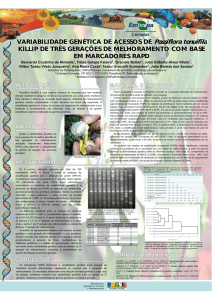
Figura 1. Gel de agarose a 1%, contendo fragmentos de
DNA amplificados com o marcador RAPD OPAA 03, a
partir do DNA de 24 acessos e 2 cultivares de melancia.
Mossoró – RN, UFERSA, 2013.
Os resultados de diferenciação das amostras avaliadas dos 26 genótipos
de melancia foram analisados, a partir de distâncias genéticas derivadas
dos perfis de amplificação gerados pelos primers RAPD.
A análise de agrupamento pelo método UPGMA com base nesses
marcadores, distribuiu os acessos e as duas cultivares em 10 grupos a
um corte de 60% (Figura 2).
7
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
Figura 2. Análise de agrupamento dos 26 genótipos de melancia por meio do
marcador RAPD, obtida pelo método de agrupamento UPGMA, utilizando o índice de
Dice. Mossoró-RN, UFERSA, 2013.
O grupo I foi constituído pelos acessos M1 e M7. O grupo II foi
formado pelos acessos M6, M17, M3 e M16. Já os grupos III e IV
englobaram apenas um único acesso cada, os quais foram M9 e M11
respectivamente, este resultado demonstra que estes genótipos são
8
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
geneticamente diferentes de todos os demais. O grupo V foi formado
pelos acessos M18, M23, M12 e M20, enquanto que o grupo VI foi
constituído pelos acessos M2 e M15. Já os grupos MVII e MVIII foram
formados pelos acessos M3, M13 e M5; M10, M19 E M8
respectivamente, enquanto que o grupo IX englobou os acessos M14 e
M21. O último grupo formado, ou seja, o grupo X englobou as duas
cultivares (Crimson Sweet e Olímpia), bem como os acesso M22 e
M24, vale salientar que de acordo com o dendrograma o acesso M22 e a
cultivar Olímpia partilham muitos genes idênticos assim como, o acesso
M24
e
a
cultivar
Crimson,
sendo
provavelmente
materiais
geneticamente iguais.
CONCLUSÃO
Os marcadores RAPD foram satisfatórios em permitir a detecção de
polimorfismo entre acessos e cultivares de melancia. Existe
variabilidade genética entre os genótipos pertencentes a coleção de
acessos de melancia da UFERSA. Os acessos da coleção de
germoplasma de melancia da UFERSA possuem alta diversidade para
ser explorada em programas de melhoramento.
9
Resumos Expandidos do I CONICBIO / II CONABIO / VI SIMCBIO (v.2)
Universidade Católica de Pernambuco - Recife - PE - Brasil - 11 a 14 de novembro de 2013
REFERÊNCIAS
CONTE, R.; REIS, M.S.; VENCOVSKY, R. Effects of management on the genetic
structure of Euterpe edulis Mart. Populations based on microsatellites. Scientia
Forestalis, Piracicaba, v.72, p.81-88, 2006.
CRUZ, C.D. Programa Genes: Biometria. Editora UFV. Viçosa (MG). 382p. 2006
DOYLE, I.J.; DOYLE, J.L. Isolation of plant from fresh tissue. Focus, Rockville,
v.12, p.13-15, 1990.
GRATTAPAGLIA, D. Aplicações operacionais de marcadores moleculares. In:
BORÉM, A. (Ed). Biotecnologia florestal. Viçosa, MG: UFV, 2007. P.175-200.
FLEIRO, F.G.; LOPES, U.V.; YAMADA, M.M.; PIRES, J.L.; BAHIA, R.C.S.;
SANTOS, R.C.; GOMES, L.M.C.; ARAÚJO, I.S.; FALEIRO, A.S.G.; GRAMACHO,
K.P.; MELO, G.R.P.; MONTEIRO, W.R.; VALE, R.R. Caracterização de variedades
clonais de Theobroma cação L com base em marcadores RAPD, AFLP e
microssatélites. Agrotrópica, Salvador, v.1, p. 86-88, 2001.
SILVA, M.L.; QUEIROZ, M.A.; FERREIRA, M.A.F.da F.; BUSO, G.S.C.
Caracterização morfológica de acessos de melancia. Horticultura Brasileira. Recife.
v.24, p.405-409, 2006.
VIDAL, R. A.; LAMEGO, F. P.; NUNES, A. L.; Otimização do número de primers
empregados em RAPD para detectar variabilidade genética entre acessos de picãopreto. Scientia Agrária. Curitiba, v.6, p71-77, 2005.
RAMOS SRR; QUEIROZ MA. 2005. Recursos genéticos de abóbora no Nordeste
brasileiro.In: MOURA MCCL. Recursos genéticos de hortaliças:
riquezas
naturais.São Luís: Instituto Interamericano de Cooperação para a Agricultura, 99-116.
10