ESTUDO DO PAPEL DE GRÂNULOS DE RNA E DE EXOSSOMOS NA
INTERAÇÃO PATÓGENO-HOSPEDEIRO
PROJETO APRESENTADO À FUNDAÇÃO ARAUCÁRIA – EDITAL PRONEX
ROTEIRO DESCRITIVO
1 – Sobre o Núcleo de Excelência
a- Descrição do Núcleo e sua origem:
O Núcleo é formado por pesquisadores do Instituto Carlos Chagas (FiocruzParaná), pesquisadores da Universidade Estadual de Londrina (UEL) e
pesquisadores da Pontifícia Universidade Católica do Paraná (PUCPR). Na
verdade parte deste Núcleo já esteve organizado em outro projeto do Pronex
promovido pela Fundação Araucária e parte do Núcleo colabora intensamente
desde 2005 e integra o Núcleo de Células Tronco e Terapia Celular sediado no
Paraná. Participam ainda do Núcleo, na qualidade de colaboradores, um
pesquisador da Embrapa e dois pesquisadores do Uruguai (vinculados ao
Instituto Pasteur de Montevidéu e ao Instituto Clemente Estabile), todos
vinculados a atividades de análise bioinformática. É importante mencionar que
os projetos acima referenciados propiciaram um forte vínculo entre alguns dos
pesquisadores do ICC e os pesquisadores da UEL e da PUCPR, que perduram
desde então que podem, através deste projeto, aumentar de intensidade. O
foco do Núcleo proposto ao PRONEX é o estudo da interação patógenohospedeiro usando modelos experimentais bem estabelecidos em nossas
instituições, os patógenos Trypanosoma cruzi e vírus da dengue e os modelos
biológicos de células tronco e células dendriticas. O estudo da interação
patógeno-hospedeiro será feito através da análise de exossomos e de grânulos
de RNA, seu papel na imuno-modulação e seu papel na regulação da
expressão gênica tanto dos patógenos como das células hospedeiras. Esta é
uma abordagem original e moderna , e conhecimentos fundamentais serão
adquiridos através deste projeto, propiciando uma excelente oportunidade e
ambiência para a formação de recursos humanos qualificados.
a- Adequação da equipe:
A equipe é adequada pela qualidade de sua formação profissional (como
facilmente comprovado pelos curricula em anexo) e a multi-disciplinaridade
requerida dentro da área de Ciências Biológicas. Tanto é assim que a equipe
domina a grande plêiade de técnicas que serão utilizadas, como comprovado
pela própria produtividade científica dos diferentes componentes do Núcleo. Os
membros do Núcleo são pesquisadores ativos e produtivos e todos com
bastante experiência no desenvolvimento de projetos de pesquisa (como
constatado abaixo pela descrição dos projetos da equipe nos últimos três
anos). A equipe é formada por:
Nome
Função
Instituição
http://lattes.cnpq.br/
Samuel Goldenberg
Coordenador
ICC e IBMP
6917479410354653
Alejandro Correa
Pesquisador
ICC
0502314179930901
Alexandra Senegaglia
Pesquisador
PUC-PR
5130849139417489
Bruno Dallagiovanna
Pesquisador
ICC
5038843270685960
Carmen Rebelatto
Pesquisador
PUC-PR
9258812501826236
Claudia N. Duarte dos Santos
Pesquisador
ICC
4598537010242642
Fabiola Barbieri Holetz
Pesquisador
ICC
6996846049865794
Juliano Bordignon
Pesquisador
ICC
4831386727923397
Lysangela Ronalte Alves
Pesquisador
ICC
2213128430343323
Marco Augusto Stimamiglio
Pesquisador
ICC
2641327012811390
Maurilio José Soares
Pesquisador
ICC
1880268584104636
Nilson Zanchin
Pesquisador
ICC
4325584779249968
Phileno Pinge Filho
Pesquisador
UEL
1339724757657824
Sueli F. Yamada-Ogatta
Pesquisador
UEL
1783272660304122
Francisco Pereira Lobo
Colaborador
Embrapa
9614758933055047
Hugo Naya
Colaborador
Estrangeiro
José Sotelo-Silveira
Colaborador
Alessandra Melo de Aguiar
Tecnologista
Inst. Pasteur
Montevideu
Inst. Clemente
Estable,
Uruguai
ICC
Guilherme Silveira
Doutorando
ICC
0631237520300349
Pryscilla Fanini Wowk
Pós-Doutoranda
ICC
3170471417515903
Sharon de Toledo Martins
Doutoranda
ICC
4000443506010803
Cryscielli Kuligoski
Técnica
ICC/IBMP
4570976955280649
Giovanny A.C. A. Mazzarotto
Tecnologista
ICC
4491426793431771
Estrangeiro
8168088417330193
b- Descrição da infra-estrutura disponível
A maior parte das atividades experimentais serão realizadas no Instituto Carlos
Chagas. O ICC ocupa uma área de 2500 m2 no campus do Tecpar da Cidade
Industrial de Curitiba com perspectiva de expansão da área para quase 4 mil
metros quadrados até o final de 2012 com a reforma de áreas internas e a
construção de um grande laboratório de experimentação animal (500m2), um
almoxarifado central e reforma de áreas internas de expansão. O ICC é dotado
de moderna e excelente infra-estrutura envolvendo cinco laboratórios para
cultivo de bactérias, fungos e leveduras, parasitas, vírus e células tronco
(incluindo um área para transformação com lentivírus), um laboratório de nível
de segurança biológica 3 (NB3), duas salas de equipamentos comuns (com
centrífugas refrigeradas, ultra-centrífugas, ultra-freezers, máquinas de PCR
convencional e em tempo real), equipamentos de fotodocumentação,
cromatógrafos, microscópios de fluorescência e invertidos com câmeras de alta
resolução,
micromanipulador
de
células
e
um
microscópio
confocal.
Recentemente foi aprovado pelo programa CT-Infra apresentado pela Fiocruz a
aquisição de um microscópio eletrônico. O ICC possui ainda três plataformas
tecnológicas, uma para citometria de fluxo (consistindo de um citômetro FACS
Aria II e um citômetro FACS Canto II), uma para seqüenciamento de ácidos
nucléicos (consistindo de um seqüenciador SoLID IV) e uma plataforma de
espectrometria de massas (Orbitrap LQ). O ICC conta ainda com excelente
infra-estrutura
para
processamento
e
armazenamento
de
dados
e
investimentos deverão ser feitos pela Fiocruz ao longo de 2012 para
aperfeiçoamento de tecnologias da informação em geral. Somando-se à infraestrutura em equipamentos, o ICC conta com infra-estrutura em termos de
técnicos e tecnologistas para dar o importante suporte para as atividades de
pesquisa
(preparo
de
materiais,
confecção
de
meios,
operação
de
equipamentos das plataformas, preparação de células e de antisoros
policlonais e monoclonais).
O
Laboratório
de
Biologia
Molecular
de
Microrganismos,
lotado
no
Departamento de Microbiologia do Centro de Ciências Biológicas da
Universidade Estadual de Londrina, possui 45m2 e conta com os seguintes
equipamentos: Microscópio de luz, Fluxo laminar, Incubadora com agitação,
Microcentrífuga,
Centrifuga
refrigerada,
Estufa
bacteriológica,
Balança
analítica, Termociclador, Fontes de eletroforese, Sistema de fotodocumentação
de géis de agarose e poliacrilamida, Sistema de transferência de géis de
poliacrilamida, Máquina de gelo, pH metro, Autoclave, Universal Microplate
Reader ELx 800, Estufa incubadora de CO2, Computadores, Geladeiras e
Freezers (-20 e -80 oC). Ainda estão disponíveis no CCB, Ultracentrífuga,
Sistema de água ultrapura, Sistema de eletroforese em campo de pulsos
alternados, Microscópio eletrônico de transmissão e Biotério de criação e
manipulação que serão úteis no desenvolvimento do projeto.
O Laboratório de Imunopatologia Experimental do CCB da UEL (LIE-CCB-UEL)
é coordenado pelo Dr. Phileno Pinge Filho. O LIE-CCB-UEL possui 45 m2 de
área construída disponibilizará os seguintes equipamentos: Estufa de cultivo
celular,cabine de Fluxo laminar, Microscópio invertido, 2 refrigeradores, 2
congeladores, Balança analítica, estufa de secagem de material, forno para
esterilização de vidraria, 2 computadores de mesa, 2 Microscópios binoculares,
Luminômetro para placas, Centrífuga de tubos com capacidade para 1,5 e 15
ml, não refrigerada, centrífuga para capilares indicadores de hematócrito e
destilador de água de bancada.
O Núcleo de Cardiomioplastia Celular é formado pelo Laboratório Experimental
de Cultivo Celular, onde são desenvolvidos os projetos de pesquisa básica e
pré-clínica, pelo Laboratório de Cirurgia Experimental e pelo Laboratório do
Núcleo de Cardiomioplastia Celular, onde estão sendo desenvolvidos os
projetos clínicos. O Laboratório do Núcleo de Cardiomioplastia Celular está
localizado no segundo andar do Laboratório de Engenharia e Transplante
Celular no Parque Tecnológico do campus da Pontifícia Universidade Católica
do Paraná. Este espaço conta com um sistema de ar condicionado filtrado,
câmara-fria e núcleo de limpeza e esterilização, obedecendo a todas as
normas preconizadas na RDC 210 de Boas Práticas para a Fabricação de
Produtos Biológicos da Agência Nacional de Vigilância Sanitária e as normas
internacionais para o cultivo e expansão de células destinadas ao transplante
em seres humanos. O laboratório possui uma área de 67,61m² que está
dividida da seguinte maneira: 01 sala de recepção do material; 01 sala de
preparo de materiais (classe 100.000); 02 salas de cultivo celular (classe
10.000); 01 almoxarifado e 01 sala administrativa. O laboratório conta com os
seguintes equipamentos: 02 fluxos laminares, 02 incubadoras de CO 2, 03
microscópios, 02 centrífugas refrigeradas, 02 refrigeradores, 02 freezeres, rede
de CO2, 01 container para armazenamento de amostras congeladas, 02
balanças, 01 banho-maria, 01 estufa bacteriológica, 01 pHmetro, 01 citômetro
de fluxo e 01 microscópio de imunofluorescência. O Centro de Tecnologia
Celular tem uma área de 148,22m² com uma estrutura de 04 salas de cultivo
celular (classe 10.000), sendo que cada sala contem 02 estufas de CO 2, 01
fluxo laminar e 01 microscópio; 02 salas de preparo de materiais (classe
100.000), 01 sala de congelamento de
amostras, 01 entrada para
paramentação, 01 saída para desparamentação, 01 almoxarifado, 01 sala
administrativa e 01 sala de apoio e 01 central de esterilização.
A Unidade de Bioinformática do Instituto Pasteur de Montevideo (IPM)
administra conjuntamente com o Grupo de Simulações Biomoleculares do IPM
um cluster BULL de 80 nodos para cálculo. Conta com 15 computadores de
escritório, espaço físico adequado, acesso à rede CLARA, capacidade de
armazenamento e suporte de informação centralizado. O software utilizado é
de acesso livre, contando com licenças de suítes de edição gráfica. Para o
presente projeto se disponibilizarão 15.000 horas nodo/mês em média.
O Laboratório de Neurobiologia Celular y Molecular do Instituto de
Investigaciones Biológicas Clemente Estable possui uma área de 30 m².
Sistema de hibridação para Microarranjos Agilent. Equipamentos de PCR em
Tempo Real. Facilidades para microscopia e análises de estrutura celular
como: Microscópio Confocal FV300 Olympus, Microscópio Eletrônico Jeol e
Fuji Bas-FLA5000 High Resolution Imager. Para a análise do transcriptoma e
resultados de seqüenciamento em larga escala se dispõe de processadores
Tetra Quad com 32 GB de memória e 10 TB de Hard Drive.
c- Listagem dos projetos de pesquisa desenvolvidos pela equipe nos
últimos 3 anos (agência, vigência, recursos e resultados obtidos)
- Vírus da dengue: caracterização genética, identificação de assinaturas
moleculares de virulência e resposta do hospedeiro humano à infecção, CNPq,
Proc. 558369/2008-1, Edital PNPD, R$36.000,00, resp. Samuel Goldenberg.
Esse projeto teve como objetivo buscar uma maior compreensão da patogênese da dengue
através do estudo da interação entre o vírus e seu hospedeiro mamífero. Para tanto foram
utilizadas diferentes abordagens como a caracterização genética de diferentes isolados virais
através de sequenciamento nucleotídico, estudo da resposta do hospedeiro à infecção em
modelos in vivo (modelos murinos) e ex vivo (culturas primárias de células humanas) e
desenvolvimento de ferramentas de genética reversa (clones infeccioso e replicons) para
avaliar o papel de mutações pontuais ou em conjunto na virulência de determinadas cepas
virais. Essa abordagem permitiu que identificássemos a circulação de diferentes genótipos de
DENV-3 no Brasil, com importantes diferenças em termos de patogênese, que podem estar
relacionadas a uma inibição da via de sinalização do IFN-tipo I pelo genótipo III, que se
mostrou mais virulento.
- Análise epigenética de células tronco mesenquimais da medula óssea na
indução adipogênica, CNPq, Proc. 573520/2008-9, Edital Terapia Celular,
R$174.000,00, resp. Samuel Goldenberg.
Investigamos o perfil epigenético que caracteriza a diferenciação adipogênica de célulastronco adultas. Investigamos o efeito da acetilação de histonas no processo de diferenciação
adipogênica de CTMs utilizando o inibidor de histona desacetilases tricostatina A (TSA). As
análises mostraram que o tratamento com TSA interfere na expressão gênica, na diferenciação
adipogênica e no crescimento celular. O tratamento parece ter efeito intrínseco à amostra
biológica (doador), mas de modo geral levou à diminuição da diferenciação adipogênica.
A análise da arquitetura nuclear pela presença de determinadas modificações em histonas
através de imunomarcações em CTMs-MO induzidas à adipogênese mostrou que há
diminuição de H3K4 metilada em células diferenciadas em comparação às células que estão
iniciando o processo de diferenciação
- INCT para Diagnóstico em Saúde Pública, CNPq, Proc. 573791/2008-2, Edital
INCT, R$ 4.792.361,91, Resp. Samuel Goldenberg.
O projeto está em andamento e busca desenvolver novos reagentes e novas plataformas para
o diagnóstico das doenças de diagnóstico compulsório nas transfusões. As instituições
envolvidas são, FIOCRUZ, IBMP, UFPR, UTFPR, UFSC, UFRGS, Tecpar.
- Análise ribonômica e caracterização de complexos mRNP em Trypanosoma
cruzi, CNPq, Proc. 403484/2008-1, Edital Encomendas Papes/FIOCRUZ Papes V, R$ 60.000,00, Resp. Samuel Goldenberg.
Os resultados obtidos levaram ao isolamento dos complexos mRNA-proteínas (mRNPs)
associados aos P-bodies bem como complexos mRNPs não associados a estas estruturas.
Caracterizamos proteínas de mRNPs e mRNAs associados aos P-bodies a o outros complexos
mRNP utilizando micro-arranjo de DNA e sequenciamento de última geração. Entre as
proteínas de mRNP evidenciadas, caracterizamos uma proteína de ligação a RNA com motivo
de dedo de zinco CCCH e os resultados indicam que dependendo do estado fisiológico da
célula diferentes mRNAs encontram-se associados a esta proteína. O conjunto dos resultados
mostra que a mobilização de mRNAs para os grânulos de RNA na forma de mRNPs é
extremamente dinâmico, respondendo rapidamente às alterações de meio a que o parasita é
submetido, portanto indicando que mRNPs não são partículas estáticas para mera estocagem
e degradação de mRNAs mas que devem participar do fluxo e funcionalidade dos mRNAs na
célula.
- Caracterização funcional de complexos mRNP em Trypanosoma cruzi e seu
papel na modulação da expressão gênica do parasita, Proc. 478516/2010-0,
Edital MCT/CNPq 14/2010 - Universal - Faixa C, R$140.000,00, Resp. Samuel
Goldenberg.
Projeto em andamento e visa caracterizar, utilizando análise proteômica e sequenciamento de
ácidos nucleicos em larga escala, proteínas e RNAs associados em complexos mRNP .
- Caracterização funcional de complexos mRNP em Trypanosoma cruzi e seu
papel na modulação da expressão gênica, Proc. 301730/2011-3, Edital
Produtividade em Pesquisa - PQ – 2011, Nível 1A R$ 168.000,00, Resp.
Samuel Goldenberg.
Solicitação de bolsa de produtividade récem aprovada e com inicio em março de 2012. Visa
estudar os componentes proteico e de ácidos nucleicos de mRNPs de T.cruzi.
- Estabelecimento de uma metodologia de indução da diferenciação de células
tronco adultas à cardiomiócitos através do método de cultivo com meio
condicionado por explantes cardíacos humanos para utilização em terapia
celular cardíaca, CNPq, EDITAL - MCT/CNPq 14/2009 - Universal / Edital
MCT/CNPq 14/2009 – Universal, R$ 27.000,00, Resp. Alejandro Correa
Resultados: Foram identificados vários componentes solúveis presentes no meio condicionado
de explantes cardíacos de coração humano, utilizando arranjos de anticorpos e 2DGel/espectrometria de massas. As células tronco adultas quando colocadas a crescer na
presença do meio condicionado aumentaram a taxa de proliferação e expressaram
marcadores típicos de cardiomiócitos.
- Isolamento, caracterização, ativação e diferenciação em linhagens
mesenquimais de pericitos cardíacos humanos, CNPq, Edital Encomendas
Papes/FIOCRUZ - Papes V, R$ 40.000,00, Resp. Alejandro Correa
Resultados: Foi isolada uma população de células do músculo cardíaco humano positivas para
Fosfatase Alcalina, possivelmente pericitos. Estas células mostraram características
imunofenotípicas de células tronco mesenquimais e foram capazes de diferenciar nas três
linhagens mesodérmicas (adipócitos, osteoblastos e condrócitos). A diferenciação desta
população positiva para a Fosfatase Alcalina é mais eficiente que das células-tronco
mesenquimais da medula ou tecido adiposo, pelo menos do ponto de vista molecular.
- Estabelecimento de uma metodologia de indução da diferenciação de células
tronco adultas à cardiomiócitos através do método de cultivo com meio
condicionado e com matriz tecidual para utilização em terapia celular cardíaca,
Fundação Araucária, Projeto de Pesquisa para o SUS: Gestão Compartilhada
em Saúde PPSUS – 2008/2009. Chamada de Projetos 08/2009, R$ 49.786,54,
Resp. Alejandro Correa.
Resultados: Foi padronizado um protocolo para isolamento de matriz extracelular de coração
humano. Neste projeto estão sendo cultivadas células tronco adultas em meio condicionado
de explantes cardíacos de coração humano em combinação com a matriz extracelular. Os
resultados estão sendo analisados.
- Efeito das proteínas 1 e 2 de ligação ao fator de crescimento semelhante à
insulina (IGFBP-1 e 2) sobre a atividade de células-tronco mesenquimais,
CNPq, EDITAL MCT/CNPq N º 14/2010 – Universal, R$ 15.000,00, Resp.
Marco A. Stimamiglio.
Trata-se de projeto em desenvolvimento que tem como objetivos analisar funcionalmente o
papel das proteínas IGFBP-1 e 2 nos processos de proliferação, citoproteção e diferenciação
de células-tronco mesenquimais. Resultados preliminares indicam a possível modulação da
proliferação das células-tronco através da adição de concentrações crescentes das proteínas
IGFBP avaliadas. Por outro lado, não foram observadas até o momento diferenças na indução
da morte celular nos cultivos de células-tronco sob o estímulo destas proteínas.
- Efeito das proteínas IGFBP-1 e 2 sobre a atividade de células-tronco
mesenquimais, Fundação Araucária, Chamada de Projetos n º 14/2009 –
Programa de Apoio à Pesquisa Básica e Aplicada, R$ 20.000,00, Resp. Marco
A. Stimamiglio.
Trata-se de projeto em desenvolvimento, iniciado em julho de 2010, que tem como objetivos
analisar funcionalmente o papel do microambiente tecidual cardíaco e das proteínas IGFBP-1 e
2, presentes neste microambiente, na indução da diferenciação cardiomiogênica de célulastronco mesenquimais humanas. Resultados preliminares indicam a necessidade da associação
de outras proteínas encontradas no meio condicionado pelo cultivo de explantes cardíacos
para a indução da pré-diferenciação destas células-tronco
- Imunobiologia farmacêutica na fase aguda da doença de chagas
experimental: papel dos eicosanóides, Fundação Araucária (Vigência 20072009, R$ R$ 27.561,00), Resp. Phileno Pinge Filho.
Neste projeto demonstramos que a ciclooxigenase e a 5-lipoxigenase estão envolvidas no
controle da carga parasitária e contribuem para o desenvolvimento do estresse oxidativo de
eritrócitos que ocorre na fase aguda da infecção por Trypanosoma cruzi.
- Impacto da biodisponibilidade de óxido nítrico por AINES-NO sobre os níveis
de citocinas pró-inflamatórias na cardiopatia chagásica experimental. CNPq
(2009-2010, R$ 20.000,00). Resp. Phileno Pinge Filho.
Neste projeto demonstramos que a 5-lipoxigenase participa da produção de citocinas, óxido
nítico e expressão da óxido nítrico sintase (iNOS) no tecido cardíaco na fase aguda da infecção
por T. cruzi.
- Mecanismos de infecção por formas tripomastigotas metacíclicas do
Trypanosoma cruzi: participação da enzima óxido nítrico sintase constitutiva
(cNOS) e dos eicosanóides, Auxílio Financeiro da Fundação Araucária (2009,
R$ 37.775,00) Resp. Dr. Phileno Pinge Filho.
- Mecanismos de ativação e invasão de fagócitos mononucleares pelo
protozoário Trypanosoma cruzi: relações entre eicosanóides, óxido nítrico e cAMP, Auxílio Financeiro do CNPq, Edital Universal (2012, R$ 17.800,00),
Resp. Phileno Pinge Filho.
- Interação de células dendríticas humanas e vírus da dengue: papel do
interferon tipo I e da maturação celular. Edital MCT/CNPq 14/2009 - Universal /
Edital MCT/CNPq 14/2009 - Universal - Faixa A – R$ 14.000,00, Resp. Juliano
Bordignon.
Neste projeto foi demonstrado que cepas de DENV3 genótipo III são capazes de inibir a
ativação da via de IFN tipo I o que possibilita sua maior replicação em células dendríticas
(mdDCs), bem como, maior produção de citocinas inflamatórias. Adicionalmente, demonstrouse que a ativação de mdDCs após a infecção protege as células da apoptose.
- Produção, caracterização e aplicabilidade de anticorpos monoclonais contra
cepas de vírus dengue-1, -2 e -3 circulantes no Brasil. Chamada de Projetos
08/2010 Programa de Pesquisa para o SUS: Gestão Compartilhada em Saúde
PPSUS – 2008/2009 – R$ 49.500,00, Reps. Juliano Bordignon.
Foram desenvolvidos hibridomas secretores de anticorpos monoclonais contra cepas de
DENV-1, -2 e -3 circulantes no Brasil. No momento estes anticorpos estão sendo
caracterizados e avaliados quanto sua utilidade para o desenvolvimento de testes
imunoenzimático para diagnósitco da dengue.
- Identificação da atividade anti-dengue e anti-inflamatória de oleos essências.
Chamada de Projetos 14/2009 Programa de Apoio a Pesquisa Básica e
Aplicada. RS 15.900,00, Resp. Juliano Bordignon.
Neste projeto foi desenvolvido um teste ELISA in situ para triagem da atividade
anti-dengue de substâncias isoladas de óleos essencias. A concentração segura dos
compostos citral e timol foram determinadas por ensaios de MTT/vermelho neutro,
e no momento, esta sendo avaliada a atividade anti-dengue dos mesmos pelo teste
de ELISA in situ.
- Identificação e Produção de Marcadores Celulares de Endocitose e Exocitose
em Trypanosoma cruzi (Euglenozoa: Kinetoplastea). CNPq, Edital Universal
CNPq MCT / CNPq 14/2010 (Processo 471928/2010-0 ), Recursos:
R$25.000,00 (Edital Universal, faixa B), Vigência: 01/10/2010 a 01/10/2012
Resp.: Maurilio José Soares
Resultados obtidos, Artigo publicado no tema:
Barboza NR, Cardoso J, Lima CVP, Soares MJ, Gradia DF, Hangai NS, Bahia MT, Lana M,
Krieger MA, Sá RG. Expression profile and subcellular localization of HslV, the proteasome
related protease from Trypanosoma cruzi. Experimental Parasitology, 130: 171-177, 2012.
- Programa Institucional de Iniciação Científica (PIBIC), Fundação Araucária
Convênio Protocolo 18602 Vigência: 01/09/2011 , Valor: R$ 36.000 Resp.
Bruno Dallagiovanna
Resultados: Dez bolsas de iniciação científica, em andamento.
- Caracterização de redes pós-transcricionais de regulação gênica em
Trypanosoma cruzi, CNPq Edital Universal 473828/2009-0, R$ 20.000 ,
03/08/2009- 2011, Resp. Bruno Dallagiovanna
Resultados: Foram identificados os membros da família de proteínas RRM em T. cruzi.
Caracterizamos funcionalmente a proteína TcRBP40 específica de T. cruzi identificando a rede
gênica regulada por ela.
- Mecanismos da regulação pós-transcricionais da auto-renovação em célulastronco mesenquimais humanas. Análise funcional das proteínas Pumilio2 e
DZIP1, Edital MCT/CNPq/CT-Saúde/MS/SCTIE/DECIT nº 17/2008 - Terapia
Celular, Valor: R$ 100.000, 09/2008 – 2010, Resp. Bruno Dallagiovanna
Resultados: Foram identificadas as redes gênicas reguladas por Pumilio2 e DZIP1 e seu papel
no controle da proliferação celular e da diferenciação de MSCs .
- Identificação e caracterização funcional de RNAs associados aos homólogos
de PABP (PABP1-3) em Leishmania, Edital Universal CNPq 14/2009 – Faixa A
Processo número 474052/2009-5, Valor aprovado: R$ 20.000,00, Resp.
Fabíola Barbieri Holetz
Foi possível identificar grupos distintos de mRNAs que se associam às diferentes PABPs.
Verificamos diferenças significativas nos mRNAs que se associam a proteína PABP1 daqueles
que estão associados as proteínas PABP2 e 3 e não observamos diferenças entre os mRNAs
associados as proteínas PABP2 e 3. A proteína PABP1 está associada aos RNAs que
codificam, em sua maioria, proteínas ribossomais, enquanto as proteínas PABP2 e 3 associamse preferencialmente a RNAs que codificam proteínas ligadoras de DNA, entre elas as histonas
e proteína de ligação a minicírculos, além das proteínas enolase, RBP 5 e Hsp70 Estes dados
corroboram a participação da proteína PABP1 no processo de tradução, e a participação das
proteínas PABP2 e 3 no transporte de mRNAs do núcleo ao citoplasma.
- Identificação e caracterização funcional de mRNAs associados aos fatores
eIF4E1-6 de iniciação da traduação em Trypanosoma brucei, FACEPE Processo APQ-0090-2.13/09, R$ 33.000,00, 2009-2011, Resp. Fabíola Barbieri
Holetz.
Identificamos diferenças significativas nos RNAs que se associam aos fatores de início de
tradução em T. brucei, corroborando resultados prévios do grupo, onde se sugere a existência
de dois complexos de iniciação da tradução em tripanosomatídeos: um deles formado pelos
fatores eIF4E3 e eIF4G4, e o outro formado pelos fatores eIF4E4 e eIF4G3, além das demais
proteínas envolvidas no início deste processo.
- Expressão e Análise Molecular e Funcional de Proteínas Relacionadas a
Doenças Genéticas – Agência financiadora: CNPq (479779/2010-4), vigência
12/2010-11/2012. Valor R$ 35.000,00. Responsável: Nilson Zanchin
Resultados obtidos: Caracterização funcional das proteínas humanas NIP7 e FTSJ3 .
- Investigação dos Mecanismos de Síntese e Regulação da Função de
Ribossomos Associados a Doenças Genéticas e Proliferação Celular - Agência
financiadora: CNPq (473551/2008-0), vigência 01/2009-12/2010. Valor R$
30.160,00. Responsável: Nilson Zanchin
Resultados obtidos: Caracterização funcional das proteínas SBDS, Septinas e ISG95 humanas
e do exossomo de RNA de Archaea.
- Análise Funcional de Proteínas Envolvidas em Síntese de Ribossomos e
Associadas a Doenças Genéticas - Agência financiadora: CNPq, 309215/20110, edital produtividade em pesquisa nível 1C, vigência Março 2012-Fevereiro
2016, valor R$ 105.600,00. Responsável: Nilson Zanchin
Resultados obtidos: Caracterização funcional das proteínas SBDS e SPP1 humanas e do
exossomo de RNA de Archaea.
- Estudo da Estrutura de Proteínas Componentes e Reguladoras do Exossomo
de Archaea e de Levedura. Modalidade “Projeto Temático”. Agência
financiadora FAPESP 2010/51842-3 – FAPESP valor R$, 480.000,00, vigência
01/01/2011 a 31/12/2014. Responsável, Carla Columbano de Oliveira,
pesquisador participante: Nilson I. T. Zanchin.
Resultados obtidos: Caracterização funcional do exossomo de RNA de Archaea e de levedura.
- The NIP7 protein is required for accurate pre-rRNA processing in human cells.
Agencia FAPESP - 2011/00309-6 - Vigência, 01/03/11 a 31/08/11. Valor
R$ 5. 259,29. Responsável: Nilson Zanchin
Resultados obtidos: publicação de artigo na revista Nucleic Acids Research.
- Investigação dos Mecanismos de Síntese e Regulação da Função de
Ribossomos Associados a Doenças Genéticas e Proliferação Celular. Agência
financiadora: CNPq 304578/2008-8, edital produtividade em pesquisa nível 1D.
vigência Março 2008-Fevereiro 2011, valor R$ 79.200,00.
Resultados obtidos: Caracterização funcional das proteínas SBDS, Septinas e ISG95 humanas
e do exossomo de RNA de Archaea.
- Caracterização funcional de proteínas envolvidas no controle da expressão
gênica. Auxílio individual regular (FAPESP 06/02083-7), coordenador, vigência:
01/08/2006 – 29/02/2009. Valor R$ R$ 222.471,25 e US$ 57.160,00.
Responsável: Nilson Zanchin
Resultados obtidos: Caracterização funcional das proteínas humanas NIP7, FTSJ3, SBDS,
Septinas e ISG95 humanas e do exossomo de RNA de Archaea.
- Vigilância e caracterização molecular e sorológica de arbovirus circulantes na
Argentina, Brasil, Paraguai, Uruguai e Venezuela, CAPES, R$150.000,00
(bolsas de longa duração, cooperação internacional), Resp. Claudia Nunes
Duarte dos Santos.
- Desenvolvimento de testes imunoenzimáticos utilizando antígenos
recombinantes para detecção de patógenos virais visando certificação sanitária
de camundongos SPF. CNPq Edital Universal 2011, R$85.500,00, Resp.
Claudia Nunes Duarte dos Santos.
Resultados: Desenvolvimento de uma série de imunobiológicos para rápido diagnóstico de
patógenos virais comuns em camundongos de biotério, visando certificação sanitária dos
mesmos.
- Caracterização molecular de hantavírus e seus potenciais reservatórios:
Desenvolvimento de insumos e contribuições para programas de vigilância
epidemiológica no Estado do Paraná. PPSUS-Fundação Araucária, R$
99.945,00, Resp. Claudia Nunes Duarte dos Santos.
Resultados: identificação precisa de áreas de risco para os seres humanos, e na
implementação de estratégias de controle desta zoonose no estado. Ademais, aumentará a
nossa compreensão sobre os aspectos básicos da biologia desses agentes, contribuindo para
a melhoria das técnicas de diagnóstico, e proporcionarão informações úteis para a concepção
de vacinas e antivirais.
- Desenvolvimento e validação de insumos para diagnóstico e monitoramento
de arboviroses no Brasil, Secretaria de Vigilância em Saúde/Ministério da
Saúde, R$ 158.000,00, Resp. Claudia Nunes Duarte dos Santos.
Resultados: Desenvolvimento de insumos para o diagnóstico sorológico de febre amarela,
Chikungunya e Encefalite de Saint Louis em amostras humanas.
- Produção de reagentes para kits de diagnóstico sorológico humano de IgM e
IgG para hantavirose, CGLAB/ Ministério da Saúde, R$ 134.000,00, Resp.
Claudia Nunes Duarte dos Santos.
Resultados: Produção e distribuição de kits HANTEC EIA para o diagnóstico de infecções
agudas e convalescentes por hantavírus em humanos.
Manutenção dos Laboratórios de Referência da Fiocruz integrantes da Rede
Nacional de Laboratórios de Vigilância Ambiental em Saúde, CGLAB –
Ministério da Saúde, R$ 107.619,66, Resp. Claudia Nunes Duarte dos Santos.
-
Resultados: Prestar serviço de referência no diagnóstico de infecções por hantavirus em
amostras humanas e de roedores reservatórios
- Vigilância e caracterização molecular e sorológica de arbovirus circulantes na
Argentina, Brasil, Paraguai, Uruguai e Venezuela, CAPES, R$150.000,00
(bolsas de longa duração, cooperação internacional), Resp. Claudia Nunes
Duarte dos Santos.
- Hantaviroses e Arenaviroses no Brasil: Desenvolvimento de insumos para o
diagnóstico rápido em roedores silvestres através da plataforma de
cromatografia de fluxo lateral, PROBIO II – Banco Mundial, R$355.400,00,
Resp. Claudia Nunes Duarte dos Santos.
Resultados: Desenvolvimento de insumos para o diagnóstico sorológico rápido de hanta e
arenavirus em roedores silvestres através da tecnologia de cromatografia de fluxo lateral.
- Interação vírus dengue e hospedeiro: busca por marcadores moleculares de
virulência e análise da resposta imune antiviral nos hospedeiros vertebrados e
invertebrados, PROCAD-CNPq, R$333.513,21, Resp. Claudia Nunes Duarte
dos Santos.
Resultados: Estudo da interação entre os vírus da dengue e o hospedeiro vertebrado e
invertebrado, o papel dos componentes salivares do mosquito nessa interação, bem como
mapear mutações no genoma viral envolvidas nas diferenças de estimulação da resposta
imune.
- Candida spp. isoladas de mucosa bucal de pacientes portadores de HIV:
determinação dos fatores de virulência, formação de biofilme e sensibilidade
aos antifúngicos, PPSUS – 2008/2009 Fundação Araucária, R$ 81.291,85,
Resp. Sueli Fumie Yamada Ogatta.
Resultados:Neste projeto foram isoladas 123 leveduras do gênero Candida que foram
caracterizadas quanto à sensibilidade aos antifúngicos fluconazol e nistatina, e à expressão de
fatores de virulência. Esses isolados foram utilizados para seleção de substâncias de origem
natural quanto a atividade antifúngica, principalmente antibiofilme. Os resultados geraram uma
dissertação de mestrado e dois trabalhos que serão submetidos para publicação.
- Phytomonas serpens: caracterização do gene que codifica proteina P
ribossomal e sua participação na reatividade antigênica com Trypanosoma
cruzi. Programa de Apoio à Pesquisa Básica e Aplicada, Fundação Araucária,
R$ 24.896,00, Resp. Sueli Fumie Yamada Ogatta.
Resultados: Os resultados parciais deste projeto permitiram a caracterização do gene que
codifica a proteína P ribossomal de Phytomonas serpens.
O uso de células-tronco mesenquimais autólogas da medula óssea no
tratamento de lesões da cartilagem articular. Nº 461/2010/14939 financiado
pela Fundação Araucária. Resp. Alexandra Cristina Senegaglia. Valor:
R$ 17.567,86.
-
Resultados: O projeto encontra-se na sua fase inicial, até o momento foram validadas as
técnicas e selecionados os candidatos em potencial para participar do ensaio clínico. Ainda
este ano pretendemos realizar os isolamento/expansão das células mesenquimais da medula
óssea dos pacientes e proceder com as infusões.
- Avaliação da interface osso/implante em mandíbula de miniporcos irradiados
e o uso de células tronco na osseointegração. Nº 461/2010/15658 financiado
pela Fundação Araucária. Resp.: Carmen Lúcia Kuniyoshi Rebelatto Valor: R$
19.861,00.
Resultados: Neste estudo foi realizada a padronização do isolamento e cultivo das célulastronco mesenquimais de miniporcos BR-1. Esta primeira parte do estudo “Characterization and
in vitro differentiation of mesenchymal stem cells derived from bone marrow of Brazilian
minipig. A protocol for basic research and tissue engineering” foi submetida para publicação no
periódico International Journal of oral Science. Serão realizadas as análises histológicas dos
implantes dentários dos animais que receberam transplante de células-tronco e será avaliado a
influência destas células na osteointegração.
2 – Sobre o Coordenador e Pesquisadores Principais
a- Experiência e competência comprovada e compatíveis com o Projeto;
O coordenador da proposta é pesquisador 1A do CNPq desde 1988 e tem
ampla experiência na coordenação de projetos tendo coordenado projetos
individuais nas diferentes agências de fomento do país (CNPq , PADCT e
FINEP), agências de fomento regionais (FAPERJ e Fundação Araucária), foi
“Pesquisador do nosso Estado” (FAPERJ), coordenou projetos individuais
Internacionais (Organização Mundial da Saúde, três projetos incluindo um
Research
Strengthening
Grant),
coordenou
projetos
de
cooperação
internacional (CAPES-COFECUB, CABIO-CBAB, DAAD, Amsud, PasteurFiocruz, Fiocruz-CNRS, Fiocruz-Inserm), coordenou projetos em rede nacionais
em rede destacando-se dois projetos do Pronex (um nacional no primeiro edital
do CNPq e outro pela Fundação Araucária), um projeto da rede Genoma
Nacional do CNPq e atualmente coordena o INCT Para Diagóstico em Saúde
Pública, um dos dois INCTs instalados no Estado do Paraná.
Os
pesquisadores principais também têm experiência comprovada como pode ser
observado pelo grande número de publicações, orientações e captação de
recursos, sendo alguns bolsistas de produtividade do CNPq (Claudia Nunes
Duarte dos Santos, Bruno Dallagiovanna, Maurilio José Soares, Nilson
Zanchin).
b- Vínculo dos pesquisadores:
Os pesquisadores são tanto vinculados à Fiocruz, seja como servidores do
Sistema Jurídico Único (Samuel Goldenberg, Claudia Nunes Duarte dos
Santos, Bruno Dallagiovanna, Alejandro Correa, Juliano Bordignon, Maurilio
José Soares), servidores terceirizados da Fiocruz (Fabiola Barbieri Holetz) ou
servidores récem concursados aguardando posse (Nilson Zanchin, Lysangela
Ronalte Alves e Marco Augusto Stimamiglio). Dois pesquisadores principais
são professores da Universidade Estadual de Londrina (Sueli Fumie YamadaOgatta e Phileno Pinge Filho), dois pesquisadores são professores da PUCPR
(Carmem Rebelatto, Alexandra Senegaglia) e os pesquisadores colaboradores
são servidores da Embrapa (Francisco Lobo), Instituto Pasteur de Montevideu
(Hugo Naya) e Instituto Clemente Estable do Uruguai (Jose Sotello-Silveira).
c- Qualidade e regularidade da produção científica
Os pesquisadores principais apresentam em seus curricula publicações
regulares em periódicos de impacto reconhecido. Várias destas publicações
envolvem as principais técnicas que serão utilizadas para o desenvolvimento
do presente projeto e os modelos experimentais aqui utilizados (Trypanosoma
cruzi, vírus dengue, células dendriticas, células tronco)
d- Experiência prévia na formação de pesquisadores:
A capacidade dos pesquisadores participantes desta proposta em formar
recursos humanos é inconteste haja vista o grande número de dissertações e
teses orientadas, além de orientações de iniciação científica e pós-doutorado.
É importante mencionar que o projeto descrito nesta proposta tem um grande
potencial para a formação de recursos humanos, dando continuidade a este
importante processo.
e- Experiência de intercâmbio com instituições e pesquisadores do Brasil e
de outros países
Vários dos pesquisadores participantes desta proposta participaram de projetos
e redes de cooperação internacional e algumas destas cooperações ainda
estão em vigência, notadamente com pesquisadores do Instituto Pasteur de
Montevidéu. É nosso objetivo buscarmos fonte de financiamento com a
Universidade de Lund na Suécia em vista de estreitarmos colaboração para o
estudo de microRNAs e seqüenciamento envolvendo a plataforma Ilumina. O
coordenador da presente proposta coordenou vários projetos de cooperação
internacional, organizou cursos internacionais e congressos e workshops
envolvendo a participação de pesquisadores de outros países.
3 – Sobre a Equipe Técnica e de apoio:
a- Qualificação dos técnicos de apoio
Os técnicos são bem treinados e conhecem bem a rotina do trabalho. Grande
parte dos técnicos tem curso superior e três tecnologistas (todos com pósgraduação) ocupam-se da operação e manutenção dos equipamentos mais
sofisticados (os citômetros Facs Aria e Canto, o seqüenciador SoLID IV e o
espectrômetro de massas Orbitrap funcionam como plataformas tecnológicas e
têm técnicos que auxiliam na sua operação) . É importante ressaltar que como
a maior parte das técnicas estão bem estabelecidas, as mesmas são de
domínio do pessoal técnico.
b- Nível e fonte de financiamento dos estudantes e estagiários
Os estudantes e estagiários são financiados com bolsas do CNPq, CAPES,
Fundação Araucária e Fundação Oswaldo Cruz. Tanto o ICC como a UEL e a
PUCPR têm cursos de pós-graduação e atividades de PIBIC em suas
respectivas sedes e por consequência disponibilidade de bolsas.
c- Perfil de pessoal a ser eventualmente recrutado para o Núcleo
O pessoal a ser recrutado são estudantes nos diferentes níveis (iniciação
científica, pós-graduação e pós-doutorado) e o recrutamento será feito de
acordo com a dinâmica do projeto. Vale ressaltar que já existe o envolvimento
de estudantes e estagiários no projeto, mas em vista da riqueza e da
potencialidade desta proposta outros ingressos realizar-se-ão ao longo dos 36
meses de execução.
4- Sobre o conteúdo do projeto:
PROJETO: ESTUDO DO PAPEL DE GRÂNULOS DE RNA E DE
EXOSSOMOS NA INTERAÇÃO PATÓGENO-HOSPEDEIRO
a- Coerência temática, foco e articulação das atividades propostas
As atividades de pesquisa propostas serão centradas nos modelos biológicos
já utilizados pela equipe nos seus estudos mas novas perguntas serão
abordadas neste projeto. O Trypanosoma cruzi tem sido estudado pela equipe
há quase 30 anos (mais de 85 publicações), havendo o interesse no estudo
dos mecanismos de diferenciação deste parasita e nos mecanismos de
regulação da expressão gênica. Mais recentemente foram realizados estudos
relativos à caracterização de grânulos de RNA e proteínas associadas na
modulação da expressão gênica em T. cruzi. Os conhecimentos adquiridos
com os estudos de grânulos de RNA e a percepção da ubiquidade desses
grânulos na regulação da expressão gênica em eucariotos leva-nos ao estudo
dos mesmos em células tronco, outro importante modelo de estudo por
membros da equipe. A proposta aqui apresentada é bastante original pois
embora existam vários relatos na literatura sobre grânulos de RNA, nada existe
sobre os mesmos nas células tronco e de que maneira regulam o programa de
diferenciação destas células. De maneira análoga, estudos com vírus dengue
(mais de 15 publicações) demonstram a proficiência da equipe com este
modelo biológico e um dos aspectos originais desta proposta é a realização de
estudos referentes à presença e papel de grânulos de RNA utilizando este
modelo biológico.
Atividades de pesquisa envolvendo exossomos são um componente novo nas
atividades
da
equipe,
mas
resultados
preliminares
já
levaram
ao
estabelecimento da metodologia para isolamento e análise dos exossomos (o
projeto de tese da doutoranda Sharon Martins Toledo trata do estudo de
exossomos em células dendriticas infectadas e não infectadas com vírus
dengue). O conjunto de atividades, estudos de caracterização biológica e
funcional de grânulos de RNA e de exossomos em células tronco e células
dendriticas, infectadas ou não com os patógenos T.cruzi e vírus dengue,
permitirá a geração de novos conhecimentos para a melhor compreensão dos
mecanismos de patogênese e de regulação da expressão gênica nos modelos
estudados.
b- Estado atual de conhecimentos no domínio da pesquisa
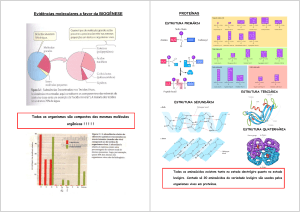
1 - Grânulos de RNA
Nas células eucarióticas, a regulação da expressão gênica ocorre em
diferentes níveis e os mecanismos pós-transcricionais têm um papel crucial no
desenvolvimento, diferenciação e sinalização celular. A biogênese e a
funcionalidade de uma determinada molécula de RNA mensageiro (mRNA)
depende da ação de um conjunto de proteínas ligadoras de RNA (RNA binding
proteins) que participam de todas as etapas de processamento do transcrito
desde o capeamento, passando pelo splicing, a poliadenilação, a exportação
nuclear, a associação com ribossomos e em última etapa a degradação.
Proteínas específicas ligam-se às moléculas de mRNA e a dinâmica da
interação está diretamente associada à funcionalidade dos mRNAs na célula. A
combinação única de fatores que acompanham um mRNA específico, bem
como suas posições relativas neste transcrito, determinam o destino deste
mRNA no citoplasma, ou seja, tradução, estocagem para posterior tradução ou
degradação (Moore, 2005). Em células de mamíferos, os RNAs que não estão
sendo traduzidos, ou aqueles que estão destinados para a degradação, são
compartimentalizados em estruturas citoplasmáticas distintas denominadas
genericamente de “Grânulos de RNA” que podem ser classificados em
Processing bodies (P-bodies) e grânulos de estresse (GS), em função da
presença de proteínas específicas, e têm papel fundamental na regulação póstranscricional da expressão gênica. Os grânulos de estresse e P-bodies são
estruturas
citoplasmáticas
dinâmicas
que
participam
ativamente
no
metabolismo de RNA determinando o destino de mRNAs não envolvidos na
maquinaria de tradução, seja mantendo-os estabilizados, seja levando-os às
vias
de
degradação.
Estas
partículas
por
serem
espacialmente
e
funcionalmente ligadas, apresentam semelhanças, por exemplo, ambas são
induzidas por estresse e contém mRNAs não traduzidos provenientes da
dissociação dos polissomos (Kedersha & Anderson, 2002; Sheth & Parker,
2003; Teixeira et al., 2005; Holetz et al., 2007). Entretanto, estes grânulos
também diferem em vários aspectos, entre eles podemos ressaltar que
somente os P-bodies são observados em células em fase exponencial de
crescimento e em células não submetidas a estresse. Além disso, o estresse
induzido pela fosforilação do fator de tradução eIF2α afeta somente a formação
dos grânulos de estresse e não tem efeito sobre os P-bodies . De forma geral,
os grânulos de estresse são definidos pela presença de fatores de iniciação da
tradução enquanto os P-bodies são definidos pela presença de componentes
da maquinaria de degradação. Vários estudos, utilizando diferentes modelos,
como por exemplo, células de mamíferos, leveduras e protozoários, têm
revelado que muitas proteínas e RNAs com funções celulares distintas estão
integrados com os grânulos de RNA, indicando que as funções destas
estruturas vão além da triagem de mRNAs entre a tradução e a degradação
(Anderson & Kedersha, 2006; Anderson & Kedersha, 2007; Holetz et al., 2010).
Resultados recentes e ainda não publicados por membros de nosso núcleo
demonstraram que em células tronco adultas ocorre um aumento significativo
de grânulos que contém a proteína TIA-1 (marcadora de grânulos de estresse),
quando comparadas com as células diferenciadas, mesmo na ausência de
estresse. Este dado é mais um indicativo da diversidade de atuação e da
importância destas estruturas na modulação da expressão gênica, ressaltando
a necessidade de se ampliar os estudos acerca da dinâmica de formação e da
composição destes grânulos em diferentes tipos celulares, como também frente
a diferentes situações biológicas enfrentadas pelas células.
Adicionalmente, alguns estudos indicam que proteínas de grânulos RNA
podem favorecer ou limitar uma infecção viral e que determinados vírus
interagem com estas proteínas utilizando diferentes mecanismos para
manipular a montagem destas estruturas na célula hospedeira (Li et al., 2002).
Por exemplo, o vírus West Nile (WNV) codifica um RNA contendo uma
estrutura em forma de stem-loop que interage com a proteína TIAR, uma
proteína presente nos grânulos de estresse (Anderson & Kedersha, 2007; Li et
al., 2002). Durante a infecção, TIAR é seqüestrada em grânulos de replicação
viral e a montagem dos grânulos de RNA é inibida na célula hospedeira (Emara
& Brinton, 2007). Neste mesmo estudo, foi demonstrada uma redução do
número de P-bodies
nas células infectadas com WNV e com o vírus da
Dengue, sendo postulado que a interferência dos flavivírus na montagem dos
grânulos de RNA beneficia a replicação viral por impedir a repressão
traducional das proteínas da célula hospedeira, através da inibição da
formação dos grânulos de estresse, e por proteger o RNA viral da degradação,
através da redução do número de P-bodies . Em contraste com os flavivirus, o
poliovirus e o SFV (Semliki Forest Virus) interagem com proteínas dos grânulos
de estresse utilizando estas estruturas para favorecer a repressão traducional
das proteínas celulares e a replicação viral (McInerney et al ., 2005). Estes
estudos demonstraram que além das proteínas e RNAs virais, proteínas de
defesa antiviral da célula hospedeira também se acumulam nestes grânulos.
Mediante o exposto, fica evidente que os grânulos de RNA presentes nas
células hospedeiras, não atuam somente no controle da degradação,
estocagem ou tradução de seus próprios mRNAs, mas participam ativamente e
seletivamente no mecanismo de defesa da célula frente a uma situação
patológica.
Com base nos dados publicados na literatura, pode-se especular que
grânulos de RNA estejam envolvidos na modulação da resposta imunológica
em virtude de uma infecção viral. Neste caso, não é novidade que existe uma
extensa modulação de genes da célula hospedeira. Resultados recentes do
nosso grupo demonstraram que a infecção do sistema nervoso central (SNC)
de camundongos por cepas neuroadaptadas de DENV-1 induz a modulação de
genes da imunidade inata com atividade antiviral estimuladas por IFN-I (ISGs).
No entanto, esta resposta antiviral não é suficiente para conter a infecção, e os
animais desenvolvem encefalite e morrem dentro de duas semanas. Os
principais genes modulados são: genes da família OAS (Oligoadenilato
sintetase, que funciona como uma RNAseL degradando RNA no citoplasma);
família de genes de resistência a Mixovírus (Mx1 e 2), IFIT, Usp18, MHC-I e II
(apresentação de antígenos), entre outros. Além dos genes com atividade
antiviral observam-se também genes inflamatórios, como as quimiocinas
quimioatrativas, Ccl4, Ccl5, Cxcl10, Ccl9, entre outros (Bordignon et. al., 2008).
Da mesma forma, os experimentos realizados ex vivo utilizando células
dendríticas derivadas de monócitos humanos (mdDCs), confirmaram a extensa
modulação de genes da resposta antiviral estimulada por IFN-I, receptores de
imunidade inata (toll like), e apresentação de antígenos. Alguns genes superexpressos na infecção por DENV-3 são: APOBEC, IFIT, OAS, MX, STAT1 e 2,
CCL8, CXCL10, ISG15 e 20, HERC, e os suprimidos são: CD177, CCL3,
CD180, CLEC6A (receptor envolvido na patogênese da dengue), entre outros
(Silveira et al., 2011). Neste caso novamente foi observado que a superexpressão de genes com atividade antiviral não foi capaz de conter a infecção
e o mecanismo de evasão viral por DENV-3 em mdDCs permanece
desconhecido. Sabe-se que alguns flavivirus bloqueiam a sinalização por
receptor de reconhecimento de padrão (PRRs), e desta forma, escapam do
sistema imune.
Recentemente, foram isoladas por um dos laboratórios do núcleo, duas
cepas de DENV-3, uma do genótipo III e outra do V, apresentando
comportamentos bastante distintos nestas células, o do genótipo III (mais
patogênico) inibe a via de sinalização de IFN-I por impedir a fosforilação da
STAT1, ao contrário do genótipo V, menos patogênico, que ativa a via, e
modula a expressão de muitos genes, não se replicando de forma eficiente.
Este exemplo corrobora os dados obtidos recentemente pelo grupo em
camundongos nocaute para IFN-I, que mostram que a resposta de IFN-I é vital
para o controle da infecção por dengue. No entanto, o que faz com que
algumas cepas bloqueiem esta via ou ainda, ativem esta via, mas escapem da
resposta por ela induzida, permanece obscuro, o que estimula a investigação
do envolvimento de grânulos de RNA durante a resposta do hospedeiro.
Embora existam estudos demonstrando a interação de proteínas e RNAs
virais com proteínas de P-bodies e grânulos de estresse, nada se conhece
acerca da dinâmica de formação e a composição destes grânulos em células
de mamíferos infectadas com outros organismos patogênicos.
Esta proposta científica é inédita e propomos investigar como os grânulos de
RNA presentes nas células humanas poderiam modular a expressão gênica e
interagir com moléculas virais, e proteínas celulares da resposta imune inata,
influenciando o mecanismo de defesa das células frente a uma situação
patológica. Neste estudo serão utilizadas como modelo células apresentadoras
de antígenos (células dendríticas) e células tronco humanas infectadas com
diferentes patógenos. A inclusão de células tronco deve-se ao fato de que a
geração de conhecimento para a caracterização funcional de células tronco é
primordial no avanço das pesquisas visando à aplicação terapêutica destas
células. Os patógenos utilizados para a infecção das células serão o
protozoário T. cruzi e o vírus da Dengue, como explicitado anteriormente.
2 – Exossomos
Exossomos são pequenas vesículas contidas por membrana (30 a
100nm) e de origem endocítica que as células eucarióticas secretam no meio
extracelular. Foram inicialmente descritas como vesículas para a eliminação de
receptor de ferritina em reticulócitos (Harding et al., 1983) e só depois foram
descritas de maneira mais ubiquitária. O exossomos são formados através de
brotamento
de
endossomos
tardios
formando
corpos
multivesiculares
(multivesicular bodies, MVB) e são liberados no meio após a fusão destes MVB
com a membrana plasmática. Embora descritos inicialmente em reticulócitos,
exossomos foram posteriormente descritos em linfócitos B (Raposo et. al.,
1996), tendo um papel na apresentação de antígenos e consequentemente na
indução de resposta de linfócitos T. Desde então, exossomos foram descritos
em vários tipos celulares incluindo células tumorais (Taylor & Gercel-Taylor,
2011), células tronco mesenquimais (Lai et al., 2011, Huang et al., 2012),
células endoteliais (Dignat-George & Boulanger, 2011) e também em diferentes
fluidos biológicos como plasma, urina, saliva, líquido amniótico, etc (Simpson
et. al., 2009). Além de mediarem a comunicação intercelular, os exossomos
também estão envolvidos em enfermidades como câncer e doenças
degenerativas (Ludwig & Giebel B, 2012).
A composição protéica dos exossomos é variável de acordo com o tipo
celular do qual se originam (Simpson et. al., 2009). No entanto, existe um
núcleo de proteínas que são comuns a diferentes exossomos derivados de
células dendríticas, linfócitos B e células epiteliais intestinais. Dentre essas
proteínas comuns, estão aquelas associadas com a biogênese do exossomo,
adesão celular e fusão de membranas (como anexinas e proteínas Rab),
transdução de sinal, proteínas do citoesqueleto (como actina e tubulina). Os
exossomos apresentam também um conteúdo enriquecido em colesterol,
esfingomielina
e
GM3,
que
estão
envolvidos
na
manutenção
dos
microdomínios lipídicos que são essenciais na interação entre as células
(Février e Raposo, 2004; Li et al., 2006). As proteínas associadas à transdução
de sinal mais comumente encontradas são as proteínas-quinase, proteínas G
heterotriméricas, HSP70 e HSP90. Exossomos possuem ainda moléculas MHC
classe I e tetraspaninas, sendo que os membros CD3, CD63, CD9, CD81 e
CD82 são os mais encontradas nos exossomos. Acredita-se que essas
tetraspaninas interajam com integrinas e MHC classe II, e ainda participem na
ativação celular dos exossomos (Février e Raposo, 2004; Li et al., 2006). As
proteínas que compõem os exossomos são selecionadas por uma série de
proteínas que se ligam à ubiquitina, chamadas Vps (vacuolar protein sorting),
que constituem o complexo proteíco ESCRT 0, I, II e III (Complexo endossomal
de escolha protéica para o transporte) (Bishop, 2003; Vella et al., 2008).
Uma característica dos exossomos é que eles podem ser transferidos
entre diferentes tipos de células dendríticas e linfócitos B (Février e Raposo,
2004). Podem também estar associados à indução de respostas anti-tumorais.
Exossomos derivados de células dendríticas sensibilizadas com peptídeos
tumorais foram capazes de ativar respostas anti-tumorais mediadas por células
T citotóxicas, levando à regressão de tumores em modelos animais. Essas
observações levaram a estudos que tentam utilizar os exossomos como
imunoterapia para tratamento do câncer (Azevedo, 2007; Mignot et al., 2006).
Foi ainda demonstrado que os exossomos podem induzir tolerância
imunológica. Células epiteliais intestinais secretam exossomos em condições
inflamatórias, carregando MHC II na circulação. Esses exossomos, juntamente
com as células T CD4+ regulatórias, essenciais para a indução e manutenção
de tolerância periférica a antígenos do conteúdo intestinal (Li et al., 2006). As
plaquetas também secretam exossomos ricos em CD63, e são provavelmente
liberados no local da injúria vascular, podendo funcionar no ambiente direto de
adesão plaquetária (Denzer et al., 2000).
Recentemente foi descrito que os exossomos podem transferir proteínas
entre células sem a necessidade de contato direto entre as mesmas, e gerar
diferentes estímulos biológicos. Exossomos derivados de células dendríticas
podem modular a imunidade de células T (Li et al., 2006), além de produzirem
respostas anti-inflamatórias, resultando na diminuição de doenças auto-imunes
em modelos animais (Bianco et al., 2007; Kim et al., 2007). Estudos
demonstraram o papel dos exossomos no tráfego do vírus HIV (Loomis et al.,
2006), bem como no tráfego de proteínas neurodegenerativas, como príon
(Vella et al., 2007) e precursores mielóides (Rajendran et al., 2006). Esses
dados recentes da literatura indicam que as células utilizam os exossomos para
comunicação entre si em situações patológicas, fornecendo uma nova rota de
comunicação celular e possivelmente de disseminação de um patógeno (Cho
et al., 2005).
3- Células dendriticas
As células dendríticas (DCs) foram isoladas na década de 1970 do baço
de camundongos, apresentando uma morfologia semelhante a dêndritos
neuronais e aderência ao vidro, assim como os macrófagos (Steinman e Cohn,
1973). Atualmente, estas células são conhecidas como as principais células
apresentadoras de antígenos (APCs), realizando o mapeamento do ambiente
em busca de antígenos estranhos, os quais quando encontrados são
capturados, processados e apresentandos aos linfócitos. Desta forma, as DCs
são fundamentais na ativação da resposta imune adaptativa (Banchereau et al.,
2000). Além da ativação da resposta imune adaptativa, recentemente, outras
funções têm sido atribuídas às DCs, como o controle da migração de linfócitos
aos linfonodos (Moussin e Girard, 2011). As células dendríticas (DCs) formam
uma população heterogênea de células que apresentam subtipos com
diferenças no fenótipo de superfície, função e localização (Shortman e Naik,
2007). As células dendríticas respondem com plasticidade a diversos estímulos
endógenos, como citocinas, mediadores inflamatórios, neuropeptídeos e
hormônios. As modificações decorrentes de tais estímulos e a ampla
distribuição pelos tecidos têm levado a uma classificação ainda mais complexa
das DCs, que podem ser subdivididas com base em características fenotípicas,
de migração, presença na inflamação, estágios de desenvolvimento, entre
outros. Os estágios de desenvolvimento, por exemplo, fazem a distinção entre
células denominadas pré-DCs, DCs imaturas e DCs ativadas (Steinman, 1991;
Naik, 2008). As células plasmacitóides são exemplos de pré-DCs, pois
precisam de estímulo (vírus, bactérias) para apresentar a forma e a função
típicas de células dendríticas. Estas células têm como característica peculiar a
capacidade de produzir grandes quantidades de interferons do tipo I (O’Keefe
et al., 2002).
Baseando-se nestes aspectos as DCs representam atualmente as
células de escolha para estudos de patogênese de agentes infecciosos,
resposta a vacinas, câncer e outras doenças imuno-mediadas (Kooten et al.,
2011; Silveira et al., 2011; Hayden et al., 2009; Palucka et al., 2011).
Adicionalmente, a imunoterapia baseada em células dendríticas vem ganhando
importância, especialmente no desenvovlimento de vacinas anti-tumorais,
aumentando a importância dessas células para a imunologia e a medicina
(Hayden et al., 2009).
3.1 - Células dendriticas e dengue
Na infecção pelo vírus da dengue, as células dendríticas (DC), mais
especificamente as células de Langerhans da pele, representam o alvo
primário da infecção pelo vírus (Wu et al., 2000). Foi demonstrado que DCs
ativadas após a infecção pelo vírus dengue secretam fator de necrose tumoral
alfa (TNF-α), interferon (IFN) tipo I (alfa/beta) e IFN-γ, além de expressarem
marcadores de maturação como o HLA-DR, CD11b e CD83 (Libraty et al.,
2001). Enquanto o IFN tipo I pode conter diretamente a disseminação viral
(Umareddy et al., 2008), a produção de citocinas e a maturação das DCs são
fundamentais para a estimulação de linfócitos T DENV-específicos. Deste
modo, pode-se dizer que as DCs são responsáveis por iniciarem a resposta
imune adaptativa contra o DENV, bem como, constituem a primeira linha de
defesa contra a infecção (Wu et al., 2000; Navarro-Sánchez et al., 2005).
Finalmente, diferenças na capacidade das DCs em controlar a infecção podem
estar relacionadas com a variabilidade na patogenia da dengue (NavarroSanchez et al., 2005; Silveira et al., 2011).
É importante mencionar que a resposta de DCs na infecção pelo DENV
também podem contribuir para a febre hemorrágica. Foi demonstrado por
Luplertlop e colaboradores (2006) que as DCs produzem metaloproteinases -9
e -2 (MMP-9 e MMP-2) após a infecção pelo DENV de forma dose-dependente.
MMP-9 e MMP-2 aumentam a permeabilidade endotelial através da redução na
expressão de PECAM-1 e VE-caderina, além da redistribuição das fibras de
actina F (Luplertlop et al., 2006).
Ainda mais, o número absoluto de células dendríticas plasmocitóides e
mielóides mostrou-se reduzido no sangue periférico de indivíduos infectados
com DENV em comparação aos controles saudáveis. Apesar disso, não foi
observado diferenças significativas no número de DCs em pacientes com febre
da dengue e febre hemorrágica da dengue. Além disso, a viremia não foi
correlacionada com a severidade da doença, apesar de mostrar relação com a
redução de DCs plasmocitóides e mielóides no sangue, isto é, quanto maior a
viremia menor o numero de DCs, sugerindo um importante papel das DCs no
controle inicial da viremia pelo DENV (De Carvalho et al., 2011).
Finalmente, as DCs contribuem para a eliminação direta do vírus, ou
ainda para a estimulação de linfócitos T e B DENV-específicos. Por outro lado,
sabe-se que sub-produtos da infecção das DCs pelo DENV podem contribuir
para a hemorragia observada na dengue hemorrágica. Desta modo, em virtude
deste duplo papel na infecção pelo DENV, bem como, no seu importante papel
na resposta imune inata e adaptativa, as DCs constituem em um importante
modelo para estudo da patogenia da dengue.
3.2 – Células dendríticas e doença de Chagas
A doença de Chagas, descoberta e descrita por Carlos Chagas há mais
de 100 anos ainda é um grave problema de saúde pública afetando uma
população superior a 10 milhões de indivíduos na America Latina. O primeiro
mecanismo de resistência à infecção por T. cruzi é a fagocitose. As formas
tripomastigotas metacíclicas replicam-se dentro dos macrófagos, e embora
muitos parasitas acabem destruídos dentro dos vacúolos fagocíticos, as formas
que evadem conseguem se multiplicar no citoplasma como amastigotas. Os
macrófagos não ativados controlam a infecção se a razão parasitas/célula
infectada for 5:1, no entanto os macrófagos são destruídos se forem infectados
na razão 10:1 (Kress et al., 1975).
Fatores associados à imunidade inata como citocinas, proteínas
catiônicas, transferrinas e proteínas do sistema complemento são responsáveis
pela lise do protozoário, resistência à infecção e redução da carga parasitária.
Semelhante a resposta às infecções virais e de outros protozoários, a infecção
por T. cruzi induz a expressão de interferons (IFN) e outras citocinas
produzidas por fagócitos mononucleares e células natural killer (NK) (Hatcher &
Kuhn, 1982).
Os níveis de interferons sérico estão correlacionados com a atividade de
células NK do baço durante a infecção por T. cruzi em modelo murino (James
et al., 1982). Inoculação diária de interferons aumenta a resistência de
camundongos infectados com T cruzi quando comparados aos animais
inoculados com placebo. Estes achados sugerem que a atividade das células
NK induzidas por IFN- é responsável pela resistência e sobrevivência dos
animais infectados (Hatcher & Kuhn, 1981; Hatcher et al., 1981; Sanderson et
al., 1977). No entanto, citocinas não afetam diretamente a viabilidade,
motilidade, infectividade e virulência dos parasitas (James et al., 1982;
Kierszenbaum & Sonnenfeld, 1982).
A imunidade protetora à infecção por T cruzi parece ser dependente de
linfócitos T CD4 e CD8 secretores de IFN- (de Alencar et al., 2009; Rodrigues
et al., 2009). No entanto, a geração de células T CD8 parasita-específicas não
é afetada na ausência de sinalização via IFN do tipo I (Bixby & Tarleton,, 2008;
Martin et al., 2010). Por sua vez, o papel das células T reguladoras parece ser
limitado na infecção por T cruzi (Sales et al., 2008). Vale ressaltar que os
diferentes estágios de infecção, aguda e crônica, parecem estar sob diferente
regulação de interações complexas entre o patógeno e as células hospedeiras,
consequentemente, vários padrões de ativação de células do sistema imune
influenciam a produção de citocinas e o equilíbrio parasita-hospedeiro (Goronzy
& Weyand, 2009).
A participação de células T CD4 (Th1) é essencial na resposta imune ao
T. cruzi, uma vez que recrutam e ativam outras células da resposta imune,
como macrófagos, células B, mastócitos, neutrófilos, basófilos, eosinófilos e os
próprios linfócitos T CD8 (Sanderson & de Souza, 2009). Porém, a
sobreposição de células produtoras de citocinas tanto do padrão Th1 quanto do
padrão Th2 em ambos os estágios da progressão da infecção por T.cruzi
demonstram não haver um padrão dominante de produção das mesmas
(Zhang & Tarleton, 1996).
Células dendríticas são reconhecidas como células apresentadoras de
antígeno profissionais responsáveis pelo início da resposta imune devido à
capacidade de reconhecer PAMPs (Pathogen Associated Molecular Patterns)
(Manickasingham et al., 2003). Porém, diferentes estudos demonstraram que a
infecção por T. cruzi desencadeia mecanismos reguladores capazes de reduzir
a resposta imune (Abrahamsohn & Coffman, 1995; Kierszenbaum et al., 1999;
Tarleton, 1991). Em modelos murinos foi demonstrado que a infecção com T.
cruzi regula negativamente a expressão de moléculas MHC de classe II e de
moléculas coestimuladoras assim como prejudicam a capacidade de células T
estimularem células dendríticas do baço (Alba Soto et al., 2003). Experimentos
in vitro com tripomastigotas (forma infectiva do T.cruzi), vivos ou mortos por
aquecimento, adicionados de LPS (lipopolissacarídeo) induzem propriedades
tolerogênicas com produção de altos níveis de IL-10 e inibição da capacidade
das células apresentadoras de antígenos em induzir linfoproliferação (Poncini
et al., 2008; Poncini et al., 2010). Além disso, foi demonstrado que o aumento
na produção de IL-10 pelas DCs é independente de TLR2 e envolve interações
parasita-DC via TLR4 ainda não caracterizadas (Poncini et al., 2010).
A produção de IL-10 na infecção com T. cruzi parece modular a função
das DCs através de mecanismos que resultam em estimulação da resposta
imune insuficiente para a erradicação do parasita. Desta forma, estratégias que
reduzam esta modulação podem ser essenciais no desenvolvimento da
imunidade protetora contra T. cruzi (Alba Soto et al., 2010).
4 - Células-Tronco Mesenquimais
As células tronco mesenquimais (CTMs) são células indiferenciadas,
auto-renováveis, com alta capacidade proliferativa e que podem diferenciar-se
em linhagens celulares maduras como osteócitos, adipócitos e condrócitos
(Dominici et. al., 2006). Estas células podem ser isoladas de diversos tecidos
adultos, dentre eles a medula óssea e o tecido adiposo (Rebelatto et. al.,
2008). Acredita-se que as CTMs presentes nos tecidos adultos sejam capazes
de se diferenciar nas linhagens celulares de seu tecido de origem como forma
de manutenção da integridade destes tecidos e órgãos (Verfaillie, 2002). Por
outro lado, além de seu potencial de diferenciação celular, as CTMs atuam na
homeostase tecidual através da secreção de fatores solúveis e da ação
parácrina destes fatores sobre os constituintes teciduais (Singer & Caplan,
2011). Neste sentido, já foi demonstrado que as CTMs são capazes de
efetivamente suprimir a proliferação clonal e a expansão de células T primadas
(Di Nicola et. al., 2002), migrando para os sítios de inflamação e induzindo a
regeneração tecidual (Singer & Caplan, 2011).
Neste contexto, os efeitos parácrinos induzidos pelas CTMs podem ser
divididos
em:
efeitos
tróficos;
imunomodulatórios;
anti-cicatriciais;
e
quimioatraentes. Tais efeitos tróficos podem regular ações celulares antiapoptóticas (VEGF, HGF, IGF-1, TGFb, GM-CSF), proliferativas (SCF, LIF,IL6, M-CSF, SDF-1), de diferenciação (GM-CSF, SCF, HGF) e angiogênicas
(bFGF, VEGF, PIGF, MCP-1, ECM) (Meirtelles et. al., 2009). A produção e
secreção desta gama de moléculas bioativas fazem das CTMs ferramentas
promissoras para o tratamento de diferentes enfermidades. Entretanto, os
mecanismos utilizados pelas CTMs que promovem tais ações e, desta forma,
mantém a homeostase tecidual ainda não são conhecidos (Di Nicola et. al.,
2002). Neste sentido, tem sido descrito como um novo mecanismo de
comunicação célula-célula a transferência de moléculas bioativas através de
microvesículas, sendo este mecanismo um potencial efetor das ações
biológicas características das CTMs (Morel et. al., 2004).
O mecanismo de sinalização celular através da secreção de
exossomos/microvesículas no microambiente extracelular, descrito em célulastronco, foi inicialmente relacionado às células-tronco embrionárias. Neste
trabalho, Ratajczak e colaboradores demonstram o papel deste tipo de
sinalização como indutor da reprogramação de progenitores hematopoiéticos
através da transferência de mRNAs (Ratajczak et. al., 2006). Trabalhos mais
recentes sugerem que alguns dos efeitos regenerativos induzidos pelas CTMs
seriam mediados através da secreção de exossomos (Bruno et. al., 2009; Lai
et. al., 2010). Neste contexto, foi demonstrado que microvesículas derivadas
de CTMs humanas são capazes de estimular in vitro proliferação e maior
resistência
contra
apoptose
em
células
tubulares
epiteliais.
Quando
administradas in vivo em camundongos SCID com lesão renal aguda, as
microvesículas aceleram a recuperação funcional e morfológica do órgão
afetado (Bruno et. al., 2009). Por outro lado, a análise proteômica do meio
condicionado derivado do cultivo com CTMs revelou que os efeitos
terapêuticos deste meio condicionado são, ao menos em parte, relacionados a
ação de proteínas de membrana e intracelulares, possivelmente presentes nos
exossomos derivados destas células (Sze et. al., 2007).
Acredita-se que os exossomos derivados das CTMs poderiam
redirecionar funções alteradas de células alvo durante processos de lesão
tecidual, sugerindo que estas microvesículas bioativas poderiam servir como
terapia alternativa não baseada em células e, assim, serem exploradas na
medicina regenerativa para reparar tecidos lesionados (Camussi et. al., 2010).
De qualquer forma, os mecanismos envolvidos na regeneração tecidual
induzida pela administração de CTMs ou de seus exossomos permanecem
obscuros.
5 – Panorama das questões propostas no projeto
O projeto que propomos desenvolver visa estudar o papel de exossomos
e de grânulos de RNA durante o processo de infecção de duas células alvo
com patógenos diferentes, um vírus e um protozoário. Trata-se de um projeto
original pois não há qualquer descrição na literatura do papel de exossomos
secretados por células infectadas com o vírus dengue ou com T.cruzi no
processo de infecção de outras células. Adicionalmente, não encontramos na
literatura qualquer descrição do papel da infecção por estes patógenos na
diferenciação de células tronco mesenquimais. Finalmente, é desconhecido se
os exossomos normalmente secretados por células dendriticas têm sua
composição alterada quando da infecção pelos patógenos estudados e como
os exossomos de células infectadas se comparam aos exossomos normais de
células tronco e células dendriticas. Na outra vertente, resta a ser determinado
de que maneira são alterados e o papel de grânulos de RNA (sejam P-bodies
ou grânulos de estresse) no processo de patogênese. Estes estudos serão
realizados utilizando sequenciamento em massa de RNAs (RNAseq) e análise
proteômica das proteínas constituintes dos
exossomos e dos grânulos de
RNA, permitindo a visão holística dos genes e vias metabólicas e de regulação
envolvidas no processo de patogênese.
6 – Modelo experimentais de patógenos.
Neste projeto propomos estudos com dois modelos experimentais de
patógenos
bem
estabelecidos
nas
atividades
de
grande
parte
dos
pesquisadores do Núcleo. As doenças causadas por estes patógenos são de
alta relevância para a saúde pública (dengue e doença de Chagas) e os
modelos são descritos abaixo.
6.1 – Modelo experimental com vírus dengue.
Duas cepas do vírus da dengue (DENV) sorotipo 3 foram isoladas por
pesquisadores do Núcleo e serão utilizadas neste projeto. Uma foi denominada
BR DENV/3/290-02 (GeneBank EF629369), proveniente de um paciente
residente na cidade de Curitiba, que viajou para a cidade do Rio de Janeiro
onde se infectou. O diagnóstico clínico desse paciente foi o de dengue clássica
não fatal, durante uma epidemia em 2002 (LACEN, Curitiba-PR). A outra cepa
foi isolada de um caso visceral ocorrido no Paraguai em 2007 (PAR
DENV/3/5532-07). Essa cepa foi isolada de um caso fatal de febre da dengue
aguda primária com complicações hepáticas e cardíacas na cidade de
Lambaré, região metropolitana de Assunção, Paraguai, durante uma epidemia
em 2007.
Análises moleculares revelaram que ambas as cepas pertencem ao genótipo III
do sorotipo 3, o que possibilita o uso dessas cepas em estudos de interação
células dendriticas humanas – DENV,
permitindo mimetizar a resposta do
paciente frente a infecção por dengue 3 com diferentes casos clínicos. A
comparação da sequência de aminoácidos na região codificadora do genoma
das cepas DENV/3/290 e DENV/3/5532, revelou 12 substituições localizadas
nas regiões estruturais e não estruturais e não foram observadas alterações na
estrutura secundária da região 3' não traduzida do RNA das cepas em estudo.
O primeiro alvo celular para a infecção pelo DENV são as DCs residentes da
pele, células de Langherans (LC) e as células dendriticas dermais (DDC)
(Navarro-Sánchez er al., 2005). Os resultados obtidos por Silveira e
colaboradores (2011), demonstraram que a cepa DENV/3/5532 associada a um
caso fatal de dengue, é capaz de infectar de maneira mais eficiente mdDCs
(células dendriticas derivadas de monócitos), modula mais intensamente genes
envolvidos na resposta imune inata, induz a uma maior apoptose e uma maior
secreção de citocinas pró-inflamatórias. Esse conjunto de fatores pode ter
contribuído para a miocardite, hepatite e morte por choque do paciente. Já foi
demonstrado que a patogenia da dengue está associada com a carga viral
(Libraty et al., 2002), onde foram detectados picos de viremia 100 a 1000
vezes maiores em paciente que desenvolveram febre hemorrágica da dengue
em comparação aos pacientes que apresentaram febre da dengue (Vaughn et
al., 2000). Corroborando os dados da literatura, a cepa DENV/3/5532 infectou
mais eficientemente as mdDCs do que a cepa DENV/3/290, produzindo altos
títulos de vírus no sobrenadante de cultura (Silveira et al., 2011). A infecção
das mdDCs com a cepa DENV/3/5532 induziu uma maior produção de
citocinas TNF- e IL-6 em comparação à infecção com a cepa DENV/3/290 em
72h pós-infecção; também foram observados elevados níveis de apoptose nas
mdDCs infectadas com DENV/3/5532.
A comparação entre cepas de DENV que apresentem diferentes casos clínicos
podem ajudar a elucidar mecanismos implicados numa maior virulência de uma
cepa em relação a outra e contribuir para o prognóstico de casos graves de
dengue.
6.2 – Modelo experimental com Trypanosoma cruzi.
Será utilizado o T.cruzi Dm28c. Trata-se de um clone bem caracterizado e
isolado por pesquisadores do Núcleo (Contreras et. al., 1988), sendo hoje uma
cepa de referência para trabalhos com o T.cruzi. As diferentes formas
(epimastigotas, tripomastigotas e amastigotas) serão obtidas por cultivo
axênico (Contreras et al., 1985, Bonaldo et. al., 1988) ou então a partir de
células Vero infectadas com tripomastigotas metacíclicos provenientes de meio
axênico ou tripomastigotas isolados do sangue de animais infectados.
c- Plano geral de trabalho: atividades a serem desenvolvidas,
descrição dos experimentos e da metodologia, parâmetros de
avaliação do desempenho
Isolamento e cultura de células dendriticas derivadas de monócitos:
Células dendriticas derivadas de monócitos (mdDCs) serão obtidas utilizandose sangue periférico (120 mL) de voluntários saudáveis, após consentimento
informado, com a aprovação do Comitê de Ética em Pesquisa da FIOCRUZ
(número 514/09). Para tanto, células mononucleares serão separadas pela
técnica de gradiente de densidade utilizando-se o reagente Lymphocyte
Separation Medium (Lonza, Walkersville, USA), e destas, será isolada a
população de células CD14+, utilizando-se o método de imuno-separação
magnética por esferas revestidas com anticorpos anti- CD14 (Miltenyi Biotec,
Auburn, CA), de acordo com as recomendações do fabricante. Estas células
CD14+ serão cultivadas por 6-7 dias em meio RPMI 1640 contendo 100 ng/mL
de Interleucina-4 (IL-4) e 50 ng/mL Fator Estimulante de Colônias de Monócitos
e Granulócitos (GM-CSF; PeproTech, Rocky Hill, NJ), 10% de soro fetal bovino
(FBS), 100 IU/mL de Penicilina, 100 µg/mL Estreptomicina e 2 mM de LGlutamina (reagentes da marca Gibco-BRL, Grand Island, NY), sendo
realizando uma troca de meio no dia 3- 4 de cultivo. Após este período, as
culturas serão avaliadas para determinar o fenótipo de mdDCs utilizando-se a
técnica de citometria de fluxo (FACS, Becton Dickson – BD). Para tanto, serão
utilizados seis marcadores distintos: CD14, CD1a, CD11b, CD11c, CD209 e
HLA-DR. As células dentríticas isoladas serão cultivadas sobre lamínulas em
densidade de 4 x 105 células/cm2 em meio RPMI 1640.
Parâmetros de avaliação do desempenho: Isolamento e cultivo de células
dendriticas, análise e caracterização das células isoladas.
Isolamento e cultura de células tronco de tecido adiposo: As células
tronco adultas do tecido adiposo serão removidas de material excedente de
cirurgia bariátrica. O material será coletado no Instituto de Medicina e Cirurgia
do Paraná, em pacientes submetidos à cirurgia bariátrica, após consentimento
livre e esclarecido dos doadores, com a aprovação do Comitê de Ética em
Pesquisa da FIOCRUZ (número 419/07). Logo após a retirada do tecido
adiposo, este será lavado com solução salina para remover resíduos
contaminantes e hemácias. Em seguida o material será digerido com 0,02
mg/mL de colagenase A (Roche Diagnostic) em meio HAM-F12 e meio de
Dulbecco´s (DMEM-F12, GIBCO), contendo 2% de albumina bovina por 45
minutos a 37ºC. O tecido digerido será filtrado duas vezes com um filtro de
100μm e outro de 25μm de membrana de nylon, para eliminar os fragmentos
não digeridos. Em seguida o material será centrifugado a 600 g por 10 minutos
e as células que formam a fração vascular do estroma do tecido adiposo serão
coletadas e as células vermelhas lisadas com um tampão contendo 155mmol/L
de NH4CL, 20 mmol/L Tris pH 7,6, durante 5 minutos.
As células estromais isoladas serão cultivadas sobre lamínulas em
densidade de 2 x 104 células/cm2 em meio DMEM-F12 (1:1), suplementado
com 5% de soro bovino fetal, 100μg/mL de ácido pantotênico, 100μmol/L de
ácido ascórbico, 16μmol/L de biotina, 250 μg/mL de anfotericina, 5 μg/mL de
estreptomicina e 5 U/mL de penicilina. Após 24 horas as células não aderentes
serão removidas e o meio será trocado duas vezes por semana.
Parâmetros de avaliação do desempenho: Isolamento de adipócitos,
caracterização fenotípica das células e cultivo das mesmas.
Indução de estresse celular por arsenito de sódio: Células dendriticas e
células tronco serão tratadas com concentrações crescentes de arsenito de
sódio (0,5 – 2 mM) por diferentes tempos de incubação (10 – 60 minutos). Com
esta abordagem, esperamos estabelecer o melhor protocolo para a indução de
estresse e verificar a presença e a dinâmica de formação dos grânulos de RNA
nos diferentes tipos celulares.
Parâmetros de avaliação do desempenho: Observação da formação de
grânulos de RNA em resposta ao tratamento, estabelecimento do protocolo
experimental.
Infecção das células:
Infecção com o protozoário T. cruzi: Células dendriticas e células tronco,
previamente cultivadas sobre lamínulas em placas de 24 poços, serão
infectadas com as formas tripomastigotas de T. cruzi provenientes de cultivo
celular na proporção de dez parasitas por célula. Após quatro horas, as
monocamadas serão lavadas com PBS para remoção dos parasitas não
interiorizados e mantidas por 6, 12, 24, 48 e 72 horas em 500 l/poço de
DMEM ou RPMI suplementado com SFB 10% a 37C e 5% de CO2. Serão
realizados experimentos de infecção celular com e sem indução de estresse.
Para a indução do estresse as células infectadas serão incubadas com arsenito
de sódio nas condições pré-estabelecidas de acordo com o item anterior. Para
se certificar de que a diminuição do número de células infectadas não tenha
ocorrido pela lise da célula hospedeira, decorrente do ciclo de multiplicação
intracelular, as monocamadas serão observadas em microscópio invertido para
verificar a presença de tripomastigotas no sobrenadante. As lamínulas serão
lavadas com PBS e processadas para os ensaios de imunofluorescência
indireta.
Infecção com o vírus da Dengue: Células dendriticas e células tronco,
previamente cultivadas sobre lamínulas em placas de 24 poços, serão
infectadas com os vírus DENV/3/290 e DENV/3/5532 (MOI = 5) por 2 horas a
37C e 5% de CO2. As células serão lavadas com meio fresco para remoção
dos vírus não interiorizados e mantidas por 6, 12, 24, 48 e 72 horas em 500
l/poço de DMEM ou RPMI + SFB 10% a 37C e 5% de CO2. Nos tempos de
incubação estabelecidos, serão retiradas alíquotas do sobrenadante das
culturas para determinar a presença de citocinas e quimiocinas inflamatórias
através da tecnologia CBA (Cytometric Bead Array) de acordo com instruções
do fabricante. Serão realizados experimentos de infecção celular com e sem
indução de estresse. Para a indução do estresse as células infectadas serão
incubadas com arsenito de sódio nas condições pré-estabelecidas de acordo
com o item anterior. As lamínulas serão lavadas com PBS e processadas para
os ensaios de imunofluorescência indireta.
Parâmetros de avaliação do desempenho: Estabelecimento da cinética de
infecção com cada patógeno.
Detecção das proteínas dos grânulos de RNA, de proteínas virais e de
proteínas envolvidas com a resposta imune inata por ensaios de
imunofluorescência
indireta:
Células
dendriticas
e
células
tronco
previamente cultivadas em lamínulas, não infectadas ou infectadas com os
diferentes patógenos e submetidas ou não ao estresse com arsenito de sódio,
serão fixadas com paraformaldeído 4% em PBS por 10 minutos em
temperatura ambiente, permeabilizadas com Triton X-100 0,5% por 5 minutos,
lavadas três vezes com PBS e incubadas com solução de bloqueio (PBS
contendo 5% de soro fetal bovino e 1% de soro humano) por 1 hora em
temperatura ambiente. Os anticorpos primários serão diluídos na solução de
bloqueio e incubados com as células a 37ºC por 1 hora. As células serão
lavadas 3 vezes por 10 minutos com PBS e incubadas com os anticorpos
secundários conjugados aos fluoróforos Alexa 488 ou Alexa 546 e as etapas de
incubação e lavagem serão repetidas. Os núcleos das células serão corados
durante 15 minutos com DAPI, diluído em solução de bloqueio na concentração
final de 1 µg/µl. Após esta etapa as lamínulas serão lavadas cinco vezes por 10
minutos e montadas sobre lâminas de vidro com 10 µL de n-propil-galato na
concentração de 200 µg/ml. A fluorescência será observada em microscópio
confocal.
Os anticorpos primários utilizados para a detecção das proteínas dos
grânulos de estresse e P-bodies serão anti-TIA1/TIAR e anti-Dcp1a,
respectivamente. As proteínas virais serão detectadas com o uso dos
anticorpos anti-NS3 e anti-flavivírus (4G2). A presença e/ou ausência das
proteínas envolvidas com a resposta imune inata nos grânulos de RNA serão
evidenciadas com o uso de anticorpos direcionados contra citocinas próinflamatórias (TNF- e IL-12) e antiinflamatórias (IL-10).
Ensaios de coimunoprecipitação: Células dendriticas e células tronco
não infectadas e infectadas com os diferentes patógenos serão lisadas em um
mililitro de tampão de lise (KCl 100 mM; MgCl2 5 mM; Hepes 10 mM pH 7,0;
NP-40 1% e inibidores de proteases) por 1 hora a 4°C sob agitação. As células
serão centrifugadas a 2.000 g por 5 minutos a 4°C e o sobrenadante será
incubado com proteína A/G Sepharose por 30 minutos a 4°C. A mistura será
centrifugada nas mesmas condições acima e o sobrenadante será incubado
com anti-TIA1/TIAR e com anti-Dcp1a por 1 hora a 4°C sob agitação. Após
esta etapa será adicionada a resina de proteína A/G Sepharose e a mistura
será incubada por 1 hora nas mesmas condições. A resina será coletada por
centrifugação e lavada 5 vezes com tampão de lise. Para as análises do
conteúdo protéico presente nas frações imunoprecipitadas com as proteínas
dos grânulos de RNA, a resina será fervida por 5 minutos e as proteínas serão
separadas em gel SDS-PAGE, transferidas para membrana de nitrocelulose e
analisadas
por
Western
blotting
utilizando
os
anticorpos
primários
especificados no item anterior. Os RNAs presentes nos imunocomplexos serão
purificados utilizando o kit RNeasy (Qiagen) de acordo com as instruções do
fabricante e identificados através da técnica de RT-PCR, como descrito a
seguir.
Reação da transcriptase reversa seguida da reação da polimerase em
cadeia (RT-PCR). Para verificar a presença de RNA viral e de RNAs celulares
dos genes envolvidos na resposta imune inata (por exemplo, IFIT, Usp18,
MHC-I e II, APOBEC, entre outros) os RNAs presentes nos imunocomplexos
serão concentrados em microcon e submetidos à transcrição reversa para
obtenção do cDNA correspondente, utilizando oligonucleotídeos degenerados,
dNTPs (GE Healthcare) e transcriptase reversa M-MLV (Promega). As reações
serão incubadas a 42 °C durante 60 minutos. Para as amplificações (PCRs)
será utilizado 1/10 do cDNA obtido, oligonucleotídeos iniciadores específicos
para cada gene, DNA polimerase Pfx Platinum® (Invitrogen) e condições de
amplificação previamente padronizadas. Os produtos obtidos serão resolvidos
em gel de agarose contendo brometo de etídio (Sigma) e visualizados em
transiluminador UV.
Obtenção de exossomos.
O sobrenadante das culturas de células
dendriticas, correspondente a 1x106 cel/mL, infectadas com T. cruzi ou com o
vírus da dengue será coletado e centrifugado à 2500 X g por 5 minutos. Em
seguida esse sobrenadante será filtrado em 0,22 mM e submetido a uma
ultracentrifugação à 100.000 X g por 3 horas. O sedimento será ressuspenso
PBS 1X e estocado à -80C até o momento do uso. Procedimento semelhante
será utilizado para o isolamento de exossomos a partir de cultivos axênicos de
T.cruzi. As células serão sedimentadas e o sobrenadante tratado.
Avaliação da captura de exossomos por células dendríticas
Uma alíquota contendo exossomos secretados por T. cruzi será processada
com o reagente Alexa Fluor kit ULYSIS Alexa Fluor 594 de acordo com as
recomendações do fabricante, a fim de promover a formação de um complexo
exossoma - Alexa Fluor. Os exossomos marcados serão ressuspensos em
PBS
para
posterior
estimulação
de
células
dendriticas
previamente
diferenciados. Aproximadamente 2 x 105 células (células dendriticas) serão
incubadas em cada câmara de uma lâmina (Lab-Tek Chamber Slide System,
Nalge Nunc Internacional, Rochester, NY) contendo 300 L de meio RPMI
suplementado. Estas células serão estimuladas com exossomos complexados
ao Alexa Fluor durante 4 horas. Decorrido este período, a lâmina será lavada
gentilmente com PBS, três vezes, para retirada de material excedente.
Posteriormente,
as
culturas
serão
fotografadas
em
microscópio
de
fluorescência Nikon Eclipse E800 (Nikon Instruments Inc., Melville, NY). As
imagens serão adquiridas com câmera digital Nikon DXM-1200 (Nikon
Instruments Inc., Melville, NY) conectada ao microscópio, para a avaliação da
captura dos exossomos.
Ensaios distintos serão realizados com células dendriticas (2 x 105), cultivadas
em placas de 96 poços, fundo reto (Corning, NY, USA), em 200 L de meio
RPMI suplementado, estimuladas com exossomos complexados ao Alexa Fluor
durante 4 horas. Como controle negativo, células serão estimuladas com
exossomos não marcados. Decorridas quatro horas de cultura, a placa será
centrifugada a 400 x g, durante 10 minutos para retirada do sobrenadante.
Posteriormente, as células aderentes serão removidas da placa com solução
PBS gelada e transferidas para tubos utilizados na citometria de fluxo e
centrifugadas a 400 x g durante 10 minutos para remoção do PBS, sendo em
seguida, marcadas com o anticorpo monoclonal contra CD11c, CD86 e HLADR. Serão adquiridos 25.000 eventos por amostra para a realização da análise.
Inicialmente, as células serão selecionadas através dos parâmetros de
tamanho (FSC) e granularidade (SSC), e na população selecionada, será
analisada a porcentagem de células com dupla marcação positiva: para a
molécula CD11c e para Alexa Flúor com intuito de se determinar a
porcentagem de células que capturarão os exossomos, bem como se a captura
dos exossomos é capaz de induzir a maturação das DCs (CD86 e HLA-DR).
Cultivo de células dendríticas para avaliação da resposta inata.
Células dendriticas previamente diferenciados a partir de PBMC em placas de
cultura de 48 poços (Corning; NY, USA) contendo meio RPMI suplementado, e
incubadas a 37ºC, com atmosfera de 5% de CO2 serão estimuladas com
exossomos secretados por T. cruzi, tendo como controle negativo células
cultivadas apenas com meio de cultura. Decorridas 12, 24 e 48 horas de
estimulação, os sobrenadantes serão coletados e armazenados a – 70ºC para
posterior dosagem de citocinas. As células cultivadas também serão avaliadas
por citometria de fluxo para determinação da expressão da molécula coestimuladora CD86 e da molécula de classe-II HLA-DR. Serão adquiridas 5.000
células dentro da população CD11c por amostra.
Co-cultivo de células apresentadoras de antígeno estimuladas com
exossomos provenientes de T. cruzi e linfócitos TCD4+ ou CD8+
Avaliação da proliferação celular - Linfócitos TCD4+ ou TCD8+ (ou células não
aderentes), com concentrações celulares previamente determinadas, serão
ressuspensos em 1 mL de PBS para cada 106 células, e acrescidas de 1 mL de
uma solução CFSE (5-(and-6)-carboxyfluorescein diacetate, succinimidyl Ester)
(Invitrogen, Molecular Probes) a 2,5 M. As células serão incubadas ao abrigo
da luz, durante 5 minutos a temperatura ambiente, sob agitação periódica. A
reação de incorporação de CFSE será interrompida com a adição de 5% de
SBF. Proceder-se-á a lavagem com 10 mL de PBS, seguida de centrifugação a
400 x g, a 4C, durante 10 minutos. Após novo procedimento de lavagem, as
células serão ressuspensas em meio RPMI suplementado com 10% de plasma
autólogo, penicilina (100U/mL), estreptomicina (100 g/mL), gentamicina (10
g/mL) e polimixina B (30 g/mL). A viabilidade e a concentração celular serão
novamente determinadas em câmara de Neubauer com Azul de Trypan.
Aproximadamente 1 x 105 células dendriticas e estimuladas com
exossomos secretados por T. cruzi, serão co-cultivados com 5 x 105 linfócitos
TCD4+ ou TCD8+ previamente marcados com CFSE, em placas de cultura de
96 poços (Corning; NY, USA) contendo meio RPMI suplementado e incubadas
a 37ºC, com atmosfera de 5% de CO2. Como controle da fluorescência da
marcação com CFSE também serão realizadas co-culturas com linfócitos não
marcados. Como controle negativo, as células serão cultivadas apenas em
presença de meio de cultura, e como controle positivo, as células serão
estimuladas com PHA (1%) (Gibco, Grand Island, NY. As culturas serão
mantidas durante 5 a 7 dias.
Decorrido o período de cultura, as células serão transferidas para tubos
de poliestireno (Falcon, BD, USA). Adicionar-se-á 100 L de EDTA 2 mM, e
após homogeneização, as células serão incubadas durante 10 minutos, à
temperatura ambiente, ao abrigo da luz. Posteriormente, serão adicionados 4
mL de solução PBS acrescida de 0,5% de BSA e 0,1% de azida sódica (PBSWash), e as células serão centrifugadas a 400 x g, durante 10 minutos, a 4C.
O sedimento celular será ressuspenso em 100 L de PBS-Wash (PBS-W) , e
no mesmo dia, proceder-se-á à marcação celular com anticorpos monoclonais
específicos para os receptores de superfície celular e posterior análise da
proliferação celular por citometria de fluxo (50.000 eventos adquiridos).
Inicialmente, os linfócitos serão selecionados através dos parâmetros de
tamanho (FSC) e granularidade (SSC). Em seguida, dentro da população
selecionada, correspondente aos linfócitos, será avaliada a porcentagem de
células positivas para os receptores CD4 ou CD8. A avaliação da capacidade
de proliferação das células estimuladas será obtida através da análise de
histogramas das populações positivas para CD4 ou CD8. Células que
apresentarem grande intensidade da marcação com CFSE corresponderão às
células que não proliferarão após cultivo. Células com diminuição na
intensidade de marcação para CFSE corresponderão às células que
proliferarão
após
estimulação.
Os
resultados
serão
expressos
pela
porcentagem das células que proliferaram.
Determinação
da
freqüência
de
células
produtoras
de
citocinas
-
5
Aproximadamente 1 x 10 células dendriticas previamente diferenciados a partir
de PBMC e estimulados com exossomos secretados por T. cruzi, serão cocultivados com 5 x 105 linfócitos TCD4+ ou TCD8+, em placas de cultura de 96
poços (Corning; NY, USA) contendo meio RPMI suplementado e incubadas a
37ºC, com atmosfera de 5% de CO2. As culturas deverão ser realizadas em
triplicata para obtenção de número suficiente de células para a análise. Como
controle negativo, as células serão cultivadas apenas em presença de meio de
cultura, e como controle positivo, as células serão estimuladas com PHA (1%).
Decorridos 24 e 48 horas de cultura, o transporte de proteínas para
vesículas intracelulares será abolido com a adição de 10 g/mL de Brefeldina A
(Sigma, St Louis, USA). As células permanecerão em cultura por mais 4 horas
e, em seguida, serão centrifugadas a 400 x g, durante 10 minutos, a 4C. Os
sobrenadantes serão coletados e armazenados a –70ºC para posterior
dosagem de citocinas secretadas. A marcação dos receptores CD4 e CD8,
assim como de citocinas intracitoplasmáticas IFN-, IL-4, IL-10, IL-17 e Foxp3,
será realizada de acordo com Sathler-Avelar [74]. A aquisição das amostras
será realizada no citômetro de fluxo, FACS Canto II (Becton & Dickinson), e a
análise realizada no programa Cell Quest a partir de 50.000 eventos
adquiridos.
Avaliação da produção de citocinas. A detecção de citocinas IFN-, IL-10,
TNF-, IL-5, IL-17, IL-12p70 nos sobrenadantes das culturas será realizada por
ensaio imunoenzimático (ELISA) ou por CBA (Cytometric Bead Array).
Utilizaremos o Human Th1/Th2/Th17 Cytokine Kit (Becton & Dickinson), que
quantifica a concentração de IFN-, TNF-, IL-17, IL-10, IL-6, IL-4 e IL-2. A
leitura do ensaio será realizada no citômetro de fluxo (BD FACS Canto II) de
acordo com as recomendações do fabricante. A análise dos dados será
realizada utilizando-se software específico denominado FCAP Array (Becton &
Dickinson), por meio da obtenção de curvas de calibração obtidas com os
padrões de citocinas do kit. Após a construção das curvas, a concentração das
amostras será determinada em pg/mL, a partir dos valores obtidos na leitura
referentes à Intensidade Média de Fluorescência (MFI).
Extração do RNA total. O RNA total das células dendriticas, cultivadas na
presença ou na ausência de exossomos provenientes de T. cruzi será extraído
utilizando-se o kit RNeasy, sendo estocado a –80ºC, até o momento de uso
Quantificação e avaliação da integridade das amostras de RNA. A
quantificação do RNA total bem como a integridade do mesmo será verificada
por eletroforese capilar utilizando o equipamento Bioanalyzer (Agilent).
Análise da expressão gênica relativa por PCR em tempo real. A partir do
RNA mensageiro serão obtidas amostras de cDNA para realização da PCR em
tempo real (Real Time – PCR). A quantificação de cDNA será realizada em
espectrofotômetro (NanoDrop, Thermo Fisher Scientific, USA) a partir de uma
solução de cDNA (50 ng/L) em um volume de 100 L. Serão realizados
ensaios para amplificação dos genes constitutivos GAPDH (Glyceraldehyde-3phosphate dehydrogenase) e HuPO (human ribosomal protein) e para os genes
de interesse, relacionados à resposta imune. As reações serão realizadas no
aparelho AB 7500 Real-Time PCR System (Applied Biosystems). Em paralelo,
serão realizadas amplificações de genes constitutivos utilizados para a
normalização dos dados de expressão gênica. Os dados serão normalizados
de acordo com a expressão dos genes GAPDH e HuPO de cada amostra,
utilizando o método do Ct (cycle threshold) e a equação 2-CT [75].
Sequenciamento em massa dos RNAs isolados dos exossomos.
Para o sequenciamento das amostras provenientes do isolamento dos
exossomos ou amostras tratadas com essas vesículas, utilizaremos 100 ng do
RNA purificado. Os RNAs serão fragmentados com a utilização de 1 U de
RNase III por 5 minutos à 37 ºC para obtenção de fragmentos de 150 a 250
nucleotídeos. Após a incubação a reação será parada com a adição de água
RNase free e as amostras serão imediatamente acondicionadas em gelo. A
purificação dos RNAs é feita com o kit RiboMinusTM Concentration Module
(Invitrogen) de acordo com as recomendações do fabricante. Após a
purificação, será feita uma reação de hibridização por 16 horas à 16 ºC, na qual
um adaptador é ligado na extremidade 3’. O adaptador é utilizado como
oligonucleotídeo iniciador numa reação de transcrição reversa para a síntese
de uma fita de cDNA. A fita simples de cDNA será purificada com o kit MinElute
PCR purification kit (Qiagen) de acordo com as recomendações do fabricante.
Após a purificação, o cDNA fita simples será submetido a uma eletroforese em
gel de acrilamida 6% (NovexR – Invitrogen) por 25 minutos à 180 V. O gel será
então corado com SYBR Gold por 5 minutos e posteriormente os fragmentos
de 150 a 250 nucleotídeos serão excisados do gel. Para a obtenção da
biblioteca de cDNA a amplificação será feita a partir da banda cortada do gel,
sendo que, nesse momento cada amostra recebe dois adaptadores (P1 e P2),
sendo que o P1 é específico e único que funciona como um código de barras
que identifica cada amostra. Após a amplificação do cDNA este será purificado
com o kit PureLinkTM PCR Micro kit (Invitrogen) de acordo com as
recomendações do fabricante. O tamanho e a concentração das amostras
serão analisados no equipamento Agilent 2100 Bioanalyzer com o kit DNA
1000. A PCR em emulsão (ePCR) tem como objetivo a amplificação clonal dos
fragmentos de DNA modificados e sua ligação às esferas (beads) de
sequenciamento pelo adaptador P1. A escala de preparação da PCR em
emulsão (ePCR) será a full scale que gera entre 400 e 500 milhões de esferas.
Para a reação de ePCR, o cDNA obtido cada amostra será misturado gerando
uma biblioteca que contém até 16 amostras diferentes, cada uma
individualizada pelo adaptador que confere o código de barras (P1). Essa
biblioteca será diluída para duas titulações, 0,5 e 1,0 pM, para cada biblioteca.
Assim sendo, serão feitas duas ePCRs para cada biblioteca, permitindo
selecionar qual titulação produz melhores resultados, tanto no que se refere à
quantidade de esferas quanto à qualidade da sequência. Após a ePCR, as
esferas passam por um processo de enriquecimento para maximizar a
quantidade de esferas P2 positivos, ou seja, que possuem fragmentos de DNA
completos contendo os adaptadores P1, interno e P2. Desse modo as esferas
sem produtos de PCR serão eliminadas. A contagem final de esferas após o
enriquecimento será feita em câmara de Neubauer e encaminhadas para o
sequenciamento. As lâminas utilizadas pelo sequenciador SOLiD apresentam
as configurações full (1 poço), quad (4 poços) e octeto (8 poços). A
configuração quad permite um número máximo de 60 milhões de esferas. O
tempo total de sequenciamento é de aproximadamente sete dias, sendo que no
primeiro dia será sequenciado adaptador código de barras e nos últimos 6 dias
o fragmento de 50 pb referente ás amostras. As análises das sequências
geradas pelo sequenciador SOLiD serão realizadas com base no mapeamento
das sequências obtidas no genoma de referência utilizando-se o programa CLC
Genomics WorkbenchTM.
Identificação do conteúdo protéico dos exossomos. As proteínas que
compões os exossomos serão solubilizadas em tampão de desnaturação (uréia
6 M / tiouréia 2 M em HEPES 10 mM (pH 8,0)). Em seguida será adicionado 1
μg (1 μl) de DTT para 50 μg de proteína por 30 minutos à temperatura
ambiente. Posteriormente adiciona-se 5 μg (1 μl) de iodoacetamida (IAA) para
50 μg de proteína e incuba-se por 20 minutos à temperatura ambiente, seguido
de diluição das amostras com 4 volumes de tampão de digestão. Para
obtenção dos peptídeos, adiciona-se 1 μg (0,5 μl) tripsina e incuba-se durante
a noite a temperatura ambiente, a reação é interrompida por acidificação a pH
menor que 2,5 com 100% de TFA (concentração final 0,5% TFA). Os peptídeos
são purificados usando C18-StageTips previamente equilibrados em tampão A.
Os peptídeos são lavados em tampão A e posteriormente eluídos em tampão
B. Após a secagem das amostras as mesmas são ressuspensas em solução A
e analisadas no espectrômetro de massas de acordo com as recomendações
do fabricante.
Clonagem, expressão e purificação de antígenos recombinantes.
Os dados obtidos a partir do seqüenciamento massivo e das análises
proteômicas
permitirão
evidenciar
alvos
de
interesse
para
estudos
individualizados. Para produção de antissoros e para caracterização funcional e
estrutural das proteínas de interesse será necessária a produção de proteínas
recombinantes. Inicialmente as proteínas de interesse serão analisadas quanto
à complexidade estrutural e a escolha do sistema de expressão será feita
conforme a necessidade de cada caso. Proteínas monoméricas ou domínios
extracelulares e intracelulares de estrutura menos complexa poderão ser
expressos em sistemas de Escherichia coli utilizando os vetores da série pET
(Novagen) baseados na RNA polimerase do fago T7 (Studier et al., 1986). A
produção de proteínas de membrana pode ser de particular importância para
este projeto. Para expressão deste tipo de proteína primeiramente serão
testadas as cepas de E. coli C41, C43 ou Lemo21(DE3) (Miroux & Walker
1996; Wagner et al., 2008) que foram selecionadas para produção de altos
níveis de proteínas de membrana e para proteínas tóxicas. Para as proteínas
complexas e para as proteínas cuja expressão não seja viável em E. coli, será
usado o sistema de baculovírus e células de inseto modificado a partir do
sistema “pFastBacDual” (Invitrogen). Este sistema permite expressar duas
proteínas recombinantes simultaneamente, uma sob o promotor da proteína
P10 e outra sob o promotor da polihedrina. As principais dificuldades deste
sistema são, primeiro determinar a taxa de infecção das células de inseto pelo
baculovírus recombinante e, segundo, determinar os níveis de expressão da
proteína de interesse. Visando contornar a primeira dificuldade, clonamos a
proteína GFP, a qual serve como gene marcador, sendo fácil de visualizar e
quantificar o número de células infectadas em microscópio de fluorescência.
Visando contornar a segunda dificuldade, as proteínas de interesse são
clonadas em fusão com uma sequência de seis histidinas que servem tanto
para detecção por immunoblotting como para purificação por cromatografia de
afinidade. Assim, a etapa inicial de purificação será por cromatografia de
afinidade a metal imobilizado. As etapas subsequentes de purificação serão
definidas de acordo com a necessidade de cada caso, e a resolução das
colunas cromatográficas seguindo uma ordem de prioridade de interação
iônica, interação hidrofóbica e exclusão por tamanho. A sequencia das etapas
cromatográficas seguirá a lógica utilizada em estudos anteriores (Smetana et
al., 2006, Navarro et al., 2008).
Produção de antisoros.
As proteínas recombinantes de interesse serão
utilizadas para a inoculação de camundongos, seguindo protocolos padrão,
visando a produção de antissoros policlonais e monoclonais. Para a confecção
de antissoros policlonais os animais (Balb/C) serão inoculados de 10 a 50
g/dose/animal em volume 1:1 com adjuvante hidróxido de alumínio (Alu-Gel-S;
Serva Heidelberg, Germany). Serão realizadas de 4 a 6 doses por via
intraperitoneal em intervalos de 7 a 15 dias entre as mesmas, sendo a última
dose por via endovenosa. Três dias após a última dose os animais serão
sacrificados e o soro coletado por punção cardíaca. Após a coagulação do
sangue, o soro policlonal será separado por centrifugação (1500 g por 5
minuto), aliquotado e armazenado a -20C até sua análise. Para a confecção
dos anticorpos monoclonais será utilizado o baço dos animais imunizados. O
baço será dissociado entre duas lâminas de vidro, e os esplenócitos serão
filtrados em membranas de nylon. Após a separação dos esplenócitos, será
realizada a lise dos eritrócitos residuais com solução de cloreto de amônio (166
mM de NH4Cl, 9 M de EDTA (Sigma-Aldrich, St. Louis, USA) e 95 mM de
NaHCO3 (Merck, Rio de Janeiro, Brasil) 5 minutos no gelo. As células serão
lavadas duas vezes em PBS 1 X e fusionadas com o mieloma murino
Ag8XP3653 com a utilização de polietilenoglicol (Sigma-Aldrich, St. Louis,
USA) 1:1 em meio RPMI sem SFB por 2 minutos. Após a fusão, as células
serão lavadas duas vezes com PBS 1 X e plaqueadas na confluência de 2.5 x
106 células/mL em placas de 96 orifícios (100 L/orifício). Após 24h de cultivo
será adicionado ao meio a droga de seleção dos hibridomas (esplenócitos +
mieloma) Hipoxantina-Aminopterina-Timidina (HAT; Sigma-Aldrich, St. Louis,
USA) e a seleção irá ocorrer por 12 dias com troca de metade do meio a cada
48 h. Após a seleção com meio HAT será adicionado o meio HipoxantinaTimidina (HT; Sigma-Aldrich, St. Louis, USA) por mais 4 dias, com trocas a
cada 48h. Finalmente, o cultivo seguirá com meio RPMI + 20% de SFB, 100
UI/mL de penicilina, 100 g/mL de estreptomicina, 2.5 g/mL de anfotericina B,
2 mM de glutamina e 1 mM de piruvato de sódio (Sigma-Aldrich, St. Louis,
USA). A triagem de clones positivos será realizada pela técnica de ELISA,
assim como, a isotipagem dos clones positivos após diluição limitante (SBA
Clonotyping HRP System, Southern Biotech, Birminghan, USA).
Ensaios de microscopia eletrônica. Para os ensaios de microscopia eletrônica
de transmissão, serão utilizados exossomos isolados e purificados bem como as
células dendríticas e tronco não infectadas e infectadas com dengue ou T. cruzi.
Após lavagem com PBS os exossomos ou células serão fixados uma solução de
glutaraldeído 0,1% / paraformaldeído 2% em tampão cacodilato de sódio 0,1 M,
pH 7,2. Após incubação por 60 minutos à temperatura ambiente os parasitas
serão lavados com tampão cacodilato 0,1 M, e submetidos a diversas etapas de
desidratação em temperaturas e tempos variáveis: etanol 30% a 4ºC por 30 min;
etanol 50% a -20ºC por 60 min; etanol 70% a -20ºC por 60 min; etanol 90% a 20ºC por 60 min; e por fim etanol 100% a -20ºC por 60 min. Em seguida, o
material será infiltrado em solução de metanol 100% e resina Lowicryl MonoStep
na proporção de 2:1 e o material incubado a -20ºC, por 16 horas. Em seguida, a
proporção de metanol será diminuída, e seguiu-se incubação em solução
metanol 100% e resina na proporção 1:1 a -20ºC por 16 horas. Após as etapas
de infiltração, o material será emblocado em resina pura e submetido a
incubação a -20ºC pelo período de 48 a 72 horas sob incidência de
luz
ultravioleta UVA (360 nm), para que ocorra a polimerização da resina. As
amostras serão então cortadas em um em um ultramicrótomo Leica modelo M6,
de modo a se obter cortes ultrafinos que foram coletados em grades de níquel.
Para a marcação das grades com anticorpo de interesse, estas serão incubadas
por 30 min em solução de cloreto de amônio 50 mM em PBS. Em seguida as
grades
serão
incubadas
por
30
min
em
solução
de
bloqueio
para
imunolocalização e incubadas por 1 hora nesta mesma solução contendo o
anticorpo primário diluído 1:20. Após duas lavagens, o material será incubado
por 1 hora com o anticorpo secundário anti IgG de coelho conjugado a partículas
de ouro diluido 1:20. Em seguida as grades serão lavadas em solução de
bloqueio para imunolocalização e em água destilada. Finalmente as grades
serão contrastadas por 30 min com acetato de uranila (5% em água) e por 2 min
em citrato de chumbo e então observadas no Microscópio Eletrônico de
Transmissão JEOL modelo 1200EXII de 80 kV (Centro de Microscopia
Eletrônica, UFPR).
Análise estatística. Para as análises estatísticas serão empregados testes T
não
pareados,
pareados
não-paramétrico
(teste
de
Wilcoxon)
para
comparações dentro do mesmo grupo, e o teste não pareado não-paramétrico
(teste de Mann-Whitney) para comparação entre os diferentes grupos, de
acordo com a necessidade. Os dados serão analisados no programa de
computador PRISM (versão 4.0, GraphPad, San Diego, USA). Valores de p<
0,05 serão considerados significativos.
d- Relação da pesquisa com trabalhos realizados anteriormente
Trabalhos anteriores de grande parte do Núcleo foram centrados nos
mecanismos de regulação da expressão gênica em T.cruzi, destacando-se o
papel de grânulos de RNA na modulação pós-transcricional. Desta forma o
estudo de grânulos de RNA, seja grânulos de estresse seja P-bodies, passou a
ser um interesse da equipe em outros modelos de estudo no Núcleo (por
exemplo, células tronco) e no processo de infecção de células de mamífero
pelo T.cruzi. Dados preliminares indicam que células tronco apresentam um
comportamento bastante peculiar no sentido em que grânulos de estresse são
muito mais abundantes do que em células diferenciadas. Considerando o
importante papel de grânulos de RNA na modulação pós-transcricional da
expressão gênica é interessante determinar seu papel no processo de
infecção. Assim, a comparação de um patógeno que utiliza a maquinaria
celular (vírus dengue) e outro que utiliza sua própria maquinaria (T.cruzi) para a
síntese protéica proverá novos dados para o nosso conhecimento do processo
de patogênese na dengue e na doença de Chagas. Soma-se ainda o interesse
de determinar o efeito espacial da ocupação do citoplasma por estes
patógenos na geração de grânulos de RNA e consequentemente na modulação
da expressão gênica celular. A análise ribonômica (sequenciamento dos RNAs)
contidos nas células infectadas e não infectadas permitirá determinar se vias
metabólicas específicas são afetadas pelo processo de patogênese.
No caso da pesquisa envolvendo os exossomos, a pesquisa é nova para os
componentes do Núcleo, mas os modelos biológicos por nós estudados
prestam-se de maneira muito positiva para a compreensão do papel dos
exossomos no processo de patogênese tanto da dengue como na doença de
Chagas. Resultados preliminares permitiram estabelecer a metodologia para
isolamento de exossomos de células dendriticas e assim a comparação de
células infectadas e não infectadas abre novas perspectivas para a
compreensão do papel de exossomos como mediadores do processo de
infecção (teriam os exossomos um papel parácrino na infecção?). No caso
particular do modelo dengue, esta questão é bastante estimulante em vista das
diferenças biológicas das cepas que serão estudadas.
Assim, a presente proposta é baseada essencialmente nos modelos de
trabalho dos pesquisadores do Núcleo, mas trazendo novos aspectos e
metodologias à pesquisa. É importante destacar que a maior parte dos
aspectos abordados na pesquisa proposta são originais e não se tratam de
simples contribuições aditivas a dados já existentes na literatura.
e- Impactos
ambientais,
econômicos,
tecnológicos e/ou de inovação
avanços
científicos,
A presente proposta não apresenta qualquer impacto ambiental ou econômico
(pelo menos a curto prazo). Todavia carrega embutida uma grande perspectiva
de avanços científicos tanto para a compreensão dos processos de patogênese
da dengue e do T.cruzi como na relação patógeno-hospedeiro. Há também o
grande potencial de geração de novos conhecimentos sobre o papel de
grânulos de RNA na modulação da expressão gênica de células de mamíferos
infectadas com diferentes patógenos. Adicionalmente, a área que trata do
estudo dos exossomos e seu papel na interação célula-célula no hospedeiro e
na interação célula do hospedeiro-patógeno, será bastante enriquecida.
É importante destacar que esta proposta apresenta um grande efeito
multiplicador no tocante à formação de recursos humanos capacitados nas
tecnologias de ponta da biologia moderna, e seu impacto regional não pode ser
negligenciado em virtude da carência de grupos de pesquisa nas áreas de
estudo no Estado do Paraná.
f- Proposta orçamentária e justificativa
A maior parte dos recursos solicitados refere-se a despesas de custeio, sejam
materiais de consumo, sejam passagens e diárias para as atividades do Núcleo
ou então serviços de terceiros para eventuais reparos nos equipamentos
danificados e despesas acessórias de importação. A destinação da maior parte
dos recursos para consumo é justificada em virtude do elevado custo dos
reagentes e kits para sequenciamento e amplificação de ácidos nucleicos,
anticorpos marcados para citometria e microscopia confocal e reagentes para
análise proteômica, sendo que a maior parte destes reagentes será adquirida
por importação direta. Assim, previmos gastos relativos às despesas
acessórias de importação. Parte dos insumos será adquirida diretamente no
mercado nacional, principalmente vidraria descartável para cultivo de células,
ponteiras, tubos de centrifugação, meios de cultivo, soro fetal bovino de padrão
cultura de tecidos, reagentes químicos diversos para o preparo de meios e
soluções. A necessidade de equipamentos é importante e prioritária para a
equipe da Universidade Estadual de Londrina, mais especificamente a
aquisição de um citômetro de fluxo, o que permitiria que parte dos
experimentos fosse executado diretamente in loco, e um PCR em tempo real
(Real Time PCR) que permitiria que os trabalhos de quantificação de mRNAs
específicos fossem feitos em Londrina, sem a necessidade de deslocamento
dos pesquisadores até Maringá ou até Curitiba (que é o que ocorre
atualmente).É importante ressaltar que estes dois equipamentos não existem
na UEL e certamente beneficiariam outros grupos de pesquisa não associados
diretamente ao Núcleo desta proposta ao Pronex.
g- Cronograma detalhado de execução do projeto
ATIVIDADE DESENVOLVIDA
Isolamento de células dendriticas
Isolamento e diferenciação de células tronco
Infecção de células com T. cruzi
Infecção de células com vírus dengue
Caracterização de grânulos de RNA em células
dendriticas
Caracterização de grânulos de RNA em células tronco
Isolamento de exossomos de células não infectadas
Isolamento de exossomos de células infectadas
Infecção de células nas presença de exossomos
Sequenciamento dos RNAs extraídos de exossomos
Análise proteômica (Orbitrap) dos exossomos
Seleção dos genes de interesse e análise bioinformática
Clonagem e expressão dos genes de interesse
Produção de antisoros (policlonais e monoclonais) contra
as proteínas de interesse
Estudos de localização celular e ultra-estrutura por
microscopia confocal de fluorescência e por microscopia
de transmissão dos exossomos.
*INICIO PREVISTO JULHO DE 2012
SEMESTRE*
1 2 3 4 5 6
REFERÊNCIAS BIBLIOGRÁFICAS
Abrahamsohn IA, Coffman RL. Cytokine and nitric oxide regulation of the
immunosuppression in Trypanosoma cruzi infection. J Immunol; 155:3955-63,
1995.
Alba Soto CD, Mirkin GA, Solana ME, Gonzalez Cappa SM. Trypanosoma cruzi
infection modulates in vivo expression of major histocompatibility complex class
II molecules on antigen-presenting cells and T-cell stimulatory activity of
dendritic cells in a strain-dependent manner. Infect Immun; 71:1194-9, 2003
Alba Soto CD, Solana ME, Poncini CV, Pino-Martinez AM, Tekiel V, GonzalezCappa SM. Dendritic cells devoid of IL-10 induce protective immunity against
the protozoan parasite Trypanosoma cruzi. Vaccine; 28:7407-13, 2010.
Anderson P & Kedersha N. Stress granules: the Tao of RNA triage. J Cell Biol
172, 803 – 808, 2006.
Anderson P & Kedersha N. Stress granules: the Tao of RNA triage. Trends
Biochem Sci 33, 141 – 150, 2007.
Azevedo LC, Pedro MA, Laurindo FR. Circulating microparticles as therapeutic
targets in cardiovascular diseases. Recent Pat Cardiovasc Drug Discov.
2(1):41-51, 2007.
Banchereau J, Pulendran B, Steinman R, Palucka K. Will the making of
plasmacytoid dendritic cells in vitro help unravel their mysteries? J Exp Med.
192(12):F39-44, 2000.
Bianco NR, Kim SH, Morelli AE, Robbins PD.Modulation of the immune
response using dendritic cell-derived exosomes. Methods Mol Biol. 380:443-55,
2007
Bishop NE. Dynamics of endosomal sorting. Int Rev Cytol. 232:1-57, 2003.
Bixby LM, Tarleton RL. Stable CD8+ T cell memory during persistent
Trypanosoma cruzi infection. J Immunol, 181:2644-50, 2008.
Bonaldo M.C.; Souto-Padron T.; de Souza W.; Goldenberg S. Cell-substrate
adhesion during Trypanosoma cruzi differentiation. J. Cell Biol., 106: 13491358, 1988.
Bordignon J, Probst CM, Mosimann AL, et. al. Expression profile of interferon
stimulated genes in central nervous system of mice infected with dengue virus
Type-1. Virology 377, 319-329, 2008.
Bruno S, Grange C, Deregibus MC, Calogero RA, Saviozzi S, Collino R,
Morando L, Busca A, Falda M, Bussolati B, Tetta C, Camussi G. Mesenchymal
stem cell-derived microvesicles protect against acute tubular injury. J Am Soc
Nephrol 20:1053-67, 2009.
Camussi G, Deregibus MC, Tetta C. Paracrine/endocrine mechanism of stem
cells on kidney repair: role of microvesiclemediated transfer of genetic
information. Curr Opin Nephrol Hypertens 19:7-12, 2010.
Cho JA, Yeo DJ, Son HY, Kim HW, Jung DS, Ko JK, Koh JS, Kim YN, Kim CW.
Exosomes: a new delivery system for tumor antigens in cancer immunotherapy.
Int J Cancer. 20;114(4):613-22, 2005.
Contreras VT, Araujo-Jorge TC, Bonaldo MC, Thomaz N, Barbosa HS,
Meirelles Mde N, et al. Biological aspects of the Dm 28c clone of Trypanosoma
cruzi after metacyclogenesis in chemically defined media. Mem Inst Oswaldo
Cruz; 83:123-33, 1988.
Contreras VT, Salles JM, Thomas N, Morel CM, Goldenberg S. In vitro
differentiation of Trypanosoma cruzi under chemically defined conditions. Mol
Biochem Parasitol; 16:315-27, 1985.
de Alencar BC, Persechini PM, Haolla FA, de Oliveira G, Silverio JC, LannesVieira J, et al. Perforin and gamma interferon expression are required for CD4+
and CD8+ T-cell-dependent protective immunity against a human parasite,
Trypanosoma cruzi, elicited by heterologous plasmid DNA prime-recombinant
adenovirus 5 boost vaccination. Infect Immun. 77:4383-95, 2009.
De Carvalho Bittencourt M, Martial J, Cabié A, Thomas L, Césaire R.
Decreased peripheral dendritic cell numbers in dengue virus infection. J Clin
Immunol. 32(1):161-72, 2012.
Denzer K, van Eijk M, Kleijmeer MJ, Jakobson E, de Groot C, Geuze HJ.
Follicular dendritic cells carry MHC class II-expressing microvesicles at their
surface. J Immunol. 1;165(3):1259-65, 2000.
Di Nicola M, Carlo-Stella C, Magni M, Milanesi M, Longoni PD, Matteucci P,
Grisanti S, Gianni AM. Human bone marrow stromal cells suppress Tlymphocyte proliferation induced by cellular or nonspecific mitogenic stimuli.
Blood 99:3838–43, 2002.
Dignat-George F, Boulanger CM. The many faces of endothelial microparticles.
Arterioscler Thromb Vasc Biol. 31(1):27-33, 2011.
Dominici M.; Le Blanc K; Mueller I; Slaper-Cortenbach I; Marini F; Krause D;
Deans R; Keating A; Prockop DJ; Horwitz E. Minimal criteria for defining
multipotent mesenchymal stromal cells. The International Society for Cellular
Therapy position statement. Cytotherapy 8(4):315-7, 2006.
Emara MM & Brinton MA. Interaction of TIA-1/TIAR with West Nile and dengue
virus products in infected cells interferes with stress granule formation and
processing body assembly. PNAS 104, 9041-9046, 2007.
Février B, Raposo G. Exosomes: endosomal-derived vesicles shipping
extracellular messages. Curr Opin Cell Biol. 16(4):415-21, 2004.
Goronzy JJ, Weyand CM. Developments in the scientific understanding of
rheumatoid arthritis. Arthritis Res Ther, 11:249, 2009.
Harding C, Heuser J, Stahl P. Receptor-mediated endocytosis of transferrin and
recycling of the transferrin receptor in rat reticulocytes. J Cell Biol. 97(2):329-39,
1983.
Hatcher FM, Kuhn RE, Cerrone MC, Burton RC. Increased natural killer cell
activity in experimental American trypanosomiasis. J Immunol 127:1126-30,
1981.
Hatcher FM, Kuhn RE. Destruction of Trypanosoma cruzi by Natural killer cells.
Science 218:295-6, 1982.
Hayden H, Friedl J, Dettke M, Sachet M, Hassler M, Dubsky P, BachleitnerHofmann T, Gnant M, Stift A. Cryopreservation of monocytes is superior to
cryopreservation of immature or semi-mature dendritic cells for dendritic cellbased immunotherapy. J Immunother. 32(6):638-54, 2009.
Holetz FB, Alves LR, Probst CM, et. al. Protein and mRNA content of TcDHH1containing mRNPs in Trypanosoma cruzi. FEBS Journal 277, 3415- 3426,
2010.
Holetz FB, Correa A, Avila AR, et. al. Evidence of P-body-like structures in
Trypanosoma cruzi. BBRC 356, 1062-1067, 2007.
James SL, Kipnis TL, Sher A, Hoff R. Enhanced resistance to acute infection
with Trypanosoma cruzi in mice treated with an interferon inducer. Infect Immun
35:588-93, 1982.
Kedersha N & Anderson P. Stress granules: sites of mRNA triage that regulate
mRNA stability and translatability. Biochem. Soc. Trans. 30, 963 – 969, 2002.
Kierszenbaum F, de Diego JL, Fresno M, Sztein MB. Inhibitory effects of the
Trypanosoma cruzi membrane glycoprotein AGC10 on the expression of IL-2
receptor chains and secretion of cytokines by subpopulations of activated
human T lymphocytes. Eur J Immunol; 29:1684-91, 1999.
Kierszenbaum F, Sonnenfeld G. Characterization of the antiviral activity
produced during Trypanosoma cruzi infection and protective effects of
exogenous interferon against experimental Chagas' disease. J Parasitol;
68:194-8, 1982.
Kim SH, Bianco NR, Shufesky WJ, Morelli AE, Robbins PD. Effective treatment
of inflammatory disease models with exosomes derived from dendritic cells
genetically modified to express IL-4. J Immunol. 179(4):2242-9, 2007.
Kress Y, Bloom BR, Wittner M, Rowen A, Tanowitz H. Resistance of
Trypanosoma cruzi to killing by macrophages. Nature; 257:394-6, 1975.
Lai RC, Arslan F, Lee MM, Sze Ns, Choo A, Chen T, Salto-Tellez M, Timmers
L, Lee CN, El Oakley R, Pasterkamp G, De Kleijn D, Lim Sk. Exosomes
secreted by MSC reduces myocardial ischemia/reperfusion injury. Stem Cell
Res 4:214-22, 2010.
Lai RC, Chen TS, Lim SK. Mesenchymal stem cell exosome: a novel stem cellbased therapy for cardiovascular disease. Regen Med. 6(4):481-92, 2011.
Li W, Li Y, Kedersha N, et. al. Cell proteins TIA-1 and TIAR interact with the 3'
stem-loop of the West Nile virus complementary minus-strand RNA and
facilitate virus replication. J Virol 76, 11989-12000, 2002.
Li XB, Zhang ZR, Schluesener HJ, Xu SQ. Role of exosomes in immune
regulation. J Cell Mol Med. 10(2):364-75, 2006.
Libraty DH, Pichyangkul S, Ajariyakhajorn C, Endy TP, Ennis FA.Human
dendritic cells are activated by dengue virus infection: enhancement by gamma
interferon and implications for disease pathogenesis. J Virol. 75(8):3501-8,
2001.
Loomis RJ, Holmes DA, Elms A, Solski PA, Der CJ, Su L. Citron kinase, a
RhoA effector, enhances HIV-1 virion production by modulating exocytosis.
Traffic. 7(12):1643-53, 2006.
Ludwig AK, Giebel B. Exosomes: small vesicles participating in intercellular
communication. Int J Biochem Cell Biol. 44(1):11-5, 2012.
Luplertlop N, Missé D, Bray D, Deleuze V, Gonzalez JP, Leardkamolkarn V,
Yssel H, Veas F. Dengue-virus-infected dendritic cells trigger vascular leakage
through metalloproteinase overproduction. EMBO Rep. Nov;7(11):1176-81,
2006. Erratum in: EMBO Rep. 2006 Dec;7(12):1290. Luplerdlop, Natthanej
[corrected to Luplertlop, Natthanej].
Manickasingham SP, Edwards AD, Schulz O, Reis e Sousa C. The ability of
murine dendritic cell subsets to direct T helper cell differentiation is dependent
on microbial signals. Eur J Immunol, 33:101-7, 2003.
Martin DL, Murali-Krishna K, Tarleton RL. Generation of Trypanosoma cruzispecific CD8+ T-cell immunity is unaffected by the absence of type I interferon
signaling. Infect Immun; 78:3154-9, 2010.
McInerney GM, Kedersha NL, Kaufman RJ, et. al. Importance of eIF2alpha
phosphorylation and stress granule assembly in alphavirus translation
regulation. Mol Bio Cell 16, 3753-3763, 2005.
Meirelles LS, Fontes AM, Covas DT, Caplan AI. Mechanisms involved in the
therapeutic properties of mesenchymal stem cells. Cytok Growth Fac Rev,
20:419-427, 2009.
Mignot G, Roux S, Thery C, Ségura E, Zitvogel L. Prospects for exosomes in
immunotherapy of cancer. J Cell Mol Med. 10(2):376-88, 2006
Miroux B, Walker JE. Over-production of proteins in Escherichia coli: Mutant
hosts that allow synthesis of some membrane proteins and globular proteins at
high levels. J. Mol. Biol. 260:289–298, 1996.
Moore MJ. From birth to death: the complex lives of eukaryotic mRNAs.
Science 309, 1514 – 1518, 2005.
Morel O, Toti F, Hugel B, Freyssinet JM. Cellular microparticles: a disseminated
storage pool of bioactive vascular effectors. Curr Opin Hematol 11:156-64,
2004.
Moussion C, Girard JP. Dendritic cells control lymphocyte entry to lymph nodes
through high endothelial venules. Nature. 479(7374):542-6, 2011.
Naik SH. Demystifying the development of dendritic cell subtypes, a little.
Immunol Cell Biol. 86(5):439-52, 2008.
Navarro M, Oliveira CC, Zanchin NIT, Guimaraes BG. Insights into the
Mechanism of Progressive RNA Degradation by the Archaeal Exosome. J. Biol.
Chem., 283(20):14120-14131, 2008.
Navarro-Sánchez E, Desprès P, Cedillo-Barrón L Innate immune responses to
dengue virus. Arch Med Res. 36(5):425-35, 2005.
O'Keeffe M, Hochrein H, Vremec D, Caminschi I, Miller JL, Anders EM, Wu L,
Lahoud MH, Henri S, Scott B, Hertzog P, Tatarczuch L, Shortman K. Mouse
plasmacytoid cells: long-lived cells, heterogeneous in surface phenotype and
function, that differentiate into CD8(+) dendritic cells only after microbial
stimulus J Exp Med. 196(10):1307-19, 2002.
Palucka K, Ueno H, Fay J, Banchereau J.Dendritic cells and immunity against
cancer. J Intern Med. 269(1):64-73, 2011.
Poncini CV, Alba Soto CD, Batalla E, Solana ME, Gonzalez Cappa SM.
Trypanosoma cruzi induces regulatory dendritic cells in vitro. Infect Immun;
76:2633-41, 2008.
Poncini CV, Gimenez G, Pontillo CA, Alba-Soto CD, de Isola EL, Piazzon I, et
al. Central role of extracellular signal-regulated kinase and Toll-like receptor 4 in
IL-10 production in regulatory dendritic cells induced by Trypanosoma cruzi. Mol
Immunol; 47:1981-8, 2010.
Rajendran L, Honsho M, Zahn TR, Keller P, Geiger KD, Verkade P, Simons K.
Alzheimer's disease beta-amyloid peptides are released in association with
exosomes. Proc Natl Acad Sci U S A. 25;103(30):11172-7, 2006.
Raposo G, Nijman HW, Stoorvogel W, Liejendekker R, Harding CV, Melief CJ,
Geuze HJ. B lymphocytes secrete antigen-presenting vesicles. J Exp Med.
183(3):1161-72, 1996.
Ratajczak J, Miekus K, Kucia M, Zhang J, Reca R, Dvorak P, Ratajczak Mz.
Embryonic stem cell-derived microvesicles reprogram hematopoietic
progenitors: evidence for horizontal transfer of mRNA and protein delivery.
Leukemia 20: 847-56, 2006.
Rebelatto, CK; Aguiar AM; Moretão MP; Senegaglia AC; Hansen P; Barchiki F;
Oliveira J; Martins J; Kuligovski C; Masur F; Christofis A; Amaral VF; Brofman
PS; Goldenberg S; Nakao LS; Correa A. Dissimilar differentiation of
mesenchymal stem cells from bone marrow, umbilical cord blood, and adipose
tissue. Exp Biol Med 233(7):901-13, 2008.
Rodrigues MM, Alencar BC, Vasconcelos JR. Acquired immunity against
Trypanosoma cruzi infection and vaccine development. In A. R. L. Teixeira, M.
C. Vinaud, and A. M. Castro (ed.). Emerging Chagas disease Bentham Science
Publishers, New York, NY; 1st ed. Bentham Science Publishers, New York,
NY:94–103, 2009.
Sales PA, Jr., Golgher D, Oliveira RV, Vieira V, Arantes RM, Lannes-Vieira J, et
al. The regulatory CD4+CD25+ T cells have a limited role on pathogenesis of
infection with Trypanosoma cruzi. Microbes Infect; 10:680-8, 2008.
Sanderson CJ, de Souza W. A morphological study of the interaction between
Trypanosoma cruzi and rat eosinophils, neutrophils and macrophages in vitro. J
Cell Sci; 37:275-86, 1979.
Sanderson CJ, Lopez AF, Moreno MM. Eosinophils and not lymphoid K cells kill
Trypanosoma cruzi epimastigotes. Nature; 268:340-1, 1977.
Sheth U & Parker R. Decapping and decay of messenger RNA occur in
cytoplasmic processing bodies. Science 300, 805 -808, 2003.
Shortman K, Naik SH. Steady-state and inflammatory
development. Nat Rev Immunol. 7(1):19-30, 2007.
dendritic-cell
Silveira GF, Meyer F, Delfraro A, Mosimann AL, Coluchi N, Vasquez C, Probst
CM, Báfica A, Bordignon J, Dos Santos CN. Dengue virus type 3 isolated from
a fatal case with visceral complications induces enhanced proinflammatory
responses and apoptosis of human dendritic cells. J Virol 85, 5374-5383, 2011.
Simpson RJ, Lim JW, Moritz RL, Mathivanan S. Exosomes: proteomic insights
and diagnostic potential. Expert Rev Proteomics. 6(3):267-83, 2009.
Singer NG; Caplan AI. Mesenchymal stem cells: mechanisms of inflammation.
Annu Rev Pathol Mech Dis 6:457-78, 2011.
Smetana JHC, Oliveira CLP, Jablonka W, Carneiro FRG, Montero-Lomeli M,
Torriani I, Zanchin NIT. Low resolution structure of the human α4 protein
(IgBP1) and stability of α4 and of its yeast orthologue Tap42. BBA Protein and
Proteomics, 1764(4): 724-734, 2006.
Steinman RM, Cohn ZA. Identification of a novel cell type in peripheral lymphoid
organs of mice. I. Morphology, quantitation, tissue distribution. J Exp Med.
137(5):1142-62, 1973.
Steinman RM. The dendritic cell system and its role in immunogenicity. Annu
Rev Immunol. 9:271-96, 1991.
Studier FW, Moffatt BA. Use of bacteriophage T7 RNA polymerase to direct
selective high-level expression of cloned genes. J. Mol. Biol. 189(1):113-130,
1986.
Sze Sk, DE Kleijn DP, Lai RC, Khia Way Tan E, Zhao H, Yeo KS, Low Ty, Lian
Q, Lee CN, Mitchell W, El Oakley RM, Lim SK. Elucidating the secretion
proteome of human embryonic stem cell-derived mesenchymal stem cells. Mol
Cell Proteom 6:1680-89, 2007.
Tarleton RL. Regulation of immunity in Trypanosoma cruzi infection. Exp
Parasitol; 73:106-9, 1991.
Taylor DD, Gercel-Taylor C. Exosomes/microvesicles: mediators of cancerassociated immunosuppressive microenvironments. Semin Immunopathol.
33(5):441-54, 2011.
Teixeira D, Sheth, U, Valencia-Sanchez, MA, et. al. Processing bodies require
RNA for assembly and contain nontranslating mRNAs. RNA 11, 371-382, 2005.
Umareddy I, Tang KF, Vasudevan SG, Devi S, Hibberd ML, Gu F. Dengue virus
regulates type I interferon signalling in a strain-dependent manner in human cell
lines. J Gen Virol. 89(Pt 12):3052-62, 2008.
van Kooten C, Lombardi G, Gelderman KA, Sagoo P, Buckland M, Lechler R,
Cuturi MC. Dendritic cells as a tool to induce transplantation tolerance:
obstacles and opportunities. Transplantation. 91(1):2-7, 2011.
Vella LJ, Sharples RA, Lawson VA, Masters CL, Cappai R, Hill AF. Packaging
of prions into exosomes is associated with a novel pathway of PrP processing. J
Pathol. 211(5):582-90, 2007.
Vella LJ, Sharples RA, Nisbet RM, Cappai R, Hill AF. The role of exosomes in
the processing of proteins associated with neurodegenerative diseases. Eur
Biophys J. 37(3):323-32, 2008.
Verfaillie C M. Adult stem cells: assessing the case for pluripotency. Trends Cell
Biol 12(11):502-8, 2002.
Wagner S, Klepsch MM, Schlegel S, Appel A, Draheim R, Tarry M, Högbom M,
van Wijk KJ, Slotboom DJ, Persson JO, de Gier JW. Tuning Escherichia coli for
membrane protein overexpression. Proc Natl Acad Sci U S A. 105(38):1437114376, 2008.
Wu SJ, Grouard-Vogel G, Sun W, Mascola JR, Brachtel E, Putvatana R, Louder
MK, Filgueira L, Marovich MA, Wong HK, Blauvelt A, Murphy GS, Robb ML,
Innes BL, Birx DL, Hayes CG, Frankel SS. Human skin Langerhans cells are
targets of dengue virus infection. Nat Med. 6(7):816-20, 2000.
Zhang L, Tarleton RL. Characterization of cytokine production in murine
Trypanosoma cruzi infection by in situ immunocytochemistry: lack of association
between susceptibility and type 2 cytokine production. Eur J Immunol; 26:102-9,
1996.
Zhu W, Huang L, Li Y, Zhang X, Gu J, Yan Y, Xu X, Wang M, Qian H, Xu W.
Exosomes derived from human bone marrow mesenchymal stem cells promote
tumor growth in vivo. Cancer Lett. 1;315(1):28-37, 2011.