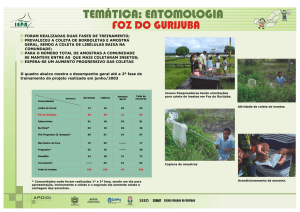
27 e 28 de setembro de 2012
Porto Alegre, RS
DIFERENCIAÇÃO ENTRE OS SUBTIPOS DE HERPESVÍRUS BOVINO TIPO
5 POR AMPLIFICAÇÃO DE UM FRAGMENTO DO GENE UL27
Felipe Fontoura¹, Gustavo Strelczuk², Fabrício Campos³, Paulo Roehe4(orient)
¹Bolsista Probic/Fapergs, Fepagro Saúde Animal – Eldorado do Sul, Graduando em
Biomedicina - Universidade Federal de Ciências da Saúde de Porto Alegre (UFCSPA);
²Bolsista Probic/UFRGS, Instituto de Ciências Básicas da Saúde – Porto Alegre,
Graduando em Ciências Veterinárias – Universidade Federal do Rio Grande do Sul
(UFRGS); ³Bolsista CAPES, Instituto de Ciências Básicas da Saúde – Porto Alegre,
Doutorando em Ciências Veterinárias - Universidade Federal do Rio Grande do Sul
(UFRGS); 4Pesquisador, Fepagro Saúde Animal – Eldorado do Sul;
E-mail: [email protected], [email protected]
O herpesvírus bovino tipo 5 (BoHV-5) é um alfaherpesvírus associado a meningoencefalites de curso geralmente fatal em bovinos Previamente classificado como um
subtipo do herpesvírus bovino tipo 1 (BoHV-1), o BoHV-5 foi reclassificado com base
em características biológicas e genômicas que o distinguem dos isolados respiratórios e
genitais do BoHV-1. Surtos de meningo-encefalite pelo BoHV-5 têm sido
frequentemente descritos, principalmente no Brasil e Argentina. Os sinais clínicos da
enfermidade incluem tremores, andar em círculos, incoordenação, nistagmo, bruxismo,
ataxia, convulsões e depressão profunda seguida de morte. As amostras de BoHV-5 são
subdivididas em três subtipos, a, b e c. Até o momento, não existe uma associação
conhecida entre patogenicidade e subtipos de BoHV-5. Entretanto, essa subdivisão é
importante para que possa ser ampliado o conhecimento sobre o comportamento de
diferentes subtipos do vírus em sua interrelação com o hospedeiro. Para isso, é
necessário que sejam construídos instrumentos que permitam efetuar a subtipificação de
amostras com praticidade. Este trabalho tem como objetivo analisar um fragmento do
gene UL27 que em estudos prévios revelou um “hot spot” em algumas amostras de
BoHV-5 e avaliar seu potencial para permitir a discriminação entre subtipos. Os
isolados de BoHV-5 utilizados no trabalho foram provenientes do banco de amostras do
Laboratório de Virologia da UFRGS e IPVDF. A multiplicação das amostras de vírus
foi realizada utilizando células da linhagem de rim de bovino “CRIB”. A extração de
DNA foi feita com fenol seguindo protocolos usuais. A padronização da reação em
cadeia da polimerase (PCR) foi realizada utilizando amostras de BoHV-5 anteriormente
caracterizadas. Após a padronização, todas as amostras foram amplificadas e os
produtos de PCR, com o fragmento de tamanho esperado (549 pares de base) foram
clonados e sequenciados. A presença do “hot spot” foi confirmada nas amostras dos
subtipos a e c; as amostras do subtipo b não apresentaram tal sítio. Até o momento,
nove de dez amostras analisadas foram classificadas nos subtipos “a” ou “c”, apenas
uma foi classificada como pertencente ao subtipo “b”. Estudos com base em outras
regiões do genoma serão a seguir realizados buscando a diferenciação entre os subtipos
“a” e “c”.
(Apoio: FAPERGS, FINEP & CNPq)