‐ UNIVERSIDADE DE SÃO PAULO ‐– ‐PÓS‐
GRADUAÇÃO GENÉTICA ‐ SCI1, UM NOVO INIBIDOR TECIDO‐ESPECÍFICO DA PROLIFERAÇÃO CELULAR RELACIONADO À SINALIZAÇÃO POR AUXINA Tese de Doutorado | Henrique C De Paoli Junho - 2010
SCI1, UM NOVO INIBIDOR
TECIDO-ESPECÍFICO DA PROLIFERAÇÃO
CELULAR RELACIONADO
À SINALIZAÇÃO POR AUXINA
Henrique Cestari De Paoli
Orientação, Profa. Dra. Maria Helena S. Goldman (USP-RP/BRASIL)
Co-Orientação, Prof. Yunde Zhao – PhD. (UCSD/USA)
Universidade de São Paulo
Faculdade de Medicina de Ribeirão Preto
Programa de Pós-Graduação em Genética
TESE DE DOUTORADO apresentada ao programa de Pós‐graduação da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo, como parte dos requisitos para a obtenção do Título de DOUTOR EM CIÊNCIAS, área de concentração Genética Geral Ribeirão Preto – SP – BRASIL
Junho - 2010
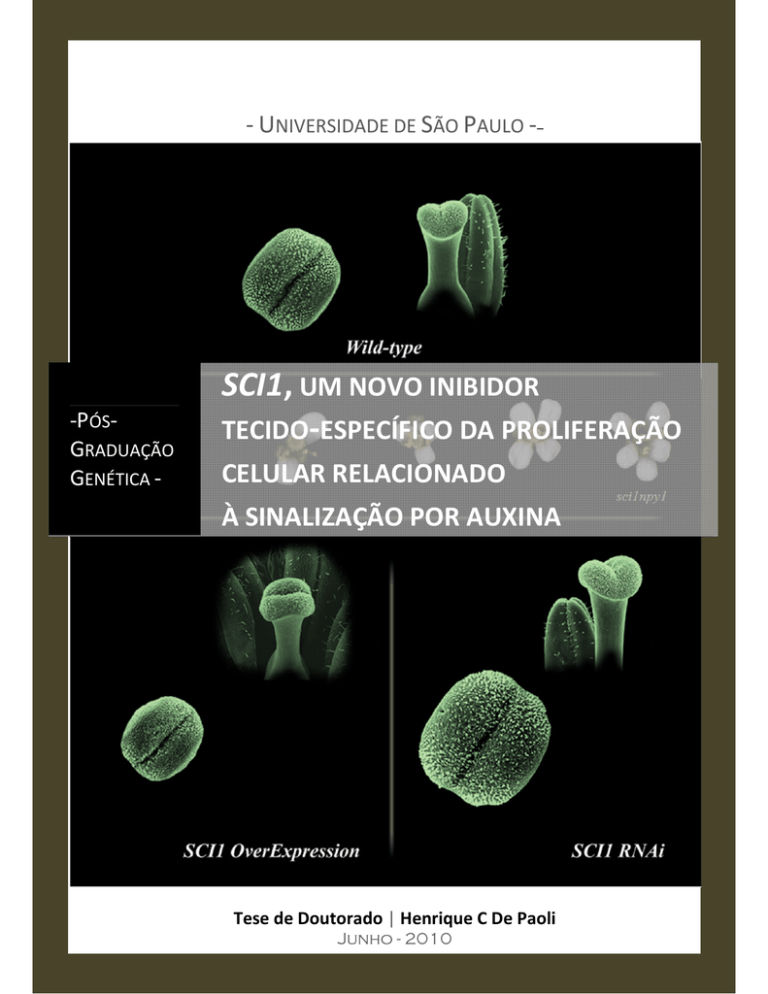
Ficha Catalográfica Henrique Cestari De Paoli DePaoli, H.C. Título: SCI1, UM NOVO INIBIDOR TECIDO‐ESPECÍFICO DA PROLIFERAÇÃO CELULAR RELACIONADO À SINALIZAÇÃO POR AUXINA. Ribeirão Preto: FMRP, 2010 Número de Páginas: #183# Tese de Doutorado apresentada ao Departamento de Genética da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo – USP Orientadora: Profa. Dra. Maria Helena S. Goldman Co‐Orientador: Prof. Yunde Zhao, PhD. As atividades apresentadas nesta Tese foram realizadas no Laboratório de Biologia Molecular de Plantas, da Faculdade de Filosofia Ciências e Letras da Universidade de São Paulo/BRASIL; e no Laboratório do Prof. Yunde Zhao, na “Section of Cell and Developmental Biology, University of California San Diego/CA‐USA”. Descrição da Capa A imagem da capa mostra uma microscopia eletrônica de varredura de estigmas no estágio ‐2 e pistilos no estágio 1 de plantas de Nicotiana tabacum selvagens (no topo; “wild‐type”), superexpressando SCI1 (canto inferior‐esquerdo; “SCI1 overexpression”) e silenciadas para SCI1 (canto inferior‐direito; SCI1 RNAi). Embora os estigmas estejam no mesmo estágio do desenvolvimento, os de SCI1 RNAi mostraram um aumento marcante do tecido estigmático, comparado com o selvagem, enquanto que um fenótipo oposto foi observado nas plantas de superexpressão. Adicionalmente, nas plantas de RNAi, o estilete elonga mais que nas plantas selvagens, posicionando o estigma acima das anteras e introduzindo em nossas plantas o que chamamos de heterostilia. Propõe‐
se que SCI1 seja um novo regulador tecido‐específico do ciclo celular que provavelmente modula a atividade de CDK, na parte superior do pistilo, através da interação com ciclinas do tipo A. As pequenas flores por detrás do título são de Arabidopsis thaliana e representam algumas variações florais de duas, três, quatro ou cinco pétalas que, às vezes, foram observadas no duplo mutante sci1npy1. The heavens declare the glory of GOD; the skies proclaim the work of His hands, day after day they pour forth speech; night after night they display KNOWLEDGE” Psalm 19:1‐2 A
Aggrraaddeecciim
meennttooss
Ao DEUS DE ISAQUE, ABRAÃO E JACÓ, que triunamente trabalhou em meu favor e me capacitou a
vencer cada etapa para esta vitória;
Aos meus pais, Fernando Antônio De Paoli e Mari Angela Cestari De Paoli pelo amor, incentivo,
por direcionar minhas decisões e, principalmente, pela compreensão e por serem porto seguro em
minha vida, amo vocês;
À Jaqueline Moisés Tobal, “carne da minha carne, osso dos meus ossos”, sem a qual meu ser
ainda seria manco, por cada incentivo, pelo apoio, pela compreensão, pela paciência, paciência,
paciência, paciência, amor e por ser “a melhor do mundo!”;
À Profa. Maria Helena Goldman, mais que uma orientadora, pelos ensinamentos e oportunidade
na Ciência, pelo zelo pessoal, pelas lutas em busca de sonhos, pela paciência e compreensão às
minhas limitações e, é claro, pelas discontrações;
Ao Prof. Dr. Yunde Zhao, que um dia disse “sim” a um simples email, e mudou a minha vida, por
cada ensinamento, pelo apoio e, principalmente, pela oportunidade e paciência;
À minha família, Carolina, Samuel e Leonardo Bortoletto, Felipe Cestari De Paoli, avôs, avós,
tios, tias, primos e primas, que torceram por mim, mesmo que de longe;
Aos Professores e ex-orientadores, que se empenharam para o meu crescimento, em especial ao
Dr. José Edson Fontes Figueiredo, instrutor dos meus primeiros passos, e às oportunidades do
Prof. Dr. Marcelo C. Dornelas e Dra. Maria Cristina Pranchevicius;
Aos membros do LBMP/USP do passado, pelo incentivo; aos do presente, pela participação ativa
no meu crescimento, pela amizade, companheirismo e paciência, em especial pela dedicação da
Dra. Andréa C. Quiapim no suporte ao sequenciamento de clones e; aos que chegam, com
contribuições modestas, meu muito obrigado e boa sorte, façam bom proveito do que
conquistamos;
Aos meus amigos de Rep, aos Manos da Bio, aos colegas de Departamento na Genética e na
Biologia, aos parceiros da Salinha e aos Futebolistas (Prof. Abel), que me permitiram aliviar as
pressões do dia a dia e se tornaram companheiros de uma jornada;
Ao Goldman´s Team, especialmente ao Prof. Dr. Gustavo Henrique Goldman, por ceder
equipamentos e representar um alicerce, sem o qual meu trabalho e conhecimento jamais seriam o
mesmo;
Aos membros do Laboratório do Prof. Yunde Zhao, pela receptividade, companheirismo, apoio e
carinho;
Aos amigos Brazucas, em San Diego/CA, os quais não tenho palavras para descrever minha
gratidão e carinho, em especial, à Katlin Massirer e Leonardo Karan Teixeira;
Aos Professores Lieven De Veylder (Ghent Univeristy); Mark Estelle, Julian Schroeder, Martin F.
Yanofsky, Laurie Smith, Robert Schmidt (UCSD); Elliot Meyerowitz (CalTech); Wagner Ferreira
dos Santos, Maria Cristina Roque Barreira, Constance Oliver, Maria Célia Jamur, Zilá Luz
Paulino Simões, Roy Edward Larson, Simone de Pádua Teixeira, Carlos Alberto Martinez y
Huaman, John Campbell McNamara (USP), por gentilmente cederem parte de seus equipamentos
e espaços físicos, apoiando a realização deste trabalho;
Aos Professores Luis Lamberti P da Silva, Adriana Hemerly, Jen Sheen, Joanne Chory, Jeffrey
Long, James Umen, Anthony Hunter e aos revisores do periódico The Plant Cell, por sugestões e
estimularem a discussão do trabalho;
Às agências de fomento FAPESP, CNPq, CAPES, Genética/FMRP, FAEPA e Biologia/FFCLRP,
por cada centavo que, suado, foi atenciosamente concedido, em fé de que seria bem aproveitado, e
aos que contribuíram de alguma forma nesta jornada, agradeço sem palavras.
ÍÍnnddiiccee
CONTEÚDO
PÁGINAS
Resumo da Tese
Thesis Abstract
1
4
Introdução
As espécies Nicotiana tabacum e Arabidopsis thaliana.
A reprodução de plantas no modelo vegetal N. tabacum
O transcriptoma no estigma e estilete
O desenvolvimento floral: enfoque no carpelo de N. tabacum.
A maquinaria da divisão celular em plantas
Uma visão da atuação da auxina no desenvolvimento do pistilo
7
9
9
13
15
18
23
Objetivos
27
Material e Métodos
Material Vegetal
Sequenciamento, primers e análises computacionais
Manipulação de ácidos nucléicos e construções gênicas
Extração de RNA
RT-PCR quantitativo (qRT-PCR), “semi-quantitativo” (RT-PCR) e
hibridização in situ
Importação dos dados de Microarranjo
Transformação de Plantas
Testes de interação genética
Experimentos de Microscopia
Coloração com GUS
Números de acesso de seqüências utilizadas
Mutantes de T-DNA de Arabidopsis analisados
30
32
32
34
35
Resultados e Discussão
Capítulo I: Um novo gene de estigma/estilete de Nicotiana tabacum, SCI1,
que codifica uma proteína rica em lisina e controla a divisão e diferenciação
celular durante o desenvolvimento
Resultados
Identificação do gene SCI1 e análise de sua localização subcelular
SCI1 é um gene específico de estigma/estilete regulado ao longo do
desenvolvimento
O gene SCI1 é expresso exclusivamente nos tecidos especializados do
estigma/estilete relacionados com a reprodução de plantas
O produto do gene SCI1 controla o tamanho do estigma
A proteína SCI1 é um regulador negativo do ciclo celular
SCI1 é expresso nos estágios iniciais do desenvolvimento do estigma/estilete
SCI1 controla a proliferação/diferenciação celular e influencia o
momento do desenvolvimento do estigma/estilete
43
36
38
38
39
40
41
41
42
44
45
45
50
50
52
55
58
61
A proteína SCI1 tem as Ciclinas do tipo A como suas parceiras de interação
SCI1 influencia os níveis de transcrição de alguns genes regulados por auxina
Discussão
SCI1 é um regulador tecido-específico do ciclo celular que influencia o
desenvolvimento do pistilo
SCI1 é um regulador do ciclo celular distinto dos inibidores de CDK
já descritos em plantas
SCI1 conecta a sinalização de auxina e o controle da
divisão/diferenciação celular?
Capítulo II: Caracterização do gene SCI1 na via de sinalização de auxina
do pistilo
Resultados
Identificação e análise de SCI1 em Arabidopsis: AtSCI1
Caracterização do locus At1g79200 (AtSCI1) de Arabidopsis thaliana
Análise da região promotora e de expressão do gene AtSCI1
SCI1 atua no desenvolvimento, dependente de auxina, do pistilo
Interação genética entre sci1 e, yuc2yuc6, pid336 e npy1 em Arabidopsis
Discussão
SCI1 em diferentes espécies vegetais: N. tabacum e A. thaliana
SCI1 e a via de sinalização de auxina no pistilo
SCI1 no controle do ciclo celular dependente de auxina
64
65
67
67
70
73
76
77
77
78
80
83
85
87
87
89
91
Considerações Finais
93
Referências
96
Anexos
109
Anexo 1 - Artigo submetido para publicação no periódico “The Plant Cell”
Anexo 2 - Curriculum Vitae
DePaoli,H.C
2010
DePaoli, H.C., 2010.
SCI1, UM NOVO INIBIDOR
TECIDO-ESPECÍFICO DA PROLIFERAÇÃO
CELULAR RELACIONADO
À SINALIZAÇÃO POR AUXINA
O
sucesso da reprodução de plantas depende do desenvolvimento apropriado dos
órgãos reprodutivos, o qual envolve redes regulatórias específicas. Realizou-se a
caracterização de um gene previamente desconhecido, identificado em uma
biblioteca subtrativa de estigmas/estiletes de Nicotiana tabacum. Esse gene codifica uma proteína
pequena, rica em lisina, a qual mostrou-se ser específica de pistilo e, mais precisamente, transcrita
nos tecidos especializados do estigma/estilete: zona secretória do estigma (SSZ) e o tecido
transmissor do estilete (STT). A seqüência da proteína, deduzida do cDNA, demonstrou ter dois
domínios putativos de interação com ciclina, 15 sítios preditos de fosforilação (p ≥ 96%) e um
putativo sinal de localização nuclear, que foi confirmado pela localização da proteína de fusão
com GFP na região intercromática/corpos nucleares. O maior nível de transcritos ocorre nos
estágios bem jovens do desenvolvimento floral, em que o estigma/estilete está se diferenciando.
Plantas transgênicas de RNAi e superexpressão (OE) de N. tabacum resultaram, notoriamente, em
estigmas com áreas aumentadas e reduzidas, respectivamente. Cortes longitudinais do
estigma/estilete maduro demonstraram que essa alteração em tamanho é uma conseqüência da
alteração no número de células, que está aumentado nas plantas de RNAi e diminuído nas plantas
OE. Juntamente com a demonstração de que esta proteína interage com ciclinas do tipo A, in vitro
e in vivo, pôde-se concluir que esta proteína é um regulador negativo do ciclo celular, que
provavelmente regula a atividade de CDK de forma tecido-específica. Com base nestes
resultados, este gene foi denominado SCI1, a partir de “Stigma/style Cell-cycle Inhibitor 1”. SCI1
é distinto dos inibidores de CDK já descritos em plantas e animais, propondo-se assim que este
gene seja o primeiro membro de uma nova classe de inibidores de CDK, aqui definida com TICK
(“Tissue-specific Inhibitors of CDK). As diferenças na divisão celular causadas por SCI1
afetaram o momento da diferenciação das células papilares, mostrando que sua diferenciação é
desencadeamento da diferenciação através do controle da proliferação celular. Baseado na
similaridade fenotípica entre as plantas de RNAi de N. tabacum e os pistilos de Arabidopsis
Resumo acoplada às divisões celulares que ocorrem no estigma, consistente com um papel de SCI1 no
‐ 2 ‐ DePaoli,H.C
2010
tratados com um inibidor do transporte polar de auxina (NPA), decidiu-se analisar a expressão de
três genes relacionados à via de auxina, ARF8, Aux/IAA13 e Aux/IAA19, em quatro plantas
transgênicas independentes de RNAi e OE. Todos os três genes foram significativamente
alterados, em até 5,1 vezes para Aux/IAA19, mostrando que SCI1 influencia a regulação
transcricional de alguns genes de resposta rápida à auxina. A caracterização do mutante sci1, uma
linhagem de inserção de T-DNA em Arabidopsis, também revelou um tecido estigmático maior
devido a um aumento no número de células, como ocorre em plantas de RNAi de N. tabacum.
Surpreendentemente, os pistilos de sci1 são bem semelhantes aos do duplo mutante yuc2yuc6, que
perderam a habilidade de sintetisar auxina, de forma adequada, neste tecido. Cortes longitudinais
e imagens de contraste de fase (DIC) mostraram que a aumentada estrutura estigmática de
yuc2yuc6, assim também como a aumentada estrutura estigmática dos sinalizadores de auxina
pid336 e npy1, são todas conseqüências do aumento no número de células. Coerentemente, o gene
AtSCI1 tem sua expressão diminuída nos mutantes yuc2yuc6, pid336 e npy1. Testes de interação
genética mostraram que sci1, npy1 e sci1npy1 têm uma estrutura estigmática bastante semelhante
entre si, enquanto que sci1 mostrou uma interação sinergística com yuc2yuc6 e pid336. Esses
resultados consistentemente enquadram SCI1 na sinalização de auxina, que controla a divisão
celular na parte superior do pistilo. Em conjunto, os resultados mostraram que SCI1 é um novo
inibidor de CDK, que atua como componente da via de transdução de sinal de auxina no
estigma/estilete, para controlar a proliferação e diferenciação celular, representando um efetor
Resumo molecular deste hormônio no desenvolvimento do pistilo.
‐ 3 ‐ DePaoli,H.C
2010
DePaoli, H.C., 2010.
SCI1, A NOVEL TISSUE-SPECIFIC
INHIBITOR OF CELL PROLIFERATION
RELATED TO
AUXIN SIGNALING
T
he success of plant reproduction depends on the appropriate development of the
reproductive organs that involve specific regulatory networks. We have undertaken
the characterization of apreviously unknown gene identified in a N. tabacum
stigma/style (S/S) subtracted cDNA library. This gene encodes a small lysine-rich protein which
was shown to be pistil-specific and, more precisely, transcribed in the reproductive specialized
tissues: stigmatic secretory zone - SSZ- and stylar transmitting tract - STT. The deduced protein
sequence has two putative cyclin interaction domains, 15 predicted phosphorylation sites (p≥96%)
and a putative nuclear localization signal, which was confirmed by localization of the GFP fusion
protein in the interchromatin/nuclear bodies. The highest transcript levels occur at the very early
stages of flower development, in which the S/S is differentiating. Transgenic RNAi and
overexpression (OE) N. tabacum plants resulted in stigmas with remarkably enlarged and reduced
areas, respectively. Longitudinal sections of the mature S/S clearly showed that this change in size
occurs as a consequence of the alteration in cell numbers, which is increased in RNAi plants and
decreased in OE plants. Together with the in vitro and in vivo interaction of this protein with Atype cyclinsit was possible to conclude that this protein is a negative cell cycle regulator, that
probably regulates the CDK activity I a tissue specific manner. Based on these results, this gene
was named SCI1, from Stigma/style Cell-cycle Inhibitor 1. SCI1 is distinct from the plant and
animals CDK inhibitors already described, and so it is proposed that it is the first member of a
new class of CDK inhibitors, here defined as TICK (Tissue-specific Inhibitors of CDK).
Differences on cell division caused by SCI1 affected the differentiation timing of the papillar
cells, showing that its differentiation is coupled to stigmatic cell division, which is consistent with
a role for SCI1 in triggering cell differentiation through cell proliferation control. Based on the
phenotypic similarity between the N. tabacum RNAi plants and the Arabidopsis pistils treated
three auxin related genes, ARF8, Aux/IAA13 and Aux/IAA19, in four independent RNAi and OE
plants. All three genes were significantly altered, up to 5.1 fold for Aux/IAA19, showing that
SCI1 influences the transcriptional regulation of some early auxin responsive genes. The
Abstract with an inhibitor of polar auxin transport (NPA), it has been decided to analyze the expression of
‐ 5 ‐ DePaoli,H.C
2010
characterization of the sci1 mutant, an Arabidopsis T-DNA insertion line, also revealed a bigger
stigmatic tissue due to an increased cell number, like the N. tabacum RNAi plants. Surprisingly,
the sci1 pistils are very similar to those of the yuc2yuc6 double mutants, which lack proper auxin
synthesis on this tissue. Longitudinal sections and DIC images showed the yuc2yuc6 enlarged
stigmatic tissue, as well as the enlarged stigmas of the auxin signalers pid336 and npy1 mutants,
are all consequences of the increased cell number. Coherently, the AtSCI1 gene is down-regulated
on yuc2yuc6, pid336 and npy1 mutants. Genetic interactions tests showed that sci1, npy1 and
sci1npy1 have very similar pistil structures, while sci1 showed synergistic interaction with
yuc2yuc6 and pid336. These results consistently enframe SCI1 in the auxin signaling that controls
cell division in the upper pistil. Taken together, the results showed that SCI1 is a new CDK
inhibitor, which acts as a component of the auxin signalling transduction pathway in S/S to
control cell proliferation/differentiation, representing a molecular effector of this hormone on
Abstract pistil development.
‐ 6 ‐ DePaoli,H.C.
2010
Introdução Geral
Henrique C. DePaoli 1,2 e Maria Helena S. Goldman 1
1
Introdução Departamento de Biologia, Faculdade de Filosofia, Ciências e Letras de Ribeirão Preto, Universidade de São Paulo,
Brazil 14040-901
2
PPG Genética, Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Brazil; 14049-900
‐ 8 ‐ DePaoli,H.C.
2010
As espécies Nicotiana tabacum e Arabidopsis thaliana. O termo angiosperma tem origem grega e vem da fusão de duas palavras: angeion, que
significa vaso, e sperma, que significa semente (Raven et al., 1996). As angiospermas são um
grupo de plantas com sementes distintas dos demais filos por terem um ciclo de vida
característico, e pela presença de flores e frutos. A maioria das plantas ao nosso redor é de
angiospermas, com uma grande variedade de espécies diferentes, que podem ser divididas em
monocotiledôneas (com apenas um cotilédone), eudicotiledôneas (dicotiledôneas – com dois
cotilédones) e as magnolideas (angiospermas basais com um ou mais cotilédones).
Dentre as famílias de eudicotiledôneas, encontram-se exemplares distintos, modelos de
estudo em genética de plantas, como Nicotiana tabacum e Arabidopsis thaliana. O modelo
vegetal Nicotiana tabacum, tem um genoma relativamente grande (de aproximadamente
4.500Mb), parcialmente sequenciado (NCBI ID: 13234), tem um ciclo de vida relativamente curto
(em média 6 meses de semente a semente), possui flores relativamente grandes e que permitem
auto-polinização, e cresce bem em climas tropicais. Em paralelo, o modelo vegetal Arabidopsis
thaliana, tem um genoma pequeno (125Mb), totalmente sequenciado e anotado (NCBI ID:9506),
a disponibilidade de um banco de mutantes de perda de função gênica (por inserção de T-DNA),
tem um ciclo de vida curto (em média 2 meses de semente a semente), possui flores bem
pequenas e que permitem a auto-polinização, e cresce bem em climas temperados. Essas
características fazem dessas plantas os dois principais modelos vegetais de estudo no mundo,
dependendo das vantagens e desvantagens associadas aos alvos de cada pesquisa.
A reprodução de plantas no modelo vegetal N. tabacum Em plantas, o desenvolvimento ocorre, em vários aspectos, após a embriogênese, com
diferentes órgãos/tecidos se diferenciando ao longo do seu ciclo de vida. Para tanto, existem
populações de células tronco, capazes de gerar outros tipos celulares, localizadas nos meristemas
da raiz e apical. No meristema apical, ocorre a diferenciação do meristema vegetativo em
meristema floral (detalhes no tópico “O desenvolvimento floral: enfoque no pistilo de N.
tabacum” abaixo) que originará a flor. Esta é responsável pela reprodução, transmitindo as
características da planta ao longo de um número ilimitado de gerações (Curtis, 1976).
As flores são compostas de órgãos estéreis e órgãos férteis. Há dois grupos de órgãos
estéreis, que são as sépalas e as pétalas (Curtis, 1976). O conjunto de sépalas é denominado cálice
e o conjunto de pétalas, corola (Esau, 1977). Os órgãos férteis são os estames e os pistilos. O
androceu é o conjunto de estames que, por sua vez, são constituídos pelo filete e pela antera,
1977; Figura 1). Os pistilos, órgãos reprodutivos femininos, são coletivamente chamados de
gineceu. Em N. tabacum o gineceu é formado a partir da fusão de dois carpelos, sendo
Introdução responsável pela formação e disseminação dos gametófitos masculinos (grãos-de-pólen) (Esau,
‐ 9 ‐ DePaoli,H.C.
2010
denominado pistilo, formado por estigma, estilete e ovário (Figura 2). O estigma, do tipo úmido, é
bilobado, sendo formado, em sua superfície, por uma epiderme com células papilares (Figura 2).
Logo abaixo da epiderme, observa-se uma zona secretória (SSZ) (Bell & Hicks, 1976) (Figura 2)
responsável pela produção do exudato, um secretado viscoso rico em açúcar, aminoácidos,
lipídeos e glicoproteínas (Labarca et al., 1970). Dando continuidade à zona secretória do estigma,
observa-se o tecido transmissor do estilete (STT), que conduzirá o tubo polínico, no processo de
fecundação, até o óvulo. Em torno do tecido transmissor, é encontrado um tecido parenquimatoso,
comumente denominado córtex (Figura 2). O córtex, tem a função de suporte (Raven et al., 1996).
Cada pistilo, em N. tabacum, possui um único feixe vascular localizado paralelamente ao tecido
transmissor no estilete.
O ovário, por sua vez, representa a porção mais densa das três partes do pistilo. Sua
epiderme não apresenta modificações especializadas como no estigma, e freqüentemente
apresenta estômatos (Mauseth, 1988). Algumas famílias apresentam uma hipoderme, além de um
considerável mesófilo, juntamente com o tecido vascular. Em N. tabacum, aproximadamente
2000 óvulos são produzidos pelo ovário (Huang & Russell, 1992), e se apresentam dispostos ao
longo de uma camada de células denominada placenta. Cada óvulo é unido à placenta por um
grupo limitado e estreito de células, denominado funículo, que determina a posição do óvulo em
relação à placenta. A partir do funículo, próximo à base do nucelo, duas camadas finas de células
se projetam acima deste, rodeando-o, denominadas integumento. Na extremidade dos
integumentos, uma pequena abertura é encontrada, denominada micrópila, por onde ocorre a
penetração do tubo polínico (Fahn, 1982). Uma célula do nucelo, usualmente uma das que estão
abaixo da camada de células mais externa, próxima à micrópila, se diferencia na célula mãe do
megásporo (Fahn, 1982). Esta passa por diferentes processos de crescimento, diferenciação e
Introdução divisão celular até atingir a maturação completa.
‐ 10 ‐ DePaoli,H.C.
2010
Figura 1 – Esquema do ciclo reprodutivo e da morfologia floral de uma angiosperma, discriminando as diferentes
estruturas florais. (Modificado de www.portalsaofrancisco.com.br).
A reprodução nas angiospermas ocorre através de um ciclo do tipo haplodiplobionte, com
alternância de gerações. O primeiro processo que ocorre é a polinização, que se dá quando as
anteras se abrem e liberam o grão-de-pólen que, com a ajuda de diferentes vetores, como insetos,
mamíferos, pássaros, vento, água, entre outros, são depositados na superfície do estigma. Os grãos
de pólen, ao entrar em contato com a superfície do estigma, dão início às interações pólen-pistilo,
ou seja, uma série de processos que incluem o reconhecimento e a aceitação ou rejeição do grão
de pólen, feito pelos tecidos especializados do pistilo.
Com isso, o pistilo discrimina entre os diferentes tipos de grãos-de-pólen recebidos, e,
além disso, é o responsável por produzir o gametófito feminino no óvulo, tendo assim uma dupla
função (Cornish et al., 1988). Em geral, cruzamentos intergenéricos e interespecíficos não
ocorrem. Os mecanismos que impedem os grãos de pólen de efetuar a fertilização em pistilos de
lado, os cruzamentos intraespecíficos geralmente são bem sucedidos, e têm a base molecular do
seu funcionamento representada por interações proteína-proteína, como, por exemplo, proteínas
ligantes a kinases de sinalização, ou recrutar moléculas mais simples como água, cálcio, lipídeos e
Introdução gêneros ou espécies diferentes constituem a incongruidade (Linskens, 1983; 1986). Por outro
‐ 11 ‐ DePaoli,H.C.
2010
açúcares (Gaude & McCormick, 1999). Uma exceção a este sucesso acontece quando os genes do
sistema de auto-incompatibilidade impedem a fertilização (Takayama & Isogai, 2005). A autoincompatibilidade é um mecanismo de reconhecimento que resulta na rejeição, pelos tecidos
especializados do pistilo, de grãos-de-pólen que exibem a expressão dos mesmos genes do
sistema de auto-incompatibilidade da planta receptora (Takayama & Isogai, 2005). A
conseqüência mais direta da auto-incompatibilidade é que este mecanismo impede a autofecundação, aumentando desta forma a variabilidade genética. A base molecular da autoincompatibilidade tem sido amplamente estudada (para revisão ver Kao & Tsukamoto, 2004). O
principal locus envolvido nesse mecanismo é o locus S que, no sistema gametofítico, codifica os
genes de S-RNase, suficientes para a rejeição do grão de pólen incompatível em algumas espécies
do gênero Nicotiana (Lee et al., 1994). Além disso, há outros fatores envolvidos no processo de
auto-incompatibilidade, que atuam em paralelo ao locus S (Mcclure et al., 2000). A ausência de
um ou mais elementos-chaves, dentre esses fatores, pode justificar a autocompatibilidade
encontrada em N. tabacum.
O grão de pólen que, em N. tabacum é binucleado, chega à superfície do estigma, se hidrata
e germina, formando assim o tubo polínico. Uma célula geradora (n) é a responsável por se
dividir, mitoticamente, e formar duas células espermáticas, responsáveis pela dupla fecundação do
óvulo. O estigma e o estilete sofrem algumas mudanças estruturais e fisiológicas necessárias para
a realização desses processos. Na maioria das angiospermas os estiletes são sólidos com uma ou
mais faixas de tecido transmissor se estendendo do estigma até o óvulo (Raven et al., 1996), onde
os tubos polínicos podem crescer entre as células do tecido transmissor ou dentro das paredes
espessas das células. Na planta do fumo, especificamente, o tubo polínico cresce através dos
espaços intercelulares da zona secretória do estigma e do tecido transmissor do estilete em direção
ao ovário, transportando os dois núcleos espermáticos até o saco embrionário (gametófito
feminino) (Bell & Hicks, 1976; Sanchez et al., 2004). Além disso, a nutrição e o crescimento
direcionado do tubo polínico até o ovário ocorrem sob influência de compostos presentes no
exudato (Cheung et al., 1993; Goldman et al., 1994; Cheung et al., 1995; Wolter-Arts et al.,
1998). O tubo polínico penetra no óvulo através da micrópila, uma abertura na extremidade deste
formada pela fresta na camada do tegumento externo do óvulo, e libera os núcleos espermáticos
no saco embrionário. Um núcleo espermático fecunda a oosfera, originando o zigoto 2n, enquanto
o outro se une aos núcleos polares, dando origem ao endosperma, que na maioria das vezes é 3n.
Após a dupla fertilização, os tegumentos do óvulo se desenvolvem no envoltório da semente, e as
Em resumo, a reprodução sexual de plantas depende de interações específicas entre os
grãos-de-pólen e o pistilo, sendo que este último, e principalmente os tecidos especializados do
estigma e estilete, têm um papel decisivo na determinação do genótipo dos futuros embriões e
Introdução paredes do ovário e estruturas associadas se desenvolvem no fruto.
plantas (Linskens, 1983).
‐ 12 ‐ DePaoli,H.C.
2010
Figura 2 – Desenho da morfologia do pistilo de Nicotiana tabacum, discriminando as diferentes estruturas do estigma,
estilete e ovário (Adaptado de Kandasamy et al., 1990).
O transcriptoma no estigma e estilete O transcriptoma (o conjunto de transcritos de uma célula) é a base do controle de
diferentes processos dentro da célula, como a formação dos órgãos/tecidos e, na reprodução de
plantas, no controle das interações específicas entre os grãos-de-pólen e o pistilo.
Subseqüentemente, o transcriptoma dará origem ao proteoma (o conjunto de proteínas), que
representa o motor molecular do metabolismo celular. Com isso, desvendar e conhecer os
mecanismos moleculares envolvidos no desenvolvimento e no processo reprodutivo são a chave
para o melhor entendimento desses eventos nas plantas.
O modo predominante da regulação tecido específica é transcricional (Wickens et al.,
2000). Há uma série de outros mecanismos para a regulação nas vias de expressão gênica, como a
elongação da transcrição, estabilidade do RNA mensageiro (RNAm), “splicing” do pré-RNAm, a
poliadenilação, transporte do RNAm do núcleo para o citoplasma e controles traducionais. Porém,
pode-se considerar que o método “mais econômico” para a célula modular a expressão de um
gene é controlar a taxa de início da transcrição (Carey & Smale, 2000). Existem distintas
vantagens em se utilizar outros mecanismos para regular certas classes de genes, porém, se o
se o gene não for transcrito. Esse processo se faz necessário principalmente pelo fato das células
serem idênticas quanto a sua seqüência de DNA, mas não necessariamente possuírem os mesmos
genes sendo transcritos. É neste contexto de expressão coordenada de genes que se desenvolve a
Introdução produto gênico não é necessário em um determinado tipo celular, então energia será economizada
diferenciação celular.
‐ 13 ‐ DePaoli,H.C.
2010
Feitas essas considerações, não é surpreendente que o início da transcrição seja o
principal modo de regulação da maioria dos genes (Carey & Smale, 2000) e, assim, analisar o
transcriptoma pode nos fornecer dados específicos, acerca das necessidades fisiológicas e
bioquímicas da célula, diretamente relacionados com a função exercida pelo tecido/órgão. Nesse
âmbito, vários genes vêm sendo caracterizados e, atualmente, são conhecidos muitos genes que se
expressam de forma específica ou predominante no pistilo, envolvidos no correto
desenvolvimento dos órgãos reprodutivos, a partir do meristema floral, e também no
funcionamento do pistilo, para exercer as atividades ncessárias durante a interação pólen-pistilo.
Uma excelente estratégia é a identificação e análise de genes em larga escala, a qual tem
se tornado uma ferramenta útil para obter uma visão global do processo de desenvolvimento e
preparação do pistilo para a fecundação, junto com sua interação e crescimento do tubo polínico.
Yoshida et al. (2005) utilizaram microarranjos de cDNA para realizar uma análise da expressão
gênica no pistilo de arroz durante a polinização, elongação do tubo polínico, fertilização e o início
da embriogênese. Os genes identificados foram agrupados de acordo com o padrão de expressão,
revelando vários genes que não haviam sido descritos como específicos de pistilo,
desempenhando um papel na patogenicidade, germinação do pólen, elongação do tubo polínico e
degradação de amido. Tung et al. (2005) foram mais adiante e utilizaram microarranjos
comerciais de Arabidopsis para comparar o padrão de expressão do estigma e estilete da planta
selvagem, com o estigma e estilete de uma planta transgênica, sem as células da epiderme do
estigma e do tecido transmissor do estilete. Além disso, compararam o padrão de expressão do
estigma e estilete com o perfil transcricional do ovário em plantas selvagens. Swanson et al.
(2005) utilizaram microarranjos de cDNA e biblioteca subtrativa para, em conjunto, analisar o
padrão de expressão no estigma de Arabidopsis. Dentre 24.000 genes analisados, foram
identificados 11.403 genes expressos no tecido estigmático. O preparo do estigma pôde ser
evidenciado por classes de genes envolvidos em modificação da parede celular e metabolismo de
lipídeos, assim como genes potencialmente envolvidos nas vias de interação pólen-pistilo. Em N.
tabacum, DePaoli (2006) e Quiapim et al. (2009) utilizaram diferentes estratégias para a análise
em larga escala para identificar genes expressos de forma específica/preferencial em
estigmas/estiletes durante o desenvolvimento e a preparação para os eventos da polinização. Foi
criado um banco de dados, TOBEST (TOBacco EST), com 11.216 etiquetas de seqüências
expressas (ESTs) organizadas em 6177 agrupamentos (“contigs”) distintos, onde 51,2% são
transcritos/genes do estigma/estilete de função desconhecida. Destes, 782 agrupamentos foram
estigma/estilete, dos quais 10 genes foram analisados por RT-PCR em tempo real, confirmando a
expressão pistilo-específica de 9 deles (Quiapim et al., 2009). Para complementar o banco de
dados TOBEST, DePaoli et al. (manuscrito em preparação) construíram duas bibliotecas
Introdução analisados por macroarranjo, revelando 46 transcritos preferencialmente expressos no
subtrativas de cDNAs de estigma/estilete para identificar transcritos preferencial/especificamente
‐ 14 ‐ DePaoli,H.C.
2010
expressos nesses tecidos, quando comparados com raiz, caule, folha, sépala, pétala, anteras e
estigmas/estiletes de plantas STIG1::barnase (as quais não possuem a zona secretória do estigma;
Goldman et al., 1994). Foram isoladas 1101 ESTs, que agrupadas geraram 882 agrupamentos, dos
quais 585 (66,3%) não haviam sido identificados no TOBEST. Dessa forma, juntamente com
algumas análises de genes individualmente caracterizados na reprodução de plantas (para revisão
em Solanaceas ver DePaoli, 2006), a categorização dos genes identificados na análise em larga
escala pode nos dar uma visão de como os mecanismos intracelulares se coordenam em diferentes
vias para promover o desenvolvimento e o preparo do estigma/estilete para a polinização.
O desenvolvimento floral: enfoque no carpelo de N. tabacum O desenvolvimento dos órgãos reprodutivos em plantas é iniciado a partir do meristema
floral. Nos estágios iniciais, a ativação de alguns genes, que codificam fatores de transcrição,
determina a identidade de cada órgão floral. Portanto, estes genes são conhecidos como genes de
identidade floral. Ao longo dos anos, análises genéticas realizadas com mutantes para estes genes,
originaram o modelo ABC, que evolui recentemente para ABCE (Figura 3a-e). Este modelo
postula que quatro classes de genes, A (APETALA1, APETALA2), B (APETALA3,
PISTILLATA),
C
(AGAMOUS)
e
E
(SEPALATA1,
SEPALATA2,
SEPALATA3,
SEPALATA4), trabalham de forma combinada em complexos protéicos entre eles (Figura 3
painel inferior), para determinar o órgão que se desenvolverá em cada um dos quatro verticilos
florais, os quais são sépalas (Figura 3a1), pétalas (Figura 3a2), estames (Figura 3a3) e pistilos
Introdução (Figura 3a4) (Krizek & Fletcher, 2005).
‐ 15 ‐ DePaoli,H.C.
2010
Krizek and Fletcher, Nature Reviews Genetics (2005)
Figura 3 – O modelo ABCE proposto para a padronização dos órgãos florais (painel superior, a-e) com o modelo
molecular de complexos proteícos que os formam (painel inferior). se/Sepals, sépalas; pe/Petals, pétalas; st/Stamens,
estames; ca/Carpels, carpelos.
(a) As flores de Arabidopsis thaliana (e também de N. tabacum) consistem de quarto verticilos florais. A identidade das
sépalas (se) é conferida no primeiro verticilo pela atividade dos genes de classe A, a identidade das pétalas (pe) é
conferida no segundo verticilo pela atividade de genes das classes A e B, a identidade dos estames (st) é conferida no
terceiro verticilo pela atividade dos genes das classes B e C e a identidade dos carpelos (ca) é conferida no quarto
verticilo pela atividade do gene da classe C. A atividade da classe E é necessária para a especificação de cada tipo de
órgão. (b) Flor do mutante apetala 2 (ap2) que consiste em carpelos no primeiro e quarto verticilos e estames no
segundo e terceiro verticilos. Esse mutante não tem atividade de genes da classe A, resultando em expressão da
atividade de genes da classe C nos quatro verticilos. (c) Flor do mutante pistillata (pi) que consiste em sépalas no
primeiro e segundo verticilos, e carpelos no terceiro e quarto verticilos. O mutante não tem atividade do gene da classe
B. (d) Flor do mutante agamous (ag) que consiste de sépalas no primeiro verticilo, pétalas no segundo e terceiro
verticilos, e repetição deste padrão nas camadas interiores, indefinidamente. O mutante não possui atividade do gene da
classe C, resultando em expansão da atividade da classe A e perda da determinação floral. (e) Uma flor mutante para os
quatro genes SEPALLATA (sep1 sep2 sep3 sep4) que consiste em verticilos reincidentes de estruturas semelhantes a
folhas. O mutante quádruplo não possui a atividade dos genes da classe E, resultando em perda de função das classes A,
B e C e perda da determinação floral. Painel inferior: Os tetrâmeros se formam através da interação proteína-proteína
entre os dímeros de proteínas “MADS Box”. A proposta de composição dos tetrâmeros em cada um dos quatro
verticilos é: AP1–AP1–SEP–SEP no verticilo 1 para especificar sépalas; AP1–SEP–AP3–PI no verticilo 2 para
especificar pétalas; AG–SEP–AP3–PI no verticilo 3 para especificar estames; e AG–AG–SEP–SEP no verticilo 4 para
especificar carpelos. AG, AGAMOUS; AP1, APETALA 1; AP3, APETALA 3; PI, PISTILLATA; SEP, SEPALLATA
Introdução .
‐ 16 ‐ DePaoli,H.C.
2010
Em especial, o gene AGAMOUS, além de determinar o quarto verticilo em carpelos, tem
duas outras funções fundamentais: determinar o final da diferenciação dos órgãos florais (o
carpelo é o último órgão a se diferenciar) e; ativar, juntamente com outros genes semelhantes ao
agamous (“agamous-like”), a expressão de genes da classe D, que determinam a identidade do
óvulo.
O ápice do meristema floral é constituído de três camadas de células germinativas
independentes, designadas L1, L2 e L3. O início dos primórdios do pistilo são determinados por
divisões celulares da camada mais interna, a camada L3, em vários planos (Satina, 1944). Este
momento foi definido como o estágio -7 do desenvolvimento floral (Figura 4; Koltunow et al.,
1990). Em N. tabacum, o pistilo é constituído pela fusão de dois carpelos, um conjunto de células
que crescem lado a lado, formando uma espécie de “cone”, que se funde no topo próximo ao
estágio -4 formando dois lóculos. Neste momento, começa a diferenciação do estigma/estilete a
partir da camada celular L1, que tem origem epidérmica (Satina, 1944). A camada L2 dará origem
ao córtex (tecido parenquimático) no estilete, e as células provenientes da camada L3 continuam
se desenvolvendo e vão originar, também, os feixes vasculares do estilete. Logo após a fusão dos
carpelos no estágio -3, as papilas estigmáticas começam a se diferenciar no estágio -2 e,
gradualmente, aumentam em número mas sem se desenvolver na extremidade externa dos lóbulos
(Wolters-Arts et al., 1996). Durante estes estágios bem jovens do desenvolvimento do pistilo, que
vai do -2 ao 2, os tecidos especializados do estigma/estilete (SSZ e STT) se expandem devido às
divisões celulares, com um indice mitótico 5 vezes maior nos estágios -2 e -1 que nos estágios 1 e
2 (Wolters-Arts et al., 1996), quando o pistilo já está totalmente diferenciado em estigma, estilete
e ovário (Koltunow et al., 1990). A partir daí, o índice mitótico, ou seja, a velocidade com que as
células se dividem, diminui até o estágio 6, quando as células não se dividem mais e somente a
elongação celular ocorre. No estágio 12, ocorre a antese e a flor de N. tabacum está madura e
pronta para fornecer e receber os grãos de pólen, culminando com a fertilização (Figura 4). Ao
final, é fato que a regulação dos mecanismos desenvolvimentais está associada com o controle da
Introdução proliferação/diferenciação e, portanto, envolvem a maquinaria de controle do ciclo celular.
‐ 17 ‐ DePaoli,H.C.
2010
Figura 4 – Os estágios de desenvolvimento floral em N. tabacum (modificado de KOLTUNOW et al., 1990). O início
da formação dos verticilos florais se dá no estágio -7, diferenciando os órgãos florais até o estágio 1, onde a
diferenciação dos órgãos florais está completa (à excessão dos gametófitos masculino, grãos de pólen, e feminino,
ovócitos). O estigma/estilete se prepara, dos estágios 1 ao 11, para os eventos da polinização, que ocorrerá no estágio 12
(antese). Os tamanhos médios (mm) de cada botão, do estágio -7 ao 12, são, respectivamente, 0,75, 1,5, 3, 4, 5, 6, 7, 8,
11, 14, 16, 20, 22, 28, 39, 43, 45, 47 e 46.
A maquinaria da divisão celular em plantas É esperado que o controle do ciclo celular tenha uma função ímpar no crescimento e
desenvolvimento vegetal. Como em outros eucariotos, quinases dependentes de ciclina (CDK)
governam o ciclo celular em plantas. A chave do desenvolvimento vegetal, controlado pela
proliferação (controle do ciclo celular, quiescência e morte celular) e diferenciação celular, é
exatamente a atividade de CDK nos tecidos/órgãos em questão (De Veylder et al., 2007). Como o
próprio nome diz, para serem funcionais, as CDKs precisam se complexar com as ciclinas,
proteínas que determinam os “ciclos” da célula. Dessa forma, os diferentes complexos
ciclina/CDK irão fosforilar uma série de fatores ao longo das fases do ciclo celular, G1-S-G2-M
(iniciais de gap1, síntese, gap2 e mitose, respectivamente), promovendo a divisão celular (Figura
5) (Menges et al., 2005; Inzé & De Veylder, 2006). Embora o controle do ciclo celular em plantas
tenha os mesmos mecanismos fundamentais que outros eucariotos, elas desenvolveram novas
CDKF), 15 CKLs (“CDK-like kinases”, CKL1 a CKL15), e, ao menos, 49 ciclinas diferentes
(ciclinas e “cyclin-like”), que irão determinar a especificidade da divisão celular
unidirecionalmente, e, muito provavelmente, permitir a flexibilidade do desenvolvimento pósembrionário observado nas plantas (Menges et al., 2005; De Veylder et al., 2007). Como as
Introdução moléculas, orquestrando o controle do ciclo. Em Arabidopsis, pode-se citar 12 CDKs (CDKA a
‐ 18 ‐ DePaoli,H.C.
2010
CDKs, as ciclinas também são classificadas em diferentes grupos. As ciclinas do tipo D (CYC-D),
desempenham um papel na transição G1-para-S, na etapa inicial do controle do ciclo celular; as
ciclinas do tipo A (CYC-A), têm um papel previsto durante a fase S e no ponto de controle de Spara -M; e as ciclinas do tipo B (CYC-B) controlam a saída de G2 e entrada na fase M, além de
exercer funções na fase M (para uma revisão ver Inzé & De Veylder, 2006). Adicionalmente à
associação de CDKs com ciclinas, outros níveis de regulação da atividade de CDK são sua
própria fosforilação por quinases que ativam a atividade de CDKs (“CDKs-activating kinases”, as
clássicas e o gene WEE1) e a inibição da atividade de CDK por inibidores de CDK (CKI – “CDK
inhibitors”) (De Veylder et al., 2003; Dewitte & Murray, 2003; Inzé, 2005; Verkest et al., 2005).
Introdução Menges et al., Plant Journal (2005)
Figura 5 – Visão combinada da regulação transcricional no controle do ciclo celular em Arabidopsis, descrito por
MENGES et al. (2005). Várias alterações pós-traducionais também ocorrem e são tão importantes quanto à ativação
transcricional. Em amarelo, estão as fases do ciclo celular (G0 e G1-S-G2-M). CDK, quinases dependentes de ciclina;
CYC, ciclina; KRP, “Kip-related proteins”; Rb, “retinoblastome protein”; WEE1, uma “CDK-activating kinase”.
‐ 19 ‐ DePaoli,H.C.
2010
As CKIs inibem diretamente a atividade de CDK por interagirem com o complexo
ciclina/CDK. Em mamíferos, duas famílias de CKIs foram identificadas: INK4 (Inhibitors of
CDK4/6) e Cip/Kip (CDK-interacting protein/CDK-inhibitor proteins). Os inibidores da família
INK4 (p16INK4A, p15INK4B, p18INK4C, p19INK4D) se ligam especificamente a CDK4 e CDK6,
complexos específicos da fase G1, e são caracterizados pela presença de uma sequência de
aminoácidos repetida de 4 a 5 vezes, denominada “ankyrin” (“ankyrin repeats”). Até hoje,
nenhum inibidor de planta semelhante à família INK4 foi identificado.
Os membros de Cip/Kip foram os primeiros a serem identificados em humanos (p21CIP1,
p27KIP1 e p57KIP2) e se ligam a uma variedade de complexos de CDKs, com uma preferência para
complexos das fases G1 e S. Todos os inibidores da família Kip/Cip têm um domínio aminoterminal envolvido na ligação de CDKs e ciclinas (Pei & Xiong, 2005; Verkest et al., 2005).
Embora os membros da família INK4 tenham mostrado alguma regulação ao longo do
desenvolvimento (expressão em diferentes tecidos/órgãos/tipos celulares), os membros das duas
famílias, INK4 e Kip/Cip, são amplamente expressos e suas funções específicas propostas são
mais prováveis de serem relacionadas a modificações pós-traducionais que a uma expressão e/ou
alvo(s) tecido específicos. (Cánepa et al., 2007).
Em plantas, todas as CKIs que foram identificadas compartilham uma similaridade,
embora limitada, com o inibidor de mamíferos p27KIP1. Por esta razão, as CKIs de plantas foram
denominadas de KRPs (“Kip-related proteins”) (Verkest et al., 2005). Porém, Barroco et al.
(2006), também as definiu como ICKs (“interactors of CDC2 kinases”), existindo assim na
literatura dois nomes diferentes para se referir à mesma classe de proteínas (CKIs de plantas). Ao
contrário dos inibidores de mamíferos, os domínios conservados de ligação à ciclina/CDK nas
KRPs está localizado na porção carboxi-terminal da proteína (Figura 6). Em Arabidopsis, sete
KRPs (KRP1-7) foram identificadas através de ensaios de duplo híbrido e análises in silico (De
Veylder et al., 2001). Em N. tabacum, uma proteína semelhante às KRPs, denominada NtKIS1a,
foi isolada e mostrou ser capaz de reduzir a atividade de CDK in planta. Também foram
identificadas ICKs de plantas em tomate (LeKRP1, Bisbis et al., 2006), milho (Zeama;KRP1-2,
Coelho et al., 2005) e arroz (Oryza;KRP1, Barroco et al., 2006). Mais recentemente foi
identificada uma nova classe de ICK específica de plantas em Arabidopsis, SIM (SIAMESE e os
“siamese-related” SMR1-3). A família SIM/SMR tem 5 domínios curtos conservados e, embora o
domínio 4 (≈ 10aa) compartilhe uma alta similaridade com a região carboxi-terminal das
CKI/KRPs (Churchman et al., 2006), de uma forma geral, esta família não apresenta similaridade
endoreplicação, processo de duplicação do material genético sem o processo de divisão celular
(Churchman et al., 2006). Esta proteína é capaz de interagir com ciclinas do tipo D e CDKA, mas
não com as CDKB, específicas de plantas.
Introdução com as CKIs já descritas. O produto do gene SIM suprime a mitose direcionando a célula para
‐ 20 ‐ DePaoli,H.C.
2010
Em plantas, a organogênese requer um aumento no número de células e, portanto, uma
ativação da maquinaria de ciclo celular. Ao mesmo tempo, as células precisam se diferenciar e,
portanto, sair do ciclo celular. Com isso, devido à necessidade de controlar esse equilíbrio entre
proliferação/diferenciação, a atividade de CDK acaba por ser modulada a nível pós-traducional,
como relatado acima, com sua ativação e inibição ocorrendo por diferentes classes de proteínas.
Dessa forma, não poderia ser diferente que as CKIs tivessem uma proposta de estar envolvidas
como integradoras dos sinais desenvolvimentais e o controle do ciclo celular (revisto em Verkest
et al., 2005). Embora alguns membros sejam mostrados como sendo ativados e/ou inibidos por
diferentes classes de hormônios (ex., KRP1 é induzida por ABA – “abscisic acid”, Wang et al.,
1998; e KRP2 é reprimida por auxina, Himanen et al., 2002), ainda são necessários maiores
estudos para preencher a ponte sinalizadora entre a maquinaria do ciclo celular e os fatores
exógenos de resposta ao ambiente e a hormônios que alteram o desenvolvimento vegetal. A tabela
1 relaciona alguns controladores do ciclo celular que, quando modificados, também provocam
Figura 6 – Estrutura dos membros da família KRP em Arabidopsis e um membro de N. tabacum, NtKIS1, descritos por
JASINSKI et al. (2002). Os exons (caixas) e introns (linhas) de cada gene são indicados. As caixas pretas representam a
região de homologia entre CKIs de plantas e de animais (Kip/Cip). A parte hachurada (juntamente com a caixa preta)
representa a região de homologia entre as CKIs de plantas. (A) KRPs de Arabidopsis e N. tabacum com os respectivos
nomes e números de acesso. (B) NtKIS1a, e sua variante de “splicing” do último exon NtKIS1b. NtKIS1b perde a
habilidade de ligar ciclinas e CDKs in vitro, e inibe a capacidade de NtKIS1a de inibir a atividade de CDK in vitro.
Introdução alterações de desenvolvimento/organogênese em plantas.
‐ 21 ‐ DePaoli,H.C.
2010
Tabela 1 – Relação de alguns controladores do ciclo celular com as vias/fatores que os relacionam à
organogênese/desenvolvimento em plantas.
GENE
DESCRIÇÃO
REFERÊNCIA
CDKA;1
CDKB1;1
CDKE
CYCA2:2
CYCA3:2
CYCB1:2
CYCD2
CYCD3
E2Fa/DPa
E2Fc/Dpb
Zhang et al. (1996)
Hemerly et al. (1995).
Boudolf et al. (2004).
Wang e Chen (2004).
Roudier et al. (2003)
Yu, et al. (2003).
Schnittger, et al. (2002).
Cockcroft et al., (2000).
DeVeylder et al. (2001);
Wang et al. (2000);
Zhou et al. (2003)
Soni et al. (1995)
Riou-Khamlichi, et al.
(1999); Dewitte, et al.
(2003).
Goda et al. (2002)
DeVeylder et al. (2002)
del Pozo et al. (2006)
Wang et al. (2000)
Himanen et al., (2002)
Introdução KRP1
KRP2
Auxina induz a expressão de CDKA, mas sua atividade de quinase com
subsequente entrada na mitose é dependente de citocina, via
desfosforilação;
Superexpressão de AtCDKA não produz mudanças macroscópicas; a
expressão de uma forma dominante negativa (semelhante à perda de
função) inibe a divisão, mas não a morfogênese.
Uma diminuição da atividade de CDKB1;1 prejudica o desenvolvimento
de estômatos pelo bloqueio da divisão celular (neste caso a diferenciação
é desvinculada da divisão celular)
Brassinosteróides induzem a expressão de CDKB1;1
Perda de função em CDKE (mutantes HUA ENHANCER 3), tem
especificação imprópria de órgãos florais e término da atividade de
células tronco no meristema floral
Expressão de CYCA2;2 é induzida por auxina durante a iniciação e
elongação de raiz lateral . Consistentemente, CYCA2 tem sido envolvida
com ciclos mitóticos, mas não com endociclos ou diferenciação celular
Redução na expressão de CYCA3;2 produz embriões defeituosos e
prejudica a formação de calo;
A superexpressão leva à redução do potencial de diferenciação celular e
previne a regeneração da planta a partir de discos foliares.
A superexpressão de CYCB1;2 gera tricomas multicelulares
Superexpressão de CYCD2 aumenta a taxa de crescimento pela
diminuição do período que a célula fica em G1, em meristemas
Superexpressão de CYCD3 parcialmente restaura o fenótipo causado pela
superexpressão de KRP1/2;
O aumento no tamanho do órgão por superexpressão do gene
aintegumenta (ANT; Mizukami and Fischer, 2000) ou o gene ARGOS
(induzido por auxina, Hu et al., 2003) é resultado da proliferação celular
que ocorre através do aumento na expressão de CYCD3;
A ativação transcricional de CYCD3 por citocinina é a evidência
principal do envolvimento de citocinina na regulação de G1-S;
Superexpressão de CYCD induziu o crescimento independente de
citocinina (em calos de Arabidopsis) e divisões celulares ectópicas,
produzindo folhas com mais células, porém menores;
A expressão de CYCD3 é aumentada no tratamento com Brassinolídeo
Dentro da classe de ativadores transcricionais do grupo E2F/DP, o
dímero E2Fa/DPa induz a divisão celular quando superexpresso
Atuam como repressores transcricionais no equilíbrio da proliferação
celular e desenvolvimetno das células do periciclo;
Apresenta anomalias no desenvolvimento (folhas serrilhadas)
KRP2 parece regular o desenvolvimento inicial do ramo lateral da raiz
pelo bloqueio da transição de G1 para S;
É regulado transcricionalmente por auxina.
‐ 22 ‐ DePaoli,H.C.
2010
Uma visão da atuação da auxina no desenvolvimento do pistilo O primórdio do pistilo é um pequeno grupo de células capaz e comprometido a dividir e
diferenciar para gerar os tecidos do pistilo. Tal processo desenvolvimental inclui principalmente a
organogênese mediada por hormônio associada à divisão celular, crescimento e sinais de
diferenciação. Frequentemente, alteração no tamanho final do órgão é uma das conseqüências de
uma sinalização por fitohormônio modificada. Neste processo, o principal fitohormônio para o
crescimento vegetal é o ácido indol-acético (IAA), o hormônio auxina mais frequente nas plantas.
Auxina é um efetor da divisão e expansão celular bem conhecido (Leyser, 2002;
Vandenbussche and Van Der Straeten, 2004) e seu transporte polar adequado é essencial para a
iniciação do meristema floral (Okada et al., 1991), assim como também para o desenvolvimento
do pistilo e, mais diretamente, na diferenciação do estigma, estilete e tecido transmissor (Figura 7;
Nemhauser et al. 2000; Sundberg & Østergaard, 2009). O local da síntese, o nível da síntese e o
sentido do transporte do hormônio precisam ser finamente regulados para que o desenvolvimento
correto de cada órgão possa ocorrer. Nemhauser et al. (2000) propuseram um modelo no qual
altos níveis de auxina na região apical do pistilo promovem o desenvolvimento do estilete e do
estigma, enquanto níveis intermediários e baixos de auxina especificariam o ovário e o ginóforo,
respectivamente. Neste modelo, a síntese de auxina ocorreria preferencialmente no ápice e o
hormônio seria transportado do ápice para a base do órgão. De forma coerente com este modelo,
genes propostos como sendo reguladores da biossíntese de auxina (STYLISH [STY]/SHORT
INTERNODES [SHI], NGATHA [NGA]) e seus alvos (p.ex.: os genes de biossíntese de auxina
YUC2 (yucca), e YUC4) são todos expressos na extremidade apical do pistilo em
desenvolvimento (Cheng et al. 2006; Kuusk et al. 2006; Sohlberg et al. 2006; Alvarez et al. 2009;
Trigueros et al. 2009). Mais especificamente, o desenvolvimento do tecido transmissor também é
altamente dependente da atividade de genes relacionados à produção de auxina ou de resposta à
auxina, tais como os genes HECATE (HEC), SPATULA (SPT), STYLISH (STY)/SHORT
INTERNODES (SHI) e ETTIN (ETT)/AUXIN RESPONSE FACTOR (ARF3) (Heisler et al.
2001; Alvarez & Smyth 2002; Kuusk et al. 2006; Sohlberg et al. 2006; Gremski et al. 2007;
Crawford & Yanofsky, 2008).
Além dos genes envolvidos na síntese e/ou resposta de auxina, também podem ser citados
os produtos dos genes PINFORMED (PIN; Krecek et al., 2009), AUX1 (Pickett et al., 1990),
PINOID (PID; Bennett et al., 1995) e NPY (naked pins in yucca mutants; Cheng et al., 2007), que
atuam no transporte (Figura 7) e/ou transdução do sinal deste hormônio (Figura 8). Mais
papel fundamental no desenvolvimento floral, principalmente na formação do pistilo. Plantas de
Arabidopsis carregando diferentes mutações de perda de função no gene PID geram desde pistilos
alterados (fenótipo mais brando) até inflorescências em forma de pino, ou seja, sem verticilos
Introdução especificamente, a proteína PINOID é uma quinase que regula a sinalização de auxina e tem um
‐ 23 ‐ DePaoli,H.C.
2010
florais (mutante pid; fenótipo mais severo) (Bennett et al., 1995; Christensen et al., 2000). Foi
isolado recentemente, um alelo parcialmente funcional no locus do gene PID, o PID336 (Genji et
al., comunicação pessoal). pid336 não apresenta grandes alterações fenotípicas por si só em
plantas de Arabidopsis, mas sim quando combinado com outro(s) mutante(s) relacionado à
síntese/sinalização de auxina nos tecidos em que o gene PID se expressa. Com isso, a variante
PID336 é uma ferramenta poderosa em experimentos de interação genética, para identificar genes
envolvidos na via de auxina, principalmente no pistilo, o primeiro órgão a apresentar alterações
fenotípicas nos mutantes pid (Bennett et al., 1995). De forma semelhante, mutantes para o gene
NPY1 (mutantes npy1) desenvolvem inflorescências semelhantes às apresentadas pelo mutante
pid somente quando no “background” do duplo mutante yuc1yuc4, um mutante deficiente na
síntese de auxina (Cheng et al., 2007). Adicionalmente, foi demonstrado que o produto do gene
NPY1 trabalha junto com a proteína quinase PID, colocando em uma mesma via genética os
genes de síntese de auxina da família YUC e os genes de sinalização de auxina PID e NPY1
(Cheng et al., 2007). Dessa forma, esta via parece ser fundamental para o correto
desenvolvimento de órgãos florais, especialmente o pistilo.
No que diz respeito à sinalização intracelular do hormônio, as famílias de fatores de
transcrição Aux/IAA (“Auxin/Indole-3-Acetic Acid”; Figura 8A) e ARF (“auxin response factor,
Figura 8A) representam reguladores chaves com funções bem caracterizadas nesta resposta à
auxina (Benjamins e Scheres, 2008). As proteínas Aux/IAA se dimerizam com proteínas ARFs,
que têm a habilidade de se ligar no elemento de resposta à auxina (AREs) em promotores, para
reprimir a transcrição gênica dependente de auxina. Quando a concentração de auxina intracelular
aumenta, as proteínas Aux/IAA são degradadas, desligando-se, portanto, das proteínas ARF.
Estas, por sua vez, podem então regular a transcrição gênica dependente de auxina (Figura 8B)
(Teale et al., 2006). Essa resposta ocorre de forma relativamente rápida e, em minutos, os genes
de resposta à auxina estão sendo modulados. Por serem membros de uma família gênica, aos
proteínas ARF e Aux/IAA (23 e 29 membros em Arabidopsis, respectivamente) (Paponov et al.,
2009) podem possuir uma certa sobreposição de funções (ex. ARF6 e ARF8; Nagpal et al., 2005).
Ao mesmo tempo, é interessante notar que mutantes de perda de função dos genes ARF3, ARF5 e
ARF7 geram fenótipos bem distintos (Berleth e Jürgens, 1993; Liscum e Briggs, 1995; Sessions e
Zambryski, 1995; Ruegger et al., 1997; Watahiki e Yamamoto, 1997), o que sugere também que
alguns deles controlam processos morfológicos e desenvolvimentais diferentes, com pouca
sobreposição funcional. Conforme citado para alguns genes descritos na Tabela 1, o hormônio
assim, que a síntese, transporte e quantidade do hormônio são percebidas pela sinalização
intracelular, para molecularmente controlar a transcrição de diferentes genes, promovendo assim a
correta organogênese vegetal.
Introdução auxina pode influenciar alguns membros da maquinaria de controle do ciclo celular. Percebe-se,
‐ 24 ‐ DePaoli,H.C.
2010
A B C
D E
F Introdução Figura 7: Morfologia do pistilo (“gynoecium”) de Arabidopsis e a teoria do gradiente de auxina. O primórdio do pistilo
se desenvolve como um cilindro aberto (A) que se fusiona na parte apical para formar o estilete e o estigma (B) e,
internamente, para formar o septum (cinza) e a placenta com os óvulos (A, cinza). Foi sugerido que o padrão apicalbasal depende de um gradiente de auxina (Nemhauser et al. 2000) e, de acordo com o modelo proposto (C), uma alta
concentração de auxina na parte apical (amarelo) resulta na proliferação do estilete e do estigma, enquanto níveis
intermediários de auxina são necessários para a formação do ovário e baixos níveis para a formação do ginóforo.
Redução no transporte polar de auxina (PAT) resultaria no aprisionamento da auxina produzida apicalmente e em um
gradiente de auxina mais abrupto (E), alterando as delimitações das estruturas do gineceu (F). Um modelo alternativo
(D), proposto por Sundberg & Østergaard (2009), sugere que o ovário é formado em uma região de baixas
concentrações de auxina (amarelo) e citocinina (laranja), enquanto que altas concentrações de auxina induzem estilete e
estigma, e altas concentrações de citocinina, o ginóforo (retirado de Sundberg & Østergaard, 2009).
‐ 25 ‐ DePaoli,H.C.
2010
Figura 8 – Fatores e sinalização intracelular da via de auxina em células vegetais.
(A) Esquema representativo da família ARF e Aux/IAA de fatores de transcrição, com seus respectivos domínios (B-3 e
I-IV). O domínio B-3 é o domínio de ligação a AREs nos membros da família ARF. Os domínios III e IV são
conservados entre ARF e Aux/IAA e são os responsáveis por suas heterodimerizações. Os domínios I e II são
característicos da família Aux/IAA e estão envolvidos com a repressão da transcrição na heterodimerização com ARFs
e degradação da proteína, respectivamente. Os membros da família ARF possuem domínios ricos em determinados
aminoácidos na região entre B-3 e o domínio III. Essas diferenças é que caracterizam se um determinado membro da
família ARF atua como repressor (ex. Região rica em Serina – S) ou ativador (ex. Região rica em glutamina – Q) da
transcrição (Quint e Gray, 2006). (B) Há quatro etapas distintas na regulação da expressão gênica mediada por auxina
(1 a 4). Membros da familia ARF, se ligam a elementos de resposta à auxina (AREs), em promotores de genes de
resposta primária à auxina, e medeiam a transcrição destes (etapa 1). Aux/IAAs são proteínas de resposta rápida a
auxina, que dimerizam consigo mesmas e com membros da família ARF, e, como conseqüência, inibem a transcrição
de genes mediados por ARE (etapa 2). As proteínas Aux/IAAs são alvos de degradação mediada pelo proteasoma 26S,
e essa degradação é direcionada pela poliubiquitinação (Ub) das Aux/IAAs; uma reação que é catalisada pelo complexo
SCF, que possui a capacidade de ubiquitinar proteínas (“E3 ubiquitin ligase”) (etapa 3). A proteólise das Aux/IAAs
do “SCF E3 ligase“, que especificamente reconhece as proteínas a serem degradadas (etapa 4). IAA, “indole-3-acetic
acid”. (A) Paponov et al., 2009. (B) Teale et al., 2006.
Introdução mediada pela sua ubiquitinação é estimulada pela ligação do hormônio auxina na proteína F-box TIR1, um componente
‐ 26 ‐ DePaoli,H.C.
2010
Objetivos
Henrique C. DePaoli 1,2,3,4, Yunde Zhao3,4 e Maria Helena S. Goldman 1
Departamento de Biologia, Faculdade de Filosofia, Ciências e Letras de Ribeirão Preto, Universidade de São Paulo,
Brazil 14040-901
2
PPG Genética, Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Brazil 14049-900
3
Department of Cell & Developmental Biology, University of California San Diego, La Jolla, CA/USA 92093
4
SALK Institute for Biological Studies, La Jolla, CA/USA 92037
Objetivos 1
‐ 28 ‐ DePaoli,H.C.
2010
OBJETIVOS Para entender o processo de desenvolvimento e os eventos relacionados à fecundação,
faz-se necessária a identificação e caracterização de genes expressos de forma preferencial e/ou
específica nos tecidos reprodutivos das plantas. Nesse contexto, esta tese teve como objetivo
iniciar a caracterização do clone HS1-002E06 (SCI1), identificado em uma biblioteca subtrativa
de cDNAs de estigmas/estiletes (DePaoli, 2006), explorando sua função no órgão reprodutor
feminino das plantas, utilizando dois modelos vegetais diferentes: Nicotiana tabacum e
Arabidopsis thaliana.
Os objetivos específicos foram:
I.
Examinar a expressão do gene, referente ao clone HS1-002E06 (NtSCI1), nos
diferentes órgãos/tecidos da planta de N. tabacum, através das técnicas de RTPCR em tempo real e hibridização in situ;
II.
Realizar experimentos de localização subcelular e de interação de proteínas
(BiFC) in planta, através da fusão de SCI1 com proteínas fluorescentes;
III.
Identificar a função do gene através da análise dos fenótipos de plantas
transgênicas de N. tabacum que superexpressam o gene ou que tenham o gene
silenciando, pela técnica de RNA de interferência (RNAi);
IV.
Caracterizar o homólogo de NtSCI1 em A. thaliana, AtSCI1 (At1g79200),
através da análise dos fenótipos de plantas que superexpressam o gene ou de
mutantes de inserção de T-DNA (perda de função) disponíveis para compra no
“Arabidopsis Biological Resource Center”;
V.
Estudar a expressão de AtSCI1 em diferentes tecidos/órgãos de A. thaliana,
através dos dados de microarranjo disponíveis em banco de dados internacional e
da caracterização da sua região promotora, em fusão com os genes repórteres
GFP e GUS;
VI.
Entender o envolvimento de SCI1 na sinalização de auxina, pela análise de
interação genética com mutantes de auxina (pid336, npy1 e yuc2yuc6), análise do
nível de expressão de SCI1 nestes mesmos mutantes e pela indução do promotor
Objetivos sintético de resposta à auxina (DR5), em plantas mutantes de SCI1 (sci1).
‐ 29 ‐ DePaoli,H.C
2010
Métodos Material e Métodos
‐ 31 ‐ DePaoli,H.C
2010
Material Vegetal Nicotiana tabacum cv Petit Havana SR-1 e as plantas transgênicas foram crescidas sobre
condições padrão de casa de vegetação em Ribeirão Preto – SP, Brasil (Latitude - 21º 10'24" S,
Longitude - 47º 48'24" W, com média de temperatura de 22˚C no inverno e 27˚C no verão; a
diferença entre dia longos e curtos é de menos de duas horas). Amostras de raiz, caule, folha,
sépala, pétala, estame, estigma/estilete e ovário foram coletadas em nitrogênio líquido e estocadas
a -70˚C para a extração de RNA. Para os estudos de expressão no desenvolvimento floral,
estigmas/estiletes foram excisados de flores nos estágios -4 a 12 de flores de N. tabacum,
conforme descrito por Koltunow et al. (1990).
Sementes de Arabidopsis thaliana Col-0 e de cada um dos mutantes sci1-1, sci1-2, sci1-3,
sci1-4, sci1-5, yuc2yuc6, npy1 e pid336 e de plantas DR5:GUS, foram esterilizadas (5minutos em
etanol 75% e mais 5minutos em etanol absoluto), semeadas em meio MS (Murashige & Skoog,
1962) com 3% de sacarose e 0,7% de ágar, por uma semana, transplantadas para o solo e mantidas
em camara de crescimento (fotoperíodo de 16/8horas, luz/escuro respectivamente, a 24ºC). Foram
coletados meristemas florais e flores jovens, e armazenadas a -70˚C para a extração de RNA. Os
cruzamentos para obter duplos e triplos mutantes foram feitos polinizando flores jovens de
Arabidopsis emasculadas.
Sequenciamento, “primers” e análises computacionais Os clones HS1-002E06 de uma biblioteca subtrativa de cDNA de estigma/estilete de N.
tabacum (DePaoli 2006; DePaoli et al., manuscrito em preparação) e TOB068F10 do projeto
TOBEST foram completamente seqüenciados, utilizando a metodologia previamente descrita
(Quiapim et al., 2009). Uma vez que eram clones parciais, mas complementares, suas seqüências
foram usadas para definir “primers” para amplificar a seqüência codante completa de SCI1. O
fragmento NtSCI1-ORF foi amplificado diretamente de cDNAs de estigma/estilete (conforme
preparado
para
qRT-PCR,
veja
abaixo),
utilizando
(5’gcaggcttcaccATGGGGAGCGATAAGAAGAC3’)
os
e
primers
Ct11attB1FW
Ct11attB2RW
(5’aagctgggtcTTACTTTTTGATATTCCAAGCGTG3’), que contêm parte dos sítios de
recombinação (mostrados em letras minúsculas). As condições do PCR foram: 94ºC por 4 min; 35
ciclos de 94ºC, 50ºC e 72ºC por 1 min cada; e 72ºC por 10 min. Uma alíquota de 5µl do produto
deste PCR foi utilizada para uma segunda reação de PCR, nas mesmas condições (exceto pela
(5’ggggacaagtttgtacaaaaaagcaggcttc3’)
e
BP2
(5’ggggaccactttgtacaagaaagctgggtc3’).
Uma
alíquota de 10µl deste segundo produto de PCR foi utilizado para a recombinação no vetor de
entrada pDONR201 (Gateway, Invitrogen, USA) utilizando a “BP clonase” (Invitrogen, USA), de
acordo com as intruções do fabricante. Uma alíquota de 8µl (de um total de 20µl) da reação BP,
Métodos temperatura de pareamento, que nesta segunda reação foi de 45ºC), utilizando os primers BP1
‐ 32 ‐ DePaoli,H.C
2010
foi usada para transformar células DH5α de E. coli, químico-competentes (Sambrook and Russell,
2001).O clone resultante, denominado NtSCI1-ORF, foi completamente sequenciado.
De forma semelhante, a região codante completa de AtSCI1 (SCI1FL) foi amplificada
utilizando os primers AtSCI1OxFW (5’gcaggcttcATGGTAAGCGAACGGAGCTCCA3’) e
AtSCI1OxRW (5’aagctgggtcTCACCTACGCTTGATCATCCAGTC3’), e a região codante que
inclui os exons 1, 2 e 3 (AtSCI1123) utilizando os primers AtSCI1OxFW e AtSCI1123OxRW
(5’aagctgggtcTCATAGCTTGTGGTCGCTTTCCGTATG3’). Estes primers contêm parte dos
sítios de recombinação (mostrados em letras minúsculas). Em seguida, foi então realizada a
reação de PCR com os primers BP1/BP2. Igualmente, os primers AtSCI1PROM1FW (5’
gcaggcttcTACTTTGCTTGTAGTAAATACTTTTCTG3’)
e
AtSCI1PROM1RW
(5’aagctgggtcTATTTCCCGACGGACTAAAA3’) foram utilizados para amplificar a região
promotora de 287pb de AtSCI1, e os primers AtSCI1PROM1FW e AtSCI1PROM2RW (5’
aagctgggtcCTTCCATTGATAGCTCTGGAATT3’) para amplificar a sequência genômica de
1392pb de AtSCI1, ambos seguidos da reação de PCR com os primers BP1/BP2, gerando os
clones de entrada pENTRYAtSCI1PROM1 e pENTRYAtSCI1PROM2. A citosina em negrito e
sublinhada, no “primer” AtSCI1PROM2RW, foi inserida para manter a fase de leitura entre a
proteína SCI1 de fusão, incluída no promotor2, com a GFP-GUS do plasmídeo de destino,
baseado na seqüência disponível para o plasmídeo pKGWFS7 (Karimi et al., 2002). As reações de
PCR e BP foram realizadas conforme descrito acima para NtSCI1-ORF, e posteriormente
sequenciadas. Os mutantes de T-DNA foram genotipados por reações de PCR (5 minutos a 95ºC;
45 ciclos de 15segundos a 95ºC, 30segundos a 60ºC e 1minuto a 72ºC) em volume de 10µl,
utilizando os “primers” já descritos para yuc2, yuc6, npy1 e pid (Cheng et al., 2006; Cheng et al.,
2007), sendo que para pid336, após a amplificação, seguiu-se à digestão com a enzima RsaI, a
qual digere o alelo selvagem, mas não o mutante, por 4 horas a 37ºC (10µl de tampão 2x com
0,1U
de
enzima);
e
os
“primers”
5’GCAGTCTCTTGTCTTTGTAGACCAAATGCG3’
e
5’ATTGATGGCATTTCTCGGGAATCGGAAGA3’
para
5’CAGCAAAGAAGTCCAAGGATGATAAGCCG3’
5’AGCTCTGGAATTCCCTCTTTCTGGA3’
Atsci1-1FW
Atsci1-2FW
sci1-1,
e
Atsci1-2
para
5’ACAACGAGTTTGCTACATGGCTGAAAGA3’
Atsci1-1RW
RW
Atsci1-3FW
sci1-2,
e
Atsci1-U-RW
5’AAACGGTCTATTTCTCCCTTAACCGGA3’ para sci1-3, sci1-4 e sci1-5. Os “primers”
localizados
no
T-DNA
foram
os
“primers”
universais
JMLB1
GCCTTTTCAGAAATGGATAAATAGCCTTGCTTCC-3′), para sci1-2. Os produtos de PCR de
cada mutante do locus At1g79200, dos alelos selvagem e mutante (sci1-x), foram sequenciados
(ETON BioSciences Company, La Jolla/CA-USA; http://www.etonbio.com) direto do produto de
PCR (250ng) para confirmar a posição de inserção do T-DNA.
Métodos (5’GCGTGGACCGCTTGCTGCAACT3’), para sci1-1, sci1-3, sci1-4 e sci1-5, e SAIL-LB1 (5′-
‐ 33 ‐ DePaoli,H.C
2010
As seqüências de nucleotídeos e aminoácidos foram analisadas por vários programas de
computador: “Translate” no “ExPASy Proteomics Tools” (www.expasy.org), BLAST no NCBI
(http://blast.ncbi.nlm.nih.gov/Blast.cgi)
e
SOL
Genomics
(http://sgn.cornell.edu/solanaceae-project/),
(http://www.ebi.ac.uk/Tools/clustalw2/index.html),
bin/motif_scan),
PSORT
Network
ClustalW
MotifScan
(http://myhits.isb-sib.ch/cgi-
(http://psort.ims.u-tokyo.ac.jp/form.html),
PredictNLS
(http://cubic.bioc.columbia.edu/predictNLS/), “ELM prediction” (http://elm.eu.org/), NetPhos e
NetPhosK
(http://www.cbs.dtu.dk/services/),
ustc.org/pail/prediction.php),
“Protein
Molecular
PAIL
Weight”
(http://bioinformatics.lcde
“Isoelectric
Point
with
phosphorylation” (http://scansite.mit.edu/calc_mw_pi.html), T-Coffee (www.ebi.ac.uk/t-coffee/),
Multalign (http://www.expasy.org/tools/#align) e GPS (Group-based prediction system; Xue et
al., 2008; http://gps.biocuckoo.org/).
Manipulação de ácidos nucléicos e construções gênicas Todas as manipulações de ácidos nucléicos foram feitas conforme descrito em Sambrook
e Russell (2001). Para obter a construção 35Spro:SCI1-GFP, a seqüência codante de NtSCI1 foi
amplificada a partir do clone NtSCI1-ORF, utilizando os “primers” Ct11attB1FW e
Ct11attB2GFPRW (5’aagctgggtcCTTTTTGATATTCCAAGCGTG3’), que exclui o códon de
término da tradução da seqüência SCI1-ORF criando, coincidentemente, um sítio de restrição para
AvaII (negrito e sublinhado). O produto do PCR foi recombinado no vetor de entrada pDONR201
conforme descrito acima para NtSCI1-ORF. O clone resultante, pENTRY SCI1-ORFss (sem
“stop codon”) foi confirmado por digestão com AvaII e posterior sequenciamento. Depois, 300ng
do DNA positivo foi utilizado para a reação de recombinação LR, com o vetor de expressão
pK7FWG2 (Karimi et al., 2002) via LR clonase (Invitrogen, USA). A construção foi sequenciada
para confirmar que mutações indesejadas não ocorreram. Para se obter as construções de RNAi e
superexpressão, 300ng de DNA de NtSCI1-ORF original (com códon de término da tradução) foi
recombinado nos vetores de expressão pK7GWIWG2(I) e pK7WG2 (Karimi et al., 2002), para
gerar as construções SCI1Ri e SCI1OE, respectivamente. Essas reações de recombinação foram
realizadas utilizando a LR clonase (Invitrogen, USA), conforme as instruções do fabricante. De
forma semelhante, obteve-se a superexpressão de AtSCI1 (AtSCI1FL e AtSCI1123) recombinando
os respectivos clones de entrada, via LR clonase, no plasmídeo pK7WG2. A construção
fluorescent protein”), foi obtida via LR clonase do pENTRY AtSCI1FL no vetor pK7WGY2,
conforme descrito acima. A construção SCI1PROM1:GFP-GUS e SCI1PROM2:GFP-GUS foi obtida
via LR clonase do clone pENTRYAtSCI1PROM1 e pENTRYAtSCI1PROM2 no vetor pKGWFS7.
Métodos 35Spro:YFP-SCI1, com o gene AtSCI1 fusionado à YFP (proteína amarela fluorescente – “yellow
‐ 34 ‐ DePaoli,H.C
2010
Cada vetor de expressão de planta foi introduzido separadamente em Agrobacterium tumefaciens
C58C1RifR (pGV2260) por eletroporação.
As seqüências de NtCYCA1;1 e NtCYCA2;2, clonadas no vetor pDONR221 (Strini,
2010)
foram
reamplificadas
utilizando
os
primers
CYCA1.1FW
(5’agcaggcttcGAAGGAGATAGAACCATGGCGACGACYCAGAATAG3’) e CYCA1.1ssRV
(5’aagctgggtcGCAGCTTATGTTCTGGAAG3’),
e
(5’agcaggcttcGAAGGAGATAGAACCATGAGGCATGCAAATATAAAAC3’)
CYCA2.2FW
e
CYCA2.2ssRV (5’aagctgggtcGAACAGTGATTGGACTGGTTTTG3’), respectivamente, para
retirar o códon de término da tradução. Os sítios de recombinação attB são mostrados em letras
minúsculas e os sítios de ligação ao ribossomo estão sublinhados. Os produtos desta primeira
PCR foram reamplificados (0,5µl) com os primers BP1 e BP2, e inseridos no vetor de entrada
pDONR201, conforme já descrito acima. Os clones foram sequenciados para confirmar a ausência
do códon de término da tradução. A seqüência NtCYCA2;296-398 (sem o “destruction-box”) foi
obtida por PCR a partir de NtCYCA2;21-398 (ORF completa), utilizando os primers
CYCA2.2DelFW (5’agcaggcttcGAAGGAGATAGAACCATGAATTGCATCAACGGAAG3’) e
CYCA2.2ssRV.
Para se obter NtSCI1 fusionado à “cabeça” da GFP (aminoácidos 1 a 149)
(35Spro:NtSCI1-hGFP), foi recombinado o pENTRY p35S - attL4/R1, o pENTRY hGFP attR2/L3 e o pENTRY SCI1-ORF (no caso L1/L2) sem o códon de término da tradução, no vetor
pK7m34GW via reação LR. Semelhantemente, para se obter NtCYCA1;1, NtCYCA2;21-398 (ORF
completa) e NtCYCA2;296-398 (sem o “destruction-box”) fusionados à tGFP (aminoácidos 150 a
238) (35Spro:NtCYCA-tGFP), recombinou-se o pENTRY p35S – attL4/R1, o pENTRY NtCYCA
– attL1/L2 apropriado, e o pENTRY tGFP - attR2/L3 no vetor pH7m34GW. Ambas as reações
foram realizadas utilizando 100ng de cada DNA e 2µl da LR clonase com tampão 5x. Neste caso,
a reação ocorreu por 16 horas a 25ºC, antes da adição de proteinase K (conforme as instruções do
fabricante). Em seguida, 1,5µl de cada reação LR foi utilizada para transformar E. coli DH10B
eletrocompetentes. Cada construção foi sequenciada a partir de 3 pontos diferentes para confirmar
que a recombinação ocorreu de forma correta e sem mutações indesejáveis. Os clones
selecionados foram separadamente introduzidos em Agrobacterium tumefaciens C58C1RifR
(pGV2260) por eletroporação.
Extração de RNA vegetal congelado, essencialmente conforme descrito por Dean et al. (1985). Para as análises das
plantas transgênicas de N. tabacum (SCI1Ri and SCI1OE) e comparações com as plantas selvagens,
quatro amostras de estigma/estilete no estágio 4 do desenvolvimento floral foram coletadas de
Métodos Para N. tabacum, o RNA total foi extraído a partir de uma quantidade grande de material
‐ 35 ‐ DePaoli,H.C
2010
cada planta e o RNA total foi extraído utilizando o “Plant RNA purification Reagent” (Invitrogen,
USA). As amostras foram quantificadas por medições feitas a OD260 e OD280. Para avaliar a
integridade do RNA, este foi resolvido em gel de agarose 1,2% com 2,2M de formaldeído, corado
com brometo de etídio em solução (1µg/ml) e visualizado sobre luz U.V. A presença de rRNA
18S e 28S intactos foi utilizada como critério para a integridade do RNA. Em Arabidopsis, o
material congelado foi macerado e extraído utilizando o kit “RNeasy" com DNAse (Quiagen),
segundo instruções do fabricante, e quantificado.
RT‐PCR quantitativo (qRT‐PCR), “semi‐quantitativo” (RT‐PCR) e hibridização in situ Em N. tabacum, aproximadamente 20µg de RNA total, de cada um dos tecidos, obtidos
como descrito acima, ou os RNAs extraídos pelo “Plant RNA purification Reagent”, foram
tratados com 5U de DNAse (Promega), em 5µl de tampão da DNase 10x, 1 µl de DTT 100mM,
20U de RNase out e água DEPC 0,01%, em um volume final de 50 µl, e deixados por 1,5 horas a
37ºC. Em seguida, foi retirada uma alíquota de 2 µl que, por sua vez, foi diluída em 8 µl de água.
Destes 10 µl, foi utilizado 1 µl para uma reação de qRT-PCR (vide abaixo), utilizando os
“primers” da actina, para constatar que todo o DNA havia sido degradado. Uma vez livre de
DNA, o RNA foi limpo com fenol (Sambrook & Russell, 2001) e utilizado na síntese de cDNA,
na presença de 150pmol de oligo dT, que foi previamente colocado a 75ºC por 10min,
adicionando, logo após, 5 µl de tampão de primeira fita 5X, 1 µl de DTT 100mM, 20U de RNase
out, 1 µl de dNTPs (10mM de cada), deixando a 42ºC por 5min. Em seguida, adicionou-se 200U
de Superscript II (Invitrogen) e incubou-se a 42ºC por mais 1 hora, completando o volume para
70µl (ou 35µl para os RNAs provenientes da extração com “Plant RNA purification Reagent”)
após o término da reação, antes de armazenar a -20ºC. As reações padrão de qPCR foram
realizadas usando uma alíquota de 1µl do cDNA, 2 µl de cada “primer” específico (Tabela 1) a
1pmol e 5 µl do “Power SYBR Green PCR Master Mix” (Applied Biosystems, UK) em um
volume final de 10µl. Os PCRs foram realizados em placas de 96 poços utilizando o “ABI
Sequence Detection System 7500 Fast” (Applied Biosystems, UK). As condições da ciclagem
foram, um passo inicial a 50ºC por 2 minutos, seguidos de 10 minutos a 95°C; 40 ciclos, com cada
ciclo consistindo de 15 segundos a 95°C e 1 minuto a 60°C e uma curva de dissociação final, com
a temperatura gradualmente aumentando de 60ºC até 95ºC. A curva padrão foi construída para
cada gene usando uma diluição seriada para calcular o número log de cópias de transcritos
baseado nos valores de Ct conforme descrito por Semighini et al. (2002). Todas as análises foram
de plantas selvagens. Em todos os experimentos, controles negativos adequados, sem conter DNA
ou RNA, foram submetidos ao mesmo processo para excluir ou detectar qualquer contaminação
possível. O nível de expressão em unidades arbitrárias corresponde ao log de cópias calculado do
Métodos realizadas em triplicatas e pelo menos duas replicatas biológicas foram feitas para a quantificação
‐ 36 ‐ DePaoli,H.C
2010
gene de interesse sobre o log de cópias calculado do controle interno da β-actina ou
poliubiquitina. Os “primers” utilizados para as reações de qRT-PCR estão na Tabela 1. As
análises estatísticas foram realizadas utilizando o programa “SigmaStat.v3.5”.
Tabela 1: “Primers” usados nas análises de expressão gênica por RT-PCR em tempo real.
Gene
Forward (5’- 3’)
Reverse (5’- 3’)
β–actina
agatgatgctccaagggctgtgtt
Poliubiquitina
SCI1
NtARF8
NtAux/IAA13
NtAux/IAA19
tccgacacaatcgacaacgtaa
gacgtactgttgtaatcggctagggtacgtc
gccacaaacataaggataaatcg
cagccacgtagcaaattcattgtt
ccatgccaaccatcacaccagt
ggcaggattgcagtgaaagctaca
ggtctgattctcttcttcccaagg
gctcacaactacctctgaagatcgca
acctcacgggtaacaaactggtca
gctgcgagctttaagcacattatcc
ggacgagtggcctctcatataatc
As seqüências de “primers” foram determinadas com base na seqüência dos seguintes
“clusters” encontrados no TOBEST (Quiapim et al., 2009): β-actina (TOBC110E12),
poliubiquitina (TOBC018H09), NtARF8 (TOB044H02), NtAux/IAA13 (TOB100E10) e
NtAux/IAA19 (TOB056F02).
Para os experimentos com Arabidopsis thaliana, os RNAs extraídos (já livres de DNA),
foram convertidos (1µg de RNA total) em cDNA (conforme descrito acima). O volume foi
completado para 50µl e 1 µl de cada reação foi utilizada em uma diluição 1:2, 1:4, 1:8, 1:16, 1:32,
1:64, 1:128 e 1:256. As diluições de 1:8 a 1:256 foram submetidas à reação de PCR (1µl cDNA;
94ºC 3minutos; 27 ciclos de 94ºC por 15 segundos, 58ºC por 30 segundos e 72ºC por 30
segundos) utilizando os “primers” AtSCI1OxFW e AtSCI1OxRW (amplicom de 480pb) ou os
“primers”
UbqFW
5’CTAGCTTGTGTGAGACCTAGCC3’
e
UbqRW
5’GAGGCACATACTGTCTAGACAAAC3’ (amplicom de 232pb), correspondentes a AtSCI1 e
AtUBQ (At5g12120), respectivamente. Os produtos dos PCRs foram resolvidos em gel de
agarose 1,5%, com brometo de etídio (0,1µg/ml) e a intensidade das bandas de ambos os
amplicons foram medidas no aparelho “ImageQuant TL vs2005”. O valor gerado, para a banda
correspondente a AtSCI1, foi dividido pelo valor gerado para a banda correspondente a AtUBQ
em cada uma das plantas, incluindo a planta selvagem, para normalizar o valor de expressão entre
as amostras. Os valores normalizados de cada amostra desconhecida foram, então, divididos pelo
valor normalizado encontrado na planta selvagem (Col-0) e multiplicados por 100 (planta
selvagem = 100%). A diluição seriada escolhida para as análises foi a de 1:32, que apresentou
realizadas 5 replicatas experimentais de uma única amostra biológica (que corresponde ao
material de várias plantas, de diferentes idades, crescidas em diferentes momentos, com material
coletado em diferentes períodos do dia).
Métodos estar na fase exponencial da amplificação do PCR, em todas as amostras comparadas. Foram
‐ 37 ‐ DePaoli,H.C
2010
Para os experimentos de hibridização in situ, estigmas/estiletes nos estágios 4, 7 e 10 do
desenvolvimento floral (Koltunow et al., 1990) foram fixados em glutaraldeído 4%, desidratados
em uma série de etanol-xilol, embebidos em parafina (TissuePrep2 - Fisher Scientific, USA),
seccionados em 8µm, hibridizados e revelados essencialmente como descrito em Jackson et al.
(1991). As sondas de ribonucleotídeos foram preparadas por transcrição in vitro utilizando o clone
HS1-002E06linearizado na extremidade 3’, sonda senso, ou 5’, sonda anti-senso. Para este
propósito, foi utilizado o Kit “DIG RNA Labeling Kit SP6/T7 (Roche, Germany) de acordo com
as instruções do fabricante e a mistura de detecção NBT/BCIP da Fisher “1-step NBT/BCIP
detection mixture” (Thermo Fisher Scientific, USA).
Importação dos dados de Microarranjo A análise de expressão gênica de AtSCI1 foi realizada importando-se os dados de
microarranjo disponíveis no “Bio-Array Resource” (http://bar.utoronto.ca/) para serem plotados
em um gráfico (Capítulo 2; Figura3). Dentro deste banco de dados, existe uma ferramenta
chamada “e-Northerns w. Expression Browser”. Ao clicar nesta ferramenta, na página principal,
se abre o “Expression Browser”, onde se pode selecionar: 1) o banco de dados de interesse; 2)
Restringir entre diferentes áreas de pesquisa (opcional) e; 3) especificar o tipo de dados de saída
(crua ou normalizada). Então, se digita o(s) locus de interesse (em nosso caso At1g79200) e
submete. No nosso caso, o banco de dados selecionado foi “AtGenExpress_Plus – Extended
Tissue Series”, restringindo os dados a serem analisados na área de pesquisa “Developmental
Baseline”. Ao submeter a busca, se abre uma janela com os arquivos de saída. Foi selecionado o
arquivo “View Graphical Representation of Clustered Data (Recommended)”, que contém os
dados de expressão já normalizados e agrupados. Ao clicar no arquivo, se abre uma nova janela
com a interface dos resultados. Para se extrair os valores que geraram a interface, clicou-se em
“get details” e, em seguida, nos links do tecido desejado (ex. Root, Flower Stage 9), que
direcionam para os dados das lâminas de arranjo, em triplicata, no “NASCArrays”. Clicou-se,
finalmente, em “Get the data!” para salvar cada uma das 3 planilhas excel (replicatas) que contêm
os valores de expressão já normalizados para todos os locus, copiando apenas o valor do locus de
interesse (At1g79200).
Transformação de Plantas Sementes de N. tabacum cv Petit Havana SR1 foram esterilizadas e plaqueadas em placas
MES, pH 5.8 e 0,7% agar (Bacto Agar). As plântulas foram crescidas em câmaras de germinação
a 27ºC (±1ºC) com fotoperíodo de 16h/8h. Para os ensaios de expressão transiente, as plantas (6
semanas de idade) foram transferidas para o solo e suas folhas foram infiltradas com uma cultura
Métodos de Petri com meio MS (Murashige and Skoog, 1962) suplementado com 3% de sacarose, 0.5 g/l
‐ 38 ‐ DePaoli,H.C
2010
de Agrobacterium contendo as construções apropriadas, conforme descrito por Sawers et al.
(2006), e foram analisadas de 2 a 5 dias após a infiltração.
Para as plantas transgênicas estáveis, discos foliares de N. tabacum foram co-cultivados
com o clone de Agrobacterium contendo as construções adequadas, de acordo com protocolo
descrito previamente (Brasileiro & Carneiro, 1998). As plantas transgênicas foram regeneradas
em meio contendo canamicina (200µg/ml) durante o processo inteiro de cultura in vitro e
transferidas para a casa de vegetação em solo (mistura de vermiculite). Para garantir que as
condições ambientais não estavam influenciando o fenótipo de nossas plantas, transferiu-se
algumas plantas durante o inverno/primavera (por exemplo, SCI1Ri4.4.1 e SCI1Ri13.2) e outras
durante o verão/outono (por exemplo, SCI1Ri14.2 e SCI1Ri16.1). Depois de 2 semanas, as plantas
foram transferidas para o solo onde cresceram até o florescimento e formação de frutos. Essas
plantas representam a primeira geração de transgênicos (T0), que foi analisada nos experimentos
de qRT-PCR e microscopia ótica. A geração seguinte (T1) foi produzida por polinização
controlada e as sementes foram esterilizadas, plaqueadas em meio MS com canamicina
(200µg/ml), e as plântulas resistentes foram transferidas para o solo, sendoa primeira florada
usada para as análises de microscopia eletrônica de varredura (MEV).
Para a transformação de plantas de Arabidopsis, plantas selvagens e mutantes (teste de
complementação) foram crescidas por 4 semanas e borrifadas (2 vezes em um período de 7 dias)
com uma solução de Agrobacterium carregando a construção de interesse. A solução de
Agrobacterium borrifada foi preparada crescendo-se, por 48 horas, 100ml de cultura da bactéria
em meio LB líquido com marcador de seleção, precipitada e ressuspendida em meio MS
(Murashige & Skoog, 1962) líquido, com 5% de sacarose e 0,032% de Silwet L77 (pode ser
utilizado tween 20). As sementes destas plantas foram coletadas após 3 e 4 semanas, esterilizadas
(5min. EtOH70% e 5min. EtOH100%) e semeadas em meio MS sólido com agente seletivo
(Kanamicina, 25µg/ml). Os transformantes (plântulas que apresentaram folhas bem verdes e bom
desenvolvimento radicular) foram transferidas para o solo após 10 dias, confirmadas quanto à
inserção do transgene por PCR de folha (vide genotipagem abaixo) e tiveram seus fenótipos
analisados. A análise das plantas foi feita pela observação de alterações no fenótipo,
acompanhada de experimentos moleculares complementares.
Testes de interação genética em Arabidopsis O mutante de perda parcial de função (“knockdown”) sci1-1 foi utilizado para as análises
pid336, npy1 e o duplo mutante yuc2yuc6 foram cultivados conforme descrito acima para plantas
selvagens de Arabidopsis. Flores maduras do mutante sci1-1 homozigoto foram dissecadas e as
anteras maduras (abertas e com os grãos de pólen expostos) foram utilizadas para polinizar
Métodos de interação genética com mutantes da via de auxina. Os mutantes homozigotos da via de auxina,
‐ 39 ‐ DePaoli,H.C
2010
pistilos de flores emasculadas de cada um dos 3 mutantes da via de auxina. As sementes foram
coletadas após 3 semanas, secas “overnight” em sílica, esterilizadas e semeadas. A geração
seguinte (F1, totalmente heterozigota) foi autopolinizada para se obter, então, a segunda geração
(F2). Nesta geração, foi feita a identificação dos duplos e triplos mutantes, por reação de PCR,
utilizando os “primers” alelo específicos citados acima a partir de DNA genômico extraído de
folhas (macerado em 125mM NaCl, 100mM TrisHCl pH7,5, 12,5mM EDTA, 0,25% SDS e 50%
isopropanol, centrifugado a 12000rpm por 10min e resuspendido em 150-200µl de água;
1µl/reação). A análise dos mutantes foi feita pela observação de alterações no fenótipo,
comparando os duplos/triplos mutantes com os mutantes simples, e acompanhados de
experimentos moleculares e/ou bioquímicos complementares. Foram realizados testes de
complementação (restaurar SCI1 selvagem nos mutantes de T-DNA), para garantir que as
alterações observadas foram causadas pela perda de função no locus At1g79200.
Experimentos de Microscopia Para a análise de localização subcelular de SCI1-GFP, folhas de N. tabacum de 6 semanas
de idade foram infiltradas como descrito em Sawers et al. (2006), cortadas em pequenos pedaços,
fixados em formaldeído 3,7%, 50mM NaH2PO4 e 0,2% Triton X-100 por 30minutos, corados com
4,6'-diamidino-2-phenylindole (DAPI) por 15 minutos (1.25µg/ml em PBS) no escuro, lavados
em PBS e montados em água logo antes da análise. Para os experimetnos de complementação de
fluorescência “Biomolecular Fluorescence Complementation” (BiFC), as folhas foram infiltradas
conforme supra citado e infiltradas com uma combinação de duas construções diferentes: NtSCI1hGFP e, NtCYCA1;1 ou NtCYCA2;21-398 ou NtCYCA2;296-398. As fluorescências da GFP e do
DAPI foram obtidas no microcópio confocal a laser “Leica TCS SP2” (Leica Microsystems). As
fatias individuais dos diferentes canais (GFP, DAPI e cloroplasto) foram montadas com Adobe
Photoshop CS (Adobe Systems, San Jose, CA). Pelo menos três análises adicionais sem fixar o
material foram realizadas para confirmar que a autofluorescência do formaldeído não estava
interferindo nos resultados. As imagens mostradas para os experimentos de BiFC representam
uma série de fatias montadas. Pelo menos três repetições biológicas foram realizadas para este
experimento.
Para as análises de microscopia ótica, estigmas/estiletes e anteras de N. tabacum e pistilos
de Arabidopsis foram dissecados de botões florais nos estágios do desenvolvimento floral citados,
fixados e embebidos em parafina conforme descrito para os experimentos de hibridização in situ.
com 0,05% de azul de toluidina em tampão fosfato pH4.4 (O'Brien et al., 1964). As fotos foram
tiradas utilizando o microscópio Leica DM4500B equipado com a câmera Leica DFC320. As
Métodos O material embebido foi cortado em seções de 8µm, montado em slides, desparafinizado e corado
‐ 40 ‐ DePaoli,H.C
2010
análises dos tecidos foram feitas usando o programa “Leica Q-Win Plus v3.4.0” (Switzerland) e
as análises estatísticas foram realizadas utilizando o programa “SigmaStat.v3.5”.
Para a microscopia eletrônica de varredura (MEV), botões florais e flores maduras de
plantas selvagens de N. tabacum (estágios -4 a 1), Arabidopsis (Col-0), transgênicas de N.
tabacum (estágios -4 a 1) e de cada mutante de T-DNA de Arabidopsis analisado, foram coletados
em FAA (formaldeído, ácido acético e etanol 50%, 1:1:18), submetidos a 3 etapas de vácuo de 15
minutos cada, mantidos no fixador por 24horas, desidratados em uma série de etanol (50, 60, 70,
80, 90, 100, 100 e 100%), submetidas à secagem por ponto crítico e cobertas com 30nm de ouro
para serem levadas ao microscópio eletrônico. Para as plantas de N. tabacum, durante a coleta dos
botões florais, uma única antera foi separada do botão floral e submetida à análise por
microscopia ótica (para confirmação dos estágios de desenvolvimento). Foram analisadas pelo
menos duas replicatas biológicas de cada estágio pra cada planta (selvagem e transgênicas) em N.
tabacum, e pelo menos cinco replicatas biológicas de cada mutante, transgene ou planta selvagem
em Arabidopsis.
Para as análises em contraste de fase (DIC), o material vegetal foi fixado em etanol 100%
e ácido acético (3:1) por 2 horas e substituído pela solução de clareamento (100g de tri-cloroacetaldeído monohidratado (“Chloral hydrate”), 5ml de glicerol 100%, 60ml de água e 7,5g de
goma arábica) “overnight”. O material foi observado no microscópio de Normasky (descrever
modelo e marca!).
Coloração com GUS Os tecidos a serem submetidos à coloração com GUS foram coletados em acetona 90%
gelada, deixados por no mínimo 25 minutos no gelo e lavados com tampão de coloração (0.2%
Triton X-100, 50mM NaPO4, 2mM Ferrocianato de Potássio, 2mM Ferricianato de Potássio). Foi,
então, adicionado tampão de coloração com X-Gluc (2mM), gelado, e o material foi infiltrado em
vácuo por três etapas de 10 minutos, até que os tecidos afundassem e a solução parasse de
borbulhar. Em seguida, as amostras foram incubadas a 37ºC overnight. Para parar a reação e
conservar o material para fotografar, foram, então, feitas lavagens com etanol 20%, 35%, FAA
(etanol 50%, 5% Formaldeído e 10% ácido acético), etanol 70% e 100% duas vezes, por 30
minutos cada.
Números de acesso de seqüências utilizadas NtIAA19 foram depositadas no GenBank sobre os números de acesso GQ272329, GQ272330,
GQ272331, GQ272332, GQ272334, GQ272333. As seqüências mencionadas nesta tese podem
ser achadas nas bibliotecas do GenBank/EMBL sobre os números de acesso: X92966 -
Métodos As seqüências de cDNA de SCI1-ORF, HS1-002E06, TOB068F10, NtARF8, NtIAA13 e
‐ 41 ‐ DePaoli,H.C
2010
NtCYCA1;1; D50736 - NtCYCA2;2; AC212309 – SlSCI1 de tomate; AC233353 – StSCI1 de
batata; AM424297, clone VV78X084703.3 – VvSCI1 de uva; AK252156, clone FLbaf149e13 –
HvSCI1 de trigo; NM_001052562 (Os02g0170200) – OsSCI1 de arroz; EU958997, clone 208741
– ZmSCI1 de milho; NM_106571 (At1g79200) – AtSCI1 de Arabidopsis; CAB85553 - SIM
(At5g04470); BAC42937 - SMR1 (At3g10525); AAF18255 - SMR2 (At1g08180); CAB85979 SMR3 (At5g02420); CAD56868 – N. tabacum NtaKIS2; CAC82732 – N. sylvestris NsKIS1;
CAC82731 – N. tomentosiformis NtKIS1a; CAC82734 – N. tomentosiformis NtKIS1b;
NP_179924 – A. thaliana AtKRP1; NP_190632 – A. thaliana AtKRP2.
Mutantes de T‐DNA de Arabidopsis analisados Locus
At1g79200:
SALK_005710.29.99
(sci1-1),
SAIL_31_D10.v1
(sci1-2),
SALK_137501.54.50.x (sci1-3), SALK_142445.50.60.x (sci1-4) e SALK_083219.53.50.x (sci1-
Métodos 5).
‐ 42 ‐ DePaoli,H.C.
2010
- Capítulo 1 -
Um novo gene de estigma/estilete de Nicotiana tabacum, SCI1, que codifica uma
proteína rica em lisina e controla a divisão e diferenciação celular
Henrique C. DePaoli 1,2 , Michael S. Brito 1,2 , Andréa C. Quiapim 1 ,Simone P. Teixeira 3, Gustavo H. Goldman 3,4 ,
Marcelo C. Dornelas 5 e Maria Helena S. Goldman 1
1
Departamento de Biologia, Faculdade de Filosofia, Ciências e Letras de Ribeirão Preto, Universidade de São Paulo,
Brazil 14040-901
2
PPG Genética, Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Brazil; 14049-900
3
Departamento de Ciências Farmacêuticas, Faculdade de Ciências Farmacêuticas de Ribeirão Preto, Universidade
de São Paulo, Brazil; 14040-903
4
Laboratório Nacional de Ciência e Tecnologia do Bioetanol (CTBE), Brazil
5
Departamento de Biologia Vegetal, Instituto de Biologia, Universidade de Campinas, Brazil; 13083-970
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 durante o desenvolvimento
‐ 44 ‐ DePaoli,H.C.
2010
RESULTADOS Identificação do gene SCI1 e análise de sua localização subcelular Foram construídas recentemente duas bibliotecas de cDNA de hibridização subtrativa
suprimida (SSH), que permitiram a identificação de vários genes especificamente ou
preferencialmente expressos no pistilo de Nicotiana tabacum (DePaoli et al., manuscrito em
preparação). Entre eles, optamos por um clone, TOBSH1-002E06, para posterior análise e
caracterização. Como este era um clone de cDNA parcial (Figura 1A), que representa a
extremidade 5' do transcrito, foi realizada uma pesquisa por BLASTn no banco de dados
TOBEST (Quiapim et al., 2009) e encontrou-se dois clones muito semelhantes com sobreposição
ao clone TOBSH1-002E06: TOB048C05 e TOB068F10. O clone TOB068F10 foi completamente
seqüenciado e demonstrou conter, além da região codificadora, uma 3’UTR e uma cauda polyA.
O alinhamento do HS1-002E06e o clone TOB068F10 pode ser visto na Figura 1.
É interessante notar que ele tem um 3'UTR excepcionalmente longo (395 pb), que pode
ser importante para regulação do transcrito, por exemplo, para influenciar a sua estabilidade. O
gene correspondente foi designado SCI1 (inibidor do ciclo celular estigma-1) com base nos
resultados mostrados abaixo.
A busca por homologia em banco de dados internacional, utilizando a seqüência de SCI1,
via TBLASTX (Altschul et al., 1997), mostrou que a proteína codificada é semelhante às
proteínas de função desconhecida presentes em diversos organismos vegetais. As seqüências com
a maior similaridade com o produto do gene SCI1 são de batata (AC233353), tomate
(AC212309), cevada (AK252156), milho (EU958997), arroz (NM_001052562) e uva
(AM424297). A suposta seqüência homóloga no genoma de Arabidopsis é At1g79200
(NM_106571), que compartilha a identidade de 65% e 86% de similaridade com SCI1. O
pequena sequência conservada no N-terminal; II) um domínio intermediário rico em lisina com
baixa conservação de seqüências e; III) domínio altamente conservado no C- terminal.
Para descobrir mais informações sobre essa seqüência de proteína putativa e orientar
nossos experimentos futuros, foi feita uma detalhada análise in silico utilizando a sequência
completa de 154 aminoácidos de SCI1. Através da combinação de vários pacotes de software de
análise de seqüência (ver Métodos), identificamos um domínio N-terminal rico em lisina (LRD)
(MotifScan, Evalue = 6,5 E-07), com resíduos de lisina previstos para serem Nε-acetilados, um
sinal de localização nuclear - NLS (PSORT, 0,992 afirmativa; PredictNLS), dois domínios de
interação com ciclina (previsão ELM) e um total previsto de 15 sítios de fosforilação (NetPhos, ≥
96%) (Figura 1B). Os domínios de interação com ciclina identificados, consenso [RK]. L. (0,1)
[FYLIVMP], estão presentes em uma ampla gama de proteínas que interagem com ciclinas e/ou
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 alinhamento dessas proteínas (Figura 2) permitiu o reconhecimento de 3 domínios: I) uma
‐ 45 ‐ DePaoli,H.C.
2010
CDKs (Takeda et al., 2001). Além disso, encontrou-se sítios de fosforilação alvos de diferentes
moléculas sinalizadoras (caseína quinase 1 - CK1; CK2 cinase 2 - caseína e MAPKs), todos os
três presentes em SCI1 (NetPhosK; previsão ELM: 23aa posições 17, 54 60aa, 109 115aa, 112
118aa para CK1 , as posições 4 10aa, 44 50aa, 53 59aa para CK2 e posições 4 10aa, 15 e 18-21aa-
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 24aa para MAPK).
‐ 46 ‐ A HS1-002E06
SCI1-ORF
TOB068F10
1
1
TCAGAGGTGC GAGTAAATGG GGAGCGATAA GAAGACGACG GAGGAGAAGA
---------- ------ATGG GGAGCGATAA GAAGACGCCG GAGGAGAAGA
---------- ---------- ---------- ---------- ----------
HS1-002E06
SCI1-ORF
TOB068F10
51
35
GGAAGCATAA GAGAAATTCG CCTTCTTCTC CACGAGATGA AGTGAAAAGC
GGAAGCATAA GAGAAGTTCG CCTTCTTCTC CACGAGATGA AGTGAAAAGC
---------- ---------- ---------- ---------- ----------
HS1-002E06
SCI1-ORF
TOB068F10
101
85
AAACGCCAGA ATATCAAAGG GGATGAAGAG CGAAGGAAAG AAAAG---GA
AAACGCCAGA ATATCAAAGG GGATGAAGAG CGAAGGAAAG AAAAGAAGGA
---------- ---------- ---------- ---------- ----------
HS1-002E06
SCI1-ORF
TOB068F10
148
135
CAAATCCAAG AAGGAGAAGC ACAAATCCCA TAAATCCAAA TGTCATTCTA
CAAATCCAAG AAGGAGAAGC ACAAATCCCA T--------- ------TCTA
---------- ---------- ---------- ---------- ----------
HS1-002E06
SCI1-ORF
TOB068F10
198
170
1
GTGAAGAGAA GAAGTCAGGG GAAAAGCACA AAACCAAGAG CCACAAACAT
GTGAAGAGAA GAAGTCAGGG GAAAAGCACA AAACCAAGAG CCACAAACAT
---------- ---------- ---------- ---------- ----------
HS1-002E06
SCI1-ORF
TOB068F10
248
220
1
AAGGATAAAT CGAAAAACAA GTTTGAAGAG TTATCAAAAG ATGATTATTT
AAGGATAAAT CGAAAAACAA GTTTGAAGAG TTATCAAAAG ATGATTATTT
---------- CGAAAAACAA GTTTGAAGAG TTATCAAAAG ATGATTATTT
HS1-002E06
SCI1-ORF
TOB068F10
298
270
41
CTCTAAGAAC AATGAATTTG CTTCTTGGCT GAAAGATAAG AAGAATTTGT
CTCTAAGAAC AATGAATTTG CTACTTGGCT GAAAGATAAG AAGAATTTGT
CTCTAAGAAC AATGAATTTG CTACTTGGCT GAAAGATAAG AAGAATTTGT
HS1-002E06
SCI1-ORF
TOB068F10
348
320
91
TCTTCTCAGA TCTTTCGTCT GAGACTGCAC GCAATTTATT CTCTGACTTT
TCTTCTCAGA TCTTTCGTCT GAGACTGCAC GCGATTTATT CTCTGACTTT
TCTTCTCAGA TCTTTCGTCT GAGACTGCAC GCGATTTATT CTCTGACTTT
HS1-002E06
SCI1-ORF
TOB068F10
398
370
141
GTCATACAAT GGAACAAGGG AAAGCTTGAC AGCCAATACT ATGAGGGAAT
GTCATACAAT GGAACAAAGG AAAGCTTGAC AGCCAATACT ATGAGGGAAT
GTCATACAAT GGAACAAAGG AAAGCTTGAC AGCCAATACT ATGAGGGAAT
HS1-002E06
SCI1-ORF
TOB068F10
448
420
191
TGCAACCGG- ---------- ---------- ---------- ---------TGCAACCGGG CCTCGGTCAT CCCACGCTTG GAATATCAAA AAGTAA---TGCAACCGGG CCTCGGTCAT CCCACGCTTG GAATATCAAA AAGTAATAGA
HS1-002E06
SCI1-ORF
TOB068F10
457
466
241
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------ATTCCATGTT TCTTCTCTTT AAGGAGCAGT TGTATTGTCT TTTTTCCAGT
HS1-002E06
SCI1-ORF
TOB068F10
457
466
291
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------TGTCCGATGG TTATGTCTTA GGCTTCTGTA TTTTTGTGTT TTCTTTTTTC
HS1-002E06
SCI1-ORF
TOB068F10
457
466
341
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------AGCAGAACAT GCTTAGTCTT ATGAAGTTAC TTTCTTGGAT AATGGTAAAA
HS1-002E06
SCI1-ORF
TOB068F10
457
466
391
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------TCACCATCTC TGTTGTAAGC TATGTTGTCC GGACTCTCCA AAATCCTGCC
HS1-002E06
SCI1-ORF
TOB068F10
457
466
441
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------GCATCCCCGT CGGACCCTCC AAAAATGCAC ATACTTTTAG AGGATCCGAC
HS1-002E06
SCI1-ORF
TOB068F10
457
466
491
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------ACGCACCTGG TAACATTTTT AGTGAGTCTG GGCAACATAG GTTGTAAGAG
HS1-002E06
SCI1-ORF
TOB068F10
457
466
541
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------TTCTATGCTA GTCCTACCAC GTGACTGCAT TTATCATGGC CTTTGGCTGC
HS1-002E06
SCI1-ORF
TOB068F10
457
466
591
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------AGAGACGGAC AATACTTCTT ATGATTCACG TAAACTCAAT AGTTTTTATT
HS1-002E06
SCI1-ORF
TOB068F10
457
466
641
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------CATACTTTCT ATATGTATTA AAAATTTATT GAATATTTAT AAATGTTAAA
HS1-002E06
SCI1-ORF
TOB068F10
457
466
691
---------- ---------- ---------- ---------- ------------ ---------- ---------- ---------- --ACTATAAATT CCAATAAAAA AAAAAAAAAA AAAAAAAAAA AAA
2010
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 DePaoli,H.C.
‐ 47 ‐ DePaoli,H.C.
2010
B atggggagcgataagaagacgccggaggagaagaggaagcataagagaagttcgccttct 60
M G S D K K T P E E K r k h k r s s p s 20
tctccacgagatgaagtgaaaagcaaacgccagaatatcaaaggggatgaagagcgaagg 120
s p r d e v k s k r q n i k g d e e r r 40
aaagaaaagaaggacaaatccaagaaggagaagcacaaatcccattctagtgaagagaag 180
k e k k d k s k k e k H K S H S S E E K 60
aagtcaggggaaaagcacaaaaccaagagccacaaacataaggataaatcgaaaaacaag 240
K S G E K H K T K S H K H K D K S K N K 80
tttgaagagttatcaaaagatgattatttctctaagaacaatgaatttgctacttggctg 300
F E E L S K D D Y F S K N N E F A T W L 100
aaagataagaagaatttgttcttctcagatctttcgtctgagactgcacgcgatttattc 360
K D K K N L F F S D L S S E T A R D L F 120
tctgactttgtcatacaatggaacaaaggaaagcttgacagccaatactatgagggaatt 420
S D F V I Q W N K G K L D S Q Y Y E G I 140
gcaaccgggcctcggtcatcccacgcttggaatatcaaaaagtaa
A T G P R S S H A W N I K K *
465
154
Figura 1 – Seqüência dos clones de cDNA de SCI1 e dos aminoácidos deduzidos destes com os domínios funcionais
identificados in silico. (A) Seqüência de nucleotídeos do clone TOBSH1-002E06, representando três seqüências
identicas identificadas por SSH (DePaoli et al., manuscrito em preparação), do clone TOB068F10, identificado na
busca por homologia no banco de dados TOBEST (Quiapim et al., 2009), e o produto de PCR da fase de leitura aberta
putativa (“SCI1-ORF”), que foi utilizada nas contruções SCI1Ri e SCI1OE, alinhadas pelo Dialign 2.2.1 (Brudno et al.,
2003). As sequencias dos primers utilizados nas reações de PCR estão sublinhados, letras em negrito e itálico
representam o 5’UTR e 3’UTR respectivamente. O ATG inicial está destacado em cinza claro, o códon de terminação
está em cinza escuro e as diferenças nucleotídicas em preto. Os gaps (-) foram inseridos para permitir o melhor
alinhamento das sequencias, que demonstraram ter deleções de códons e/ou inserções (AAG, codificando o amino ácido
Lisina; e CATAAATCC AAATGT, codificando os amino ácidos Lisina, Serina, Lisina, Cisteína e Histidina,
respectivamente). (B) Sequencias de nucleotídeos e amino ácidos deduzidos de “SCI1-ORF” mostrando o domínio rico
em lisina (LRD) em cinza claro, o sinal de localização nuclear (NLS) indicado em letras minúsculas, os sítios de
fosforilação em ser/thr/tyr (prediction ≥ 96%) são mostrados por amino ácidos negritos e sublinhados e, por fim, os dois
domínios putativos de interação com ciclinas (LIG_CYCLIN) estão destacados em cinza escuro. O primeiro
aminoácido “K” do domínio LIG_CYCLIN também pertence ao domínio LRD. Os nucleotídeos sublinhados e em
negrito representam os primers de RT-PCR em tempo real. O símbolo “*” refere-se ao codon de terminação. As
posições dos nucleotídeos e dos amino ácidos estão indicadas.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 ‐ 48 ‐ DePaoli,H.C.
2010
NtSCI1
SlSCI1
StSCI1
VvSCI1
HvSCI1
OsSCI1
ZmSCI1
AtSCI1
┌----------DOMAIN I----------┐┌------------------------DOMAIN II-----------------------------┐┌----------------------------------DOMAIN III---------------------------------┐
MGSDKKTPEEKRKHKRSSPSSPR-DEVKS-KRQNIKGD-----------EERRK-E--KKDKS-KKEKHKSHSSEEKKSGEKHKTKSH-KHKDKSKNKFEELSKDDYFSKNNEFATWLKDKKNLFFSDLSSETARDLFSDFVIQWNKGKLDSQYYEGIATGPRSSHAWNIK-K
MGSDRKRKEEKMKHKRNSPSSPQADEGRS-KRQKIKGD-----------EETSK-DKSRKEKH-KSHKSKRRSNEDRESGEKHKSKSH-KHKDKLKIKFEELSKDDYFSKNNEFATWLKDKKKLFFSDLSSEAARDLFSDFVKEWNKGKLDIQYYEGITTAPRSSHAWNIK-K
MGSDRKMKEEKIKHKRSSPSSPQADEGRS-KRQKIKGD-----------EEPSK-DKSRKDKH-KSHKSKRHSNEEKESGEKHKSKSH-KHKDKLKIKFEELSKDDYFSKNNEFATWLKEKKKLFFSDLSSEAARDLFSNFVKDWNKGKLDIQYYEGITTAPRSSHAWNIK-K
MGS-----ESKEKKRRSSPSISS-DGGKG-KRRRSVED------------K-KE-K--RKDKK-SHRHSKHHSDKEKKSEGKHKDKRH-KRDRHLILEFQELSNDDYFSKNNEFAAWLKEKRKIYFADLSTESARELFTEFVKDWNKQKLESKYYEGIVTGPRTSHKWNIK-Q
MGGG----EAKSRKRRSSPSSGEEGERERKKRRDKKESRRSGR----GSDEEEE-KRRRKKKHGDRGKDKERDSKERRSKEKDKSKRKDKDVSSLKQDIKEISKDDYFAKNNEFATWLKEEKGKYFSDLSSESARDIFLKFVKQWNKGKLPSQYYEGITTGPRSAHNWNIKKA
MGG-----EGKSRKRRSSPSSGEEEERERKKRRDKKESRRSSRDEQAEEEEEEERRRRKKKKHGDRGKDKERDSKEKHSKEKEKSKRKHKD-----DDFKEISKDDYFAKNNEFATWLKEEKGKFFSDLSSESARDLFEKFVKTWNKGKLPSQYYEGITRGPRSAHKWNIK-V
MGGG----EGKSKKRRSSTSSAEEEGGER-KRRDKKESKRRSRDD----REDDDDRHKKKGKHIDRNKGKERDSKDRHSKEKT-SKRKDKD-----AAFKEISKDDYFAKNNEFATWLKEEKGKYFSDLSSESARDLFLKFVKQWNKGKLPSQYYEGITSGPRSAHNWNIK-A
MVSERSSKEKKRSRARSEDSSSSDYEEKVKRHRGTEKD-----------DERRSRRSDKKDK--KSHKHHKSSTSKKSKDDKPKKKHT-ESDHKLKEGIPELSMEDYFSKNNEFATWLKEEKRTYFNDLTTEAARGLFSRFVKRWNRGKLESRYYEGISTAPRTAHDWMIKRR
cons
* .
: *.. *
.
:::
.
:
.
:* *
.
: :
. ..: .
*
*
:
: *:* :***:******:***:::
:* **::*:** :*
**
**: **
:*****
.**::* * **
BAD AVG GOOD Figura 2 – Alinhamento da sequencia de amino ácidos de SCI1 em tabaco (NtSCI1) com seus putativos homólogos em outras espécies de plantas. O alinhamento múltiplo foi
construído no programa T-COFFEE (versão 5.53). A s cores representam o valor do alinhamento (BAD, ruin; AVG, médio; GOOD, bom). Os domínios I, II e III propostos
estão indicados. NtSCI1 (sequencia de N. tabacum descrita aqui), SlSCI1 - tomate (AC212309), StSCI1 - batata (AC233353), VvSCI1 - uva (AM424297), HvSCI1 - cevada
(AK252156), OsSCI1 - arroz (NM_001052562), ZmSCI1 - milho (EU958997), AtSCI1 – Arabidopsis At1g79200 (NM_106571). Cons., seqüência consenso.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 * *
‐ 49 ‐ DePaoli,H.C.
2010
Para o estudo da potencial localização nuclear de SCI1, fundiu-se a proteína fluorescente
verde (GFP) ao C-terminal de SCI1 (35SPROM:SCI1-GFP) e analisou-se sua localização subcelular
in planta. A microscopia de fluorescência revelou que a proteína SCI1-GFP foi exclusivamente
localizada no núcleo (Figuras 3A-B), contrastando com 35SPROM:GFP que mostrou uma
distribuição uniforme da GFP no núcleo e no citoplasma (dados não mostrados). Sabe-se que
DAPI cora preferencialmente a heterocromatina e, curiosamente, a fluorescência de SCI1-GFP
estava presente no nucleoplasma, onde DAPI não foi detectado, correspondendo à intercromatina
e ao nucléolo (Figuras 3E-L). Ambos os padrões relatados da proteína de fusão ocorrem com
baixos e altos níveis de expressão, que foram estimados pela sua intensidade de fluorescência sob
o microscópio. Esta observação indica que a superexpressão da proteína de fusão não está
influenciando os diferentes padrões de localização observados. Na verdade, o padrão nuclear de
SCI1-GFP se assemelha aos observados pelas proteínas transducina/WD-40, que interage com
reguladores do ciclo celular, e a proteína ribossomal L27A da subunidade 60S, ambas de
Arabidopsis (Brown et al., 2005). Em conclusão, SCI1 é uma proteína nuclear que co-localiza
com o núcleo, especificamente na região intercromática.
SCI1 é um gene específico de estigma/estilete regulado ao longo do desenvolvimento Para determinar o padrão de expressão do gene SCI1 e confirmar a sua especificidade de
pistilo, foram realizados experimentos de qRT-PCR utilizando RNA a partir de raízes, caules,
folhas, sépalas, pétalas, estames, estigmas/estiletes e ovários. SCI1 é pelo menos 34 vezes mais
expresso no estigma/estilete do que em qualquer outra parte da planta (Figura 3M). Analisou-se
também o padrão de expressão temporal do gene SCI1 no estigma/estilete durante os 12 estágios
do desenvolvimento descrito por Koltunow et al. (1990). No estágio 1, o pistilo está totalmente
diferenciado e vai crescer e se preparar para a receptividade e maturidade, até o estágio 12, no
é observada no estágio 1, o estágio mais jovem investigado. Nas fases seguintes, a transcrição
diminui sua abundância e o seu nível varia durante a primeira metade do período de
desenvolvimento, onde ocorrem os níveis mais altos (estágios iniciais de 1 a 6) do que nas fases
posteriores em direção à antese (7-12) (Figura 3N). Assim, o padrão de expressão de SCI1 sugere
que o seu produto gênico esteja mais envolvido com os eventos que acontecem no início do
desenvolvimento do que nos que ocorrem no final do desenvolvimento do pistilo.
O gene SCI1 é expresso exclusivamente nos tecidos especializados do estigma/estilete relacionados com a reprodução de plantas Para verificar quais tipos de células transcreviam o gene SCI1, foram realizados
experimentos de hibridização in situ (Figuras 3O-P). No estágio 4, observou-se um sinal de
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 qual ocorre a antese e a polinização. A Figura 3N mostra que o maior nível de expressão de SCI1
‐ 50 ‐ DePaoli,H.C.
2010
hibridação na zona secretora do estigma, incluindo as células papilares, e no tecido transmissor do
estilete (Figura 3P). Um sinal fraco foi observado no feixe vascular, porém este também foi visto
nas seções de estigmas/estiletes hibridizados com a sonda senso (controle). Não havia sinal
detectável nos estágios 7 e 10 (dados não mostrados). Estes resultados estão de acordo com o
padrão de expressão do gene SCI1 ao longo do desenvolvimento determinado por qRT-PCR, em
que uma redução significativa da expressão de SCI1 foi observada nos estágios 7 e 10 quando
Figura 3- Análise de localização subcelular e de expressão gênica de SCI1.
(A-L) SCI1-GFP está localizada no núcleo in planta (folhas de tabaco), e claramente situada na região intercromática
(A, C, E e G) e no nucléolo (B, D, F e H). (A, B): expressão do 35S:SCI1-GFP; (C, D): Coloração com DAPI; (E, F):
sobreposição dos paineis de cima e do meio mais a fluorescência do cloroplasto em vermelho (merge); (G, H):
sobreposição de 10 (G) e 12 (H) seções dos sinais de GFP, DAPI e cloroplasto através da fatia celular (“3D” merge). A
imagens são representadas como cortes das seções vizinhas. (I, J, K, L): imagens representativas em escala de cinza
dos paineis E, G, F e H, respectivamente. Barra de escalas são 4μm em todos os painéis. cl, cloroplasto; is, espaço da
intercromatina; n, núcleo; nu, nucléolo. (M) Nível de expressão de SCI1 em diferentes órgãos de N. tabacum (SR-1)
determinados por qRT-PCR. Expressão em unidades arbitrárias (U. A.) correspondem à taxa do número de cópias de
cDNAs de SCI1 sobre o número de cópias do controle constitutivo do gene da β-actina em uma quantidade definida de
RNA total de cada amostra (ver Métodos). (N) Nível de expressão no stigma/estilete durante os 12 estágios do
desenvolvimento da flor de tabaco (estágios descritos em Koltunow et al., 1990) (Uma escala logarítmica foi utilizada
para permitir a visualização de baixos níveis de expressão. (O) Hibridização in situ realizada em cortes longitudinais do
stigma/estilete no estágio 4, utilizando uma sonda senso preparada a partir do clone HS1-002E06(controle negativo) e
(P) Localização dos mRNAs de SCI1 como revelado pela sonda anti-senso de TOBSH1-002E06. O fator de aumento é
de 40x; arrow cap, célula papilar; p, parênquima; ssz, zona secretória do estigma; stt, tecido transmissor do estilete;
vb, feixe vascular.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 comparados à expressão no estágio 4.
‐ 51 ‐ DePaoli,H.C.
2010
A transcrição de SCI1 é claramente mais abundante nos tecidos especializados do
estigma/estilete nos estágios iniciais do desenvolvimento do órgão. Embora esses tecidos estão
intrinsecamente relacionados à interação pólen-pistilo, a maioria dos genes relacionados a essas
interações têm um nível de expressão maior nas fases finais do desenvolvimento da flor (Goldman
et al., 1992, Wang et al., 1993; McClure et al. , 1999). Por outro lado, espera-se que os genes que
controlam a proliferação e diferenciação celular são expressos em níveis mais elevados nos
estágios iniciais do desenvolvimento da flor. Em conjunto, os padrões de expressão temporal e
espacial sugerem um envolvimento do produto do gene SCI1 no desenvolvimento morfológico
e/ou fisiológico do pistilo ao invés de contribuir diretamente para as interações pólen-pistilo.
O produto do gene SCI1 controla o tamanho do estigma Para desvendar a função da proteína SCI1, foram geradas plantas transgênicas de tabaco
com a expressão deste gene sendo silenciado por “RNA de grampo” (hairpin RNA - RNAi).
Adicionalmente, foram construídas plantas que superexpressam SCI1. Foram obtidas 13 plantas
transgênicas independentes possuindo a construção de RNAi, as quais foram designadas SCI1Ri, e
9 plantas transgênicas independentes, possuindo o cassete de superexpressão, designadas SCI1OE.
A Figura 4A mostra que o resultado do qRT-PCR no silenciamento mediado por RNAi,
demonstrando que houve uma diminuição eficiente nos níveis de transcrito (até 6,4% do nível de
transcrito do tipo selvagem para SCI1Ri17.9). As plantas com os mais baixos níveis de transcrição
mostraram um aumento nítido no tamanho do estigma (Figura 5B) e um elongamento marcante de
estilete, de até 28% na antese (Figura 5A). Este fenótipo foi observado em 9 das 13 plantas
transgênicas e RNAi e foi visível quando os níveis de transcrito de SCI1 foram abaixo de 27,8%
do nível das plantas do tipo selvagem (Figura 4A barras tracejadas). Houve uma correspondência
direta entre os níveis de mRNA (RNA mensageiro) de SCI1 e a presença dos fenótipos
expressivo (Figura 5A). As diferenças entre o tipo selvagem SR-1 e os fenótipos de RNAi foram
mais evidentes no estágio 10 do desenvolvimento floral (Figura 5A). Nesta estapa, o
estigma/estilete permanece fechado dentro da corola em plantas do tipo selvagem (Figura 5A*),
enquanto que nas plantas de RNAi o estigma/estilete é pronunciado para fora da corola (Figura
5A), fenótipo observado desde o estágio 8. De acordo com o fenótipo gerado pelo silenciamento
de SCI1, as plantas SCI1OE revelaram uma alteração oposta com relação ao estigma, ou seja, um
tamanho menor (Figura 5B). Entre 9 plantas transgênicas independentes de superexpressão, 5
transformantes apresentaram o mesmo fenótipo, que foi associada com os maiores níveis de
transcrito de SCI1 (48,8 vezes ou mais do que em plantas do tipo selvagem), conforme
demonstrado por qRT-PCR (Figura 4B). Por outro lado, não observamos alterações no
comprimento do estilete nas plantas SCI1OE, que posicionou os estigmas na mesma altura do topo
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 observados, com a planta SCI1Ri14.2 (8,4% do mRNA de SCI1) apresentando o fenótipo mais
‐ 52 ‐ DePaoli,H.C.
2010
dos estames e pétalas na antese (como ocorre nas plantas selvagens SR-1). A planta SCI1OE1.1
mostrou um fenótipo semelhante ao das plantas de RNAi e, consequentemente, o nível de mRNA
de SCI1 nesta planta transgênica foi de apenas 17,7% do nível de plantas do tipo selvagem
(Figura 4B). Este efeito de co-supressão é um mecanismo de silenciamento gênico póstranscricional conhecido, induzido em algumas plantas em resposta à tentativa de superexpressão
de um gene (Napoli et al., 1990; Taylor, 1997). Nenhum outro órgão da planta mostrou um
fenótipo alterado, macroscopicamente notável, quando se comparam as plantas do tipo selvagem e
as transgênicas (SCI1Ri e SCI1OE). Em conjunto, estes resultados implicam em um forte
envolvimento da proteína SCI1 no controle do tamanho do estigma, provavelmente através da
Figura 4 – Nível de expressão de SCI1 em estimgas/estiletes no estágio 4 da plantas transgênicas SCI1Ri and SCI1OE,
determinado por qRT-PCR.
Nível de expressão em unidades arbitraries (A.U.) corresponde à taxa do número de cópias de cDNAs do gene SCI1 em
cada planta transgênica (normalizado pelo controle interno do gene da β-actina) pelo número de cópias do cDNA do
gene SCI1 em plantas selvagens SR-1 (W.T.) (barras pretas) (igualmente normalizados pelo gene da β-actina), em uma
quantidade definida de RNA total (Métodos). As barras hachuradas representam as plantas transgênicas que
apresentaram um fenótipo alterado com estiletes elongados e superfícies estigmáticas maiores. As barras em xadrez
correspondem às plantas com tamanho do stigma reduzido, em contraste às barras vazias que representam um fenótipo
parecido com o tipo selvagem. (A) Nível de expressão em porcentagem (A.U. x100) pra cada planta de RNAi obtida.
(B) Nível de expressão em A.U. pra cada planta de superexpressão (OE) obtida (Uma escala logarítmica foi usada para
permitir a compração entre os níveis de expressão bem diferentes).
Plantas
Plantas Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 Nível de Expressão (U.A.) Nível de Expressão (%) inibição da proliferação celular e/ou inibição da expansão celular de forma tecido-específica.
‐ 53 ‐ DePaoli,H.C.
2010
Figura 5 – Análise do fenótipo das plantas transgênicas de SCI1.
(A) Flores de três transformantes independentes de RNAi mostrando o aumento na elongação do estilete nos estágios
12, 10 e 9 (da esquerda para a direita) do desenvolvimento floral. (*) Vista de cima da planta selvagem no estágio 10 do
desenvolvimento floral mostrando o stigma/estilete comletamente envolvido pela corolla. As barras representam 10mm.
As porcentagens correspondem ao nível de transcrito de SCI1 em comparação ao nível da planta selvagem, considerado
100%. (B) Vista superior do stigma no estágio 12 do desenvolvimento floral mostrando a redução no tamanho em
plantas de superexpressão (SCI1OE 11.1; Ba) e o aumento no tamanho em plantas de RNAi (SCI1Ri14.2; Bc),
comparados com a planta selvagem (W.T.; Bb). O nível de expressão está em unidades arbitrárias (U.A.) como descrito
na Figura 4. As barras de escala representam 1mm para todas as imagens. (C) Esquema de um corte longitudinal (Ca)
representando a região onde as camadas celulares da SSZ foram contadas (≡) (5D; Figura 6). A interseção entre SSZ e
STT foi definida por uma linha reta traçada nas curvas inferiores da estrutura em forma de guarda-chuva no stigma
(linha pontilhada verde em 5Ca). Em cinza claro está mostrada a area da SSZ avaliada e medida nas seções
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 Nível de Expressão (U.A.) ‐ 54 ‐ DePaoli,H.C.
2010
longitudinais das plantas transgênicas e selvagem (Figura 6; plano longitudinal 5Ec1). O quadrado (□) indica a região
em que o número de células na SSZ foi contado em uma área de 20.000μm2. O STT está representado em cinza escuro e
sua espessura foi medida a 400µm abaixo da interseção SSZ/STT (5Ca, linha pontilhada verde). O retângulo representa
a região mostrada em 5Ea/b para cada planta e na qual a espessura do STT e do número de células no parênquima
foram medidas. Seções longitudinais de estigmas/estiletes no estágio 11 e coradas com azul de toluidina (0,05%) são
mostradas em (Cb) para a planta de superexpressão SCI1OE3.1.1, (Cc) tipo selvagem SR-1 (W.T) e (Cd) planta de
RNAi SCI1Ri14.2, fornecendo evidências da diferença de área ocupada por cada SSZ. As barras de escala representam
500µm pra todas as fotos. As setas azuis e pretas indicam, respectivamente, uma junção do carpelo mais funda na planta
SCI1OE3.1.1 e o feixe vascular. (D) Seções longitudinais de estigmas/estiletes no estágio 11 em um aumento maior que
mostrado em 5C (200x), demonstrando o número de células alterado na SSZ. (E) Cortes histológicos de estiletes no
estágio 11. Seções longitudinais das plantas SCI1OE3.1.1, W.T. e SCI1Ri14.2, mostrando as diferenças na espessura no
tecido transmissor do estilete (Ea) e do parênquima (Eb), feitos no plano longitudinal indicado em Ec2. Note as
diferenças na espessura do STT de cada planta, indicando alteração no número de células desse tecido. (Ec) Seção
transversal do estilete selvagem mostrando os dois planos longitudinais onde foram determinados a área, as camadas
celulares a o número de células na SSZ, assim como a espessura do STT (Ec1; Figura 6) e onde a espessura do estilete a
o número de células do parênquima foram determinados (Ec2; Figura 6). O plano longitudinal “2” corresponde à
primeira camada de células atrás do tecido vascular. As barras de escala representam 50μm em Ea/Eb e 500μm em Ec.
p, parênquima; stt, tecido transmissor do estilete; ssz, zona secretória do estigma; vb e black arrow, feixe vascular.
A proteína SCI1 é um regulador negativo do ciclo celular Como um esforço para elucidar a função de SCI1 e distinguir entre os efeitos de
proliferação celular e/ou expansão celular, preparou-se cortes longitudinais de estigmas/estiletes
no estágio 11 do desenvolvimento floral em plantas SCI1Ri14.2, SCI1OE3.1.1 e plantas do tipo
selvagem. Avaliou-se a área da zona secretora do estigma (SSZ) (Figura 6 e Tabela 1), bem como
o número de camadas celulares na SSZ na região indicada na Figura 5Ca. A Figura 5 mostra que
o número de células é claramente maior (p <0,05) na zona secretora do estigma (Figuras 5Cd e
5D, Figura 6 e Tabela 1) como consequência da redução dos níveis de mRNA de SCI1, enquanto
que a superexpressão de SCI1 resultou em uma diminuição do número de células (p <0,05) na
SSZ (Figuras 5Cb e 5D, Figura 6 e Tabela 1). Por conseguinte, a planta transgênica SCI1Ri14.2
selvagem SR-1, que foi de 248.272 μm2 (Figura 5Cd e 5Cc, respectivamente, Figura 6 e Tabela
1). Em contrapartida, a planta transgênica SCI1OE3.1.1 mostrou uma área média de SSZ de
191.381,3 μm2, 23% menor do que a da planta selvagem SR-1 (Figura 5Cb; Figura 6 e Tabela 1).
Da mesma forma, o número de camadas de células (cl) no topo da SSZ (Figura 5Ca) foi
significativamente reduzida para cerca de 8cl em SCI1OE3.1.1, enquanto que este valor foi de 13cl
para a planta selvagem, e significativamente aumentado para 18cl em SCI1Ri14.2 (Figura 6 e
Tabela 1). Além da medição da área da SSZ e dos dados de camadas celulares (cl), também foi
determinado o número de células em uma área delimitada de 20.000 μm2 na SSZ (Figura 5Ca,
Tabela1) para estimar alterações no tamanho das células. Neste contexto, o número de células da
planta de superexpressão SCI1OE3.1.1 é menor, mas não estatisticamente diferente do tipo
selvagem e da planta SCI1Ri14.2, ambas com valores muito semelhantes (Figura 6 e Tabela 1).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 mostrou uma área média de SSZ de 367.355,3 μm2, 48% maior que a área média da planta
‐ 55 ‐ DePaoli,H.C.
2010
Não se observou o fenômeno chamado de compensação, que aumenta o tamanho da célula em
uma resposta de equilíbrio para a diminuição do número de células nas plantas de superexpressão.
No entanto, as células da SSZ na planta SCI1OE3.1.1 eram mais circulares em forma e mais
esparçadas, de forma que o espaço intercelulares foi ligeiramente aumentado, o que é consistente
com uma menor densidade celular. Além disso, uma junção carpelar mais profunda foi observada
(Figura 5Cb, seta). Por outro lado, as células da SSZ na planta SCI1Ri14.2 parecem ser
ligeiramente menores, provavelmente devido a um crescimento empacotado e/ou uma rápida reentrada no ciclo celular. No entanto, tomados como um todo, as células das plantas transgênicas e
do tipo selvagem demonstraram ter aproximadamente os mesmos tamanhos.
Uma vez que os experimentos de hibridização in situ demonstraram que SCI1 também
possui expressão no tecido transmissor do estilete (STT), analisou-se possíveis alterações neste
tecido nas plantas transgênicas. Como as células do STT são mais empacotadas, contá-las
individualmente como fêz-se para as células da SSZ é muito difícil. Assim, mediu-se a espessura
do STT em plantas transgênicas e do tipo selvagem (Figura 5Ea; Figura 6 e Tabela 1), em cortes
longitudinais do estigma/estilete a uma distância de 400μm abaixo da interseção entre a SSZ e o
STT (para ver detalhes desta definição veja Material e Métodos e Figuras 5E e 5Ca). De forma
coerente, a espessura do STT foi diminuído nas plantas SCI1OE3.1.1 (p <0,05) e aumentado nas
plantas SCI1Ri14.2 (p <0,05), quando comparados com o STT em plantas do tipo selvagem
(Figura 6 e Tabela 1), indicando, respectivamente, um menor e um maior número de células neste
tecido. É importante salientar que a planta de RNAi SCI1Ri14.2 mostrou um número maior de
células também no parênquima, quando comparado às plantas selvagem e SCI1OE3.1.1 (Figura
5Eb e Figura 6). Uma vez que SCI1 não é expresso nas células do parênquima, nossos resultados
sugerem que as células do SSZ e STT podem produzir sinais capazes de influenciar a divisão
celular nos tecidos adjacentes, mantendo a proporcionalidade certa: quanto mais células nos
espessura do estilete em plantas SCI1Ri14.2 (Figura 6 e Tabela 1) é um somatório do
supercrescimento de dois tecidos, o parênquima e o STT, enquanto que o tecido do parênquima
permanece aproximadamente o mesmo na planta de SCI1OE3.1.1. Em conjunto, estes resultados
revelam a participação da proteína SCI1 na inibição do ciclo celular de uma forma tecidoespecífica.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 tecidos especializados, maior o número de células no tecido de apoio. Resumindo, a maior
‐ 56 ‐ DePaoli,H.C.
2010
Figura 6 – Análises realizadas no estigma/estilete das plantas transgênicas e tipo selvagem.
Seções longitudinais de estigmas/estiletes de cada planta foram analizados como descrito na Figura 5Ca para a camada
de células, área e número de células (em uma área de 20.000µm2) da SSZ e espessura do estilete (µm), do STT (µm) e
do parênquima (numero de células) (n=3 para cada planta mostrada). Os valores acima representam porcentagens dos
valores em relação à planta selvagem (100%). Os dados numéricos estão descritos na Tabela 1.
* Significantemente diferente do tipo selvagem pelo teste T de Student (p<0.05).
Camada de célulasa
2 a
Área da SSZ (μm )
b
Tamanho celular na SSZ
SCI1OE 3.1.1
Wild type
SCI1Ri 14.2
*8.3 ± 0.6
13.0 ± 1.0
*17.7 ± 2.1
*191,381.3 (77%) ± 10,834
248,272.0 (100%) ± 9,441
*367,355.3 (148%) ± 13,474
29.3 ± 4.0
36.0 ± 1.0
36.7 ± 1.5
559.9 ± 20.4
*657.0 ± 37.3
*58.5 ± 7.2
88.4 ± 7.1
*113.1 ± 5.5
14.0 ± 1.0
14.3 ± 0.6
*17.3 ± 0.6
c
Espessura do estilete (μm) *510.7 ± 16.8
Espessura do STT (μm)
d
Espessura do parênquima
a
e
Como mostrado na Figura 5Ca e 5D.
Representa o número de células contadas em uma área da SSZ de 20.000μm2, Figura 5Ca e 5D.
c
Medido a 400μm abaixo da interseção SSZ/STT como mostrado na Figura 5Ca/5Eb.
d
Medido a 400μm abaixo da interseção SSZ/STT como mostrado na Figura 5Ca/5Ec1).
e
Número de camadas celulares contadas no plano longitudinal logo atrás do feixe vascular, como mostrado na Figura 5Ec2.
Os valores representam a media de três determinações individuais com desvio padrão.
*Significantemente diferente da tipo selvagem pelo teste T de Student (p<0.05).
b
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 Tabela 1 – Análise dos tecidos do estigma/estilete em plantas transgênicas e selvagem.
‐ 57 ‐ DePaoli,H.C.
2010
SCI1 é expresso nos estágios iniciais do desenvolvimento do estigma/estilete Devido ao alto nível de expressão de SCI1 no estágio 1 do desenvolvimento floral e seus
efeitos sobre o tamanho final do estigma/estilete nas plantas transgênicas, decidiu-se investigar a
sua expressão nos estágios iniciais do desenvolvimento floral (estágios -4 a -1), em que o estigma
está se diferenciando (Koltunow et al., 1990; Wolters-Arts et al., 1996). Para melhor correlacionar
a função de SCI1 com os acontecimentos que ocorrem no início do desenvolvimento do
estigma/stilete, foi realizada microscopia eletrônica de varredura (MEV) de botões florais dos
estágios -4 a 1, bem como experimentos de qRT-PCR em estigmas/estiletes nestes mesmos
estágios do desenvolvimento. Além de medir o comprimento do botão floral (característica que
diferencia cada um dos estágios), foram considerados também marcadores morfológicos de
anteras já descritos anteriormente (Koltunow et al. 1990) para distinguir, de forma confiável, entre
cada estágio do desenvolvimento (Figura 7; Métodos).
O pistilo de tabaco é formado pela fusão de dois carpelos que dão origem ao estigma em
torno do estágio -4 do desenvolvimento floral (Figuras 8Af e 9Bd). O tecido estigmático começa a
diferenciação na região de fusão do carpelo em direção às bordas do mesmo (Figura 8Ak). No
estágio -3, a superfície do estigma torna-se evidente com um aspecto áspero, contrastando com a
superfície lisa da epiderme do carpelo (Figura 8Ag), e o estilete começa a elongar (Figura 8Ab).
Além disso, esta fase é caracterizada por uma grande ampliação no tamanho da parte superior do
pistilo e uma visível diferenciação de células papilares na epiderme do estigma (Figuras 8Ag e
5AL). No estágio -2, o estilete está visivelmente elongado (Figura 8Ac), as células papilares se
transformam em estruturas em forma de dedo (“finger-like structure”) (Figura 8Am) na borda
distal do carpelo e começam a se tornar muito mais evidentes na área central do estigma (Figura
8Ah). Em uma visão superior do estigma, vemos claramente as células papilares em
desenvolvimento a partir das bordas do carpelo e margeando a junção do carpelo em direção à
nas bordas centrais do pistilo durante este estágio (Figura 8Ah e 8Bc, azul). A epiderme na ponta
do carpelo está quase ereta, como conseqüência da pressão provocada pelo crescimento da SSZ. O
estágio -1 é retratado por um estilete bem elongado (Figura 8Ad) e uma ampliação clara nas
dimensões do estigma (Figura 8Ai), com a epiderme superior do carpelo e o parênquima
começando a dobrar para trás, originando a parte abaxial do estigma (Figura 8Bd). A
diferenciação das papilas ocorrem em toda a superfície do estigma. Por fim, no estágio 1, a SSZ é
totalmente diferenciada e a epiderme do carpelo superior e do parênquima se dobrou
completamente, formando uma estrutura em forma de guarda-chuva em uma visão lateral (Figura
8Ae e 9Af). A dobra da epiderme do carpelo superior e do parênquima ocorrem principalmente
devido à aceleração da proliferação celular na região central, onde os carpelos não fundidos.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 parte externa da área central (Figuras 8Bb e 8Bc, amarelo). Além disso, a epiderme ainda é visível
‐ 58 ‐ DePaoli,H.C.
2010
Figura 7 – Análise de marcadores morfológicos em antera nos botões florais de estágios muito jovens do
desenvolvimento floral. As anteras foram coletadas dos mesmos botões florais que foram utilizados para a microscopia
eletrônica de varredura (MEV) de estigmas/estiletes, e para a extração de RNA para o RT-PCR em tempo real. As
anteras foram separadas dos botões florais nos estágios desenvolvimentais relevantes, fixadas, embebidas em parafina,
cortadas transversalmente em seções de 8µm e coradas com azul de toluidina (0,05%). Essa análise foi realizada para
confirmar a correta avaliação do estágio desenvolvimental através da comparação com os marcadores morfológicos de
antera descritos por Koltunow et al. (1990). (A) Seção transversal de anteras no estágio -4, coradas com azul de
toluidina (0,05%) mostrando a célula mãe do micrósporo (seta branca), formada dentro do saco polínico, visivelmente
discernível das células do tapete. Nesse estágio, o tapete é claramente dividido em interno (seta vermelha) e externo
(seta amarela). (B) Seção transversal da antera no estágio -3, com uma deposição de calose evidente na parede da célula
mãe do megásporo (seta marrom). (C) Antera no estágio -2, mostrando o tapete mais largo e multinuclear (setas pretas).
(D) Antera no estágio -1, detalhando a díade (seta preta e verde) resultante do final da meiose I. (E) Antera no estágio 1,
apresentando a meiose completa, evidente pela formação das tétrades (seta branca).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 ‐ 59 ‐ DePaoli,H.C.
2010
(A) Microscopia eletrônica de varredura de pistilos em estágios bem jovens do desenvolvimento. Botões florais nos
estágios indicados (estágio -4 até estágio 1, respectivamente Aa a Ae) tiveram as sépalas e pétalas removidas para
permitir a visualização do pistilo. Vemos a formação e o desenvolvimento da SSZ (Af to Aj), junto com a diferenciação
da epiderme em células papilares (Ak to Ao). As barras de escala indicam 500µm (Aa to Ae), 200µm (Af to Aj) e
50µm (Ak to Ao). (B) Um esquema ilustrativo do desenvolvimento e da formação da SSZ, desde o estágio -4 até o
estágio 1 (Ba to Be, respectivamente) do desenvolvimento floral. amarelo, tecido estigmático; azul, epiderme do
carpelo. Barra de escala corresponde a 500µm para todas as imagens. (C) Nível de expressão de SCI1 determinado por
RT-PCR em tempo real em estigmas/estiletes selvagens nos estágios -4 a 1 do desenvolvimento floral. Expressão em
unidades arbitrárias (U.A.) como descrito na Figura 3M.
Conforme mostrado na Figura 8C, SCI1 é altamente expresso em fases muito precoces do
desenvolvimento do estigma/estilete, com o nível mais elevado de expressão observado no estágio
-3, quando a SSZ inicia a sua diferenciação. O transcrito de SCI1 diminui em fases posteriores (2, -1 e 1), durante o qual o tecido estigmático está proliferando e continuamente diferenciando.
Esse padrão de expressão é consistente com um papel no controle da proliferação/diferenciação
celular na parte superior do pistilo.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 Figura 8 – Desenvolvimento e diferenciação bem inicial da parte superior do pistilo em plantas selvagens (SR-1).
‐ 60 ‐ DePaoli,H.C.
2010
SCI1 controla a proliferação/diferenciação celular e influencia o momento do desenvolvimento do estigma/estilete A fim de estudar como o produto do gene SCI1 estava afetando o tamanho do estigma e o
elongamento do estilete, decidimos comparar os estágios iniciais do desenvolvimento do
estigma/estilete em plantas do tipo selvagem e transgênicas por microscopia eletrônica de
varredura (MEV). Foram analisadas as plantas transgênicas com os fenótipos mais extremos:
SCI1Ri14.2 e SCI1OE3.1.1. Pistilos no estágio -4 da planta SCI1Ri14.2 apresentou características
semelhantes às do pistilo de tipo selvagem em estágios de desenvolvimento floral -4/-3 (Figuras
9Ba, 9Bd e 9Be). Durante as primeiras etapas da formação do estigma/estilete, a planta
SCI1Ri14.2 claramente exibiu um crescimento mais rápido do estigma através de estágios -3 e -2
(Figuras 9Bb e 9Bc, respectivamente) em relação à planta selvagem, nos mesmos estágios
(Figuras 9Be e 9Bf). De fato, no estágio -3 a epiderme do carpelo na planta SCI1Ri14.2 já começa
a se dobrar para trás (Figura 9Bb), um evento observado somente no estágio -1 da planta do tipo
selvagem (Figuras 8Ad, 8Ai e 8Bd). A diferenciação do estigma foi também avançada na planta
SCI1Ri14.2, ocorrendo assim que o número de células ia se tornando maior (Figuras 9Ca-9Cc),
mostrando que a diferenciação celular no estigma é acoplada à proliferação nas plantas
silenciadas para o gene SCI1. Além disso, os pistilos da planta SCI1Ri14.2 atingiram a estrutura de
guarda-chuva no estágio -2 (Figuras 9Ab e 9Bc), um fenótipo observado apenas no estágio 1 da
planta tipo selvagem (Figuras 8Ae, 8Aj e 9Af). Da mesma forma, a elongação do estilete segue o
crescimento acelerado do estigma, tornando-o mais longo e conduzindo a superfície do estigma a
uma posição superior em relação às anteras já no estágio 1 (Figura 9Ac), enquanto que isso não é
observado em plantas do tipo selvagem (Figura 9Af). A aceleração do desenvolvimento
observado na planta SCI1Ri14.2, com um nível reduzido de mRNA de SCI1, sugere fortemente
que o produto do gene SCI1 seja um regulador chave no controle do desenvolvimento do
selvagem, confirmando que um maior número de células devem estar presentes no estigma de
plantas de RNAi para ocupar a maior área observada. Nossos resultados mostraram que o
estímulo para o elongamento do estilete, o que gera um estilete saliente da planta SCI1Ri14.2 na
antese, começa em estágios muito precoces do seu desenvolvimento, causada pelo silenciamento
de SCI1. Em conjunto, estes resultados são uma forte evidência de que o produto do gene SCI1
influencia o tamanho final do estigma e a elongação do estilete, através do controle da divisão
celular, desde os estágios iniciais da formação do órgão em plantas do tipo selvagem.
A parte superior dos pistilos da planta de superexpressão SCI1OE3.1.1 mostrou um
fenótipo semelhante ao de pistilos do tipo selvagem no estágio -4 (Figuras 9Bg e 9Bd,
respectivamente). Contudo, foi observado um atraso no desenvolvimento do estigma/estilete da
planta SCI1OE3.1.1 nos estágios -4 a -3, com um mínimo de diferenciação das células papilares
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 estigma/estilete. O tamanho de cada célula no estigma da planta de RNAi é similar aos do tipo
‐ 61 ‐ DePaoli,H.C.
2010
(Figuras 9Cg e 9Ch). As características típicas da superfície do estigma, observadas no pistilo das
plantas do tipo selvagem no estágio -3, são visíveis apenas no estágio -2 em pistilos da planta
SCI1OE3.1.1 (Figura 9Ce e 9Ci, respectivamente). É interessante observar que a diferenciação da
SSZ está estritamente ligada à proliferação celular. Pistilos nos estágios -1 e 1 da planta de
superexpressão são morfologicamente e desenvolvimentalmente semelhantes aos estágios -2 e -1
da planta selvagem, respectivamente. Além disso, o estilete em SCI1OE3.1.1 é visível apenas no
estágio -2 (Figura 9Ah), permanecendo mais curto que o tipo selvagem no estágio 1 (Figura 9Ai).
Consistentemente, os estigmas da planta SCI1OE3.1.1 têm um tamanho menor do que os da planta
selvagem quando são comparadas lado a lado nos estágios -3, -2, -1 e 1 do desenvolvimento
floral. Em resumo, pistilos da planta de superexpressão mostram um atraso no crescimento e
diferenciação de cerca de um estágio, quando comparados com pistilos da planta selvagem.
Como pode ser observado na Figura 9Ag, o desenvolvimento da antera também foi
afetado na planta SCI1OE3.1.1, devido à expressão ectópica de SCI1 sob controle do promotor
35S. Os outros órgãos vegetativos e florais de ambas as plantas transgênicas não demonstraram
nenhuma outra alteração macroscópica. Embora a expressão ectópica de SCI1 resultou em anteras
menores nos primeiros estágios de desenvolvimento da flor (Figuras 9Ag e 9Ah), no estágio 1
elas já tinham recuperado seu tamanho normal (Figura 9Ai). No estágio -3, plantas SCI1Ri14.2 e
selvagem exibem anteras de tamanho similar (Figura 9Aa e 9Ad, respectivamente). Tomados em
conjunto, podemos propor que a proteína SCI1 inibe a divisão celular nas fases iniciais do
desenvolvimento da flor de maneira tecido-específica e é suficiente para modular o momento do
desenvolvimento do estigma/estilete. Estas observações sugerem que SCI1 pode ser o motor de
um “relógio molecular” na via de sinalização do desenvolvimento do pistilo que determina o
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 tamanho final do órgão e o número de células.
‐ 62 ‐ DePaoli,H.C.
2010
Figura 9 – Microscopia eletrônica de
varredura (MEV) de plantas transgênicas (T1) e selvagens SR-1 (W.T.).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 ‐ 63 ‐ DePaoli,H.C.
2010
(A) Comparação da elongação do estilete no começo do desenvolvimento nas plantas SCI1Ri14.2 (Aa, Ab e Ac), e
selvagem (W.T.) (Ad, Ae e Af). Observe que no estágio 1 (Ac), o estigma de SCI1Ri14.2 já está posicionado acima das
anteras. A elongação do estilete, por outro lado, é atrasada na planta de superexpressão SCI1OE3.1.1 (Ag, Ah e Ai). No
estágio -3 do desenvolvimento floral é possível observar que a expressão ectópica de SCI1 em anteras atrasou o seu
desenvolvimento. Este órgão tem o mesmo tamanho e marcadores morfológicos (Koltunow et al., 1990) nas plantas de
RNAi SCI1Ri14.2 e do tipo selvagem. (B) Aumento prematuro da parte superior do pistilo da planta de RNAi
SCI1Ri14.2 (Ba, Bb e Bc), comparada com a planta selvagem (Bd, Be e Bf), em contraste ao atraso no desenvolvimento
observado para a planta de superexpressão SCI1OE3.1.1 (Bg, Bh e Bi), nos estágios -4 (Ba, Bd e Bg), -3 (Bb, Be e Bh)
e -2 (Bc, Bf e Bi) do desenvolvimento floral. (C) Crescimento e diferenciação das células papilares, na parte superior do
pistilo, que, nas plantas SCI1Ri14.2 e tipo selvagem, se iniciam a partir do estágio -4 (Ca e Cd, respectivamente) e
desenvolve nos estágios -3 e -2 (Cb;Cc e Ce;Cf, respectivamente) em diante, em contraste à planta SCI1OE3.1.1 que
inicia o desenvolvimento da papila no estágio -3 (Ch) e desenvolve a partir do estágio -2 (Ci) em diante. As escalas são
indicadas e representam 500μm (Aa a Ai), 100μm (Ba a Bi), e 50µm (Ca a Ci).
A proteína SCI1 tem as Ciclinas do tipo A como suas parceiras de interação Devido ao seu papel previsto como um regulador negativo do ciclo celular in planta e
pela presença de dois domínios putativos de interação com ciclina, resolveu-se testar se SCI1
poderia ser um regulador direto de complexos ciclina/CDK.
Para verificar se a interação entre SCI1 e as ciclinas do tipo A ocorria in planta, foram
executados ensaios de BiFC (Biomolecular Fluorecense Complementation) com as seqüências de
CYCA1;1 (NtCYCA1;1) e CYCA2;2 (NtCYCA2;2) de N. tabacum. Para tanto, NtSCI1 foi
fusionado à “cabeça” da GFP (hGFP; aminoácidos 1-149) e NtCYCA1;1, NtCYCA2;21-398 (ORF
completa) e NtCYCA2;296-398 (ORF sem o “destruction box”) C-terminais foram fusionadas à
“cauda” da GFP (tGFP; aminoácidos 150-238). Conforme mostrado na Figura 10, NtSCI1 exibiu
interação com NtCYCA1;1, restaurando a fluorescência da GFP in planta, que ficou localizada no
núcleo conforme o esperado. Até o momento, não fomos capazes de demonstrar a interação de
NtSCI1 com ambas isoformas de NtCYCA2;2. Nossos resultados mostram que SCI1 interage
Figure 10 – Interação de SCI1 com Ciclina A1;1.
As interações foram testadas utilizando a técnica de BiFC. Células da epiderme de folhas foram transformadas com o
gene NtSCI1 fusionado à cabeça da GFP (hGFP, amino ácidos 1 a 149) e com o gene NtCYCA1;1 fusionado á calda da
GFP (tGFP, amino ácidos 150 a 238). Painéis da esquerda e central – Fluorescência da GFP restaurada, que foi
localizada no núcleo (diferentes aumentos são mostrados); painel da direita – painel do meio mesclado com o campo
claro. Barras de escala são de 10µm.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 com ciclinas do tipo-A in planta.
‐ 64 ‐ DePaoli,H.C.
2010
SCI1 influencia os níveis de transcrição de alguns genes regulados por auxina Devido à similaridade fenotípica dos pistilos das nossas plantas transgênicas de RNAi e
os pistilos de Arabidopsis tratados com ácido N-1-naphthylphthalamic (NPA; Nemhauser et al.,
2000), decidimos investigar os níveis de transcrição de três genes regulados por auxina,
identificados no banco de EST de estigma/estilete (banco de dados TOBEST; Quiapim et al.
2009), em plantas transgênicas (SCI1Ri e SCI1OE) comparando com o tipo selvagem. Os níveis dos
transcritos dos genes de tabaco homólogos aos fatores de transcrição “Fator de Resposta a
Auxina” em Arabidopsis (ARF8), e aos “auxina/ácido indol-3-acético” (Aux/IAA) Aux/IAA13 e
Aux/IAA19 foram medidos por qRT-PCR em estigmas/estiletes de plantas do tipo selvagem,
quatro transgênicos independentes de RNAi e quatro transgênicos independentes de
superexpressão (Figura 11). Estes genes foram escolhidos com base em seu envolvimento com o
desenvolvimento da flor (ARF8, Wu et al., 2006), a capacidade de alterar múltiplas respostas a
auxina (Aux/IAA19, Tatematsu et al., 2004) e por prejudicar o desenvolvimento regulado por
auxina (Aux/IAA13, Weijers et al., 2005). Para este experimento, optou-se pelas plantas
transgênicas que apresentaram o menor e o maior nível de mRNA de SCI1 (Figura 5). Aqui,
optamos por substituir o controle interno utilizado na análise de qRT-PCR. O gene da β-actina
mostrou ser o melhor controle interno para a comparação da expressão gênica entre diferentes
órgãos de tabaco (Quiapim et al., 2009) e foi utilizado nos experimentos acima. Por outro lado, o
gene da ubiquitina é sugerido como controle mais adequado quando o mesmo órgão é comparado
durante alterações no controle do ciclo celular (Eloy et al., 2006; Masuda et al., 2008). Uma vez
que SCI1 está mudando o número de células no estigma/estilete, o gene da ubiquitina foi utilizado
como controle interno para este experimento. A Figura 11A mostra a re-análise dos níveis de
expressão de SCI1 nas plantas transgênicas com o gene da ubiquitina como controle interno.
Coerentemente com a idéia de que este gene de referência pode ser mais apropriado quando os
expressão de SCI1 em plantas transgênicas de RNAi correspondem perfeitamente com o grau de
alteração do seu fenótipo no estigma/estilete.
O acúmulo de mRNA de NtARF8 não foi afetado em plantas SCI1Ri (Figura 11B). No
entanto, observamos um aumento de seus níveis de transcrição de até 2,8 vezes maior em plantas
SCI1OE (p <0,05) (Figura 11B). NtAux/IAA13 e NtAux/IAA19 também foram induzidos em
plantas de superexpressão em até 4,2 vezes e 5,1 vezes (p ≤ 0,001), respectivamente (Figura 11C
e 11D). Além disso, a transcrição de NtAux/IAA13 (Figura 11C) e NtAux/IAA19 (Figura 11D)
foram alteradas em plantas de RNAi, com a expressão de NtAux/IAA19 sendo proporcionalmente
associada à diminuição da expressão de SCI1 nessas plantas (p <0,05). Nossos resultados sugerem
que SCI1 pode influenciar a regulação da transcrição de alguns genes regulados por auxina e de
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 genes envolvidos no controle do ciclo celular são alterados, os resultados obtidos para o nível de
‐ 65 ‐ DePaoli,H.C.
2010
alguma forma estar relacionado com a sinalização de auxina no desenvolvimento da parte
1
0,1
SCI1Ri 11.3
SCI1Ri 4.4.1
SCI1Ri 17.9
0
W.T.
SCI1OE 11.1
SCI1OE 8.3
SCI1OE 6.4
SCI1OE 3.1.1
0,5
Plantas
0,01
**
**
*
0,03
0,025
0,025
0,02
0,02
0,015
0,015
0,01
0,01
*
*
**
** **
*
**
0,07
0,06
0,05
0,04
0,005
0,01
0
0
0
Plantas
*
0,02
0,005
Plantas
*
0,03
**
** ** *
SCI1OE6.4
SCI1OE3.1.1
1
**
0,035
0,03
0,04
0,035
IAA19
SCI1OE8.3
10
**
0,08
W.T.
SCI1OE11.1
1,5
0,04
D
IAA13
0,045
SCI1Ri14.2
SCI1Ri17.9
SCI1Ri11.3
SCI1Ri4.4.1
100
0,045
C
ARF8
Nível de Expressão (U.A.)
2
0,05
SCI1Ri14.2
SCI1Ri17.9
SCI1Ri11.3
SCI1Ri4.4.1
W.T.
SCI1OE11.1
SCI1OE8.3
SCI1OE6.4
SCI1OE3.1.1
1000
B
Nível de Expressão (U.A.)
2,5
10000
SCI1Ri14.2
SCI1Ri17.9
SCI1Ri11.3
SCI1Ri4.4.1
W.T.
SCI1OE11.1
SCI1OE8.3
SCI1OE6.4
SCI1OE3.1.1
SCI1/Ubiquitina
Nível de Expressão (U.A.)
3
SCI1Ri 14.2
Nível de Expressão de SCI1Ri(U.A.)
A
Nível de Expressão de SCI1OE (U.A.)
superior do pistilo.
Plantas
Figura 11 – Nível dos transcritos para cada gene indicado determinado por RT-PCR em tempo real, em
estigmas/estiletes no estágio 4 das plantas selvagem (W.T.) (barras pretas), SCI1Ri (barras hachuradas) e SCI1OE (barras
em xadrez).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 (A) gene SCI1, (B) gene NtARF8, (C) gene NtAux/IAA13 e (D) gene NtAux/IAA19. Unidades arbitrárias (U. A.) em
A está como descrita na Figura 4, exceto que foi utilizado o gene da poliubiquitina como controle interno (ver Métodos
para detalhes). O eixo Y da esquerda mostra os valores para as plantas de RNAi (SCI1Ri) e o eixo Y da direita mostra os
valores para as plantas de superexpressão (SCI1OE). Unidades arbitrárias (U. A.) em B-D como descrito na Figura 3M,
exceto pelo uso do gene da poliubiquitina como controle interno. Os asteriscos indicam as diferenças estatisticamente
significantes entre cada planta transgênica comparada à planta selvagem pelo teste T de Student (*, p<0.05; **,
p≤0.001).
‐ 66 ‐ DePaoli,H.C.
2010
DISCUSSÃO SCI1 é um regulador tecido‐específico do ciclo celular que influencia o desenvolvimento do pistilo Nós identificamos um gene que codifica uma proteína rica em lisina especificamente
expressa nos tecidos especializados do estigma/estilete durante a sua fase inicial de
desenvolvimento. Com base na análise fenotípica das plantas transgênicas de superexpressão e
silenciamento, nossos resultados indicam fortemente que SCI1 tem uma atividade inibitória na
proliferação celular. A menor SSZ observada em plantas transgênicas de superexpressão é
formada por células com aproximadamente o mesmo tamanho que as células das plantas do tipo
selvagem. Além disso, a análise em MEV dos botões florais das plantas de superexpressão
demonstrou um atraso na proliferação/diferenciação. Assim, a presença de uma SSZ menor é
conseqüência de uma redução no número total de células acumulada durante o desenvolvimento
do estigma. Por outro lado, a maior SSZ obtida em plantas de RNAi está diretamente associada
com um início mais cedo e/ou mais rápido na proliferação/diferenciação celular e não com células
maiores. Portanto, nossos resultados sugerem que SCI1 participa no controle do momento da
proliferação/diferenciação celular do estigma/estilete e parece ser um integrador entre a regulação
do ciclo celular e o programa de desenvolvimento do estigma/estilete, garantindo que o processo
de proliferação e diferenciação, que são interconectados, ocorra no momento apropriado.
Em meristemas florais, células indiferenciadas se dividem assimetricamente, dando
origem a células-filhas que transportam a informação do destino da célula. Entretanto, a
diferenciação final é adiada até que algumas rodadas de divisão celular simétrica ocorra. O
momento exato em que essas células vão sair do ciclo celular e se diferenciar, ou seja, o momento
da diferenciação, é controlado por diversos fatores, como atividade de CDK e fatores de
propuseram um modelo para o controle da proliferação/diferenciação celular baseado na atividade
de CDK, que deve cair abaixo de um limiar para que as células sofram a saída do ciclo celular e
entrem na diferenciação. A interação de SCI1 com ciclinas do tipo-A sugere que este gene
controla a diferenciação celular e o tamanho do órgão através de uma modificação direta de
atividade de CDK. Entre os repressores de crescimento conhecidos que regulam a atividade de
diferentes “fatores de restrição do crescimento”, somente DA1 muda o tamanho do órgão sem
alterar o tamanho das células (Li et al. 2008), como SCI1 faz no estigma/estilete. Considerando
que DA1 controla o tamanho do órgão em diferentes tecidos, como sementes, embriões e
cotilédones, folhas, caules e flores, é plausível de se propor que SCI1 represente um efetor à
jusante de DA1 entre os “fatores de restrição do crescimento” tecido-específicos, o que
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 crescimento (revisto em De Veylder et al., 2007). De Veylder et al. (2007) recentemente
‐ 67 ‐ DePaoli,H.C.
2010
complementaria o modelo recentemente descritos por Krizek (2009), em vias de controle do
tamanho final do órgão.
Adicionalmente
ao
momento
da
diferenciação,
um
tópico
interessante
no
desenvolvimento de plantas é se a divisão celular segue um programa desenvolvimental (teoria
“organismal”) ou se a divisão celular instrui a organogênese e o desenvolvimento (teoria celular).
Devido a resultados ambiguos descritos na literatura que podem se enquadrar em ambas as
teorias, foi proposta a “teoria neo-celular”. (Tsukaya, 2002; Beemster et al. 2003). Nesta teoria,
um modelo integrativo entre crescimento e divisão celular é proposto. Nas plantas transgênicas de
SCI1, as alterações na taxa de divisão celular nos tecidos do estigma/estilete afetam tanto o
tamanho final do orgão quanto o momento da diferenciação, o que apoia a teoria celular.
Entretanto, enquanto que a grossura do STT é modificado em ambas as plantas de SCI1 (SCI1Ri e
SCI1OE) devido à taxa de divisão celular alterada, o comprimento foi modificado apenas nas
plantas de RNAi. Nas plantas de superexpressão o estilete se elonga até um comprimento
parecido com o da planta selvagem. Este fenótipo representa um programa desenvolvimental
inalterado, que não foi influenciado pela redução na taxa de divisão celular nos tecidos
especializados do estigma/estilete nas plantas SCI1OE, o que apoia a teoria “organismal”. A
deleção das células da SSZ em plantas STIG1 (plantas transgênicas STIG1pro:barnase; Goldman
et al., 1994) gerou estiletes mais curtos e não teve alteração nas células do STT. Em conjunto,
estas observações sugerem que o programa de elongamento de estilete é um resultado de
diferentes sinais, incluindo os provenientes do tecido do estigma (SSZ). Assim, uma possível
explicação para o fenótipo das plantas transgênicas é que o menor número de células em estigmas
SCI1OE produziu um “sinal de elongamento estigma-dependente” suficiente para elongar os
estiletes nestas plantas de forma semelhante ao que ocorre na planta selvagem. Por outro lado, o
estilete projetado para fora da corola nas plantas SCI1Ri seria uma consequência do aumento do
estigma-dependente” ou, alternativamente, começaram a produzí-lo antes. De acordo com a idéia
de um sinal que pode se mover entre as células, observou-se nos estiletes de plantas SCI1Ri um
aumento do número de células do parênquima, um tecido que não expressa SCI1. As
conseqüências da modulação de SCI1 no tamanho do estigma e no comprimento do estilete
sugerem que o desenvolvimento do pistilo é regido por uma variedade de sinais, em concordância
com a proposta de que os processos de crescimento são implementados a nível celular, mas
também envolvem a troca de informações entre os níveis organizacionais, e não simplesmente
seguindo as teorias celular ou “organismal”. Considerando esse ponto de vista, como é que os
resultados de SCI1 se encaixam na “teoria neo-celular”? Em nossas plantas SCI1Ri, um aumento
na divisão celular no estigma e uma diferenciação prematura são observados. Assim, propõe-se
que a divisão celular inibe a diferenciação celular até que o número de células no estigma atinja
um limiar acima do qual se acumulam os sinais de diferenciação e, em seguida, a taxa de divisão
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 número de células do estigma, que por sua vez produziram mais do “sinal de elongamento
‐ 68 ‐ DePaoli,H.C.
2010
celular diminui e será acompanhada da diferenciação celular (Figura 12). Coerentemente com esta
proposta, as plantas SCI1OE, em que observou-se um excesso de inibição da divisão celular no
estigma, começam o processo de diferenciação do estigma/estilete depois da planta do tipo
selvagem, mas as etapas subseqüentes da diferenciação acompanham a taxa de desenvolvimento
da planta selvagem (Figura 12). Nossos resultados mostram que o gene SCI1 codifica um
regulador tecido-específico importante no controle do ciclo celular que influencia o crescimento e
o desenvolvimento da parte superior do pistilo, sem alterar o tamanho das células.
A regulação dos níveis de expressão de SCI1 é suficiente para influenciar o tamanho do
estigma e o comprimento do estilete, que podem ter consequências importantes na reprodução das
plantas. A diminuição do nível de SCI1 resultou em estigmas posicionados acima das anteras e
em heterostilia na nossa população de plantas transgênicas de tabaco. Na natureza, a regulação
dos níveis de expressão das seqüências homólogas de SCI1 pode contribuir para o heterostilia
observada em algumas espécies com flores perfeitas (bi-sexuais), e ter consequências sobre as
Figura 12 - Modelo proposto para o mecanismo de ação de SCI1 no controle da proliferação da parte superior do
pistilo ao longo do desenvolvimento.
Depois que os dois carpelos se formam e fundem, as células posicionadas no topo do pistilo começam a se dividir na
linha de junção do carpelo. Quando um número apropriado de células é alcançado, elas iniciam a diferenciação em zona
secretória do estigma (SSZ) e tecido transmissor do estilete (STT) (a). Na planta selvagem, o nível de SCI1 regula a
proliferação celular e determina o estágio desenvolvimental correto em que a diferenciação ocorre. Nas plantas de
RNAi (SCI1Ri), o nível de SCI1 reduzido permite que as células comecem a se proliferar mais rápido e/ou mais cedo,
alcançando o limiar de diferenciação em um estágio mais jovem do desenvolvimento floral. Como estas células vão
parar de dividir no mesmo momento do desenvolvimento que a planta selvagem, provavelmente sobre o controle de um
fator ainda desconhecido, elas vão acumular um número maior de células. Nas plantas de superexpressão (SCI1OE), o
alto nível de SCI1 vai reduzir e/ou atrasar as divisões celulares, que consequentemente vão diferenciar mais tarde.
Denovo, o fator desconhecido, que é independente de SCI1, iria regular o momento em que essas células iriam parar a
proliferação, resultando em um número reduzido de células especializadas no estigma/estilete (SSZ and STT).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 taxas de auto-polinização e de polinização cruzada.
‐ 69 ‐ DePaoli,H.C.
2010
SCI1 é um regulador do ciclo celular distinto dos inibidores de CDK já descritos em plantas Diferentes aspectos do desenvolvimento da planta têm sido estudados para descobrir a
rede molecular por detrás do crescimento e desenvolvimento dos órgãos, culminando com a
descoberta de diferentes classes de genes, como aqueles envolvidos no controle da divisão celular.
Para alcançar esse controle, a célula reconhece diferentes sinais de uma variedade de fontes, que
são transduzidas pelos reguladores do ciclo celular para provocar a ativação ou inibição do ciclo
celular. As CKI/KRPs de plantas, que têm semelhança com os inibidores Kip/CIP de mamíferos,
interagem diretamente com CDK e/ou ciclina e constituem os principais reguladores negativos
destes complexos (ciclina/CDK) para controlar o ciclo celular em plantas (De Veylder et al.
2007). Apesar do seu efeito na inibição de proteínas do ciclo celular, SCI1 não tem nenhuma
semelhança com a seqüência das CKI/KRPs, mesmo quando as regiões mais conservadas do Cterminal são utilizados em programas de busca (BlastX) ou alinhados de forma independente. Por
outro lado, SCI1 tem uma semelhança limitada, mas reconhecível, com o produto do gene SIM
(Figura 13A), uma nova classe de inibidores de CDK, em uma pequena região que coincide com a
região de semelhança entre SIM e CKI/KRPs (Figura 13B). Foi demonstrado que esta região age
como um domínio de interação com ciclina (Peres et al., 2007). Assim, utilizando o sistema de
complementação de fluorescência (BiFC) encontrou-se que SCI1 interage com ciclina A
(CYCA1;1) in planta, em contraste com as CKIs/KRPs e SIMs que associam com ciclinas do tipo
D (Wang et al., 2000; De Veylder et al., 2001; Jasinski et al., 2002a; Zhou et al., 2002;
Churchman et al., 2006). O significado biológico da especificidade de ligação de SCI1 com essa
classe de ciclinas é desconhecida neste momento.
SCI1 é uma nova classe de inibidores de CDK? Os inibidores de CDK em planta
costumam ter um pequeno peso molecular, contêm motivos conservados, como os domínios de
ligação a ciclina (Tabela 3), e estão exclusivamente presentes no núcleo das células (Wang et al.
complexar com ciclinas, e foi mostrado que SCI1 tem uma ligação direta com a maquinaria do
ciclo celular pela sua capacidade de interagir com ciclinas do tipo A. Assim, com base nos
fenótipos das plantas transgênicas de SCI1, nos resultados de interação proteína-proteína e em
conformidade com o modelo atual para o controle da proliferação/diferenciação celular (De
Veylder et al. 2007), é plausível sugerir que a atividade de CDK está sendo alterado em plantas
SCI1OE e SCI1Ri. No entanto, ainda é um mistério se SCI1 seqüestra as ciclinas do tipo A ou
forma
um
complexo
com
os
heterodímeros
de
ciclina/CDK
para
inibir
a
proliferação/diferenciação tecido-específica.
Todas as CKI/KRPs de plantas superexpressas até agora causaram um crescimento global
reduzido, folhas serrilhadas e flores anormais (Wang et al., 2000; De Veylder et al., 2001; Jasinski
et al., 2002a; Zhou et al., 2002; Ren et al., 2008). Em contraste com CKI/KRPs e SIMs, plantas
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 2008), características presentes em SCI1. Na verdade, as CDKs se tornam ativas somente após
‐ 70 ‐ DePaoli,H.C.
2010
transgênicas de SCI1 apresentaram um fenótipo tecido-específico. Apenas pistilos e anteras foram
afetados como conseqüência da superexpressão conduzida pelo promotor 35S, o que significa que
SCI1 depende de um “background” celular específico para inibir o ciclo celular e, em seguida,
regular a organogênese. Como as ciclinas do tipo A são amplamente expressas na planta inteira
(Menges et al. 2005), esse controle é provavelmente dependente de modificações pós-traducionais
tecido-específicas (por exemplo, eventos de fosforilação e desfosforilação, Figura 1B) e/ou
ocorrer através de interação(ões) com um alvo(s) tecido-específico(s). Desvendar um regulador do
ciclo celular que atua de forma tecido-específica sugere a existência de proteínas com funções
semelhantes em outros tecidos vegetais. Assim, propomos que SCI1 representa uma nova classe
de inibidores de CDK, distinta das CKI/KRPs e SIMs, e a existência então de pelo menos três
classes de inibidores de CDK: a) CKI/KRPs (inibidores de CDK e proteínas relacionadas a KIP),
b) SIMs (proteínas SIAMESE e relacionadas à SIAMESE); ambas amplamente expressas nos
tecidos da planta e com uma alteração direta na atividade de CDK e; c) TICK (inibidores de CDK
tecido-específicos – “Tissue-specific Inhibitors of CDK”), que têm tecido-especificidade e
interagem com ciclinas, provavelmente regulando a atividade de CDK de forma indireta. SCI1
Figura 13 – Análise de seqüências entre SCI1 e as classes de reguladores do ciclo celular mais relacionados. (A)
Similaridade entre NtSCI1 e AtSIM revelada por BLASTp de duas sequencias. A região sublinhada em negrito
representa o domínio conservado entre AtSIM e a classe de CKI/KRPs conforme descrito por Churchman et al. (2006).
Essa região inclui um dos domínios de interação com ciclina reconhecidos na sequencia da proteína NtSCI1. (B)
Alinhamento entre NtSCI1, AtSIM e os membros da família de CKI/KRPs mais relacionados, na região conservada
entre os inibidores de CDK (descrita por Churchman et al., 2006). O alinhamento foi produzido pelo programa
Multalign (http://www.expasy.org/tools/#align). Note os dois resíduos de Fenilalanina (F) conservados e a região
bastante carregada antes dela presente em todas as proteínas alinhadas. (C) Árvore filogenética gerada pelo programa
ClustalW2, feita a partir das sequencias mostradas em B. As cores foram inseridas para facilitar a visualização das três
classes diferentes de reguladores do ciclo celular (CKI/KRPs, SIM e SCI1).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 seria assim o primeiro membro da classe TICK (Figura 13C).
‐ 71 ‐ DePaoli,H.C.
2010
Tabela 3 – Sequencias de amino ácidos de cada domínio de interação depositado no ELM (Eukaryotic Linear Motif)
identificados nas proteínas NtSCI1 e nos inibidores de CDK, descritos em N. tabacum e outros organismos relevantes.
Domínios ELM MOD LIG MAPK_1
ProDKin_1 DKKTPEE KDKKNLFF
KRSSPSS SPSSPRD LIG CYCLIN_1 KNLFF RDLF MOD_CDK
NtSCI1 LIG_APCC D/KEN box ‐ SIM ‐ RKLM ‐
GCTTPTS TATTPPP PPSTPSS ‐
SRM1 RKLLV KLLV NFPTPIK
RSCKRKLLV
SRM2 KDLRF NFPTPIK RCSTPTS VVDSPPP SLCTPTS
SRM3 RKLTP NtaKIS2 EREVLE SRRRLT PKENA KSLDL ‐
SLVTPTS
ELSSPKE KSKGIGEMTV
NsKIS1 LKEND ‐ RERTPEK
‐
NtKIS1* ‐ ‐ RQRTPEK
AtKRP1 ‐ KELI RKLF ‐
KLISPAL VGDTPAG NLVSPSG RERTPEK KLMSPPL VGDTPAG SLVSPSS RQRTPEK ‐
AtKRP2 SRIILS SRRRLR LRKSLH LRNKLL ‐ ‐
‐
GCKTPTS RKLTPVN IILSPCV RRDSPPV RKRPREISL KRPREISL KKTRLSKDLRF KTRLSKDLRF KRKLTPVNV RKLTPVNV MOD CK1_1 MOD CK2_1 SSPSSPR SPSSPRD SHSSEEK SDLSSET SSETARD STPSSLG SSTTTAI STTTAIT SFRSCSR SKTSKTK STPTSQE SAPSATA SLCTPTS
DKKTPEE KDKSKKE KSHSSEE SSGTKRK SLVTPTS SELSSPK SNGSSKS SSKSLDL SKESIKA SCCSRNG SAESKEA TSLSKYE STPTSQE LLVSTCE LLETLEE KPRSQEE QDSSKKE RLPTRPE IPDSDHE EDPTVNE GEMTVME CVRSHSE ELSSPKE PSETDLE RNGSTEE KLSSERE KMPSEKE RKCKGIEEVTI KCKGIEEVTI SLVSPSS SPSSSVN SAESKEA EEVTIME RNGSSEE KMPSEKE RSRRIVYV RSEKSSSVSV KKKELIHL KKELIHL KRRKMEEEVDL RRKMEEEVDL RKMEEEVDL KSKRRIEFVDL KRRIEFVDL RRIEFVDL SEKSSSV SSVSVVG SSCSGSN GRKTMEE ETVTAEE EMPTESE PTESEIE SCCSTSE CCSTSEE LRKSLHE LHETVKE SETSVVI SSVSCCS SCCSTSE SEESMNM Nta, Nicotiana tabacum; Ns, Nicotiana sylvestris; Nt, Nicotiana tomentosiformis; At, Arabidopsis thaliana.
*As isoformas NtKIS1a e NtKIS1b (variantes de splicing) têm os mesmos domínios identificados. O símbolo “-”
significa que o domínio em questão está ausente ou não obteve um valor acima do limiar estabelecido pelo ELM para
ser significativo.
LIG_APCC Dbox_1/KENbox_2, motivos contendo RXXL e KEN, respectivamente, que ligan nos componentes do
complex APC/C, Cdh1 e/ou Cdc20, direcionando a proteína para degradação dependente do ciclo celular através do
proteassoma 26S. Dbox .R..L. , KEN .KEN.;
LIG CYCLIN_1, Sítio de reconhecimento do substrato que interage com ciclina e aumenta a fosforilação por
complexos ciclina/CDK. A protein também deve ter o sítio MOD_CDK, que também é usado por inibidores de ciclinas
[RK].L.{0,1}[FYLIVMP];
MOD_CDK, Motivo do substrato para fosforilação por CDK, ...([ST])P.[KR]
MOD ProDKin_1, Sítio de fosforilação pela quinase direcionada por prolina (Proline-Directed Kinase, e.g. MAPK,
CDK-like kinases) em eucariotos superiores, ...([ST])P..;
LIG MAPK_1, Moléculas de interação com MAPK (e.g. MAPKKs, substratos, fosfatases) carregam o domínio de
ligação que ajudam a regular interações específicas na cascada de MAPK,o motivo clássico se aproxima a
(R/K)xxxx#x# onde # é um resíduo hidrofóbico, [KR]{0,2}[KR].{0,2}[KR].{2,4}[ILVM].[ILVF];
MOD CK1_1 e MOD CK2_1, Sítios de fosforilação por CK1 e CK2, S..([ST])... e ...([ST])..E , respectivamente.
Números de acesso: SIM (At5g04470); SMR1 (At3g10525); SMR2 (At1g08180); SMR3 (At5g02420); N. tabacum
NtaKIS2 (CAD56868); N. sylvestris NsKIS1 (CAC82732); N. tomentosiformis NtKIS1a (CAC82731); N.
tomentosiformis NtKIS1b (CAC82734); A. thaliana AtKRP1 (NP_179924); A. thaliana AtKRP2 (NP_190632).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 Genes ‐ 72 ‐ DePaoli,H.C.
2010
SCI1 está localizada no núcleo, como demonstrado aqui em células de folhas
transientemente transformadas com o gene quimérico 35SPROM:SCI1-GFP. Portanto, a sua
localização nuclear é independente da interação (s) com o proteínas específicas do estigma/estilete
ou fosforilações resultantes da transdução de sinais específicos do estigma/estilete. A proteína de
fusão SCI1-GFP confirmou a presença do NLS previsto e exibiu duas localizações distintas:
predominantemente no interior do nucléolo, e distribuída no nucléolo e em corpos nucleares,
sempre colocados na região da intercromatina. Apesar da sua participação bem conhecida na
biogênese dos RNAs ribossomais (rRNA) e dos pequenos RNAs nucleolares (snoRNA), o
nucléolo foi recentemente considerada uma estrutura nuclear multifuncional, exercendo funções
em diferentes tarefas, tais como no controle do ciclo celular (Zimber et al., 2004 ). Foi
demonstrado que alguns reguladores do ciclo celular são seqüestrados no nucléolo, o que aponta
para um papel desta organela na regulação da sua atividade (Visintin e Amon, 2000). Além disso,
demonstrou-se que as mudanças na arquitetura e organização funcional das organelas nucleares
acompanham a troca de um programa desenvolvimental e a ativação da atividade proliferativa das
células vegetais (Testillano et al., 2005; Echeverría et al. , 2007). A compartimentalização dos
corpos nucleares também foi descrita para a proteína de Arabidopsis ICK1/KRP1 (Zhou et al.,
2006) e para NtKIS1a, quando esta foi co-expressa com a proteína AtAJH1, o homólogo em
plantas de JAB1 de humanos (domínio de ativação da proteína c-Jun 1), uma proteína que
acredita-se ser a responsável por medear a degradação do inibidor de CDK p27KIP1 (Le Foll et al.,
2008). Dessa forma, propõe-se que a distribuição de SCI1-GFP no nucléolo e sua movimentação
para corpos nucleares pode estar ligada a diferentes destinos da célula ou diferentes fases do ciclo
celular durante o desenvolvimento do pistilo, o que é coerente com a participação de SCI1 no
controle da proliferação/diferenciação celular.
As células iniciais do carpelo são um grupo pequeno de células capazes e comprometidas
a se dividirem e diferenciarem para originar o pistil adulto. Tal processo desenvolvimental inclui
principalmente a organogênese mediada por hormônio associada à divisão e diferenciação
cellular. Frequentemente, a alteração do tamanho final do órgão é uma das conseqüências de uma
sinalização hormonal modificada (Nemhauser et al., 2000; Krizek, 2009). Considerando que o
fenótipo do stigma/estilete de plantas de tabaco SCI1Ri são análogos aos pistilos de Arabidopsis
tratados com NPA (Nemhauser et al., 2000), investigou-se a possibilidade de alguns genes de
resposta à auxina estarem sendo influenciados pelo nível de expressão de SCI1.
Como mencionado anteriormente, a família de fatores de transcrição Aux/IAA e ARF são
reguladores chaves com funções bem caracterizadas na resposta a auxina. Recentemente, Jurado
et al. (2008) relataram a estabilização do heterodímero E2Fc/DPB, um regulador negativo do
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 SCI1 conecta a sinalização de auxina e o controle da divisão/diferenciação celular? ‐ 73 ‐ DePaoli,H.C.
2010
ciclo celular, mostrando que divisão celular, diferenciação e a sinalização de auxina têm algumas
interseções moleculares. O mutante de Arabidopsis “auxin insensitive mutant” msg2-1, que
codifica a protein Aux/IAA19, é deficiente na indução dos genes Aux/IAA4, DFL1 (DWARF IN
LIGHT 1) e SAUR-AC1 após o tratamento com o hormônio auxina, quando comparado com a
alta indução observada em plantas selvagens (Tatematsu et al., 2004). Os genes Aux/IAA4, DFL1
and SAUR-AC1 pertencem, respectivamente, às três classes de genes de resposta rápida à auxina
(Aux/IAA, GH3 e SAUR - small auxin up RNA), que são diretamente influenciadas pelo
tratamento com auxina. Dessa forma, a expressão alterada desses três genes no mutante msg2-1
coloca o Aux/IAA19 nas etapas primárias da resposta a auxina. Alterar os níveis de SCI1 pra
baixo ou para acima do nível encontrado na planta selvagem pode alterar, respectivamente, para
baixo ou para cima, os níveis do NtAux/IAA19, indicando que SCI1 está, de alguma forma,
ligado a uma regulação positiva da expressão de NtAux/IAA19. O homólogo do NtAux/IAA19
em arroz, OsIAA3, é preferencialmente expresso em folhas e tem sua expressão diminuída em
plântulas tratadas com “2,4-dichlorophenoxyacetic acid” (2,4-D), um análogo da auxina (Jain et
al., 2006). Em concordância, nas plantas SCI1Ri, as quais mostraram um aumento no número de
células da SSZ e STT, o nível de transcritos de NtAux/IAA19 está diminuído, o que, por
conclusão, parece simular uma indução da divisão celular por auxina. Da mesma forma, o
Aux/IAA19 e o Aux/IAA13, que em Arabidopsis são reprimidos por auxina (Weijers et al., 2005),
tiveram seus níveis de transcritos aumentados nas plantas SCIOE, que mostraram um número
reduzido de células. Ao mesmo tempo, a superexpressão de SCI1 resultou no aumento de
transcritos do gene equivalente ao ARF8 de Arabidopsis. Este gene, ARF8, mostrou ser
responsável pela repressão da elongação celular através da redução dos níveis de IAA livre (Tian
et al., 2004). Com isso, a correlação entre a indução da expressão de NtAux/IAA19,
NtAux/IAA13 e NtARF8 com um número reduzido de células com tamanho normal é totalmente
crescimento do órgão. Por outro lado, a expressão inalterada de NtARF8 e NtAux/IAA13 nas
plantas SCI1Ri suggere que a ativação desses genes pode ser suprida por outros fatores na ausência
de SCI1. Em conjunto, nossos resultados sugerem que SCI1 está, de alguma forma,
interconectado com a via de sinalização por auxina durante seu controle na divisão celular e na
diferenciação do estigma/estilete (Figura 14).
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 coerente com o fenótipo observado nas plantas SCI1OE e aponta pra um contínuo equilíbrio do
‐ 74 ‐ DePaoli,H.C.
2010
Figura 14: Modelo molecular proposto para o mecanismo de ação de SCI1 no desenvolvimento da parte superior do
pistilo.
Ciclinas e CDKs devem interagir para iniciar/progredir a proliferação celular. SCI1 interage com ciclina A (CYCA)
para inibir a proliferação/diferenciação no início do desenvolvimento do pistilo (barra de ponta reta). Existe uma
contribuição da via de auxina no controle da proliferação/diferenciação (seta pontilhada), a qual acreditamos que SCI1
faça parte.
Resumidamente, nossa caracterização inicial dessa proteína rica em lisina, SCI1, revelou
uma nova classe de proteínas, capaz de inibir o ciclo celular de forma tecido específica. Nós
acreditamos que SCI1 é uma molécula sinalizadora que influencia o desenvolvimento da parte
interação com ciclinas do tipo A. A função de SCI1 é muito provavelmente modulada por
diferentes interações proteína-proteína e por eventos de fosforilação, possivelmente tendo
interseções moleculares com a via de sinalização por auxina.
Capítulo 1 – SCI1, Stigma/style Cell‐cycle Inhibitor 1 superior do pistilo, inibindo a proliferação/diferenciação celular, provavelmente através da
‐ 75 ‐ DePaoli,H.C
2010
- Capítulo 2 -
Caracterização do gene SCI1 na via de sinalização de auxina do pistilo
1
Departamento de Biologia, Faculdade de Filosofia, Ciências e Letras de Ribeirão Preto, Universidade de São Paulo,
Brazil 14040-901
2
PPG Genética, Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Brazil 14049-900
3
Department of Cell & Developmental Biology, University of California San Diego, La Jolla, CA/USA 92093
4
SALK Institute for Biological Studies, La Jolla, CA/USA 92037
Capítulo 2 – SCI1 e auxina Henrique C. DePaoli 1,2,3,4, Yunde Zhao3,4 e Maria Helena S. Goldman 1
‐ 76 ‐ DePaoli,H.C
2010
RESULTADOS Identificação e análise de SCI1 em Arabidopsis: AtSCI1 O gene NtSCI1, um gene que controla a divisão e diferenciação em estigmas/estiletes de
Nicotiana tabacum, foi descrito e caracterizado (Capítulo 1). O gene ortólogo em Arabidopsis,
AtSCI1 (At1g79200), foi identificado em uma busca por homologia em banco de dados como o
TAIR (“The Arabidopsis Information Resource”), utilizando a ferramenta BlastX (Altschul et al.,
1997), e é mostrado na Figura 1A. AtSCI1 compartilha 65% de identidade e 86% de similaridade
com NtSCI1. Esta busca demonstrou que, assim como em N. tabacum, AtSCI1 é um gene único
no genoma de Arabidopsis, não pertencendo a uma família multigênica,. No domínio II da
proteína encontram-se os putativos sinais de localização nuclear (Figura 1C, roxo), e o domínio
rico em lisina (Figura 1C, vermelho). AtSCI1 também possui vários sítios potenciais de
Figura 1 – Comparação da estrutura primária do produto gênico de NtSCI1 e AtSCI1.
(A) Alinhamento entre as seqüências de aminoácidos dos dois ortólogos NtSCI1 e AtSCI1. As identidades estão
destacadas por asteriscos e a similaridade (aminoácidos de propriedades fisico-quimica semelhantes) por pontos
(ClustalW). (B) Predição de fosforilação para as proteínas NtSCI1 e (C) AtSCI1 (NetPhos). Os possíveis sítios de
fosforilação estão indicados em azul (serinas), verde (treonina) e vermelho (tirosina), e a probabilidade de receberem o
grupo fosfato varia de 0 a 1 (100%). As setas pretas acima do gráfico representam os domínios de SCI1 (I, II e III, da
esquerda para a direita, Capítulo 1 - Figura 2). As caixas coloridas são: domínio rico em lisina (vermelho), sinal de
localização nuclear (roxo) e domínios de interação com ciclinas (azul claro). Há 2 domínios putativos de interação com
ciclinas em NtSCI1 e apenas 1 em AtSCI1, correspondendo exatamente à posição do segundo de NtSCI1 (RDLF em
NtSCI1 e RGLF em AtSCI1).
Capítulo 2 – SCI1 e auxina fosforilação, como em NtSCI1 (Figura 1B).
‐ 77 ‐ DePaoli,H.C
2010
Caracterização do locus At1g79200 (AtSCI1) de Arabidopsis thaliana O gene órtologo a NtSCI1 em Arabidopsis, At1g79200 (AtSCI1), é composto por uma
região promotora curta, seguida por 4 exons e 3 introns (Figura 2A), codificando uma proteína de
159 aminoácidos. Para comparar o fenótipo de perda de função nas plantas de Arabidopsis com as
plantas de RNAi em N. tabacum, foram analisadas todas as 5 inserções de T-DNA disponíveis
para esse locus (Atsci1-1 a Atsci1-5; Figura 2A).
A primeira linhagem (Atsci1-1; SALK_005710.29.99) mostrou ser uma inserção
localizada na região promotora, na posição -232. Essa inserção foi capaz de reduzir o nível de
transcritos de AtSCI1 pela metade em plantas homozigotas e heterozigotas (Figura 2C), quando
comparadas com a planta selvagem, e demonstrou ser haplóide insuficiente, ou seja, além da
homozigose (Atsci1-1-/-), também apresenta perda de função quando em heterozigose (Atsci1-1+/-)
(Figura 2Db). A parte superior do pistilo das plantas Atsci1-1 demonstrou um fenótipo similar ao
descrito para as plantas de RNAi de N. tabacum, com um aumento da estrutura estigmática e
elongação do estilete (Figuras 2Ba painel do meio; 2Db, 2Dc), quando comparado com a
variedade selvagem Col-0 (Figura 2Ba painel da esquerda, 2Da). Estas plantas são parcialmente
estéreis, com poucas flores se desenvolvendo em siliquas por autopolinização natural (Figura 2E).
Isto ocorre devido a diferenças no comprimento dos órgãos masculinos e femininos, uma vez que
os estigmas saem da corola antes da abertura e maturação da flor e dos estames (Figura 2E, painel
da direita). Nenhuma outra parte aérea da planta demonstrou ser fenotipicamente alterada quando
comparada macroscopicamente com a planta selvagem, variedade Col-0. Para confirmar que as
alterações fenotípicas observadas vieram da inserção do T-DNA no locus At1g79200, plantas de
Arabidopsis background Atsci1-1-/- foram transformadas com a construção 35S:SCI1FL (FL =
“full length”, exons 1,2,3 e 4), para tentar restaurar o fenótipo selvagem. Como pode ser visto na
Figura 2Dh, a superexpressão de AtSCI1 complementou totalmente o mutante Atsci1-1.
A segunda linhagem de T-DNA (Atsci1-2; SAIL_31_D10.v1) tem uma inserção
localizada no começo do terceiro íntron, na posição genômica +882 (Figura 2A). Essa inserção
atrapalha a excisão adequada do terceiro intron (Figura 2Bb, painel de cima), produzindo uma
nova proteína com os primeiros 3 exons originais (Figura 2Bb, painel de baixo) mais 41
aminoácidos provenientes da tradução do T-DNA. Surpreendentemente, os pistilos da planta
(Figura 2Ba, painel da direita), sugerindo que, de alguma forma, a proteína truncada deve ser
funcional. Para confirmar essa funcionalidade, transformou-se plantas de Arabidopsis background
Atsci1-1-/-, que têm um fenótipo de perda de função, com uma construção que superexpressava
apenas os três primeiros exons de AtSCI1, 35S:SCI1123 (123 = exons 1,2 e 3). Como pode ser
observado na Figura 2Di, a versão truncada da proteína complementou totalmente o mutante
Capítulo 2 – SCI1 e auxina Atsci1-2, assim como o restante da planta, mostraram ser muito semelhantes à planta selvagem
‐ 78 ‐ DePaoli,H.C
2010
Atsci1-1, restaurando o fenótipo selvagem, justificando a semelhança de Atsci1-2 com a planta
Figura 2 – Caracterização do gene AtSCI1.
(A) Estrutura genômica indicando as inserções de T-DNA disponíveis. (B) (a) Fenótipo dos mutantes Atsci1-1 e
Atsci1-2 comparados com a planta selvagem. Note o estigma/estilete mais desenvolvido em Atsci1-1, como observado
nas plantas de RNAi de N. tabacum (Capítulo 1). Barra de escala é de 1mm. (b) Gel de agarose 1% com o resultado de
um RT-PCR que demonstra que o mutante Atsci1-2 transcreve os exons 1,2 e 3 de AtSCI1, mas não o transcrito
completo com os 4 exons. (C) Análise de expressão por RT-PCR do gene AtSCI1 no mutante Atsci1-1+/- e Atsci1-1-/(hetero e homozigoto, respectivamente). O nível de expressão em unidades arbitrárias (U.A.) foi obtido dividindo-se o
valor de expressão do gene SCI1 nos mutantes (normalizado pelo gene da poliubiquitina), pelo valor de expressão do
gene SCI1 na planta selvagem (também normalizado pelo gene da poliubiquitina), multiplicado por 100 (%). Os
resultados são a média de 3 replicatas biológicas independentes. (D) Fenótipo do mutante Atsci1-1 (Db, Dc e Df), e de
superexpressão de SCI1 (35S:SCI1FL; Dd) e YFP-SCI1 (35S:YFP-SCI1FL; Dg). Note as alterações nas estruturas
estigmáticas, comparadas com a planta selvagem, variedade Col-0 (a e e). Observe que o mutante heterozigoto
(Atsci1+/-) possui um fenótipo parecido com o homozigoto Atsci1-1-/-, o que demonstra que esta inserção é haplóide
insuficiente. Dh e Di são os testes de complementação realizados no “background” do mutante Atsci1-1-/- (sci1),
superexpressando SCI1 “full length” (35S:SCI1FL) e truncada (exons 1,2 e 3; 35S:SCI1123), respectivamente. Note que
ambas as formas, SCI1FL e SCI1123 foram capazes de restaurar o fenótipo selvagem. Barras de escala representam
200µm. (E) Fenótipo das plantas adultas Col-0 (esquerda) e Atsci1-1 (direita), demonstrando a esterilidade parcial da
planta mutante para SCI1 (setas brancas indicando siliquas abortadas). A esterilidade parcial é causada pela projeção do
estigma para fora da corola já nos estágios iniciais do desenvolvimento floral (microscopia eletrônica de varredura,
paineis inferiores; note que na planta selvagem o estigma ainda está dentro da corola, aberta nesta imagem
artificialmente). Barras de escala são 1cm para as plantas e 200µm para a microscopia eletrônica.
Capítulo 2 – SCI1 e auxina selvagem.
‐ 79 ‐ DePaoli,H.C
2010
Conforme o esperado pelos resultados descritos acima, a inserção no exon 4, Atsci1-4
(SALK_142445.50.60.x), que tem somente os últimos 18 aminoácidos originais deletados,
mostrou um fenótipo semelhante à planta selvagem, de forma similar ao que foi observado em
Atsci1-2. As linhagens Atsci1-3 (SALK_137501.54.50.x) e Atsci1-5 (SALK_083219.53.50.x),
disponíveis no banco de dados como mutantes para este locus, nunca demonstraram uma inserção
e são totalmente selvagens (fenotípica e genotipicamente). Dessa forma, não há nenhuma planta
de perda de função (“knockout”) disponível para o locus At1g79200; apenas um mutante de perda
parcial de função (“knockdown”), Atsci1-1, que foi a selecionado para todos os nossos
experimentos, referido de agora em diante como sci1.
Para complementar as análises de perda de função no locus At1g79200, transformou-se
plantas selvagens de Arabidopsis com a construção 35S:SCI1FL. Em paralelo, transformou-se
plantas de Arabidopsis com a construção 35S:YFP-SCI1FL, que tem a proteína amarela
fluorescente, YFP (yellow fluorescent protein), fusionada à extremidade amino-terminal de SCI1.
Entre as 6 plantas de 35S:SCI1FL analisadas por microscopia eletrônica de varredura (MEV), 5
plantas demonstraram uma região estigmática reduzida, com bem poucas células papilares (Figura
2Dd). Este fenótipo é exatamente como o descrito para N. tabacum (NtSCI1OE) e corresponde ao
fenótipo contrário ao apresentado por sci1-1. É interessante notar que as plantas 35S:YFP-SCI1FL
(n= 5) mostraram um fenótipo semelhante ao das plantas 35S:SCI1FL, mas com uma menor
intensidade, sugerindo que a proteína YFP-SCI1 seja parcialmente funcional, ou seja,
parcialmente capaz de inibir a proliferação celular (Figura 2Dg).
Análise da região promotora e de expressão do gene AtSCI1 Para ter uma melhor compreensão da região promotora de AtSCI1, foram produzidas
plantas transgênicas expressando o gene quimérico GFP-GUS (fusão da proteína verde
fluorescente – GFP - com a proteína da beta-glucuronidase - GUS) sobre controle do promotor de
AtSCI1. Para determinar a região promotora de AtSCI1, analisou-se a seqüência genômica de
1924 pares de bases no programa PLACE (plant cis-acting elements), para identificar os possíveis
elementos regulatórios presentes em AtSCI1 e direcionar nossos experimentos. A região acima do
códon de início da tradução de AtSCI1 (At1g79200) e abaixo do códon de parada da tradução do
alguns elementos como TATA box (TTATTT, posições -31 e -147), o sítio de ligação ao fator de
transcrição LEAFY (CCAATGT, posição -138) e um sítio (GTTTAAGTGT, posição -97)
bastante similar ao sítio de ligação do fator de transcrição APETALA3 (GTTTAGGTGT). Em
contrapartida, alguns elementos, considerados importantes pela função conhecida do gene, foram
encontrados na região intragênica, ou seja, fora da putativa região promotora (entre as regiões
codificadoras dos genes At1g79190 e At1g79200). Estes elementos foram o sítio de ligação da
Capítulo 2 – SCI1 e auxina locus At1g79190, é composto de apenas 287 pares de bases. Nesta região foram encontrados
‐ 80 ‐ DePaoli,H.C
2010
proteína WUSCHEL (TTAATGG), encontrado na posição +540, localizada no íntron 2 de
AtSCI1. Este sítio também está presente no gene NtSCI1 (Strini, 2010), no qual localiza-se no
íntron 3. O sítio de ligação do fator de transcrição WUSCHEL está presente ainda no íntron 2 do
gene AGAMOUS, o qual é ativado por WUSCHEL (Lohmann et al., 2001). Foram também
encontrados sítios de ligação de membros da família de fatores de transcrição Myb (AACGG),
nas posições +545 e +1055, íntrons 2 e 3, respectivamente, os quais estão presentes no promotor
de ciclina B1;1, e um sítio (TGTTG) na posição +614, íntron 2, de ligação do fator de transcrição
RAV1-A, uma proteína que contém os domínios “APETALA2-like” e “B3-like”. Baseado nesses
resultados in silico, decidiu-se realizar as construções de fusão, com GFP-GUS, com duas
seqüências diferentes: SCI1PROM1, composto pelos 287 pares de base acima do códon de início da
tradução; e SCI1PROM2, composto por estes mesmos 287 pares de base, acrescidos de 1101 pares
de bases abaixo do códon de início da tradução (compreendendo os exons 1, 2, 3 e 10
aminoácidos do exon 4, e os introns localizados entre estes exons) (Figura 3Aa). Dessa forma, os
dois “promotores” foram fusionados à quimera GFP-GUS e utilizados para transformar plantas
selvagens de Arabidopsis. Dentre as 24 plantas analisadas para a construção SCI1PROM1:GFPGUS, somente 4 plantas mostraram alguma coloração azul, sendo que apenas duas tiveram
resultados semelhantes (Figura 3Ab), com alguma expressão no pistilo em estágios jovens.
Adicionalmente, essas plantas apresentaram, respectivamente, expressão em pétalas (Figura 3Ab,
painel esquerdo) e no contorno das anteras (Figura 3Ab, painel direito). Não foram obtidas plantas
com a construção SCI1PROM2:GFP-GUS até o momento.
Para estudar o padrão de expressão de AtSCI1 em plantas de Arabidopsis, utilizou-se os
dados disponíveis no “Bio-Array Resource” (BAR; Toufighi et al., 2005), um banco de dados que
reúne, entre outras informações, resultados de expressão por microarranjo. Esses resultados são
agrupados em diferentes categorias, dependendo dos padrões que foram estabelecidos com base
nas necessidades de cada estudo. Dessa forma, todos os tecidos/órgãos de Arabidopsis, que
tinham o resultado da análise de expressão do locus At1g79200 na categoria “desenvolvimento”,
foram selecionados. Os tecidos e os níveis de expressão das 25 amostras analisadas podem ser
observados na Figura 3B. Pode-se observar que o gene AtSCI1 foi transcrito nos tecidos de raiz,
meristema apical, transição de meristema apical para meristema floral, meristema floral, pétalas
não apresentam uma expressão significativa baseada no limiar proposto pelo programa BAR
(http://bar.utoronto.ca/). Dentre esses tecidos, o nível de expressão mais significativo foi em
carpelos no estágio 9, o que sugere que em Arabidopsis o gene SCI1 também é preferencialmente
expresso em pistilos. O banco de dados não possui dados de expressão para os tecidos florais em
estágios bem jovens (abaixo do 9), como foi estudado em N. tabacum (Capítulo 1, Figura 8C).
Uma vez que AtSCI1 apresentou expressão em tecidos meristemáticos, explorou-se a
possibilidade de sua expressão estar alterada em mutantes conhecidamente importantes para a
Capítulo 2 – SCI1 e auxina jovens, estames jovens, carpelo jovem e carpelo quase maduro (Figura 3Ba). Os outros tecidos
‐ 81 ‐ DePaoli,H.C
2010
formação e desenvolvimento do meristema floral. AtSCI1 não apresentou alteração transcricional
Figura 3 – Análise da expressão gênica de SCI1.
(Aa) Esquema indicando a região genômica correspondente ao “promotor 1” (SCI1PROM1) e “promotor 2” (SCI1PROM2)
(linhas/caixas pretas) dentro da estrutura genômica de AtSCI1 (linhas/caixas cinzas). As “caixas” representam exons e
as “linhas”, os introns. (Ab) Coloração de GUS para a construção SCI1PROM1:GFP-GUS. As setas amarelas ressaltam as
marcações que aparecem no pistilo. (B) Expressão gênica de AtSCI1 extraída do “Bio-Array Resource” (BAR) em (a)
diferentes tecidos e (b) meristema floral (selvagem e de alguns mutantes) em Arabidopsis. O sinal do arranjo representa
os valores associados à fluorescência de SCI1, já normalizados e filtrados, em três replicatas biológicas independentes
(para detalhes vide Material e Métodos). Cotyledon, cotilédones; hypocotyl, hipocótilo; seedling, plântula; root, raiz;
rosette leaves, folhas; stem, caule; SAM, meristema apical; SAMFM, transição para meristema floral; FM, meristema
floral; flower, flor; sepal, sépalas; petal, pétalas; stamen, estames; carpel, carpelo (estigma/estilete/ovário); st, estágios
(9, 12 e 15); lfy, LEAFY; clv, CLAVATA; ap, APETALA; ag, AGAMOUS; ufo, (UNUSUAL FLORAL ORGANS).
Capítulo 2 – SCI1 e auxina significativa em nenhum dos sete mutantes analisados (Figura 3Bb).
‐ 82 ‐ DePaoli,H.C
2010
SCI1 atua no desenvolvimento, dependente de auxina, do pistilo Após a caracterização inicial do gene AtSCI1 de Arabidopsis, foi iniciada a investigação
sobre a relação de SCI1 com a via de auxina, sugerida no final do Capítulo 1, pela semelhança das
plantas transgênicas de N. tabacum com plantas onde a via de auxina estava alterada e pela
alteração da expressão de três genes da família ARF/Aux/IAA nos pistilos de plantas transgênicas
SCIRi e SCIOE de N. tabacum.
Inicialmente, foi analisada a resposta de DR5:GUS à auxina no “background” selvagem
(Col-0) e no mutante sci1-1 (daqui para frente denominado apenas sci1). O promotor DR5:GUS
corresponde a uma região sintética de DNA que, na presença de auxina, ativa a transcrição do
gene repórter GUS (Sabatini et al., 1999). Conforme observado na Figura 4A, sci1 tem uma
resposta reduzida da expressão do repórter à auxina no meristema da raiz (4Ad), e também em
alguns pontos do pistilo jovem (4Af), mas não na formação de raiz secundária ou nas anteras
(4Ab e 4Af, respectivamente). Considerando o padrão de expressão de AtSCI1, esses dados
indicam que SCI1 é necessário para a correta ativação da sinalização por auxina nestes tecidos.
Adicionalmente, de posse do fenótipo do mutante sci1, observou-se que o duplo mutante
yuc2yuc6 (Cheng et al., 2006) tinha pistilos muito parecidos com os de sci1 (Figura 4Bc).
Adicionalmente, foram analisados dois mutantes deficientes na sinalização de auxina neste órgão,
pid336 e npy1. Os resultados demonstraram que, em flores maduras, os três mutantes
apresentaram uma estrutura estigmática aumentada (Figura 4Bc, 4Bd e 4Be) quando comparados
com a estrutura da planta selvagem (Col-0, Figura 4Ba). Entre esses três, o mutante deficiente em
síntese de auxina yuc2yuc6 foi o que apresentou a maior estrutura estigmática. Coerentemente, o
estilete também apareceu mais elongado nos mutantes yuc2yuc6, pid336 e npy1, conforme
observado em imagens de contraste de fase (DIC) (Figura 4Dc, 4Dd e 4De, respectivamente).
Para confirmar que o fenótipo apresentado em sci1 era ocasionado pela alteração do número de
células, conforme demonstrado em N. tabacum (Capítulo 1), além das análises de MEV foram
realizadas seções longitudinais no pistilo de sci1. Conforme pode ser observado na Figura 4Db e
4Eb, o número de células está bem aumentado na porção superior do pistilo de Arabidopsis.
Assim, foi observado que a redução da expressão de SCI1, em mutantes sci1, para um nível
próximo da metade do nível presente na planta selvagem, é suficiente para gerar o fenótipo de
celular. Semelhantemente, o aumento do tecido estigmático nos mutantes yuc2yuc6, pid336 e
npy1 é conseqüência de uma maior proliferação celular (Figura 4Ec, 4Ed e 4Ee, respectivamente).
De forma notável, o nível de expressão de SCI1 é reduzido nesses mutantes para um nível similar
ao observado no mutante sci1. Com isso, é possível sugerir que a redução do nível de expressão
de SCI1 nos mutantes yuc2yuc6, pid336 e npy é a causa do aumento da proliferação celular
observada na parte superior do pistilo desses mutantes. Em conjunto, esses resultados permitem o
Capítulo 2 – SCI1 e auxina aumento de tamanho do estigma/estilete, o qual é conseqüência de uma maior proliferação
‐ 83 ‐ DePaoli,H.C
2010
enquadramento do gene SCI1 na via de auxina que controla a proliferação celular no
Figura 4 – Estudo de SCI1 na via de auxina em Arabidopsis.
(A) Coloração de GUS em raiz e flores de plantas DR5:GUS no “background” selvagem (Col-0) e de sci1 (sci1+/-).
Note a redução da resposta à auxina na ponta da raiz e no pistilo (setas amarelas). (B) Microscopia eletrônica de
varredura de flores maduras das plantas selvagem (Ba), sci1 (Bb) e três mutantes relacionados à síntese de auxina,
yuc2yuc6 (Bc) e à sinalização de auxina, pid336 (Bd) e npy1 (Be). Note o aumento da superfície estigmática nos três
mutantes, conforme encontrado em sci1. Barras de escala correspondem a 500µm em todos os painéis. (C-D) Imagens
de constraste de fase de flores maduras (C) e pistilo apical (D) das plantas mostradas em B (respectivamente). Note as
Capítulo 2 – SCI1 e auxina estigma/estilete durante o seu desenvolvimento.
‐ 84 ‐ DePaoli,H.C
2010
estruturas estigmáticas e o estilete aumentados em todos os mutantes de auxina, de forma similar ao que ocorre em sci1.
Magnitude é de 100x (C) e 200x (D) para todos os painéis. (E) Cortes longitudinais do pistilo de Arabidopsis em
plantas selvagens (Ea) e nos mutantes sci1 (Eb), yuc2yuc6 (Ec), pid336 (Ed) e npy (Ee). Note que o número de células
no estigma/estilete é bem aumentado nos quatro mutantes, quando comparados à planta selvagem (os mutantes da
síntese de auxina, azul claro, e da sinalização de auxina, lilás, apresentam fenótipos adicionais à alteração do número de
células). Os painéis laterais de cada figura (a-e) mostram a análise de expressão por RT-PCR do gene AtSCI1 nos
mutantes sci1 (Eb), yuc2yuc6 (Ec), pid336 (Ed) e npy (Ee), conforme a descrição apresentada na Figura 2C. Note que
todos os quatro mutantes apresentam um nível relativo de expressão próximo à metade do nível de expressão
encontrado na planta selvagem. Magnitude é de 100x em todos os painéis.
Interação genética entre sci1 e, yuc2yuc6, pid336 e npy1 em Arabidopsis Para determinar se o gene SCI1 apresentava alguma interação genética com os genes
YUCCA (YUC2 e YUC6), PINOID (PID) e NPY (NYP1), o mutante sci1 foi cruzado com os
mutantes yuc2yuc6, pid336 e npy1. A geração de triplo e duplos mutantes foi analisada quanto ao
fenótipo do pistilo, para determinar se o efeito combinado da perda de função desses genes iria
reverter, manter ou ampliar o fenótipo observado em cada um deles separadamente.
De forma interessante, existe uma diferença entre os fenótipos das primeiras floradas e
das floradas mais tardias nas flores dos mutantes yuc2yuc6 (Figuras 4Bc e 5Ca/Cb) , pid336 (4Bd
e 5Da) e npy1 (Figuras 4Be e 5Ba). Adicionalmente ao fenótipo descrito por Cheng et al. (2006)
em flores iniciais de yuc2yuc6, observou-se que as flores tardias deste mutante apresentavam
dificuldade em estabelecer os quatro verticilos florais de forma adequada, exibindo tecidos
estigmáticos de forma ectópica (Figura 5Ca) e, em alguns casos, desenvolvendo apenas estruturas
características de pistilos (Figura 5Cb). De forma semelhante, as flores iniciais de pid336 (Figura
4Bd) apresentavam apenas uma superfície estigmática aumentada, enquanto que as flores mais
tardias passaram a apresentar flores não totalmente separadas umas das outras e com estruturas
em forma de pistilo em outros verticilos (Figura 5Da). Notóriamente as flores yuc2yuc6
apresentam anomalias mais severas que as flores pid336. As flores iniciais e tardias do mutante
npy1 se mostraram com aumento da severidade no desenvolvimetno do pistilo e de alguns
verticilos florais, mas sem expressão ectópica de órgãos/tecidos (Figura 5Ba).
A análise do duplo mutante sci1npy1 mostrou que a parte superior do pistilo se mantinha
anomalia na morfologia floral (Figura 5Bb). De forma esperada, as flores jovens do duplo
mutante também apresentaram um desenvolvimento pronunciado, projetando o estigma para fora
da corola (Figura 5Bc), como ocorre nos mutantes simples. Considerando que npy1 apresenta
outras anomalias não presentes em sci1, é possível sugerir que SCI1 não só está na mesma via que
NPY1 no controle do desenvolvimento do pistilo, mas também se encontra abaixo deste gene, sob
o controle funcional, direto ou indireto, do mesmo.
Capítulo 2 – SCI1 e auxina praticamente a mesma, quando comparada com sci1 e npy1, não apresentando outro tipo de
‐ 85 ‐ DePaoli,H.C
2010
A análise do triplo mutante sci1yuc2yuc6 mostrou efeitos ligeiramente adicionais aos
apresentados nos mutantes sci1 e yuc2yuc6, com um maior pronunciamento do pistilo nos
estágios iniciais do desenvolvimento (Figura 5Ce) e um desenvolvimento semelhante de tecido
estigmático ectópico, porém estendido para todos os verticilos florais (Figura 5Cc/Cd). Com base
nessas observações, os genes SCI1, YUC2 e YUC6 devem trabalhar juntos para proporcionar a
Figura 5 – Interações genéticas entre sci1 e três mutantes da via de auxina no pistilo.
(A) Microscopia eletrônica de varredura de flores de Col-0(Aa) e sci1(Ab) (envolvido no controle da proliferação
celular; linha laranja). (B) Flores tardias maduras de npy1 (Ba) e dos duplos mutantes sci1npy1 (Bb/Bc). (C) Flores
tardias maduras de yuc2yuc6 (Ca/Cb) e dos triplos mutantes sci1yuc2yuc6. (D) Flores tardias maduras de pid336 (Da) e
de sci1 no “background” pid336+/- (Db/Dc/Dd). Note os meristemas finais desenvolvidos apenas em pistilo (Dd). (E)
Plântulas de 9 dias de idade de sci1 (Ea), pid336 (Eb), sci1pid336+/- (Ec) e sci1pid336-/- (Ed). As plântulas dos
mutantes sci1 e pid336 se desenvolvem normalmente; as do mutante sci1 no “background” pid336+/- retardam o
desenvolvimento (Ec) e a maioria das plântulas dos duplo mutantes sci1pid336 morre sem desenvolver o primeiro par
de folhas (Ed). As setas indicam estruturas estigmáticas desenvolvidas fora do quarto verticilo (C e D). As barras de
escala são 200µm (A-D) e 5mm (E).
Capítulo 2 – SCI1 e auxina formação adequada da parte superior do pistilo.
‐ 86 ‐ DePaoli,H.C
2010
A análise do duplo mutante sci1pid336+/- mostrou que a estrutura floral foi bastante
afetada quando a flor tem a funcionalidade de SCI1 e PINOID diminuída (Figura 5Db-d). Em
alguns casos mais extremos, o meristema desenvolvia estruturas em forma de pistilo (Figura 5Dc)
ou até mesmo apenas pistilos no seu ápice (Figura 5Dd). De forma inesperada, os fenótipos
observados no desenvolvimento floral são de mutantes homozigotos para SCI1, mas heterozigotos
para PID. Quando sci1 e pid336 são colocados juntos em homozigose, a maioria das plantas
apresenta dificuldades de desenvolver os cotilédones, retardando/impedindo o desenvolvimento
das plântulas (Figura 5Ed). Em conjunto, os resultados mostram que SCI1 e PID trabalham em
conjunto para promover um desenvolvimento adequado do pistilo, durante o desenvolvimento
floral. Adicionalmente, a sinalização de auxina parece ser afetada a níveis capazes de prejudicar o
desenvolvimento do meristema apical de plântulas em Arabidopsis.
DISCUSSÃO SCI1 em diferentes espécies vegetais: N. tabacum e A. thaliana A caracterização inicial de SCI1 em N. tabacum demonstrou que este gene controla o
número de células na parte superior do pistilo, alterando a estrutura estigmática deste órgão. De
forma semelhante aos pistilos de plantas transgênicas SCI1Ri de N. tabacum, os pistilos do
mutante sci1 de Arabidopsis demonstraram uma estrutura estigmática aumentada, com aumento
do número de células, quando comparada com a planta selvagem (Col-0). Interessantemente,
somente as plantas SCI1Ri de N. tabacum, que tiveram a expressão de SCI1 diminuída para 28%
ou menos da expressão selvagem, apresentaram um fenótipo de aumento do estigma/estilete,
enquanto que nas plantas de Arabidopsis, uma diminuição de aproximadamente 50% já foi
suficiente para produzir o mesmo fenótipo. Adicionalmente, os resultados mostraram que, em
Arabidopsis, a perda de apenas um alelo funcional de SCI1 (sci1+/-) é o suficiente para reduzir o
nível deste transcrito para níveis próximos ao encontrado na planta com perda dos dois alelos
(sci1-/-). Esses resultados sugerem que Arabidopsis é mais sensível à perda de função de SCI1 que
N. tabacum.
de SCI1, e apresenta um fenótipo totalmente selvagem. Experimentos de complementação,
expressando os exons 1, 2 e 3 no mutante de perda de função sci1 (sci1-1), demonstraram que
estes exons são suficientes para permitir a inibição da proliferação celular, mantendo a
funcionalidade de SCI1 na formação da estrutura estigmática em Arabidopsis. Intrigantemente, o
exon 4 corresponde à porção mais conservada da proteína entre os ortólogos de diferentes
espécies (domínio III, Capítulo 1 – Figura 2). Le Foll et al. (2008) demonstraram que o inibidor
Capítulo 2 – SCI1 e auxina De forma bastante interessante, foi identificado o mutante sci1-2, que não possui o exon 4
‐ 87 ‐ DePaoli,H.C
2010
de CDK de N. tabacum NtKIS1a, interage com a proteína AJH1 para ser degradado, e que o
reconhecimento por AJH1 ocorre através do domínio DLS, presente nas extremidades amino ou
carboxi-terminal de diferentes inibidores de CDK, em plantas e animais. Este domínio, tem o
consenso
D/EX(20-22)LX(8-10)S/T
(onde
X
representa
qualquer
aminoácido
(n)
vezes).
Coerentemente, esse domínio pode ser encontrado em NtSCI1 (e seus ortólogos; Capítulo 1,
Figura 2) em dois sítios putativos na região carboxi-terminal, ambos dentro do domínio III
(D88L111S121; D110L132S146). Em conjunto, é plausível especular que o domínio III de SCI1 poderia
estar relacionado ao controle da degradação da proteína, mais do que diretamente relacionado
com a habilidade de SCI1 em inibir a proliferação celular.
Os estudos em N. tabacum por hibridização subtrativa e qRT-PCR mostraram que
NtSCI1 é específico de estigma/estilete (em experimentos com amostras de raiz, caule, folha,
sépala, pétala, estame, estigma/estilete e ovário; Capítulo 1, Figura 3). Ao mesmo tempo, na
espécie Arabidopsis thaliana, dados de expressão de microarranjo mostram que AtSCI1 é
preferencial de carpelo, apresentando expressão em raiz, meristemas e, pétalas e estames jovens
(Figura 3). Mesmo com expressão nestes órgãos, SCI1 demonstra alta tecido-especificidade, uma
vez que os sinais de DR5:GUS foram reduzidos no meristema apical da raiz em sci1, mas não na
formação de raízes secundárias. De forma semelhante, a ativação da expressão de AtSCI1 no
desenvolvimento do meristema floral está em concordância com os testes genéticos realizados, os
quais demonstraram alterações no desenvolvimento floral de sci1pid336 e sci1yuc2yuc6. Baseado
no fenótipo de sci1, onde os verticilos 2 e 3 (pétalas e estames) são totalmente idênticos à planta
selvagem, a presença deste transcrito nestes verticilos deve ser complementar a outros fatores
desenvolvimentais. Esses fatores teriam a capacidade de suprir a ausência de SCI1, assim como
responder ao seu excesso, como ocorrido em plantas de superexpressão de SCI1 em N. tabacum,
SCI1OE, as quais demonstraram uma alteração no desenvolvimento de anteras nos estágios bem
jovens (que foi restaurado ao longo do desenvolvimento). Dessa forma, SCI1 seria um elemento
chave e fundamental no desenvolvimento do quarto verticilo, mais especificamente na porção
apical, onde há um incomparável nível de expressão deste gene e ausência de redundância
funcional. Coerentemente, a fusão SCI1PROM1:GUS, demonstrou coloração nos verticilos 2, 3 e 4,
com uma maior coloração no verticilo 4 (pistilo), tanto em estágios jovens quanto em estágios
GUS em raiz ou meristemas, locais indicados por microarranjo como apresentando expressão de
AtSCI1. Este resultado sugere que alguns elementos regulatórios estejam faltando na região de
287pb acima do códon de início de tradução e suporta a identificação de outros elementos
identificados na região intragênica, que seriam, então, importantes para a correta expressão do
gene. Em conjunto, é possível sugerir que SCI1 possua uma região promotora complexa, para
controlar sua função de inibidor do ciclo celular tecido-específico, conservada em diferentes
espécies vegetais, conforme aqui demonstrado para N. tabacum e Arabidopsis.
Capítulo 2 – SCI1 e auxina quase maduros (ainda que de forma reduzida). Por outro lado, não se identificou coloração de
‐ 88 ‐ DePaoli,H.C
2010
SCI1 e a via de sinalização de auxina no pistilo O destino de um tecido em desenvolvimento pode ser determinado pela sensibilidade das
células em crescimento à concentração de auxina. A análise de duplos e triplos mutantes,
juntamente com a resposta de DR5:GUS no “background” sci1, demonstra que, de alguma forma,
SCI1 participa da via de sinalização de auxina. No modelo atual de desenvolvimento do pistilo,
foco do presente estudo, propõe-se a existência de um gradiente de concentração de auxina, apicobasal, que, quando alterado, modifica o desenvolvimento do órgão. De acordo com este modelo, é
necessária uma alta concentração de auxina na parte apical do pistilo para levar ao
desenvolvimento de uma estrutura estigmática (Nenhauser et al., 2000). Baseado nesse modelo,
com a super-proliferação celular e aumento da superfície estigmática em sci1, deveria-se esperar
que a quantidade de auxina estivesse aumentada. Porém, de forma intrigante, o mutante sci1 tem
uma estrutura estigmática aumentada, com maior número de células, igualmente ao duplo mutante
yuc2yuc6 (Figura 4), que é deficiente na síntese de auxina. Esses resultados geram uma aparente
contradição, mas que aponta para uma necessidade não só de se transportar direcionalmente o
hormônio, mas também de sintetizá-lo de forma específica, em alguns grupos de células, para
promover o correto desenvolvimento do órgão. Com isso, a aparente contradição que existe entre
essas respostas, permitiria um controle fino do momento em que cada grupo celular/tecido deve se
desenvolver. Dando suporte a essa proposta, existe o fato de que diferentes mutantes afetam
subgrupos específicos de tecidos, distintos morfológica e funcionalmente, que vão compor o
carpelo maduro, sugerindo que o desenvolvimento de cada tecido seja, em boa parte,
desvinculado um dos outros (Nenhauser et al., 2000; Staldal et al., 2008). Alternativamente, podese citar o controle do desenvolvimento lateral de raiz pela ação de NAC1 e SINAT5. SINAT5 é
antagônico a NAC1 e ambos são induzidos por auxina, porém com uma pequena diferença de
tempo, garantindo o fino ajuste da resposta ao hormônio (revisado em Benjamins e Scheres,
2008). Sob essa perspectiva, SCI1 e YUC2YUC6 estariam controlando o desenvolvimento de um
grupo específico de células, que seria diferente daquele controlado pelo transporte polar de auxina
no carpelo ou, alternativamente, teriam um momento de ativação/inibição diferente entre si na
cascata de sinalização dependente de auxina. Ao final, é possível sugerir que a síntese localizada,
transporte direcionado e a concentração final do hormônio nas células acontecem de forma
células sejam estimulados a promover o correto crescimento do órgão.
A proteína PINOID pertence à classe de quinases AGC em plantas. Foi demonstrado que
PINOID se autofosforila, e que esta autofosforilação é necessária para sua ativação na
fosforilação de seus alvos. Um destes alvos é a proteína PIN, que acaba por ser fosforilada por
PINOID e remanejada dentro da célula, direcionando o transporte de auxina (Friml et al., 2004).
Mutantes pid não desenvolvem inflorescências e têm o meristema apical em forma de pino
Capítulo 2 – SCI1 e auxina balanceada e tecido-específica, para que, ao longo do desenvolvimento, diferentes grupos de
‐ 89 ‐ DePaoli,H.C
2010
(Bennett et al., 1995; Christensen et al., 2000). Esses resultados mostram que a proteína PINOID
tem uma função chave no controle do transporte de auxina, e é essencial para o desenvolvimento
do meristema floral. A partir do momento que mutantes sic1 e pid336 demonstraram interação
sinergística entre si, conclui-se que eles possam trabalhar em conjunto no desenvolvimento do
pistilo. Em paralelo, o fenótipo observado em plântulas e em flores do mutante sci1pid336,
lembra os fenótipos de mutantes deficientes em auxina (npy1pid, yuc1yuc4pid, Cheng et al.,
2007). Considerando o envolvimento concreto de PINOID no transporte de auxina, e que o
mutante pid336 apresenta alterações brandas no desenvolvimento, é razoável propor que SCI1
afeta, de alguma forma, a sinalização de auxina no pistilo, uma vez que sci1pid336 têm efeitos
adicionais a pid336. Em conjunto, o transporte, mediado por PINOID, e a sinalização, dependente
de SCI1, estariam somando alterações para gerar os fenótipos observados no mutante sci1pid336.
SCI1 possui 6 sítios de fosforilação (serinas) alvos de AGC quinases (PKA e/ou PKC, homóloga
de PINOID em humanos), conservados entre os principais ortólogos (Capítulo 1, Figura 2) em
espécies vegetais, e que estão presentes na primeira parte da proteína (Domínios I e II,
codificados pelos exons 1, 2 e 3), responsáveis pela funcionalidade de SCI1 (conforme
demonstrado pelo mutante sci1-2). Com isso, uma outra alternativa seria a proteína PINOID, que
é uma quinase de relativo amplo espectro, fosforilar SCI1 e coordenar a cascata de
desenvolvimento dependente de auxina no pistilo, abaixo de SCI1.
Além da autofosforilação, a proteína PINOID depende de NPY para atuar em alvos da
cascata de sinalização por auxina e mutantes pidnpy1 falham na formação de órgãos aéreos
(Cheng et al., 2007). Quando foi analisado o mutante sci1npy1, observou-se que ele tem
praticamente a mesma estrutura estigmática que os mutantes sci1 e npy1 separadamente. Essa
observação mostra que a quebra funcional de SCI1 não modificou a quebra funcional já existente
no gene NPY, levando à conclusão de que SCI1 deva ser um dos alvos de NPY no controle do
desenvolvimento da parte superior do pistilo. Utilizando os três argumentos: a) NPY atua em
conjunto com o gene PINOID para formar os órgãos aéreos (fenótipo pidnpy1); b) SCI1 está sob
controle da cascata de NPY (fenótipo sci1npy1) e; c) PID atua na formação de diferentes órgãos
florais, mas com ajuda de SCI1 no desenvolvimento do pistilo (fenótipo sci1pid336); também é
possível que PID e NPY juntos coordenem a cascata de sinalização por auxina no pistilo através
na via YUC/PID/NPY, e o aumento observado no número de células na parte superior do pistilo
de cada um desses mutantes, é plausível propor que SCI1 esteja abaixo destes genes na via de
sinalização de auxina que estabelece o número de células do estigma/estilete durante o
desenvolvimento do pistilo.
Capítulo 2 – SCI1 e auxina de SCI1 (Figura 6). Considerando as análises dos fenótipos dos duplos/triplos mutantes de SCI1,
‐ 90 ‐ DePaoli,H.C
2010
Figura 6 – Modelo para explicar o envolvimento de SCI1 na via de auxina durante seu controle no desenvolvimento do
pistilo (verticilo 4). Auxina controla o desenvolvimento floral a partir de diferentes vias, incluindo a via YUC/PID/NPY
(Cheng et al., 2007). Neste trabalho foi mostrado que SCI1 está abaixo de NPY no controle do desenvolvimento do
pistilo. Em paralelo, os genes PID/NPY trabalham juntos para permitir a formação de órgãos florais (Cheng et al.,
2007). Uma vez que SCI1 demonstrou uma interação sinergística com PID, alternativamente, NPY estaria controlando
seus alvos, como SCI1, de uma maneira dependente de PID (seta pontilhada PID/NPY SCI1). Dessa forma, SCI1,
que controla a proliferação celular através da interação com ciclinas A, representa o motor molecular da sinalização de
auxina no controle da proliferação celular, para coordenar o desenvolvimento da parte superior do pistilo.
SCI1 no controle do ciclo celular dependente de auxina Diferentes mutantes relacionados às vias de auxina vêm demonstrando o efeito deste
hormônio no controle do tamanho dos órgãos. Mutantes dos genes TOUSLED, CRABS CLAW,
SPATULA e SEUSS, envolvidos em respostas mediadas por auxina, mostraram defeitos na
formação do carpelo com uma redução da estrutura estigmática (Roe et al., 1997; Alvarez e
Smith, 1999; Bowman e Smith, 1999; Franks et al., 2002). Entretanto, a primeria evidência
molecular de que a maquinaria da sinalização de auxina reprimia a divisão celular veio quando
número de células em óvulos, produzindo sementes maiores no mutante mnt/arf2. Recentemente,
Ishida et al. (2010) demonstraram que auxina influencia a saída da divisão celular e entrada no
endociclo (endoreplicação), um processo intimamente relacionado com a diferenciação celular.
Células de raiz em vários mutantes de síntese, sinalização e transporte de auxina desencadeiam
esse processo pela diminuição dos níveis de auxina, sentidos pelo complexo SCFTIR1 (Ishida et al.,
2010). Entretanto, os mecanismos moleculares pelo qual os limiares de auxina vão determinar a
proliferação, expansão ou endoreplicação celular, através da maquinaria do ciclo celular, ainda
Capítulo 2 – SCI1 e auxina Schruff et al. (2006) demonstraram que o gene MEGAINTEGUMENTA(MNT)/ARF2 controla o
‐ 91 ‐ DePaoli,H.C
2010
são desconhecidos. Neste estudo, foi demonstrado que SCI1 tem uma função chave no controle da
estrutura estigmática em pistilos, que ocorre, pelo menos em parte, pela correta homeostase do
hormônio auxina. Ao mesmo tempo, SCI1 é capaz de controlar a proliferação celular interagindo
diretamente com a maquinaria do ciclo celular, ciclinas A (Capítulo 1; Figura 10) e,
provavelmente, alterando a atividade de CDK, que é fundamental para o processo de divisão
celular. Em conjunto, pode-se dizer que SCI1 representa o motor molecular da via de sinalização
de auxina para controlar a progressão do ciclo celular, durante o controle que este hormônio
exerce no desenvolvimento da parte apical do pistilo. Certamente, identificar os elementos
regulatórios que coordenam a ativação/repressão deste transcrito/proteína vai contribuir para o
Capítulo 2 – SCI1 e auxina entendimento do controle de SCI1 na organogênese dependente de auxina.
‐ 92 ‐ DePaoli,H.C
2010
Considerações Finais Considerações Finais
‐ 94 ‐ DePaoli,H.C
2010
A caracterização de SCI1 revelou alguns mecanismos celulares que ocorrem durante o
desenvolvimento do órgão reprodutor feminino dependente de auxina, mais especificamente, no
seu controle da proliferação/diferenciação celular (Figura 1). Várias perguntas, então, surgem a
partir daqui: Quais os elementos que estão diretamente sendo alterados por NPY/PID para ativar
(ou reprimir) a ação de SCI1? Esta regulação ocorre ao nível transcricional ou pós-traducional? Se
transcricional, quais os elementos responsáveis pela ativação/repressão de SCI1 e quais
mecanismos os desencadeiam? Como SCI1 responde diretamente ao hormônio (auxina) a nível
transcricional e protéico? Quais domínios de SCI1 são fundamentais na sua função biológica?
Ocorre
mesmo
alguma
alteração
pós-traducional
(fosforilação/acetilação)
para
ativar/inibir/degradar/transportar a proteína? Qual elemento é o responsável pela “parada” da
proliferação celular? Existem proteínas tecido-específicas, presentes em outros órgãos, capazes de
atuar de forma semelhante a SCI1 no pistilo? Responder estas e outras perguntas vai ajudar na
compreensão dos mecanismos de regulação da organogênese reprodutiva, assim como também na
Figura 1 – Proposta final dos mecanismos moleculares de atuação/interação de SCI1 nas células do pistilo. SCI1 é uma
proteína nuclear, possivelmente regulada pós-transducionalmente. SCI1 interage com ciclinas do tipo A (CycA) para
inibir a divisão celular, a qual está em equilíbrio com a expansão e tem uma interação regulatória com a diferenciação
celular. Auxina influencia a proliferação/diferenciação através de diferentes vias, e SCI1 está envolvido com a
sinalização de auxina, conforme mostrado pelas análises com mutantes de Arabidopsis e por sua capacidade de alterar o
nível de transcrição da classe de fatores ARF/Aux/IAA, que respondem a modulações do hormônio dentro da célula.
Considerações Finais compreensão de questões mais gerais em biologia do desenvolvimento.
‐ 95 ‐ DePaoli,H.C
2010
Referências Referências
‐ 97 ‐ DePaoli,H.C
2010
Altschul, S.F., Madden, T.L., Schaffer, A.A., Zhang, J., Zhang, Z., Miller, W., Lipman, D.J.
(1997). Gapped BLAST and PSI-BLAST: A new generation of protein database search
programs. Nucl. Acids. Res. 25: 3389–3402.
Alvarez J, Smyth DR. (1999). CRABS CLAW and SPATULA, two Arabidopsis genes that
control carpel development in parallel with AGAMOUS. Development. 126:2377-2386.
Alvarez J, Smyth Dr. (2002). CRABS CLAW and SPATULA genes regulate growth and pattern
formation during gynoecium development in Arabidopsis thaliana. Int J Plant Sci 163: 17–41.
Alvarez Jp, Goldshmidt A, Efroni E, Bowman Jl, Eshed Y. (2009). The NGATHA distal organ
development genes are essential for style specification in Arabidopsis. Plant Cell 21: 1373–
1393.
Barrôco RM, Peres A, Droual AM, De Veylder L, Nguyen le SL, De Wolf J, Mironov V,
Peerbolte R, Beemster GT, Inzé D, Broekaert WF, Frankard V. (2006). The cyclindependent kinase inhibitor Orysa;KRP1 plays an important role in seed development of rice.
Plant Physiol. 142:1053-1064.
Beemster, G.T., Fiorani, F., Inzé, D. (2003). Cell cycle: the key to plant growth control? Trends
Plant Sci. 8: 154-158.
Bell J, Hicks G. (1976). Transmitting tissue in the pistil of tobacco: light and electron
microscopic observations. Planta 131: 187-200.
Benjamins, R., Scheres, B. (2008). Auxin: the looping star in plant development. Annu. Rev.
Plant Biol. 59: 443-465.
Bennett, S.R.M., Alvarez, J., Bossinger, G., Smyth, D.R.. (1995). Morphogenesis in pinoid
mutants of Arabidopsis thaliana. Plant J. 8: 505-520
Berleth T, Jürgens G. (1993). The role of the monopteros gene in organising the basal body
region of the Arabidopsis embryo. Development 118: 575–587
Bisbis B, Delmas F, Joubès J, Sicard A, Hernould M, Inzé D, Mouras A, Chevalier C.
(2006). Cyclin-dependent kinase (CDK) inhibitors regulate the CDK-cyclin complex
activities in endoreduplicating cells of developing tomato fruit. J Biol Chem. 281:7374-7383.
Boudolf V, Barrôco R, Engler Jde A, Verkest A, Beeckman T, Naudts M, Inzé D, De
Veylder L. (2004). B1-type cyclin-dependent kinases are essential for the formation of
stomatal complexes in Arabidopsis thaliana. Plant Cell. 16:945-955.
Bowman JL, Smyth DR. (1999). CRABS CLAW, a gene that regulates carpel and nectary
development in Arabidopsis, encodes a novel protein with zinc finger and helix-loop-helix
domains. Development. 126:2387-96.
Brasileiro, A.C.M., Carneiro, V.T.C. (1998). Manual de Transformação Genética de Plantas.
Editora Embrapa Produção de Informação. Brasília.
Cánepa ET, Scassa ME, Ceruti JM, Marazita MC, Carcagno AL, Sirkin PF, Ogara MF.
(2007). INK4 proteins, a family of mammalian CDK inhibitors with novel biological
functions. IUBMB Life. 59:419-426.
Carey, M., Smale, S.T. (2000). Transcriptional Regulation in Eukaryotes. Cold Spring Harbor
Laboratory Press, Cold Spring Harbor, NY.
Referências Brown, J.W., Shaw, P.J., Shaw, P., Marshall, D.F. (2005). Arabidopsis nucleolar protein
database (AtNoPDB). Nucleic Acids Res. 1: D633-636.
‐ 98 ‐ DePaoli,H.C
2010
Cheng Y, Dai X, Zhao Y. (2006). Auxin biosynthesis by the YUCCA flavin monooxygenases
controls the formation of floral organs and vascular tissues in Arabidopsis. Genes Dev. 20:
1790-1799.
Cheng Y, Qin G, Dai X, Zhao Y. (2008). NPY genes and AGC kinases define two key steps in
auxin-mediated organogenesis in Arabidopsis. Proc Natl Acad Sci U S A. 105:21017-22.
Cheng, Y., Qin, G., Dai, X., Zhao, Y. (2007) NPY1, a BTB-NPH3-like protein, plays a critical
role in auxin-regulated organogenesis in Arabidopsis. PNAS 104:18825-18829
Cheung AY, May B, Kawata EE, Gu Q, Wu HM. (1993) Characterization of cDNAs for stylar
transmitting tissue-specific proline-rich proteins in tobacco. Plant J. 3:151-60.
Cheung AY, Wang H, Wu HM. (1995) A floral transmitting tissue-specific glycoprotein attracts
pollen tubes and stimulates their growth. Cell. 82:383-93.
Christensen SK, Dagenais N, Chory J, Weigel D. (2000) Regulation of auxin response by the
protein kinase PINOID. Cell 100:469-478.
Churchman ML, Brown ML, Kato N, Kirik V, Hülskamp M, Inzé D, De Veylder L, Walker
JD, Zheng Z, Oppenheimer DG, Gwin T, Churchman J, Larkin JC. (2006) SIAMESE, a
plant-specific cell cycle regulator, controls endoreplication onset in Arabidopsis thaliana.
Plant Cell 18: 3145-3157.
Cockcroft, C.E., den Boer, B.G., Healy, J.M., Murray, J.A. (2000). Cyclin D control of growth
rate in plants. Nature 405: 575-579.
Coelho CM, Dante RA, Sabelli PA, Sun Y, Dilkes BP, Gordon-Kamm WJ, Larkins BA.
(2005). Cyclin-dependent kinase inhibitors in maize endosperm and their potential role in
endoreduplication. Plant Physiol. 138:2323-2336.
Cornish, E.C.; Anderson, M.A; and Clarke, A E. (1988). Molecular aspects of fertilization in
flowering plants. Annu. Rev. Cell Biol. 4: 209-228.
Crawford BCW, Yanofsky MF. (2008). The formation and function of the female reproductive
tract in flowering plants. Curr Biol 18: R972–R978.
Cresti, M., Went, J.L. van, Pacini, E., Willemse, M.T.M. (1976). Ultrastructure of transmitting
tissue of Lycopersicon peruvianum style: development and histochemistry. Planta 132: 305312.
Curtis,H.; Raven, P.H.; Evert, R. F. (1976). Biologia Vegetal. Editora Guanabara Dois S. A.
2ed.
Darwin, F. & Darwin, C. (1880) The Power of Movement in Plants (John Murray, London).
de Graaf, B.H.J., Derksen, J.W.M., Mariani, C. (2001). Pollen and pistil in the progamic
phase. Sex. Plant Reprod. 14: 41-55.
De Veylder L, Beeckman T, Inzé D. (2007). The ins and outs of the plant cell cycle. Nat Rev
Mol Cell Biol. 8: 655-665.
De Veylder L, Joubès J, Inzé D. (2003). Plant cell cycle transitions. Curr Opin Plant Biol. 6:536543.
Referências De Veylder L, Beeckman T, Beemster GT, de Almeida Engler J, Ormenese S, Maes S,
Naudts M, Van Der Schueren E, Jacqmard A, Engler G, Inzé D. (2002). Control of
proliferation, endoreduplication and differentiation by the Arabidopsis E2Fa-DPa
transcription factor. EMBO J. 21:1360-1368.
‐ 99 ‐ DePaoli,H.C
2010
De Veylder, L., Beeckman, T., Beemster, G.T., Krols, L., Terras, F., Landrieu, I., van der
Schueren, E., Maes, S., Naudts, M., Inzé, D. (2001). Functional analysis of cyclindependent kinase inhibitors of Arabidopsis. Plant Cell 13: 1653-1668.
Dean C, Van der Elzen P, Tamaki S, Dunsmuir P, Bedbrook J (1985) Differential expression
of the eight genes of the petunia ribulose bisphosphate carboxylase small subunit multi-gene
family. EMBO J 4: 3055-3061.
del Pozo, J.C., Diaz-Trivino, S., Cisneros, N., and Gutierrez, C. (2006). The balance between
cell division and endoreplication depends on E2FC-DPB, transcription factors regulated by
the ubiquitin-SCFSKP2A pathway. Plant Cell. 18: 2224–2235.
DePaoli HC, Bernardes, L., Brito, M.S., Quiapim, A.C., Goldman, G.H., Goldman, M.H.S.
(2010). Unraveling the specific pattern of gene expression in the wet stigma specie N.
tabacum by suppression subtractive hybridization underlies unique plant reproduction
mechanisms. Manuscrito em preparação.
DePaoli, H.C. (2006). Análise da expressão gênica no pistilo de Nicotiana tabacum: identificação
de genes específicos por hibridização subtrativa e caracterização temporal e celular. Medicina
(Ribeirão Preto) 39:474-475.
Dewitte W, Murray JA. (2003). The plant cell cycle. Annu Rev Plant Biol. 54:235-64.
Dewitte W, Riou-Khamlichi C, Scofield S, Healy JM, Jacqmard A, Kilby NJ, Murray JA.
(2003). Altered cell cycle distribution, hyperplasia, and inhibited differentiation in
Arabidopsis caused by the D-type cyclin CYCD3. Plant Cell. 15:79-92.
Disch, S., Anastasiou, E., Sharma, V.K., Laux, T., Fletcher, J.C., Lenhard, M. (2006). The E3
ubiquitin ligase BIG BROTHER controls Arabidopsis organ size in a dosage-dependent
manner. Curr. Biol. 16: 272–279.
Echeverría, O., Vázquez-Nin, G., Juárez-Chavero, S., Moreno Díaz de la Espina, S. (2007).
Firing of transcription and compartmentalization of splicing factors in tomato radicle nuclei
during germination. Biol. Cell. 99: 519-530.
Eloy NB, Coppens F, Beemster GT, Hemerly AS, Ferreira PC. (2006). The Arabidopsis
anaphase promoting complex (APC): regulation through subunit availability in plant tissues.
Cell Cycle 5: 1957-1965.
Endo M, Matsubara H, Kokubun T, Masuko H, Takahata Y, Tsuchiya T, Fukuda H,
Demura T, Watanabe M. (2002). The advantages of cDNA microarray as an effective tool
for identification of reproductive organ-specific genes in a model legume, Lotus japonicus.
FEBS Lett 514: 229-237.
Esau, K. (1977). Anatomy of Seed Plants. John Wiley, New York.
Fahn, A. (1982). Plant Anatomy.3ed. Oxford: Pergamon Press. p426-434.
Friml J, Yang X, Michniewicz M, Weijers D, Quint A, Tietz O, Benjamins R, Ouwerkerk
PB, Ljung K, Sandberg G, Hooykaas PJ, Palme K, Offringa R. (2004). A PINOIDdependent binary switch in apical-basal PIN polar targeting directs auxin efflux. Science.
306:862-865.
Gaude T, McCormick S. (1999) Signaling in pollen-pistil interactions. Semin Cell Dev Biol.
10:139-47.
Referências Franks RG, Wang C, Levin JZ, Liu Z. (2002). SEUSS, a member of a novel family of plant
regulatory proteins, represses floral homeotic gene expression with LEUNIG. Development
129: 253–263.
‐ 100 ‐ DePaoli,H.C
2010
Gietz D, St Jean A, Woods RA, Schiestl RH. (1992) Improved method for high efficiency
transformation of intact yeast cells. Nucleic Acids Res.20:1425.
Goda H, Shimada Y, Asami T, Fujioka S, Yoshida S. (2002). Microarray analysis of
brassinosteroid-regulated genes in Arabidopsis. Plant Physiol. 130:1319-1334.
Goldberg, R.B. (1988). Plants: Novel developmental processes. Science 240: 1460–1467.
Goldman, M.H., Goldberg, R.B., Mariani, C. (1994). Female sterile tobacco plants are
produced by stigma-specific cell ablation. EMBO J. 13: 2976-84.
Goldman, M.H.S., Pezzotti, M., Seurinck, J., Mariani, C. (1992). Developmental expression of
tobacco pistil-specific genes encoding novel extensin-like proteins. Plant Cell 4: 1041–1051.
Gremski K, Ditta G, Yanofsky MF. (2007). The HECATE genes regulate female reproductive
tract development in Arabidopsis thaliana. Development 134: 3593–3601.
Hase, Y., Tanaka, A., Baba, T., Watanabe, H. (2000). FRL1 is required for petal and sepal
development in Arabidopsis. Plant J. 24: 21-32.
Heisler M, Atkinson A, Bylstra Y, Walsh R, Smyth D. (2001). SPATULA, a gene that controls
development of carpel margin tissues in Arabidopsis encodes a bHLH protein. Development
128: 1089–1098
Hemerly, A., Engler, J. A., Bergounioux, C., Van Montagu, M., Engler, G., Inzé, D.,
Ferreira, P. (1995). Dominant negative mutants of the Cdc2 kinase uncouple cell division
from iterative plant development. EMBO J. 14: 3925-3936.
Heslop-Harrison, Y., SHIVANNA, K.R. (1977). The Receptive Surface of the Angiosperm
Stigma. Annals of Botany. 41:1233-1258
Himanen, K., Boucheron, E., Vanneste, S., de Almeida Engler, J., Inzé, D., Beeckman, T.
(2002). Auxin-mediated cell cycle activation during early lateral root initiation. Plant Cell 14:
2339-2351.
Huang, B.Q., Russel, S.D. (1992) Synergid degeneration in Nicotiana: a
fluorochromatic and chlorotetracycline study. Sex. Plant Reprod. 5:151-155.
quantitative,
Ingram, G.C., Goodrich, J., Wilkinson, M.D., Simon, R., Haughn, G.W., Coen, E.S. (1995).
Parallels between UNUSUAL FLORAL ORGANS and FIMBRIATA, genes controlling
flower development in Arabidopsis and Antirrhinum. Plant Cell 7: 1501-1510.
Inzé, D. (2005). Green light for the cell cycle. EMBO J. 24:657-662.
Inzé, D., Veylder, L.D. (2006). Cell Cycle Regulation in Plant Development. Annu. Rev. Genet.
40: 77-105.
Ishida T, Adachi S, Yoshimura M, Shimizu K, Umeda M, Sugimoto K. (2010) Auxin
modulates the transition from the mitotic cycle to the endocycle in Arabidopsis.
Development. 137:63-71.
Jain, M., Kaur, N., Garg, R., Thakur, J.K., Tyagi, A.K., Khurana, J.P. (2006). Structure and
expression analysis of early auxin-responsive Aux/IAA gene family in rice (Oryza sativa).
Funct. Integr. Genomics. 6: 47-59.
Jasinski, S., Leite, C.S., Domenichini, S., Stevens, R., Raynaud, C., Perennes, C.,
Bergounioux, C., Glab, N. (2003). NtKIS2, a novel tobacco cyclin-dependent kinase
Referências Jackson, D. (1991). In- situ hybridization in plants. Molecular Plant Pathology: A Practical
Approach. Eds. Bowles, D.J., S.J. Gurr and M. McPherson. Oxford University Press.
‐ 101 ‐ DePaoli,H.C
2010
inhibitor is differentially expressed during the cell cycle and plant development. Plant
Physiol. Biochem. 41: 667-676.
Jasinski, S., Perennes, C., Bergounioux, C., Glab, N. (2002b). Comparative molecular and
functional analyses of the tobacco cyclin-dependent kinase inhibitor NtKIS1a and its spliced
variant NtKIS1b. Plant Physiol. 130: 1871-1882.
Jasinski, S., Riou-Khamlichi, C., Roche, O., Perennes, C., Bergounioux, C., Glab, N. (2002a).
The CDK inhibitor NtKIS1a is involved in plant development, endoreduplication and restores
normal development of cyclin D3; 1-overexpressing plants. J. Cell. Sci. 115: 973-982.
Joubès, J., Chevalier, C., Dudits, D., Heberle-Bors, E., Inzé, D., Umeda, M., Renaudi, J.P.
(2000). CDK-related protein kinases in plants. Plant Mol. Biol. 43: 607-620.
Jurado, S., Díaz-Triviño, S., Abraham, Z., Manzano, C., Gutierrez, C., del Pozo, C. (2008).
SKP2A, an F-box protein that regulates cell division, is degraded via the ubiquitin pathway.
Plant J. 53: 828-841.
Kandasamy, M.K., Kristen, U. (1987). Developmental aspects of ultrastructure, histochemistry
and receptivity of the stigma of Nicotiana sylvestris. Ann. Bot. 60: 427-437.
Kandasamy MK, Dwyer KG, Paolillo DJ, Doney RC, Nasrallah JB, Nasrallah ME. (1990).
Brassica S-Proteins Accumulate in the Intercellular Matrix along the Path of Pollen Tubes in
Transgenic Tobacco Pistils. Plant Cell. 2:39-49.
Kao, T-h., Tsukamoto, T. (2004). The Molecular and Genetic Bases of S-RNase-Based SelfIncompatibility. The Plant Cell, 16, S72–S83.
Karimi, M., Inzé, D., Depicker, A. (2002) GATEWAY vectors for Agrobacterium mediated
plant transformation. Trends Plant Sci 7: 193–195.
Koltunow, A.M., Truettner, J., Cox, K.H., Wallroth, M., Goldberg, R.B. (1990). Different
Temporal and Spatial Gene Expression Patterns Occur during Anther Development. Plant
Cell 2: 1201-1224.
Kondorosi, E., Roudier, F., Gendreau, E. (2000). Plant cell-size control: growing by ploidy?
Curr. Opin. Plant Biol. 3: 488-492.
Krecek P, Skupa P, Libus J, Naramoto S, Tejos R, Friml J, Zazímalová E. (2009). The PINFORMED (PIN) protein family of auxin transporters.Genome Biol. 10:249.
Krizek BA, Fletcher JC. (2005). Molecular mechanisms of flower development: an armchair
guide. Nat Rev Genet. 6:688-698.
Krizek, B.A. (2009). Making bigger plants: key regulators of final organ size. Curr Opin Plant
Biol. 12: 17-22.
Kuusk S, Sohlberg JJ, Eklund DM, Sundberg E. (2006). Functionally redundant SHI family
genes regulate Arabidopsis gynoecium development in a dose-dependent manner. Plant J 47:
99–111.
Larson-Rabin, Z., Li, Z., Masson, P.H., Day, C.D. (2009). FZR2/CCS52A1 Expression Is a
Determinant of Endoreduplication and Cell Expansion In Arabidopsis. Plant Physiol. 149:
874-884.
Referências Labarca, C., Kroh, M., Loewus, F. (1970). The Composition of Stigmatic Exudate from Lilium
longiflorum. Plant Physiology 46:150-156.
‐ 102 ‐ DePaoli,H.C
2010
Le Foll, M., Blanchet S., Millan, L., Mathieu, C., Bergounioux, C., Glab, N. (2008). The plant
CDK inhibitor NtKIS1a interferes with dedifferentiation, is specifically down regulated
during development and interacts with a JAB1 homolog. Plant Science. 175:513-523.
Lee, H.-S., Huang, S. & Kao, T.-H. (1994). S proteins control rejection of incompatible pollen
in Petunia inflata. Nature 367, 560–563.
Leyser, O. (2002). Molecular genetics of auxin signaling. Annu Rev Plant Biol. 53:377-98.
Li M, Xu W, Yang W, Kong Z, Xue Y. (2007). Genome-wide gene expression profiling reveals
conserved and novel molecular functions of the stigma in rice (Oryza sativa L.). Plant Physiol
144: 1797-1812.
Li Y, Zheng L, Corke F, Smith C, Bevan MW. (2008). Control of final seed and organ size by
the DA1 gene family in Arabidopsis thaliana. Genes Dev. 22:1331-1336.
Linskens, H. F. (1983) Pollination processes: understanding fertilization and limits to
hybridization. In Strategies of Plant Reproduction, W. J. Meudt, ed. (Granada: Beltiville
Symposia in Agricultural Research) number 6, pp. 35-49.
Linskens, H. F. (1986). Recognition during the progamic phase. In: Biology of Reproduction and
Cell Motility in Plants and animals, M. Cresti, and R. Dallai, eds. (Siena: University of Siena,
Italy), pp. 21-31.
Liscum, E., Briggs, W.R. (1995). Mutations in the NPH1 locus of Arabidopsis disrupt the
perception of phototropic stimuli. PlantCell 7:473–485.
Lohmann JU, Hong RL, Hobe M, Busch MA, Parcy F, Simon R, Weigel D. (2001). A
molecular link between stem cell regulation and floral patterning in Arabidopsis. Cell
105:793-803.
Masuda HP, Cabral LM, De Veylder L, Tanurdzic M, de Almeida Engler J, Geelen D, Inzé
D, Martienssen RA, Ferreira PC, Hemerly AS. (2008). ABAP1 is a novel plant Armadillo
BTB protein involved in DNA replication and transcription. EMBO J. 27: 2746-2756.
Mauseth JD. (1988). Plant anatomy. Menlo Park: Benjamin Cummings.
McClure, B., Mou. B., Canevascini, S., Bernatzky, R. (1999). A small asparagine-rich protein
required for S-allele-specific pollen rejection in Nicotiana. Proc. Natl. Acad. Sci. 96: 1354813553.
Menges M, de Jager SM, Gruissem W, Murray JA.(2005). Global analysis of the core cell
cycle regulators of Arabidopsis identifies novel genes, reveals multiple and highly specific
profiles of expression and provides a coherent model for plant cell cycle control. Plant J.
41:546-566.
Mizukami, Y. (2001). A matter of size: developmental control of organ size in plants. Curr. Opin.
Plant Biol. 4: 533-539.
Murashige, T., and Skoog, F. (1962). A revised medium for rapid growth and bioassays with
tobacco tissue cultures. Physiol. Plant 15: 473-497.
Nagpal P, Ellis CM, Weber H, Ploense SE, Barkawi LS, Guilfoyle TJ, Hagen G, Alonso JM,
Cohen JD, Farmer EE, Ecker JR, Reed JW. (2005). Auxin response factors ARF6 and
ARF8 promote jasmonic acid production and flower maturation. Development. 132:41074118.
Referências Mizukami, Y., Fischer, R.L. (2000). Plant organ size control: AINTEGUMENTA regulates
growth and cell numbers during organogenesis. Proc. Natl. Acad. Sci. 97: 942-947.
‐ 103 ‐ DePaoli,H.C
2010
Napoli, C., Lemieux, C., Jorgensen, R. (1990). Introduction of a chimeric chalcone synthase
gene into Petunia results in reversible co-suppression of homologous genes in trans. Plant
Cell. 2: 279-289.
Nemhauser, J.L., Feldman, L.J., Zambryski, P.C. (2000). Auxin and ETTIN in Arabidopsis
gynoecium morphogenesis. Development. 127: 3877-3888.
Oakenfull EA, Riou-Khamlichi C, Murray JA. (2002). Plant D-type cyclins and the control of
G1 progression. Philos Trans R Soc Lond B Biol Sci. 357:749-60.
O'Brien, T.P., Feder, N., McCully, M.E. (1964). Polychromatic staining of plant cell walls by
toluidine blue. Protoplasma. 59: 368–373.
Okada K, Ueda J, Komaki MK, Bell CJ, Shimura Y. (1991). Requirement of the Auxin Polar
Transport System in Early Stages of Arabidopsis Floral Bud Formation. Plant Cell. 3:677684.
Paponov IA, Teale W, Lang D, Paponov M, Reski R, Rensing SA, Palme K. (2009). The
evolution of nuclear auxin signalling. BMC Evol Biol. 9:126.
Pei XH, Xiong Y. (2005). Biochemical and cellular mechanisms of mammalian CDK inhibitors:
a few unresolved issues. Oncogene. 24:2787-95.
Peres A, Churchman ML, Hariharan S, Himanen K, Verkest A, Vandepoele K, Magyar Z,
Hatzfeld Y, Van Der Schueren E, Beemster GT, Frankard V, Larkin JC, Inzé D, De
Veylder L. (2007) Novel plant-specific cyclin-dependent kinase inhibitors induced by biotic
and abiotic stresses. J Biol Chem. 282:25588-25596.
Pickett FB, Wilson AK, Estelle M. (1990). The aux1 Mutation of Arabidopsis Confers Both
Auxin and Ethylene Resistance. Plant Physiol. 94:1462-1466.
Quiapim AC, Brito MS, Bernardes LA, Dasilva I, Malavazi I, DePaoli HC, MolfettaMachado JB, Giuliatti S, Goldman GH, Goldman MH. (2009). Analysis of the Nicotiana
tabacum stigma/style transcriptome reveals gene expression differences between wet and dry
stigma species. Plant Physiol. 149: 1211-1230.
Ramirez-Parra, E., Desvoyes, B., Gutierrez, C. (2005). Balance between cell division and
differentiation during plant development. Int. J. Dev. Biol. 49: 467-477.
Raven, P.H.; Evert, R.F.; Eichhorn, S.E. (1996) Biologia Vegetal. Rio de Janeiro: Editora
Guanabara Koogan, 5ed.
Ren H, Santner A, del Pozo JC, Murray JA, Estelle M. (2008). Degradation of the cyclindependent kinase inhibitor KRP1 is regulated by two different ubiquitin E3 ligases. Plant J.
53:705-716
Riou-Khamlichi C, Huntley R, Jacqmard A, Murray JA. (1999). Cytokinin activation of
Arabidopsis cell division through a D-type cyclin. Science. 283:1541-4.
Roudier F, Fedorova E, Lebris M, Lecomte P, Györgyey J, Vaubert D, Horvath G, Abad P,
Kondorosi A, Kondorosi E. (2003). The Medicago species A2-type cyclin is auxin regulated
and involved in meristem formation but dispensable for endoreduplication-associated
developmental programs. Plant Physiol. 131:1091-103.
Ruegger M, Dewey E, Hobbie L, Brown D, Bernasconi P, Turner J, Muday G, Estelle M.
(1997). Reduced naphthylphthalamic acid binding in the tir3 mutant of Arabidopsis is
Referências Roe JL, Nemhauser JL, Zambryski PC. (1997). TOUSLED participates in apical tissue
formation during gynoecium development in Arabidopsis. Plant Cell. 9:335-353.
‐ 104 ‐ DePaoli,H.C
2010
associated with a reduction in polar auxin transport and diverse morphological defects. Plant
Cell. 9:745-57.
Sabatini S, Beis D, Wolkenfelt H, Murfett J, Guilfoyle T, Malamy J, Benfey P, Leyser O,
Bechtold N, Weisbeek P, Scheres B. (1999). An auxin-dependent distal organizer of pattern
and polarity in the Arabidopsis root. Cell. 99:463-72.
Sachs, J. (1887) Vorlesungen über Pflanzenphysiologie (Engelmann, Leipzig,) (in German).
Sambrook J., Russell, D.W. (2001) Molecular Cloning. A Laboratory Manual. Third Edition.
Cold Spring Harbor Laboratory Press, New York.
Sanchez AM, Bosch M, Bots M, Nieuwland J, Feron R, Mariani C. (2004). Pistil factors
controlling pollination. Plant Cell. 16 Suppl:S98-106.
Satina, S. (1944). Periclinal chimeras in Datuna in relation to development and structure of the
style and stigma of calyx and secretion. Am. J. Bot. 77: 1111-1124.
Sawers, R.J., Farmer, P.R., Moffett, P., Brutnell, T.P. (2006). In planta transient expression as
a system for genetic and biochemical analyses of chlorophyll biosynthesis. Plant Methods
2:15.
Schnittger A, Schöbinger U, Stierhof YD, Hülskamp M. (2002). Ectopic B-type cyclin
expression induces mitotic cycles in endoreduplicating Arabidopsis trichomes. Curr Biol.
12:415-20.
Schruff MC, Spielman M, Tiwari S, Adams S, Fenby N, Scott RJ. (2006). The AUXIN
RESPONSE FACTOR 2 gene of Arabidopsis links auxin signalling, cell division, and the size
of seeds and other organs. Development. 133:251-261.
Schruff MC, Spielman M, Tiwari S, Adams S, Fenby N, Scott RJ. (2006). The AUXIN
RESPONSE FACTOR 2 gene of Arabidopsis links auxin signalling, cell division, and the size
of seeds and other organs. Development 133:251–261.
Semighini, C.P., Marins, M., Goldman, M.H., Goldman, G.H. (2002). Quantitative analysis of
the relative transcript levels of ABC transporter Atr genes in Aspergillus nidulans by realtime reverse transcription-PCR assay. Appl. Environ. Microbiol. 68: 1351-1357.
Sessions RA, Zambryski PC. (1995). Arabidopsis gynoecium structure in the wild and in ettin
mutants. Development. 121:1519-32.
Sohlberg JJ,Myrenas M, Kuusk S, Lagercrantz U, Kowalczyk M, Sandberg G, Sundberg E.
(2006). STY1 regulates auxin homeostasis and affects apical-basal patterning of the
Arabidopsis gynoecium. Plant J 47: 112–123.
Soni R, Carmichael JP, Shah ZH, Murray JA. (1995). A family of cyclin D homologs from
plants differentially controlled by growth regulators and containing the conserved
retinoblastoma protein interaction motif. Plant Cell. 7:85-103.
Strini, E.J. (2010). SCI1, (Stigma/style cell cycle inhibitor): análise in silico da seqüência
genômica de Nicotiana tabacum e estudo de parceiros de interação proteína-proteína.
Dissertação de Mestrado, FMRP/USP, 115p.
Sundberg E, Østergaard L. (2009). Distinct and dynamic auxin activities during reproductive
development. Cold Spring Harbor Perspectives in Biology 1:a001628.
Referências Ståldal V, Sohlberg JJ, Eklund DM, Ljung K, Sundberg E. (2008). Auxin can act
independently of CRC, LUG, SEU, SPT and STY1 in style development but not apical-basal
patterning of the Arabidopsis gynoecium. New Phytol. 180:798-808.
‐ 105 ‐ DePaoli,H.C
2010
Swanson, R., Clark, T., Preuss, D. (2005). Expression profiling of Arabidopsis stigma tissue
identifies stigma-specific genes. Sex. Plant Reprod. 18: 163-171.
Takayama S, Isogai A. (2005). Self-incompatibility in plants. Annu Rev Plant Biol. 56:467-489.
Takeda, D.Y., Wohlschlegel, J.A., Dutta, A. (2001). A bipartite substrate recognition motif for
cyclin-dependent kinases. J. Biol. Chem. 276: 1993-1997.
Tatematsu, K., Kumagai, S., Muto, H., Sato, A., Watahiki, M.K., Harper, R.M., Liscum, E.,
Yamamoto, KT. (2004). MASSUGU2 encodes aux/iaa19, an auxin-regulated protein that
functions together with the transcriptional activator nph4/arf7 to regulate differential growth
responses of hypocotyl and formation of lateral roots in Arabidopsis thaliana. Plant Cell 16:
379–393.
Taylor, C.B. (1997). Comprehending cosuppression. Plant Cell 9: 1245-1249.
Teale WD, Paponov IA, Palme K. (2006) Auxin in action: signalling, transport and the control
of plant growth and development. Nat Rev Mol Cell Biol. 7:847-859.
Testillano, P.S., González-Melendi, P., Coronado, M.J., Seguí-Simarro, J.M., MorenoRisueño, M.A., Risueño, M.C. (2005). Differentiating plant cells switched to proliferation
remodel the functional organization of nuclear domains. Cytogenet. Genome Res. 109: 166174.
Tian, C.E., Muto, H., Higuchi, K., Matamura, T., Tatematsu, K., Koshiba, T., Yamamoto,
K.T. (2004). Disruption and overexpression of auxin response factor 8 gene of Arabidopsis
affect hypocotyl elongation and root growth habit, indicating its possible involvement in
auxin homeostasis in light condition. Plant J. 40: 333-343.
Toufighi K, Brady SM, Austin R, Ly E, Provart NJ. (2005). The Botany Array Resource: eNortherns, Expression Angling, and promoter analyses. Plant J. 43:153-163.
Trigueros M, Navarrete-Gomez M, Sato S, Christensen SK, Pelaz S, Weigel D, Yanofsky
MF, Ferrandiz C. (2009). The NGATHA genes direct style development in the Arabidopsis
gynoecium. Plant Cell 21: 1394–1409
Tsukaya, H. (2002). Interpretation of mutants in leaf morphology: genetic evidence for a
compensatory system in leaf morphogenesis that provides a new link between cell and
organismal theories. Int. Rev. Cytol. 217: 1-39.
Tung, C.W., Dwyer, K.G., Nasrallah, M.E., Nasrallah, J.B. (2005). Genome-wide
identification of genes expressed in Arabidopsis pistils specifically along the path of pollen
tube growth. Plant Physiol. 138: 977-89.
Vandenbussche F, Van Der Straeten D. (2004). Shaping the shoot: a circuitry that integrates
multiple signals. Trends Plant Sci. 9:499-506.
Verkest, A., Weinl, C., Inzé, D., De Veylder, L., Schnittger, A. (2005). Switching the cell
cycle. Kip-related proteins in plant cell cycle control. Plant Physiol. 139: 1099-1106.
Wang H, Qi Q, Schorr P, Cutler AJ, Crosby WL, Fowke LC. (1998). ICK1, a cyclindependent protein kinase inhibitor from Arabidopsis thaliana interacts with both Cdc2a and
CycD3, and its expression is induced by abscisic acid. 15:501-510.
Referências Visintin, R., Amon, A. (2000). The nucleolus: the magician’s hat for cell cycle tricks. Current
Opinion in Cell Biology 12: 372-377.
‐ 106 ‐ DePaoli,H.C
2010
Wang H., Wu H.M., Cheung A.Y. (1993). Development and pollination regulated accumulation
and glycosylation of a stylar transmitting tissue-specific proline-rich protein. Plant Cell 5:
1639-1650.
Wang H., Zhou Y., Bird D.A., Fowke L.C. (2008). Functions, regulation and cellular
localization of plant cyclin-dependent kinase inhibitors. J Microsc. 231:234-246.
Wang W, Chen X. (2004). HUA ENHANCER3 reveals a role for a cyclin-dependent protein
kinase in the specification of floral organ identity in Arabidopsis. Development. 131:31473156.
Wang, H., Zhou, Y., Gilmer, S., Whitwill, S., Fowke, L.C. (2000). Expression of the plant
cyclin-dependent kinase inhibitor ICK1 affects cell division, plant growth and morphology.
Plant J. 24: 613-623.
Watahiki MK, Yamamoto KT. (1997). The massugu1 mutation of Arabidopsis identified with
failure of auxin-induced growth curvature of hypocotyl confers auxin insensitivity to
hypocotyl and leaf. Plant Physiol. 115:419-26.
Weijers D, Van Hamburg JP, Van Rijn E, Hooykaas PJ, Offringa R. (2003). Diphtheria
toxin-mediated cell ablation reveals interregional communication during Arabidopsis seed
development. Plant Physiol. 133:1882-92.
Weijers, D., Benkova, E., Jäger, K.E., Schlereth, A., Hamann, T., Kientz, M., Wilmoth, J.C.,
Reed, J.W., Jürgens, G. (2005). Developmental specificity of auxin response by pairs of
ARF and Aux/IAA transcriptional regulators. EMBO J. 24: 1874-1885.
Wickens, M., Goodwin, E.B., Kimble, J., Strickland, S., Hentze, M.W. Carey, M., Smale,
S.T. (2000) Transcriptional regulation in eukaryotes: concepts, strategies and techniques.
Cap3, pg66.
Wolters-Arts, W., Derksen, J., Kooijman, J.W., Mariani, C. (1996). Stigma development in
Nicotiana tabacum. Cell death in transgenic plants as a marker to follow cell fate at high
resolution. Sex. Plant Reprod. 9: 243-254.
Wu, M.F., Tian, Q., Reed, J.W. (2006). Arabidopsis microRNA167 controls patterns of ARF6
and ARF8 expression, and regulates both female and male reproduction. Development. 133:
4211-4218.
Xue, Y., Ren, J., Gao, X., Jin, C., Wen, L., and Yao, X. (2008). GPS 2.0, a Tool to Predict
Kinase-specific Phosphorylation Sites in Hierarchy. Mol Cell Proteomics. 7:1598-1608.
Yamaguchi M, Kato H, Yoshida S, Yamamura S, Uchimiya H, Umeda M. (2003) Control of
in vitro organogenesis by cyclin-dependent kinase activities in plants. Proc Natl Acad Sci U S
A. 100:8019-8023.
Yu Y, Steinmetz A, Meyer D, Brown S, Shen WH. (2003). The tobacco A-type cyclin,
Nicta;CYCA3;2, at the nexus of cell division and differentiation. Plant Cell. 15:2763-77.
Zhang K, Letham DS, John PC. (1996). Cytokinin controls the cell cycle at mitosis by
stimulating the tyrosine dephosphorylation and activation of p34cdc2-like H1 histone kinase.
Planta. 200:2-12.
Zhou Y, Niu H, Brandizzi F, Fowke LC, Wang H. (2006) Molecular control of nuclear and
subnuclear targeting of the plant CDK inhibitor ICK1 and ICK1-mediated nuclear transport of
CDKA. Plant Mol Biol. 62:261-278.
Referências Zhang J, Nodzynski T, Pencík A, Rolcík J, Friml J. (2010). PIN phosphorylation is sufficient
to mediate PIN polarity and direct auxin transport. Proc Natl Acad Sci U S A. 107:918-22
‐ 107 ‐ DePaoli,H.C
2010
Zhou, Y., Fowke, L., and Wang, H. (2002). Plant CDK inhibitors: studies of interactions with
cell cycle regulators in the yeast two-hybrid system and functional comparisons in transgenic
Arabidopsis plants. Plant Cell Rep. 20: 967-975.
Zhou, Y., Wang, H., Gilmer, S., Whitwill, S., Fowke, L.C. (2003). Effects of co-expressing the
plant CDK inhibitor ICK1 and D-type cyclin genes on plant growth, cell size and ploidy in
Arabidopsis thaliana. Planta 216: 604-613.
Referências Zimber, A., Nguyen, Q.D., Gespach, C. (2004). Nuclear bodies and compartments: functional
roles and cellular signalling in health and disease. Cell Signal. 16: 1085-1104.
‐ 108 ‐ Anexo 1
ARTIGO SUBMETIDO PARA PUBLICAÇÃO NO PERIÓDICO
THE PLANT CELL
View Correspondence
1 de 3
http://submit.plantcell.org/submission/viewcorr?msid=PLANTCELL/2...
Home Author Area Reviewer Area Personal Info. PLANTCELL Home Sign Out
Submit New Manuscript Queue Summary Feedback Help FAQ
Decision Letter
[Return to Queue]
To: "Maria-Helena S. Goldman" &lt;[email protected]&gt;
From: [email protected]
Subject: PLANTCELL/2009/069286 - Manuscript Decision
Cc:
Dear Dr. Goldman:
We have received reviews of your manuscript entitled "A NOVEL TOBACCO
STIGMA/STYLE GENE, SCI1, ENCODING A LYSINE-RICH PROTEIN THAT CONTROLS CELL
DIVISION AND DIFFERENTIATION," which you may view by visiting
http://submit.plantcell.org. On the basis of the advice received and my own
evaluation, I regret that I must decline this version of your manuscript.
However, I would like to encourage you to consider resubmission of a
substantially revised manuscript that fully addresses the following concerns.
The reviewers and I found this to be a very interesting manuscript with some
novel findings. Unfortunately, all agree that this study is largely descriptive
and could go further to reveal the molecular mechanism of SCI1 action on cell
cycle control. In particular, it would be of interest to evaluate interactions
between SCI1 and CDKs/cyclins in vitro and in planta. The reviewers suggest
several suitable strategies to analyse these interactions. Other helpful and
important comments are found throughout the reviewer remarks, and these should
be addressed as well prior to resubmitting.
Because of the substantial nature of the revisions necessary to produce a
manuscript suitable for further consideration by The Plant Cell, any
17/05/2010 16:14
View Correspondence
2 de 3
http://submit.plantcell.org/submission/viewcorr?msid=PLANTCELL/2...
resubmission will be subject to our full review process in the same manner as a
new manuscript (unless specified otherwise above). Please note that
resubmissions that do not fully address concerns in the manuscript itself, but
instead provide arguments against making revisions in the "response to
reviewers" section, are often returned without review.
The Plant Cell considers resubmissions for up to 6 months after a manuscript has
been declined; limited extension of this timeframe is at the discretion of the
handling editor. A reminder notice will be sent to you after 5 months, at which
time you may choose to contact Annette Kessler ([email protected]) to request an
extension; if so, please provide a justification for your request. Any related
manuscript that is submitted to The Plant Cell after expiration of this deadline
would have to be provided as a new submission, though you will be able to
indicate its relationship to a previously declined manuscript in the Benchpress
system if you choose to do so. Also, because the field of plant biology is
continually evolving, The Plant Cell's expectations for novelty and significance
also evolve; thus, after a period of time, previously considered work may no
longer be deemed suitable for publication in the journal.
Please be certain to review our Instructions for Authors and ensure that any
resubmitted manuscript complies fully with journal style and policies. Note that
The Plant Cell strongly encourages authors to deposit novel materials such as
genetic stocks in an appropriate genetic stock center (e.g., ABRC/NASC for
Arabidopsis stocks) so that they will be available conveniently to the entire
scientific community.
When you log into the online submission system, this article will be listed in
the "Resubmit a Manuscript" queue. You may include an itemized list of changes
made in the revised manuscript in the "Response to Reviewers" section; please
note that reviewers do not have access to your cover letter and so will not see
17/05/2010 16:14
View Correspondence
3 de 3
http://submit.plantcell.org/submission/viewcorr?msid=PLANTCELL/2...
any comments you provide there. If you have any questions about these reviews or
the resubmission procedures, please feel free to contact me or the Manuscript
Manager, Annette Kessler, respectively.
Thank you for your interest in The Plant Cell.
Sincerely,
Chris Staiger
Coeditor, The Plant Cell
HENRIQUE DE PAOLI, University of Sao Paulo, Av Bandeirantes 3900, Cidade Universitaria
Ribeirão Preto, SP 14140-901 Brazil
Tel: (55-16) 36023852, Email: [email protected]
The Plant Cell
American Society of Plant Biologists, 15501 Monona Drive, Rockville, Maryland 20855-2768 USA
Tel: 301-251-0560; Fax: 301-279-2996; Email: [email protected]
Copyright © 2010 by American Society of Plant Biologists
17/05/2010 16:14
Ribeirão Preto – SP (Brazil), February 4th, 2010
Dear Dr. Chris Staiger,
Enclosed you will find the revised version of the manuscript entitled: “A novel
tobacco stigma/style gene, SCI1, encoding a lysine-rich protein that controls cell division
and differentiation”, to be considered for publication in The Plant Cell – Section
Development. This manuscript has not been submitted for publication elsewhere. The
manuscript cover page is complete and includes the materials distribution responsibility
footnote as well as an estimate of the length of this article, when published, from the page
calculator (20.4 pages). As the manuscript already contains a huge amount of data and is
calculated to be long, the DNA and protein sequence analyses were included as
Supplemental Figures and Tables.
As suggested in your letter and by the reviewers, we went further to reveal the
molecular mechanism of SCI1 action on cell cycle control performing a two-hybrid screen
against the core of cell cycle regulators, including cyclins and CDKs, in Arabidopsis. The
N. tabacum orthologous genes were cloned and the interaction with A-type cyclins was
validated by BiFC experiments (in planta as requested). We have addressed all the
reviewers’ comments by performing additional experiments, including new figures in the
new version of the manuscript and carefully answering all their comments. Furthermore,
the text of the manuscript has been modified, to incorporate the new results and all the
points/concerns raised by the reviewers. We truly hope you will find our manuscript
suitable for publication after all these modifications.
In case our manuscript is accepted for publication in The Plant Cell, we have a
special figure to be considered for the cover (enclosed as an Additional Supplemental file).
We would like to thank you very much for how you have handled our manuscript.
The reviewers' comments helped us to improve considerably the quality of the manuscript.
Please, feel free to contact me for any further questions.
Sincerely yours, -- Maria Helena Goldman
Address for correspondence: Dr. Maria Helena S. Goldman
Depto. Biologia - FFCLRP/USP
Av. Bandeirantes, 3900.
14040-901, Ribeirão Preto, SP – BRAZIL.
Phone: 55-16-3602-3702
FAX: 55-16-3602-4886
E-mail: [email protected]
A NOVEL TOBACCO STIGMA/STYLE GENE, SCI1, ENCODING A LYSINE-RICH
PROTEIN THAT CONTROLS CELL DIVISION AND DIFFERENTIATION
Running title: a stigma/style cell cycle inhibitor
Henrique C. DePaoli1,2, Michael S. Brito1,2, Andréa C. Quiapim1, Edward J. Strini1,2, Simone
P. Teixeira3, Lieven De Veylder4,5, Gustavo H. Goldman3,6, Marcelo C. Dornelas7 and Maria
Helena S. Goldman*1
1
Departamento de Biologia, Faculdade de Filosofia, Ciências e Letras de Ribeirão Preto,
Universidade de São Paulo, Brazil 14040-901
2
PPG Genética, Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Brazil;
14049-900
3
Departamento de Ciências Farmacêuticas, Faculdade de Ciências Farmacêuticas de Ribeirão
Preto, Universidade de São Paulo, Brazil; 14040-903
4
Department of Plant Systems Biology, Flanders Institute for Biotechnology (VIB), B-9052
Gent, Belgium
5
Department of Plant Biotechnology and Genetics, Ghent University, B-9052 Gent, Belgium
6
Laboratório Nacional de Ciência e Tecnologia do Bioetanol (CTBE), Brazil;
7
Departamento de Biologia Vegetal, Instituto de Biologia, Universidade de Campinas, Brazil;
13083-970
*
Corresponding author:
Prof. Dr. Maria Helena S. Goldman
Departamento de Biologia
Faculdade de Filosofia, Ciências e Letras de Ribeirão Preto
Universidade de São Paulo
Avenida Bandeirantes, 3900
Ribeirão Preto, SP - CEP 14040-901 BRAZIL
Phone: 55-16-3602-3702
FAX: 55-16-3602-4886
E-mail: [email protected]
The author responsible for distribution of materials integral to the findings presented in this
manuscript in accordance with the policy described in the Instructions for Authors
(www.plantcell.org) is: Maria Helena S. Goldman ([email protected]).
Estimated length of this article: 20.4 pages
1
ABSTRACT
The success of plant reproduction depends on the appropriate development of the
reproductive organs, which involves specific regulatory networks. We have characterized a
gene, SCI1 (stigma/style cell cycle inhibitor 1) that encodes a small lysine-rich protein with a
putative nuclear localization signal. Fluorescence microscopy with SCI1-GFP protein fusion
confirmed its nuclear localization, which is confined to the interchromatic region. Real time
RT-PCR and in situ hybridization experiments showed that SCI1 is stigma/style-specific and
developmentally regulated. SCI1 RNAi knockdown and overexpression plants resulted in
stigmas/styles with remarkably enlarged and reduced areas, respectively, which were
demonstrated to be the result of differences in cell numbers. These results, combined with an
observed interaction between SCI1 and A-type cyclins, suggest that SCI1 is a novel tissuespecific negative cell cycle regulator. The differences in cell division caused by SCI1
affected the differentiation timing of the papillar cells, showing that their differentiation is
coupled to stigma cell divisions, consistent with a role for SCI1 in triggering differentiation
through cell proliferation control. SCI1 also influenced the transcriptional regulation of
specific early auxin regulated genes. We conclude that SCI1 represents a novel
developmentally regulated tissue-specific gene that controls cell proliferation/differentiation,
probably as a component of a signaling pathway involved in upper pistil development.
INTRODUCTION
The efficiency of sexual plant reproduction depends on the appropriate development
of the reproductive organs, the adequate transfer of pollen grains to a receptive stigma
surface, as well as on compatible pollen-pistil interactions, which culminate in fertilization.
The pistil has fundamental roles in receiving and discriminating the pollen grains, which are
performed by its specialized tissues (mainly stigmatic secretory zone, SSZ, and stylar
transmitting tissue, STT). Recently, efforts to unravel genes that control the plethora of
events behind pistil development and function have emerged (Endo et al., 2002; Swanson et
al., 2005; Tung et al., 2005; Li et al., 2007; Quiapim et al., 2009). To exert the reproductive
associated functions the pistil cells should proliferate and differentiate properly, i.e., promote
pistil development. Pistil development in Solanaceae has been studied at ultrastructural,
microscopical and histochemical levels (Satina, 1944; Cresti et al., 1976; Kandasamy and
Kristen 1987; Goldberg, 1988; Koltunow et al., 1990; for a review see de Graaf et al., 2001).
2
In Nicotiana tabacum, wild-type and stigmaless pistils were used to study the development of
the SSZ (Goldman et al., 1994; Wolters-Arts et al. 1996).
Cell proliferation and differentiation events are important to establish organogenesis
and final organ size and disruption of these events impairs appropriate organ development.
Different factors were found to influence organ size in plants, such as endoreduplication and
polyploidy (Hase et al., 2000; Kondorosi et al., 2000; Larson-Rabin et al., 2009), changes in
organ identity genes (Ingram et al., 1995), altered phytohormone signaling (Nemhauser et al.,
2000; Krizek, 2009) and direct alterations in cell division control (Mizukami and Fischer,
2000; Mizukami, 2001). As in yeast and mammals, the plant cell cycle machinery is
governed by cyclin-dependent kinases (CDKs). The plant CDKs were classified into at least
five classes, A-E, with one of them (CDKB) being unique to plants (Joubès et al., 2000;
Wang et al., 2008). The CDKs should complex with cyclins to gain protein kinase activity
and phosphorylate targets that will regulate the progression through the cell cycle. Like
CDKs, cyclins are classified into different groups, including the D-type cyclins (CYCDs) that
play a role at the G1-to-S transition; A-type cyclins with an anticipated role during the S
phase and at the S-to-M control point; and B-type cyclins that control the G2-M switch and
intra-M phase (for a review see Inzé and De Veylder, 2006). Cell cycle regulation is also
influenced by CDK inhibitors, which bind to CDK/cyclin complexes specifically inhibiting
their activities (Verkest et al., 2005). Plant CDK inhibitors are represented by the CKI (CDK
Inhibitors)/KRP(Kip-related proteins) family and by the more recently described SIM family,
which includes the SIAMESE and SMR (SIAMESE-related) proteins (Churchman et al.,
2006; Peres et al., 2007). In Arabidopsis, the CKI/KRP family comprises 7 members (KRP17) (De Veylder et al., 2001; for a review see Verkest et al., 2005). In tobacco, two CKI/KRPs
were identified, NtKIS1a/b (splicing variants) and NtKIS2, and were shown to be expressed
in different plant tissues (Jasinski et al., 2002b; 2003). All plant CKI/KRPs share a restricted
C-terminal region of similarity among them (Η45aa) and the mammalian Kip/Cip inhibitors
(Η22aa) (Jasinski et al., 2002b; for reviews see Verkest et al., 2005; Wang et al., 2008). The
SIM/SMR family has 5 short conserved domains from which the domain 4 (Η10aa) shares a
high similarity with a region in the CKI/KRPs C-terminal (Churchman et al., 2006). The
overexpression of CKI/KRPs or SIM impairs the correct development of different organs and
leads to an overall reduced growth (Wang et al., 2000; De Veylder et al., 2001; Jasinski et al.,
2002a; Zhou et al., 2002; Churchman et al., 2006; Ren et al., 2008).
3
The level of CDK activity is essential for cell cycle entry. The main CDK/cyclin
complex involved in the initial step of cell cycle is the CDKA/CYCD (for a review see Inzé
and De Veylder, 2006). Its activation promotes cell cycle progression beyond the restriction
point of the G1 phase and activates late G1 and S phase genes. The plant CDKA is involved
in cell cycle regulation in an organogenesis-independent manner because CDKA1 dominantnegative mutants inhibit cell division without blocking morphogenesis in planta and CDKA
overexpression does not produce any macroscopical organ-changes (Hemerly et al., 1995).
Accordingly, the CYCD subfamily has well known inducers of cell division that increase
meristem growth rate when overexpressed without affecting organogenesis (Cockcroft et al.,
2000; Zhou et al., 2003). Since plant development is largely post-embryonic and cell
proliferation is intrinsically related to differentiation, there is possibly some cell cycle
regulators involved in both processes. Cell cycle regulators have been proposed as targets for
coupling cell proliferation and development in a cell type-, tissue-, and organ-specific manner
(Ramirez-Parra et al., 2005). For example, CDKB1;1 is required for stomatal divisions and
CYCA3;2 impairs differentiation of callus (Boudolf et al., 2004; Yamaguchi et al., 2003).
Loss of function studies with the G1/S phase E2F/DP class of transcriptional repressors,
E2Fc/DPb, suggested that it balances cell proliferation and development in pericycle cells
(del Pozo et al., 2006). Similarly, the CKI/KRP family has members which are proposed to
be integrators of plant development through cell division control (Himanen et al., 2002).
Recently, in parallel to the cell cycle machinery, some “growth restricting factors” have been
identified as coordinators of organ size (reviewed in Krizek, 2009). These molecular factors,
Auxin Response Factor 2 (ARF2) (Schruff et al., 2006), BIG BROTHER (BB) (Disch et al.,
2006), DA1 and DAR1 (DA1-related) (Li et al., 2008), are involved in organ size control by
cell number changes either altering or not the cell size. Though their involvement in organ
size, the molecular mechanisms by which they regulate cell proliferation/growth remains to
be established.
Thus, it is clear that cell proliferation and differentiation enclose a regulatory
interaction and represent a prerequisite for development. However, although cell cycle
regulators are known key effectors of cell proliferation, how these molecules are
activated/inhibited by the cell proliferation/differentiation control mechanisms is only poorly
understood. Identifying the molecular bridges that interconnect cell differentiation control
and development through cell cycle regulation is therefore of primary importance. Here, we
report the identification and characterization of a novel stigma/style-specific gene, which
inhibits cell proliferation and coordinately controls upper pistil differentiation timing and, for
4
this reason was denominated SCI1 (stigma/style cell cycle inhibitor 1). SCI1 protein is
distinct from the two other types of CDK inhibitors (KRP and SIM families) already
described in plants. Moreover, whereas KRPs and SIMs mainly target CYCD complexes,
SCI1 shows a binding preference for A-type cyclins. Therefore, it represents a new type of
protein capable of inhibiting the cell cycle, which acts in a tissue-specific manner. SCI1
exerts a control in tobacco upper pistil organogenesis and influences stigma/style final size,
most likely acting as a component of a signal transduction pathway, important for proper
pistil development.
RESULTS
Identification of the SCI1 gene and analysis of its protein subcellular localization
We have recently constructed two suppression subtractive hybridization (SSH) cDNA
libraries which allowed the identification of several genes specifically or preferentially
expressed in the pistil of Nicotiana tabacum (DePaoli et al., manuscript in preparation).
Among them, we chose a clone, TOBSH1-002E06, for further analysis and characterization.
As this clone was a partial cDNA (Supplemental Figure 1A), representing the 5’ end of a
transcript, we have performed a BLAST search in the TOBEST database (Quiapim et al.,
2009) and found two very similar overlapping cDNA clones (TOB048C05 and TOB068F10).
The clone TOB068F10 was completely sequenced and shown to contain, in addition to part
of the coding region, the 3’ UTR and polyA tail. The alignment of the TOBSH1-002E06 and
TOB068F10 clones can be seen in Supplemental Figure 1. It is interesting to note that it has
an unusually long 3’UTR (395 bp), which may be important for its transcript regulation, e.g.
to influence its stability. The corresponding gene was designated SCI1 (stigma cell-cycle
inhibitor 1) based on the results shown below.
The homology search, using the complete coding sequence via TBLASTX
(Altschul et al., 1997), showed that the encoded protein is similar to proteins of unknown
function present in different plant organisms. The sequences with the closest similarity to
SCI1 gene product are from potato (AC233353), tomato (AC212309), barley (AK252156),
corn (EU958997), rice (NM_001052562) and grapevine (AM424297). The putative
homologous sequence in the Arabidopsis genome is At1g79200 (NM_106571), which shares
65% identity and 86% similarity with SCI1. The alignment of these proteins (Supplemental
Figure 2) allows the recognition of 3 domains: I) a short conserved sequence at the N-
5
terminus; II) an intermediary lysine-rich domain with low sequence conservation; and III) a
highly conserved C-terminal domain.
To uncover more information about this putative protein sequence and guide our
future experiments, we performed a detailed in silico analysis using the complete sequence of
154 amino acids. Through the combination of several sequence analysis software packages
(see Methods), we identified an N-terminal lysine-rich domain (LRD) (MotifScan, evalue =
6.5e-07), with lysine residues predicted to be Nε-acetylated, a nuclear localization signal NLS (PSORT, 0.992 affirmative; PredictNLS), two cyclin interaction domains (ELM
prediction) and a total of 15 predicted phosphorylation sites (NetPhos, ≥96%) (Supplemental
Figure 1B). The identified cyclin interaction domains, consensus [RK].L.{0,1}[FYLIVMP],
are present in a wide range of cyclin/CDK interacting proteins (Takeda et al., 2001), which
commonly share different phosphorylation sites targeted by different signaling molecules
(casein kinase 1 - CK1; casein kinase 2 - CK2; and MAPKs), all three present in SCI1
(NetPhosK; ELM prediction: positions 17-23aa, 54-60aa, 109-115aa, 112-118aa for CK1,
positions 4-10aa, 44-50aa, 53-59aa for CK2 and positions 4-10aa, 15-21aa and 18-24aa for
MAPK).
To study the potential nuclear localization of SCI1, we fused the green fluorescent
protein to the SCI1 C-terminal (35Spro:SCI1-GFP) and analyzed the subcellular in planta
localization. The fluorescence microscopy revealed that the SCI1-GFP protein is exclusively
localized in the nucleus (Figures 1A-B), contrasting to the 35Spro:GFP construct that showed
a uniform distribution of GFP in both nucleus and cytoplasm (data not shown). It is known
that DAPI preferentially stain heterochromatin and, interestingly, the SCI1-GFP fluorescence
was present as spots within the nucleoplasm, where DAPI was not detected, therefore
corresponding to the interchromatin and nucleolus (Figures 1E-L). We found that the
reported patterns of the fusion protein occur with both low and high expression levels, which
were estimated by their fluorescence intensity under the microscope. This observation
indicates that the overexpression of the fusion protein is not influencing its localization.
Indeed, the nuclear pattern of the SCI1-GFP resembles the one shown for different
transducin/WD-40 repeat proteins, which interact with cell cycle regulators, and the 60S
ribosomal protein L27A both from Arabidopsis thaliana (Brown et al., 2005). In conclusion,
SCI1 is a nuclear protein that seems to co-localize with the nucleolus and the interchromatic
regions.
SCI1 is a developmentally regulated stigma/style-specific gene
6
To determine the expression pattern of SCI1 gene and confirm its pistil specificity, we
performed qRT-PCR experiments using RNA from roots, stems, leaves, sepals, petals,
stamens, stigmas/styles and ovaries. SCI1 is at least 34-fold more expressed in the
stigma/style than in any other part of the plant (Figure 1M). We also analyzed the temporal
expression pattern of SCI1 gene in the stigma/style during the 12 developmental stages
described by Koltunow et al. (1990). At stage 1, the pistil is already fully differentiated and
will grow and prepare itself for receptivity and maturity until stage 12, in which anthesis and
pollination occur. Figure 1N shows that the highest SCI1 expression level is observed at stage
1, the earliest developmental stage investigated. In the following stages, the transcript
abundance decreases and its level fluctuates during the first half period of development, with
the highest levels occurring at the early stages (from 1 to 6) than in the later stages towards
anthesis (from 7 to 12) (Figure 1N). Thus, SCI1 expression pattern suggests that its gene
product might be implicated in events taking place rather early than late in pistil
development.
The SCI1 gene is exclusively expressed at the stigma/style specialized tissues related to
plant reproduction
To verify which cell types transcribe the SCI1 gene, in situ hybridization experiments
were performed (Figures 1O-P). Sense and antisense riboprobes were hybridized to
longitudinal sections of stigmas/styles at stages 4, 7 and 10 of flower development. At stage
4, we observed a hybridization signal in the stigmatic secretory zone, including the papillar
cells, and in the stylar transmitting tissue (Figure 1P). A weak signal was observed in the
vascular bundle; however this was also seen in sections of stigmas/styles hybridized to the
sense probe (control). There was no detectable signal at stages 7 and 10 (data not shown).
These results are in agreement with the developmental expression pattern of the SCI1 gene,
as determined by qRT-PCR, in which a significant reduction of SCI1 expression was
observed in stigma/style tissues at stages 7 and 10 comparatively to the expression at stage 4.
SCI1 transcript is clearly more abundant in stigma/style specialized tissues at the
initial stages of flower development. Although these tissues are intrinsically related to pollenpistil interaction, most of the genes related to these interactions have a higher expression
level at late stages of flower development (Goldman et al., 1992; Wang et al., 1993; McClure
et al., 1999). On the other hand, it is expected that genes controlling cell proliferation and
differentiation are expressed at higher levels in the initial stages of flower development.
Taken together, the temporal and spatial expression patterns suggest an involvement of the
7
SCI1 gene product in the morphological and/or physiological development of the pistil, rather
than contributing directly to pollen-pistil interactions.
The SCI1 gene product controls stigma size
To unravel the SCI1 protein function, we generated tobacco transgenic plants with the
SCI1 gene expression being silenced by hairpin (hp) RNA (RNAi). Additionally, plants that
overexpress SCI1 were constructed. We obtained 13 independent transgenic plants possessing
the RNAi construct, which were designated SCI1Ri and 9 independent transgenic plants
harboring the overexpression cassette, designated as SCI1OE. The Figure 2A shows that the
RNAi-mediated silencing efficiently reduced the transcript levels (up to only 6.4% of the
wild-type transcript level for SCI1Ri 17.9), as monitored by qRT-PCR. The plants with the
lowest transcript levels showed a clear enlargement in stigma size (Figure 3B) and a
remarkable style elongation, up to 28% at anthesis (Figure 3A). This phenotype was observed
in 9 out of 13 RNAi transgenic plants and was visible when the SCI1 transcript levels were
below 27.8% of that of the wild-type plants (Figure 2A dashed bars). There was an overall
correspondence between the degree of the altered phenotypes and the SCI1 mRNA levels,
with the plant SCI1Ri 14.2 (8.4% of SCI1 mRNA) exhibiting the most expressive phenotype
(Figure 3A). The differences between the wild-type SR-1 and RNAi phenotypes were more
evident at stage 10 of flower development (Figure 3A). At this stage, the stigma/style remains
enclosed by the corolla limb in wild-type plants (Figure 3A*) while the RNAi plants exhibit a
protruding stigma/style (Figure 3A), apparent since stage 8 (data not shown). In accordance
to the phenotype generated by silencing SCI1, the SCI1OE plants revealed an opposite
modification on stigma, i.e., a smaller size (Figure 3B). Among 9 independent overexpression
transgenic plants, 5 transformants showed the same phenotype, which was associated with
the highest SCI1 transcript levels (48.8 fold or higher than wild-type plants), as revealed by
qRT-PCR (Figure 2B). On the other hand, we did not observe alterations in the style length
of the SCI1OE plants, which positioned the stigmas at the same height as the top of stamens
and petals at anthesis. The plant SCI1OE 1.1 showed an RNAi-like phenotype and,
accordingly, the SCI1 mRNA level in this transgenic plant was only 17.7% of the level in
wild-type plants (Figure 2B). This co-suppression effect is a known post-transcriptional gene
silencing mechanism induced in some transgenic plants as a response to the overexpression
of a gene (Napoli et al., 1990; Taylor, 1997). No other plant organ showed a macroscopically
altered phenotype when comparing wild-type and transgenic plants (SCI1Ri and SCI1OE; data
not shown). Taken together, these results strongly imply an involvement of the SCI1 protein
8
in controlling stigma size, probably by inhibiting cell proliferation and/or avoiding cell
enlargement in a tissue-specific manner.
The SCI1 protein is a negative regulator of the cell cycle
As an effort to elucidate SCI1 function and distinguish the effect on cell proliferation
and/or enlargement, we prepared longitudinal sections of stage 11 stigmas/styles from SCI1Ri
14.2, SCI1OE 3.1.1 and wild-type plants. We evaluated the stigmatic secretory zone (SSZ)
area (Figure 4 and Supplemental Table 1) as well as the number of SSZ cell layers in the
region indicated at Figure 3Ca. Figure 3 shows that the cell number is clearly increased
(p<0.05) in the stigmatic secretory zone (Figures 3Cd and 3D; Figure 4 and Supplemental
Table 1) as a consequence of reduced SCI1 mRNA levels, whereas SCI1 overexpression
resulted in a decreased cell number (p<0.05) in the SSZ (Figures 3Cb and 3D; Figure 4 and
Supplemental Table 1). Consequently, the SCI1Ri 14.2 transgenic plant showed a SSZ
average area of 367,355.3µm2, 48% larger than the average area (248,272µm2) of the wildtype plant (Figure 3Cd and 3Cc, respectively; Figure 4 and Supplemental Table 1). In
contrast, the SCI1OE 3.1.1 transgenic plant revealed an SSZ average area of 191,381.3µm2,
23% smaller than the SR-1 control plant (Figure 3Cb; Figure 4 and Supplemental Table 1).
Accordingly, the number of cell layers (cl) at the top of the SSZ (Figure 3Ca) was
significantly reduced to around 8cl in SCI1OE 3.1.1, while the SSZ of the wild-type plant
showed 13cl and of the SCI1Ri 14.2 RNAi plant increased to approximately 18cl (Figure 4
and Supplemental Table 1). In addition to the SSZ area and cl data, we also counted the cell
number in a defined area of 20,000µm2 (Figure 3Ca). The cell number of the SCI1OE 3.1.1
overexpression plant is smaller but not statistically different from the wild-type and SCI1Ri
14.2 plant, both with very similar values (Figure 4 and Supplemental Table 1). We did not
observe the phenomenon called compensation, in which cell size increases to balance for the
decrease in cell number, in the overexpression plants. However, the SCI1OE 3.1.1 SSZ cells
were more circular in shape and the intercellular spaces were slightly increased, consistent
with a lower cell density. In addition, a deeper carpel junction was observed (Figure 3Cb,
arrow). On the other hand, the cells of the SSZ seem smaller in RNAi plants, probably due to
a packed growth and/or a rapid re-entry into the cell cycle. However, taken as a whole, the
cells of the transgenic and wild-type plants were approximately of the same size.
Since the in situ hybridization experiments also revealed SCI1 expression in stylar
transmitting tissue (STT), we searched for alterations in this tissue. As the cells of the STT
are more packed, counting them individually as we have done for the SSZ cells is very
9
difficult. Thus, we measured the STT thickness in transgenic and wild-type plants (Figure
3Ea; Figure 4 and Supplemental Table 1) in longitudinal sections of stigma/style at 400µm
below the SSZ/STT intersection (for definition see Methods and Figures 3Ca and 3E).
Consistently, the STT thickness was decreased on SCI1OE 3.1.1 plant (p<0.05) and increased
in SCI1Ri 14.2 plant (p<0.05), when compared to the wild-type (Figure 4 and Supplemental
Table 1), indicating a smaller and a higher number of cells in this tissue, respectively. It is
noteworthy that the RNAi plant SCI1Ri 14.2 showed a higher cell number also in the
parenchyma tissue, when compared to wild-type and SCI1OE 3.1.1 plants (Figure 3Eb and
Figure 4). Since SCI1 is not expressed in the parenchyma cells, our results suggest that the
cells of the SSZ and STT may produce signals capable of influencing cell division in adjacent
tissues, maintaining certain proportionality: the more cells from the specialized tissues, the
more supporting tissue. Briefly, the higher SCI1Ri 14.2 style thickness (Figure 4 and
Supplemental Table 1) is a result of both, STT and parenchyma overgrowth, while the
parenchyma tissue remains approximately the same in the overexpression plant. Taken
together, these results reveal the involvement of the SCI1 protein in inhibiting the cell cycle
in a tissue-specific manner.
SCI1 is expressed at the very early stages of stigma/style development
Due to the high SCI1 expression level at stage 1 of flower development and its effects
on the final stigma/style size on transgenic plants, we decided to investigate its expression in
the very early stages of flower development (stages -4 to stage -1), in which the stigma is
differentiating (Koltunow et al., 1990; Wolters-Arts et al., 1996). In order to better correlate
the SCI1 function with the early events occurring at the stigma/style, we performed a
scanning electron microscopy (SEM) analysis of flower buds from stages -4 to stage 1, as
well as collected stigmas/styles from the same developmental stages for qRT-PCR
experiments. Besides measuring the floral bud length, we also considered floral and anther
morphological markers described previously (Koltunow et al., 1990) to reliably distinguish
among each developmental stage (Supplemental Figure 3; Methods).
The tobacco pistil is formed by the fusion of two carpels that give rise to the stigma
tissue around stage -4 of flower development (Figures 6Af and 7Bd). The stigmatic tissue
starts differentiation from the region of carpel fusion outwards (Figure 5Ak). At stage -3, the
surface of the stigma becomes evident with a rough aspect contrasting to the smooth surface
of the carpel epidermis (Figure 5Ag), and the style begins to elongate (Figure 5Ab).
Furthermore, this stage is characterized by a great enlargement in upper pistil size and a
10
visible differentiation of papillar cells at the stigma epidermis (Figures 5Ag and 5Al). At
stage -2, the style is definitely elongated (Figure 5Ac), the papillar cells turn into finger-like
structures (Figure 5Am) at the distal carpel edges and start to become much more evident at
the stigma central area (Figure 5Ah). In an upper view of the stigma, we clearly see the
papillar cells developing from the carpel edges and bordering the carpel junction towards the
outside in the central area (Figures 5Bb and 5Bc, yellow). Furthermore, the epidermis is still
visible at the central borders of the pistil at this stage (Figure 5Ah and 5Bc, blue). The
epidermis of the carpel tip is almost straight, as a consequence of the growth pressure
provoked by the stigmatic secretory zone. The stage -1 is portrayed by an extended style
(Figure 5Ad) and a clear enlargement in stigma size (Figure 5Ai), with the upper carpel
epidermis and parenchyma starting to bend backward, originating the abaxial part of the
stigma (Figure 5Bd). The papillae differentiation occurs throughout the stigma surface. At
stage 1, the SSZ is fully differentiated; the upper carpel epidermis and the parenchyma have
bended completely, forming an umbrella-like structure in a side view (Figure 5Ae and 6Af).
The bending of the upper carpel epidermis and parenchyma occurs mainly due to the
acceleration of the cell proliferation in the central region, where the carpels have fused.
As shown in Figure 5C, SCI1 gene is highly expressed at the very early stages of
stigma/style development, with the highest expression level observed at stage -3, when the
stigmatic secretory zone starts its differentiation. SCI1 transcript level decreases at later
stages (-2, -1 and 1) during which the stigmatic tissue is proliferating and continuously
differentiating. This expression pattern is consistent with a role in controlling cell
proliferation/differentiation in the upper pistil.
SCI1 controls cell proliferation/differentiation and influences the stigma/style
developmental timing
In order to study how the SCI1 gene product was affecting the stigma size and style
elongation, we decide to compare the very early stages of stigma/style development in wildtype and transgenic plants by scanning electron microscopy (SEM). We analyzed the
transgenic plants with the most striking phenotypes: the RNAi SCI1Ri 14.2 and the
overexpression SCI1OE 3.1.1. Stage -4 pistils of the RNAi SCI1Ri 14.2 plant showed features
similar to the wild-type pistil at stages -4/-3 of flower development (Figures 6Ba, 6Bd and
6Be). During the first steps of stigma/style formation, the SCI1Ri 14.2 plant clearly exhibited
a faster growth of the stigma through stages -3 and -2 (Figures 6Bb and 6Bc, respectively)
compared to the wild-type plant at the same stages (Figures 6Be and 6Bf). Indeed, at stage -3
11
the SCI1Ri 14.2 carpel epidermis already begins to bend backward (Figure 6Bb), an event
observed at stage -1 of the wild-type plant (Figures 5Ad, 5Ai and 5Bd). The stigma
differentiation was also advanced in the SCI1Ri 14.2 plant, occurring as soon as the cell
number became increased (Figures 6Ca-6Cc), showing that cell differentiation is coupled to
proliferation in SCI1 silencing plants. Additionally, pistils of the SCI1Ri 14.2 plant reached
the umbrella-like structure at stage -2 (Figures 6Ab and 6Bc), a phenotype observed only at
stage 1 of the wild-type plant (Figures 5Ae, 5Aj and 6Af). Likewise, the style elongation
follows the accelerated growth of the stigma, becoming longer and conducting the stigma
surface to a higher position in relation to the anthers already at stage 1 (Figure 6Ac), while
this is not observed in wild-type plants (Figure 6Af). The developmental acceleration
observed in the SCI1Ri 14.2 plant, with a reduced level of SCI1 mRNA, suggests that the
SCI1 gene product might be a key regulator in the stigma/style developmental control. The
size of each cell at the RNAi stigma is similar to those of the wild-type, confirming that a
higher number of cells should be present in the RNAi stigmas to occupy the larger area
observed. Our results showed that the stimuli for style elongation, which generates a
protruding style in the SCI1Ri 14.2 plant at anthesis, starts at the very early stages of its
development, caused by SCI1 silencing. Taken together, these results strongly suggest the
involvement of the SCI1 gene product in influencing the final stigma size and style
elongation, through the control of cell divisions, since the initial stages of organ formation in
wild-type plants.
The upper pistils of the overexpression SCI1OE 3.1.1 plant showed a comparable
phenotype to the wild-type pistils at stage -4 (Figures 6Bg and 6Bd, respectively). However,
the upper pistil of this transgenic plant showed a delayed development from stage -4 to stage
-3, with a minimal differentiation of papillar cells (Figures 6Cg and 6Ch). The typical stigma
surface characteristics, observed at stage -3 pistils of the wild-type plants, are visible only at
stage -2 pistils of the SCI1OE 3.1.1 plant (Figure 6Ce and 6Ci, respectively). It is interesting
to observe that the SSZ differentiation is strictly linked to cell proliferation. Pistils at stages 1 and 1 of the overexpression plant are morphologically and developmentally similar to
stages -2 and -1 of the wild-type plant, respectively. Besides, the SCI1OE 3.1.1 style is visible
only at stage -2 (Figure 6Ah), remaining shorter than the wild-type one at stage 1 (Figure
6Ai). Consistently, stigmas of the SCI1OE 3.1.1 plant have a smaller size than the ones of the
wild-type plant when they are compared side by side at stages -3, -2, -1 and 1 of flower
development. In summary, pistils of the overexpression SCI1OE 3.1.1 plant show a growth
and differentiation delay of nearly one stage when compared to pistils of the wild-type plant.
12
As can be seen at Figure 6Ag, the anther development was also affected in the SCI1OE 3.1.1
plant, due to the ectopic expression of SCI1 gene under the control of the 35S promoter,
while no other vegetative or floral organs were macroscopically altered. The SCI1 ectopic
expression resulted in smaller anthers at very early stages of flower development (Figures
6Ag and 6Ah), although at stage 1 they had already recovered their normal size (Figure 6Ai).
At stage -3, the SCI1Ri 14.2 and the wild-type plants exhibit similar anther size (Figure 6Aa
and 6Ad, respectively). Taken together, we can propose that the SCI1 protein inhibits cell
division at very early stages of flower development in a tissue-specific manner and is
sufficient to modulate the developmental timing of the stigma/style. These observations
suggest that SCI1 may be part of a molecular developmental clock pathway that influences
the final organ size and its number of cells.
SCI1 associates with A-type cyclins
Because of its anticipated role as a negative regulator of the cell cycle and the
presence of two putative cyclin-interaction domains, it was tested whether SCI1 could be a
direct regulator of CDK/cyclin complexes. As all CDKs and cyclins have been well
annotated for Arabidopsis thaliana, the orthologous SCI1 gene (AtSCI1) was cloned from this
species, and subsequently tested for interaction with 11 CDKs and 24 different cyclins using
the yeast 2-hybrid system. The interaction between two proteins was considered positive only
when two independent reporter genes were activated (the biosynthetic histidine gene HIS3,
resulting into histidine prototrophy, and the β-galactosidase-encoding gene LacZ, resulting in
blue colonies in the presence of 5-bromo-4-chloro-3-indolyl-β-d-galactopyranoside). None of
the CDKs tested displayed interaction with AtSCI1 (Supplemental Table 2). By contrast, two
related A-type cyclins scored positive, being AtCYCA1;1 and AtCYCA2;2 (Figure 7A;
Supplemental Table 2). The binding specificity of AtSCI1 towards CDKs and cyclins differs
from that of AtKRP2 (one of the founding members of the ICK/KIP family), which
associated in the yeast 2-hybrid system with D-type cyclins only. Additionally, AtKRP2 also
binds CDKA;1 (Figure 7A and Supplemental Table 3).
To check whether the interaction between SCI1 and A-type cyclins also occurs in
planta, we cloned the N. tabacum CYCA1;1 (NtCYCA1;1) and CYCA2;2 (NtCYCA2;2)
homologous sequences and performed Biomolecular Fluorescent Complementation (BiFC)
tests (see Methods). The NtSCI1 C-terminus was fused to the head of the GFP (hGFP; amino
acids 1 to 149) and the NtCYCA1;1, NtCYCA2;21-398 (full length) and NtCYCA2;296-398
(without the destruction-box) C-termini were fused to the tail of the GFP (tGFP; amino acids
13
150 to 238). As shown in Figure 7B, the NtSCI1 exhibited interaction with NtCYCA1;1,
restoring GFP fluorescence in planta, which is nuclear localized as expected. So far, we have
not been able to demonstrate interaction of NtSCI1 with the NtCYC2;2 isoforms. Our results
clearly show that both N. tabacum and Arabidopsis SCI1 proteins interact with A-type
cyclins, in vitro and in planta.
SCI1 influences the transcript levels of some auxin-regulated genes
Due to the phenotype similarity of the pistils of our RNAi transgenic plants with
Arabidopsis pistils treated with N-1-naphthylphthalamic acid (NPA; Nemhauser et al., 2000),
we decided to investigate the transcription levels of three auxin-regulated genes, identified in
the tobacco stigma/style EST pool (TOBEST database; Quiapim et al., 2009), in the
transgenic SCI1 plants (SCI1Ri and SCI1OE) comparatively to the wild-type. The transcript
levels of the tobacco genes homologous to the Arabidopsis Auxin Response Factor 8 (ARF8)
and the Auxin/Indole-3-Acetic Acid (Aux/IAA) Aux/IAA13 and Aux/IAA19 transcription
factors were measured by qRT-PCR in stigmas/styles of wild-type plants, four independent
RNAi plants and four independent overexpression plants (Figure 8). These genes were
chosen based on their involvement with flower development (ARF8, Wu et al., 2006), ability
to change multiple auxin responses (Aux/IAA19, Tatematsu et al., 2004) and to impairs
auxin-regulated development (Aux/IAA13, Weijers et al., 2005). For this experiment, we
chose the transgenic plants that displayed the lowest and the highest SCI1 mRNA levels
(Figure 2). Here, we decided to substitute the internal control on the qRT-PCR analysis. The
β-actin gene was shown to be the best internal control for gene expression comparisons
among different tobacco organs (Quiapim et al., 2009) and used on the above experiments.
On the other hand, the ubiquitin gene is suggested as the most suitable control when the same
organ is compared during alterations on cell cycle control (Eloy et al., 2006; Masuda et al.,
2008). Since SCI1 is changing the cell number on stigmas/styles, the ubiquitin gene was used
as internal control for this experiment. Figure 8A shows the re-analysis of SCI1 expression
levels in those transgenic plants using the ubiquitin gene as an internal control. Coherently
with the idea that this reference gene might be more appropriate when genes involved in cell
cycle control are altered, the results obtained for SCI1 expression level in the RNAi
transgenic plants correspond perfectly with the alteration degree of their stigma/style
phenotypes.
The NtARF8 mRNA accumulation was not affected in SCI1Ri plants (Figure 8B).
However, we observed an increase in its transcript levels of up to 2.8-fold in SCI1OE plants
14
(p<0.05) (Figure 8B). NtAux/IAA13 and NtAux/IAA19 were also upregulated in
overexpression plants, up to 4.2-fold and 5.1-fold (p≤0.001), respectively (Figure 8C and
8D). Additionally, the transcription of NtAux/IAA13 (Figure 8C) and NtAux/IAA19 (Figure
8D) were altered in RNAi plants, with the NtAux/IAA19 expression proportionally
associated to SCI1 downregulation in these plants (p<0.05). Our results suggest that SCI1
may influence the transcriptional regulation of some auxin-regulated genes and somehow be
related to auxin signaling in upper pistil development.
DISCUSSION
SCI1 is a tissue-specific cell cycle regulator that influences pistil development
We have identified a gene encoding a lysine-rich protein specifically expressed on
stigma/style specialized tissues during its early stages of development. Based on the
phenotypic analysis of the overexpression and silenced transgenic plants, our results strongly
indicate that SCI1 has a cell proliferation inhibitory activity. The smaller SSZ observed in
SCI1 overexpression transgenic plants is formed of cells with approximately the same size as
cells of wild-type plants. Additionally, the SEM analysis of the floral buds of overexpression
plants has demonstrated a delay in cell proliferation/differentiation. Thus the smaller SSZ is a
consequence of a decrease total number of cells accumulated during stigma development. On
the other hand, the larger SSZ obtained in SCI1 RNAi plants is directly associated with an
earlier start in the cell proliferation/differentiation activity and not with larger cells.
Therefore, our results suggest that SCI1 participates in the timing control of stigma/style cell
proliferation and differentiation and seems to be an integrator of cell cycle regulation to the
stigma/style developmental program, ensuring that the interconnected cell proliferation and
differentiation processes occur at the appropriate moment.
In flower meristems, undifferentiated cells divide asymmetrically giving rise to
daughter cells, which carry the cell fate information. However, the final differentiation is
postponed until some rounds of symmetric cell divisions. The right moment in which these
cells exit the cell cycle and differentiate, i.e. the differentiation timing, is controlled by
different factors, as CDK activity and growth factors (reviewed in De Veylder et al., 2007).
De Veylder et al. (2007) recently proposed a model for the control of cell
proliferation/differentiation by CDK activity, which might drop below a threshold to the cells
undergo cell cycle exit and differentiate. SCI1 interaction with A-type cyclins suggests that it
15
controls cells differentiation and organ size through a direct modification of CDK activity.
Among the known repressors of growth that regulate the activity of different growth
restricting factors, only DA1 changes organ size without changing the cell size (Li et al.,
2008), as SCI1 does on tobacco stigma/style. Considering that DA1 control organ size in
different tissues, as seeds, embryos, cotyledons, leaves, stems and flowers, it is tempting to
speculate that SCI1 represents a downstream effector of DA1 among the tissue-specific
growth restricting factors, complementing the model recently outlined by Krizek (2009) on
pathways controlling final organ size.
Additionally to the differentiation timing, an interesting issue in organ development is
whether the cell division follows a developmental program (organismal theory) or the cell
division instructs organogenesis and development (cellular theory). Due to ambiguous results
present in the literature that can fit in both theories, the “neo-cell-theory” has been proposed
(Tsukaya, 2002; Beemster et al. 2003). In this theory, an integrating model between growth
and division is supported. In SCI1 transgenic plants, alteration in cell division activity on
stigma/style tissues affects both the final organ size and the differentiation timing, which
supports the cellular theory. However, while the STT thickness is modified in both types of
SCI1 transgenic plants due to altered cell division activity, the style length is modified only in
the RNAi plants. In the overexpression plants the style elongated to a similar length as in the
wild-type. This phenotype represents an unaltered developmental program, which was not
influenced by the reduced cell division activity in the SSZ/STT of the SCI1OE plants,
supporting the organismal theory. The SSZ cell ablation led to shorter styles in
STIG1pro:barnase transgenic plants (Goldman et al., 1994), which had no alteration in STT
cells. Taken together, these observations suggest that the style elongation program is a result
of different signals, including those from stigma tissue (SSZ). Thus, one possible explanation
for the phenotypes in SCI1 transgenic plants is that the lower number of cells in SCI1OE
stigmas had produced sufficient “stigma-dependent elongation signal” for SCI1OE styles to
elongate to the wild-type length. On the other hand, the protruded style in SCI1Ri plants
would be a consequence of the increased number of stigma cells, which in turn produced
more of the “elongation signal(s)” or, alternatively, had started to produce the “elongation
signal(s)” earlier. In agreement with the idea of a signal that can move between cells, in
SCI1Ri styles we observed an increase in the parenchyma cell number, a tissue that does not
express SCI1. The consequences of SCI1 modulation on stigma size and style length suggest
that pistil development is governed by a variety of signals, in agreement with the proposal
that growth processes are implemented at the cellular level but engage crosstalk between
16
organizational levels, not simply following the organismal or the cellular theories.
Considering this point of view, how do the SCI1 results fit into the “neo-cell theory”? In our
SCI1Ri plants, an increase in cell division on stigma and a premature differentiation is
observed. Thus, we propose that cell division is inhibiting cell differentiation until the
stigmatic cell number reaches a threshold above which the differentiation signals accumulate
and, hereafter, cell division rate slows down and will be coupled with cell differentiation
(Figure 9). Coherently with this proposal, the SCI1OE plants, in which an over inhibition of
stigma cell division occurs, begin their stigma/style differentiation process later than the
wild-type plant, but the subsequent steps of differentiation follow the wild-type
developmental rate (Figure 9). Our results show that SCI1 gene encodes an important tissuespecific cell cycle regulator that influences the upper pistil growth and development, without
changing the cell size.
The regulation of SCI1 expression levels is sufficient to influence stigma size and
style length, which may have important consequences in plant reproduction. A decreased
SCI1 level has resulted in stigmas positioned above the anthers and in heterostyly in our
population of tobacco transgenic plants. In nature, the regulation of expression levels of the
SCI1 homologous sequences may contribute to the heterostyly observed in some species with
perfect (bisexual) flowers and have consequences on the rates of self- and cross-pollination.
SCI1 is a cell cycle regulator distinct from the plant CDK inhibitors already described
Different aspects of plant development have been studied to uncover the molecular
network behind organ(s) growth and development, culminating with the discovery of
different classes of genes, as those involved in cell division control. To achieve this control,
the cell recognizes different signals from a variety of pathways, which are transduced by cell
cycle regulators and result in the activation or inhibition of the cell cycle. Plant CKI/KRPs,
which have similarity with the mammalian Kip/Cip inhibitors, directly interact with CDK
and/or cyclin proteins and constitute the major negative regulators of CDK/cyclin complexes
to control cell cycle entry in plants (De Veylder et al., 2007). Despite its effect in inhibiting
the cell cycle, SCI1 protein has no evident sequence similarity with the CKI/KRPs, even
when their more conserved C-terminal regions are either searched or aligned independently.
On the other hand, SCI1 shows a limited but recognizable similarity with SIM (Supplemental
Figure 4A), a new class of CDK inhibitors, in a short region that coincides with the region of
similarity between SIM and CKI/KRPs (Supplemental Figure 4B). This region was shown to
act as a cyclin interaction domain (Peres et al., 2007). Accordingly, using the yeast 2-hybrid
17
system, SCI1 was found to interact with two different A-type cyclins (CYCA1;1, CYCA2;2)
and, using BiFC, with at least CYCA1;1. In contrast to the CKI/KRP and SIM inhibitors,
SCI1 did not associate with D-type cyclins and CDKA;1 in the yeast 2-hybrid system. The
biological significance of the binding specificity towards A-type cyclins remains elusive at
this point.
Is SCI1 a new class of CDK inhibitor? Plant CDK inhibitors usually have a small
molecular weight, contain conserved motifs, like the cyclin-binding domains (Supplemental
Table 4), and are exclusively present in the nucleus of the cell (Wang et al., 2008),
characteristics shared with SCI1. Indeed, CDKs become active only after their interaction
with cyclins, and we showed that SCI1 has a direct link with the cell cycle machinery by its
ability to bind A-type cyclins. So, based on the phenotypes of SCI1 transgenic plants, proteinprotein interaction experiments and in conformity with the current model for the control of
cell proliferation/differentiation (De Veylder et al., 2007), it is plausible to suggest that the
CDK activity is being changed on SCI1OE and SCI1Ri plants. However, whether SCI1
sequester A-type cyclins or form a complex to inhibit the CDK/cyclin heterodimers, remains
to be established.
All plant CKI/KRPs overexpressed until now caused an overall reduced growth,
serrated leaves and abnormal flowers (Wang et al., 2000; De Veylder et al., 2001; Jasinski et
al., 2002a; Zhou et al., 2002; Ren et al., 2008). In contrast to CKI/KRPs and SIMs, SCI1
transgenics showed a tissue-specific phenotype. Only pistils and anthers have been affected
as a consequence of overexpression driven by the 35S promoter, which means that SCI1
depends on a specific cellular background to inhibit the cell cycle and then regulate
organogenesis. As the A-type cyclins are broadly expressed in the whole plant (Menges et al.,
2005), this control is probably dependent on tissue-specific post-translational modifications
(e.g. phosphorylation and dephosphorylation events, Supplemental Figure 1B) and/or occurs
through interaction(s) with a tissue-specific target(s). The unraveling of a cell-cycle regulator
that acts in a very specific tissue suggests the existence of proteins with similar functions in
other plant tissues. Therefore, we propose SCI1 as a new class of CDK inhibitor, distinct
from the CKI/KRPs and SIMs, and the existence of at least three classes of CDK inhibitors:
a) CKI/KRPs (CDK inhibitors/Kip-related proteins); b) SIMs (SIAMESE and SIAMESErelated proteins), both broadly expressed among plant tissues and with a direct change of
CDK activity; and c) TICKs (Tissue-specific inhibitors of CDK), which show tissue
specificity and interact with cyclins, probably regulating CDK activity indirectly. SCI1 would
be the first member of the TICK class.
18
SCI1 is localized in the nucleus, as demonstrated here in leaf cells transiently
transformed with a 35Spro:SCI1-GFP quimeric gene. Therefore, its nuclear localization is
independent of interaction(s) with stigma/style-specific proteins and/or phosphorylations
resulting from stigma/style-specific transduction signals. The SCI1-GFP fusion protein
confirmed the presence of the predicted NLS and exhibited two distinct nuclear localizations:
predominantly inside the nucleolus; and distributed in both nucleolus and nuclear bodies,
always placed in the interchromatin region. Despite the well-known involvement of the
nucleolus in ribosomal RNA (rRNA) and small nucleolar RNA (snoRNA) biogenesis, it has
recently been considered a multifunctional nuclear structure, exerting different functions such
as in cell cycle control (Zimber et al., 2004). It has been shown that cell cycle regulators are
sequestered in the nucleolus, which points to a role for this organelle in regulating their
activity (Visintin and Amon, 2000). Additionally, in previous reports it has been shown that
changes in the architecture and functional organization of the nuclear bodies accompanied the
switch of the developmental program and the activation of the proliferative activity in plant
cells (Testillano et al., 2005; Echeverría et al., 2007). The compartmentalization to the
nuclear bodies were also described for the Arabidopsis ICK1/KRP1 protein (Zhou et al.,
2006) and for NtKIS1a protein, when the latter was co-expressed with the AtAJH1, the plant
homolog of the human JAB1 (c-Jun activation domain binding protein 1) protein that is
believed to target the degradation of the p27KIP1 CDK inhibitor (Le Foll et al., 2008). We
propose that SCI1-GFP distribution in the nucleolus and its relocation to nuclear bodies
might be linked to different cell fates or cell cycle phases during pistil development, which is
coherent with the SCI1 involvement in the cell proliferation/differentiation control.
Does SCI1 transduce signals engaging auxin signaling and cell division/differentiation?
The initial carpel cells are a small group of cells able and committed to divide and
differentiate to give rise to the adult pistil. Such developmental process includes mainly
hormone-mediated organogenesis associated to cell division and differentiation. Often,
alteration of final organ size is one of the consequences of modified phytohormone signaling
(Nemhauser et al., 2000; Krizek, 2009). Taking in consideration that the SCI1Ri tobacco
stigma/style phenotypes are analogous to those of the NPA-treated Arabidopsis pistils
(Nemhauser et al., 2000), we have investigated the possibility that some auxin responsive
genes could be influenced by SCI1 level.
The Auxin/Indole-3-Acetic Acid (Aux/IAA) and Auxin Response Factor (ARF)
families of transcription factors are key regulators with well-characterized roles in auxin
19
responses (Benjamins and Scheres, 2008). The Aux/IAA proteins dimerize with ARFs to
repress auxin-dependent gene transcription. In high intracellular auxin concentrations, the
Aux/IAA are degraded, thereby releasing the ARF proteins, which can regulate auxindependent gene transcription through their ability to bind auxin responsive elements (AREs).
Recently, Jurado et al. (2008) reported auxin stabilization of the E2Fc/DPB heterodimers, a
negative cell cycle regulator, showing that cell division, cell differentiation and auxin
signaling have some molecular intersections. The Arabidopsis auxin insensitive mutant msg21, which encodes the Aux/IAA19 protein, is defective in the induction of Aux/IAA4, DFL1
(DWARF IN LIGHT 1) and SAUR-AC1 genes after hormone treatment, when compared to
the high induction observed in wild-type plants (Tatematsu et al., 2004). The Aux/IAA4,
DFL1 and SAUR-AC1 belong, respectively, to the three Aux/IAA, GH3 and SAUR (small
auxin up RNA) classes of early auxin responsive genes, which are directly and quickly
influenced by auxin treatment. Thus, the altered expression of these genes in the msg2-1
mutant invokes the Aux/IAA19 in the primary steps of auxin response. The down- or upregulation of SCI1 levels can, respectively, down- and up-regulate the NtAux/IAA19 mRNA
levels in transgenic plants, indicating that SCI1 is somehow positively correlated to
NtAux/IAA19 gene expression. The Aux/IAA19 homolog in rice, OsIAA3, is preferentially
expressed in flowers and is down regulated in coleoptiles treated with 2,4dichlorophenoxyacetic acid (2,4-D), an auxin analogue (Jain et al., 2006). Accordingly, in the
SCI1Ri plants, which showed an increased SSZ and STT cell numbers, the NtAux/IAA19
transcription level was reduced, simulating an auxin induction of cell division. Interestingly,
the T-DNA mediated knockout of SCI1 gene in Arabidopsis has an extremely similar stigma
morphology as yuc2yuc6 stigmas (DePaoli et al., unpublished data), which are deficient in
proper auxin synthesis (Cheng et al., 2006). These observations suggest that an appropriate
auxin signaling on stigma/style might be necessary for the correct changes on gene
transcription during cell proliferation/differentiation control. Likewise, the Aux/IAA19 and
the Aux/IAA13, which in Arabidopsis are repressed by auxin (Weijers et al., 2005), had their
transcription induced in SCI1OE plants, which showed a reduced number of cells. At the same
time, the overexpression of SCI1 resulted in the up-regulation of the ARF8 homologous
transcript, which has been shown to repress tissue elongation through a reduction in the free
IAA content (Tian et al., 2004). Therefore, the correlation between the induction of
NtAux/IAA19, NtAux/IAA13 and NtARF8 expressions and a reduced cell number of unaltered
size is totally coherent with the phenotype observed in SCI1OE plants and points towards a
continuous equilibrium on organ growth. On the other hand, the unaffected expression of
20
NtARF8 and NtAux/IAA13 in SCI1Ri plants suggests that the activation of these genes can be
supplied by other factors in the absence of SCI1. Taken together, our results suggest that
SCI1 is somehow interconnected with the auxin signaling pathway during its control of the
cell division and differentiation in the stigma/style.
In summary, our characterization of this lysine-rich protein, SCI1, revealed a new
class of protein able to inhibit the cell cycle in a tissue-specific manner. We believe that SCI1
is a signaling molecule that influences the upper pistil development by inhibiting cell
proliferation/differentiation, probably through its interaction with A-type cyclins. SCI1
function is most likely modulated by different protein-protein interactions and
phosphorylation events and possibly has molecular intersections with the auxin signaling
pathway.
METHODS
Plant Material
Wild-type tobacco (Nicotiana tabacum cv Petit Havana SR-1) and tobacco transgenic
plants were grown under standard greenhouse conditions in Ribeirão Preto – SP, BRAZIL
(Latitude - 21º 10'24" S, Longitude - 47º 48'24" W, with average temperature of 22˚C in
winter and 27˚C at summer; the difference in day length between summer and winter is less
than 2 hours). Samples of roots, stems, leaves, sepals, petals, stamens, stigmas/styles and
ovaries were collected from wild-type plants, frozen in liquid nitrogen and stored at -70˚C for
RNA extraction. For the developmental expression studies, stigmas/styles were excised from
flowers at stages -4 to 12 of the tobacco flower development as described previously
(Koltunow et al., 1990).
Sequencing and computational analyses
The clones TOBSH1-002E06 from a tobacco stigma/style SSH library (DePaoli et al.,
manuscript in preparation) and TOB068F10 from the TOBEST project were completely
sequenced using the methodology previously described (Quiapim et al., 2009). As they are
partial but complementary clones, their sequences were used to define primers for amplifying
the complete SCI1 coding sequence. The SCI1-ORF fragment was amplified directly from
stigma/style cDNAs (as prepared for qRT-PCR, see below) using the Ct11attB1FW
(5’gcaggcttcaccATGGGGAGCGATAAGAAGAC3’)
and
the
Ct11attB2RW
(5’aagctgggtcTTACTTTTTGATATTCCAAGCGTG3’) primers, which contain part of the attB
recombination sites (shown in lowercase). The PCR conditions used were: 94ºC for 4 min; 35
21
cycles of 94ºC, 50ºC and 72ºC for 1 min each; and 10 min for 72ºC. An aliquot (5µL) of this
PCR product was used for a second PCR reaction, at the same conditions (except for a
temperature
of
45ºC
during
the
annealing
step),
using
(5’ggggacaagtttgtacaaaaaagcaggcttc3’)
the
and
primers
BP1
BP2
(5’ggggaccactttgtacaagaaagctgggtc3’). An aliquot (10µL) of the second PCR product
was used for a recombination into the pDONR201 entry vector (Gateway, Invitrogen, USA)
using the BP clonase (Invitrogen, USA), as manufacturer’s instructions. An aliquot (7µL) of
the BP reaction was used to transform competent DH5α Escherichia coli cells (Sambrook
and Russell, 2001). The resulting clone, denominated SCI1-ORF, was also completely
sequenced.
The nucleotide and deduced amino acid sequences were analyzed by several computer
programs: Translate at ExPASy Proteomics Tools (www.expasy.org), BLAST at NCBI
(http://blast.ncbi.nlm.nih.gov/Blast.cgi)
and
SOL
Genomics
(http://sgn.cornell.edu/solanaceae-project/),
Network
ClustalW
(http://www.ebi.ac.uk/Tools/clustalw2/index.html), MotifScan (http://myhits.isb-sib.ch/cgibin/motif_scan),
PSORT
(http://psort.ims.u-tokyo.ac.jp/form.html),
PredictNLS
(http://cubic.bioc.columbia.edu/predictNLS/), ELM prediction (http://elm.eu.org/), NetPhos
and
NetPhosK
(http://www.cbs.dtu.dk/services/),
PAIL
(http://bioinformatics.lcd-
ustc.org/pail/prediction.php), Protein Molecular Weight and Isoelectric Point with
phosphorylation
(http://scansite.mit.edu/calc_mw_pi.html),
T-Coffee
(www.ebi.ac.uk/t-
coffee/) and Multalign (http://www.expasy.org/tools/#align).
RNA Extractions
Total RNA was extracted from large samples of frozen material essentially as
described by Dean et al. (1985). For the analyses of transgenic plants (SCI1Ri and SCI1OE)
and comparisons with wild-type plants, four samples of stigmas/styles at stage 4 of flower
development were collected from each plant and total RNA was extracted using the “Plant
RNA purification Reagent” (Invitrogen, USA). The RNA samples were quantified by
measuring OD at 260 and 280nm. To evaluate the RNA integrity, RNA was fractionated in a
2.2 M formaldehyde-1.2% agarose gel, stained with ethidium bromide, and visualized with
UV light. The presence of intact 28S and 18S rRNA bands was used as the criterion for RNA
integrity.
Quantitative real time RT-PCR and in situ hybridization
22
For qRT-PCR, 20µg of total RNA from each wild-type organ or the total RNA sample of the
“Plant RNA purification Reagent” extraction were treated with 5U of DNAse (Promega,
USA) for 1,5h as manufacturer’s instructions, and checked for genomic DNA contamination
in a standard PCR reaction, using the β-actin primers (Table 1). Each DNA free RNA sample
was used as template for reverse transcription using 150 pmol of oligo dTV primer (dT18V)
and 200 U of the Superscript II enzyme (Invitrogen, USA) as manufacturer’s instructions.
After the first strand cDNA synthesis, water was added to complete a volume of 70µL. The
standard qPCR reaction was performed using an aliquot (1µL) of the cDNA, 2µL of each
gene-specific primer (Table 1) at 1pmol and 5µL of the Power SYBR Green PCR Master
Mix (Applied Biosystems, UK) in a final volume of 10µL. The PCRs were performed in 96well plates using an ABI Sequence Detection System 7500 Fast (Applied Biosystems, UK).
The PCR thermal cycling conditions were an initial step at 50°C for 2 min; followed by 10
min at 95°C; 40 cycles, with each cycle consisting of 15 s at 95°C and 1 min at 60°C and a
final dissociation curve, with the temperature gradually rising from 60ºC to 95ºC. A standard
curve was built for each gene using a serial dilution to calculate the log of copies of the
transcripts based on the Ct value as described at Semighini et al. (2002). All analyses were
performed in triplicate and at least two biological replicates were done for wild-type gene
quantification. In all experiments, appropriate negative controls containing no template DNA
or RNA were subjected to the same procedure to exclude or detect any possible
contamination or carryover. The expression level in arbitrary units corresponds to the
calculated log of copies of the gene of interest on the calculated log of copies of the β-actin or
polyubiquitin internal control (Quiapim et al., 2009). The primers used for qRT-PCR are
shown in Table 1. The statistical analyses were performed using the SigmaStat.v3.5 software.
For in situ hybridization experiments, stigma/styles at stages 4, 7 and 10 of flower
development (Koltunow et al., 1990) were fixed in 4% glutaraldehyde, run through the
alcohol-xylol series and embedded in paraffin wax (TissuePrep2 - Fisher Scientific, USA),
sliced into 8µm sections, hybridized and revealed essentially as described by Jackson et al.
(1991). The riboprobes were prepared by in vitro transcriptions using the clone TOBSH1002E06 linearized at the 3’end, sense probe, and 5’end, antisense probe. For this purpose, we
used the DIG RNA Labeling Kit SP6/T7 (Roche, Germany) as manufacturer’s instructions.
DIG-labeled probes were detected using the anti-DIG antibody conjugated with AP (Roche,
Germany) and the 1-step NBT/BCIP detection mixture (Thermo Fisher Scientific, USA).
Nucleic acid manipulations and gene constructions
23
All the basic nucleic acid manipulations were as described at Sambrook and Russell
(2001). To obtain the 35Spro:SCI1-GFP construction, the SCI1 coding sequence was
amplified from the SCI1-ORF clone, using the primers Ct11attB1FW (described above) and
the Ct11attB2GFPRW (5’aagctgggtcCTTTTTGATATTCCAAGCGTG3’), which excluded the stop
codon from the SCI1-ORF sequence, creating an AvaII restriction site (bold underlined). The
procedure was performed as described above for the SCI1-ORF, including the recombination
of the PCR fragment into the pDONR201 vector. The resulting clone, pENTRY SCI1-ORFss
(without stop codon) was confirmed by AvaII digestion. Then, 300µg DNA of a positive
clone was used in the LR recombination reaction with the pK7FWG2 (Karimi et al., 2002)
plant expression vector, using the LR clonase from Invitrogen (USA). The construction was
sequenced to confirm the absence of undesirable mutations.
To obtain the SCI1 RNAi and overexpression constructions, 300µg DNA of the
original SCI1-ORF, cloned in the pDONR201 as described above, was recombined into the
pK7GWIWG2(I) and pK7WG2 (Karimi et al., 2002) plant expression vectors to generate the
SCI1Ri and SCI1OE constructs, respectively. These recombination reactions were performed
using the LR clonase (Invitrogen, USA). Each plant expression vector was introduced
separately in the Agrobacterium tumefaciens C58C1RifR (pGV2260) by electroporation.
The NtCYCA1;1 and NtCYCA2;2 sequences were obtained by RT-PCR from
stigma/style
mRNAs,
using
the
primers
CYCA1.1FW
and
CYCA1.1ssRV
(5’agcaggcttcGAAGGAGATAGAACCATGGCGACGACYCAGAATAG3’)
(5’aagctgggtcGCAGCTTATGTTCTGGAAG3’),
CYCA2.2FW
(5’agcaggcttcGAAGGAGATAGAACCATGAGGCATGCAAATATAAAAC3’)
and
CYCA2.2ssRV
(5’aagctgggtcGAACAGTGATTGGACTGGTTTTG3’), respectively. The attB recombination sites
are shown in lowercase and the ribosome binding sites are underlined. The amplified
fragments were purified, used for a second PCR using the BP1 and BP2 primers, and cloned
into the pDONR201 entry vector by recombination, as described above. The clones were
sequenced to confirm their identity as NtCYCA1;1 and NtCYCA2;2. The sequence
NtCYCA2;296-398 (without the destruction-box) was obtained by PCR, from the NtCYCA2;21398
(full
length),
using
the
primers
CYCA2.2DelFW
(5’agcaggcttcGAAGGAGATAGAACCATGAATTGCATCAACGGAAG3’) and CYCA2.2ssRV.
To obtain the NtSCI1 fused to the hGFP (35Spro:NtSCI1-hGFP), we recombined the
pENTRY p35S - attB4/1R, the pENTRY hGFP - attB2R/3 and pENTRY SCI1-ORF without
stop codon – attB1/B2, into the pK7m34GW via LR recombination reaction. Similarly, to
24
obtain the NtCYCA1;1, NtCYCA2;21-398 (full length) and NtCYCA2;296-398 (without the
destruction-box) fused to the tGFP (35Spro:NtCYCA-tGFP) we recombined the pENTRY
p35S – attB4/1R, pENTRY tGFP - attB2R/3 and the appropriate pENTRY NtCYCA –
attB1/B2, into the pH7m34GW. Each construction was sequenced from 3 different points to
confirm the correct recombination without mutations, and individually introduced in the
Agrobacterium tumefaciens C58C1RifR (pGV2260) by electroporation.
Plant transformation
Nicotiana tabacum cv Petit Havana SR1 seeds were surface sterilized and sowed in
Petri dishes containing MS medium (Murashige and Skoog, 1962) supplemented with 3%
sucrose, 0.5 g/L MES, brought to pH 5.8 and 7% agar (Bacto Agar). Seedlings were grown in
a growth chamber at 26-27°C with a photoperiod of 16h light /8h dark. For transient
expression assays, plants (approximately 6 weeks old) were transferred to soil and their
leaves were infiltrated with the Agrobacterium culture(s) containing the appropriate
constructs, as described by Sawers et al. (2006), and were analyzed 2 to 5 days after
infiltration.
For stable transgenic plants, tobacco leaf discs were co-cultivated with the
Agrobacterium clone containing the appropriate construction, according to the protocol
previously described (Brasileiro & Carneiro, 1998). Transgenic plants were regenerated on
kanamycin containing medium during the whole in vitro procedure and then transferred to the
greenhouse in a soil:vermiculite mixture. To ensure that environmental conditions were not
influencing our plant phenotypes, we transferred some plants during the winter/spring
seasons (i.e., SCI1Ri4.4.1 and SCI1Ri13.2) and others during the summer/autumn seasons
(i.e., SCI1Ri14.2 and SCI1Ri16.1). After 2 weeks, the plants were transferred to soil where
they remain until flowering and fruit production. These plants represent the first transgenic
generation (T0), which were analyzed in the qRT-PCR and light microscopy experiments.
The next generation (T1) was produced by controlled pollination; the seeds were sowed in
MS medium containing kanamycin, the resistant seedlings were transferred to soil and the
first flowering was used for the SEM analyzes.
Microscopy experiments
For SCI1-GFP protein subcellular localization, leaves were infiltrated as described by
Sawers et al. (2006), cut in small pieces, fixed on 3,7% formaldehyde, 50mM NaH2PO4 and
0,2% Triton X-100 for 30min, stained with 4,6'-diamidino-2-phenylindole (DAPI) for 15 min
25
(1.25µg/mL on PBS) on darkness, washed in PBS and mounted on slides with water, just
prior to analyses. For Biomolecular Fluorescence Complementation (BiFC) experiments, the
leaves of 6 weeks old tobacco seedlings were agroinfiltrated as described above, with the
combination of two different constructs (NtSCI1-hGFP with NtCYCA1;1 or NtCYCA2;21-398
or NtCYCA2;296-398). GFP and DAPI fluorescences were imaged under a Leica TCS SP2
confocal laser scanning microscope (Leica Microsystems). The individual stacks were
assembled with Adobe Photoshop CS (Adobe Systems, San Jose, CA). At least three
additional analyses without fixing material were performed to confirm that the formaldehyde
self-fluorescence was not interfering in our results. The images shown for the BiFC
experiments represent a series of stacks merged.
For light microscopy analysis, stigmas/styles and anthers were dissected from floral
buds at the relevant developmental stages, fixed and embedded in paraffin as described for in
situ hybridization. The embedded material was sliced into 8µm sections, mounted on slides
and stained with 0.05% toluidine blue in phosphate buffer at pH4.4 (O'Brien et al., 1964).
The pictures were taken using a Leica DM4500B microscope equipped with a Leica DFC320
camera. The tissues analysis was done using the Leica Q-Win Plus v3.4.0 software
(Switzerland) and the statistical analyses were performed using the SigmaStat.v3.5 software.
For scanning electron microscopy (SEM), the floral buds from wild-type and T1
transgenic plants, resulting from the controlled pollination between the T0 transgenic pistils
and SR-1 pollen grains, were collected from stages -4 to 1. For each stage, one anther of each
floral bud was collected for light microscopy analysis. The remaining floral bud (pistil and 4
anthers) was fixed for 48h in formaldehyde, acetic acid and 50% ethanol (1:1:18), dehydrated
in an ethanol series, subjected to critical-point drying, and coated with 30nm of gold.
Preparations were examined using a Jeol JSM-5200 scanning electron microscope. At least
two biological replicates of each stage were analyzed from the transgenic and wild-type
plants.
Yeast two-hybrid analysis
Plasmids encoding the baits (pDEST32) and preys (pDEST22) were transformed into
the yeast strain PJ69-4α (MATα; trp1-901, leu2-3,112, ura3-52, his3-200, gal4∆, gal80∆,
LYS2::GAL1-HIS3, GAL2-ADE2, met2::GAL7-lacZ) and PJ69-4a (MATa; trp1-901, leu23,112, ura3-52, his3-200, gal4∆, gal80∆, LYS2TGAL1-HIS3, GAL2-ADE2, met2TGAL7lacZ) by the LiAc method (Gietz et al., 1992). Transformed yeast cells were selected on
synthetic dextrose (SD) plates without Leu (pDEST 32) or without Trp (pDEST22),
26
respectively. Interactions between proteins were assayed by the mating method. All pDEST32
yeast cultures were inoculated in 200 µL SD without Trp in a 96-well microtiter plate
(Falcon), while one pDEST22 yeast culture was inoculated in 50 ml SD medium without Leu.
To upscale the yeast cultures, 20 µL of each culture grown for 2 days at 30°C were added to a
microtiter plate containing 125 µL of bacto-yeast extract (10 g/l), bacto-peptone (10 g/L, and
dextrose (20%) (YPD) medium, and again grown for 24 h at 30°C. The YPD medium was
replaced by SD medium without Leu and Trp. Diploid strains grown in a 96-well microtiter
plate (NUNC) for 2 days at 30°C were diluted until OD600 = 0.2 and then added to a 96-well
microtiter-plate (Falcon) containing either 190 µL SD medium without Leu and Trp, but with
His (as control) or SD medium without Leu, Trp, and His. The OD was measured after 2 days
of incubation at 30°C. Positive interactions were scored by dividing the OD600 of the SD
medium without Leu, Trp, and His by that of the SD medium without Leu and Trp. For the
LacZ test, 3 µL of the OD600 = 0.2 dilution was spotted on a Hybond-N+ membrane (GEHealthcare) on a YPD agar plate. After incubation of 2 days at 30°C, the membrane was snap
frozen in liquid nitrogen and placed on two Whatmann papers soaked in 6 mL of Z buffer
(16.1 g Na2HPO4.7H2O, 5.5 g NaH2PO4.H2O, 0.750g KCl, and 0.246g MgSO4.7H2O) with 11
µL β-mercaptoethanol and 100 µL 4% 5-bromo-4-chloro-3-indolyl-β-d-galactopyranoside.
After an incubation of 6 h at 37°C, colonies were scored for blue staining.
Accession Numbers
The SCI1-ORF, TOBSH1-002E06, TOB068F10, NtARF8, NtIAA13 and NtIAA19
cDNA sequences have been deposited at GenBank under accession numbers GQ272329,
GQ272330, GQ272331, GQ272332, GQ272334, GQ272333. Sequences mentioned in this
article can be found in the GenBank/EMBL data libraries under the following accession
numbers: X92966 - NtCYCA1;1; D50736 - NtCYCA2;2; AC212309 – SlSCI1 from tomato;
AC233353 – StSCI1 from potato; AM424297, clone VV78X084703.3 – VvSCI1 from
grapevine; AK252156, clone FLbaf149e13 – HvSCI1 from barley; NM_001052562
(Os02g0170200) – OsSCI1 from rice; EU958997, clone 208741 – ZmSCI1 from corn;
NM_106571 (At1g79200) – AtSCI1 from Arabidopsis; CAB85553 - SIM (At5g04470);
BAC42937 - SMR1 (At3g10525); AAF18255 - SMR2 (At1g08180); CAB85979 - SMR3
(At5g02420); CAD56868 – N. tabacum NtaKIS2; CAC82732 – N. sylvestris NsKIS1;
CAC82731 – N. tomentosiformis NtKIS1a; CAC82734 – N. tomentosiformis NtKIS1b;
NP_179924 – A. thaliana AtKRP1; NP_190632 – A. thaliana AtKRP2.
27
Supplemental Data
The following materials are available in the online version of this article.
Supplemental Figure 1 - Sequences of the SCI1 cDNA clones and the deduced amino acid
sequence showing the functional domains identified by in silico analysis.
Supplemental Figure 2 - Amino acid sequence alignment of the SCI1 protein and the
putative homologous sequences found in other plant species.
Supplemental Figure 3 - Analysis of anther morphological markers from floral buds at very
early stages of flower development.
Supplemental Figure 4 - Sequence analysis between SCI1 and the most closely related
classes of cell cycle regulators.
Supplemental Table 1 - Stigma/style tissues analyses in transgenic and wild-type plants
(original raw data)
Supplemental Table 2 - Pairwise 2-hybrid interactions between Arabidopsis SCI1, and CDK
and cyclins.
Supplemental Table 3 - Pairwise 2-hybrid interactions between Arabidopsis KRP2, and
CDK and cyclins.
Supplemental Table 4 - Amino acid sequences belonging to each ELM (Eukaryotic Linear
Motif) interacting domain identified in CDK inhibitors, described in N. tabacum and other
plants, and in the NtSCI1 protein.
Acknowledgements
The authors are grateful to Prof. Dr. Maria-Cristina S Pranchevicius (FM-Universidade
Federal do Tocantins), Prof. Dr. Adriana S Hemerly (Universidade Federal do Rio de
Janeiro) and Prof. Dr. Eric Lam (Rutgers University, USA) for stimulating discussions and
suggestions; to Prof. Dr Wagner F dos Santos from FFCLRP-Universidade de São Paulo
(USP) and Marina F B Costa for their contributions in the optical microscopy; to José
Augusto Maulin and Maria Dolores S Ferreira from “Departamento de Biologia Celular e
Molecular de Bioagentes Patogênicos” of the FMRP-USP and to Rodrigo Silva from
“Departamento de Química” of the FFCLRP-USP for their technical services on electron
microscopy; to Dr. Lenaldo B Rocha from FMRP-USP and Luis A A Toledo-Filho for their
contribution in the confocal microscopy; to Hilde Van Den Daele for the yeast two-hybrid
analysis, and to CEMEQ for their help in statistical analysis. We would like to thank Gerard
28
M. van der Weerden and the Botanical Garden of the University of Nijmegen (Netherlands)
for providing us Nicotiana seeds and the Flanders Interuniversity Institute for Biotechnology
(VIB - Belgium) for providing us the Gateway vectors for Agrobacterium-mediated
transformation. This work was supported by grants from Fundação de Amparo à Pesquisa no
Estado de São Paulo - Brazil (FAPESP no. 06/54431-9) and from Conselho Nacional de
Desenvolvimento Científico e Tecnológico (CNPq) - Brazil. HC DePaoli was supported by a
fellowship from CAPES/CNPq; AC Quiapim, MS Brito and EJ Strini were supported by
fellowships from CAPES; GH Goldman and MHS Goldman are indebted to CNPq for their
research fellowships.
Figure Legends
Figure 1 – SCI1 subcellular localization and gene expression analyses.
(A-H) SCI1-GFP is nuclear localized in planta (tobacco leaves), and is clearly visible in the
interchromatic region (A, C, E and G) and in the nucleolus (B, D, F and H). (A, B):
35Spro:SCI1-GFP expression; (C, D): DAPI staining; (E, F): superposition of upper and
middle upper panels plus the chloroplast (red) fluorescence (merge); (G, H): superposition of
10 (G) and 12 (H) stacks of the GFP, DAPI and chloroplast signals through a cell slice (“3D”
merge). Images are presented as stacks of neighboring sections. (I, J, K, L): representative
image in grayscale of the panels E, G, F and H, respectively. Scale bars are 4μm in all panels.
cl, chloroplast; is, interchromatin space; n, nucleus; nu, nucleolus. (M) SCI1 expression level
in the different organs of N. tabacum (SR-1) as determined by qRT-PCR. Expression in
arbitrary units (A.U.) corresponds to the ratio of the copy number of cDNAs of the SCI1 gene
on the copy number of the constitutive control β-actin gene in a defined amount of total RNA
of each sample (see Methods). (N) Expression level in stigmas/styles during the 12 stages of
tobacco flower development (Koltunow et al., 1990) (A logarithmic scale was used to enable
the detection of the lower expression levels). (O) In situ hybridization on longitudinal
sections of stage 4 stigmas/styles, using a sense probe prepared from clone TOBSH1-002E06
(negative control) and (P) Localization of SCI1 mRNA as revealed by the TOBSH1-002E06
antisense probe. The magnification factor is 40x; arrow cap, papillar cell; p, parenchyma;
ssz, stigmatic secretory zone; stt, stylar transmitting tissue; vb, vascular bundle.
Figure 2 – Expression level of the SCI1 gene in stage 4 stigmas/styles of SCI1Ri and SCI1OE
transgenic plants, determined by qRT-PCR.
29
Expression level in arbitrary units (A.U.) corresponds to the ratio of the copy number of
cDNAs of the SCI1 gene in each transgenic plant (normalized by the internal control β-actin
gene) on the copy number of cDNAs of the SCI1 gene in wild-type plant SR-1 (black bars)
(equally normalized by the β-actin gene), in a defined amount of total RNA (Methods). The
dashed bars represent the transgenic plants that showed altered phenotypes with elongated
styles and larger stigma surface. The chess-painted bars correspond to plants with a smaller
stigma surface, in contrast to empty bars that represent the wild-type-like phenotype. (A)
Expression level in percentage (A.U. x100) for each RNAi transgenic plant obtained. (B)
Expression level in A.U. for each overexpression (O.E.) transgenic plant obtained (A
logarithmic scale was used to allow the comparison of the very different expression levels).
Figure 3 – Analyses of the SCI1 transgenic plant phenotypes.
(A) Flowers of three independent RNAi transformants showing the increased style length at
stages 12, 10 and 9 (from left to right) of flower development. (*) Upper view of wild-type
plant (W.T. SR-1), at stage 10 of flower development, showing the stigma/style completely
enclosed by the corolla. Scale bars represent 10mm. The percentages correspond to the
transcript level of the SCI1 gene in comparison to the wild-type level considered as 100%.
(B) Upper view of the stigma at stage 12 of flower development showing the decreased size
in overexpression plants (SCI1OE 11.1; Ba) and the increased size in RNAi plants
(SCI1Ri14.2; Bc), compared to the wild-type plant (SR-1; Bb). Expression level is in arbitrary
units as described in Figure 2. Scale bar represent 1mm for all images. (C) Longitudinal
section scheme (Ca) representing the region where the SSZ cell layers were counted (≡) (3D;
Supplemental Table 1). The SSZ/STT intersection was defined by a straight line drawn at the
abaxial curves of the stigmatic umbrella-like structure (green dotted line in 3Ca). In light gray
is shown the SSZ area evaluated and measured in the longitudinal sections (Supplemental
Table 1; longitudinal plane 3Ec1). The square (□) indicates the region in which the SSZ cell
numbers were counted in an area of 20,000μm2. The stylar transmitting tissue is represented
in dark gray and its thickness was measured at 400µm below the SSZ/STT intersection (3Ca
red line). The rectangle represents the region shown in 3Ea/b for each plant and in which the
STT thickness and parenchyma cell number were measured. Longitudinal sections of stage
11 stigmas/styles stained with 0.05% toluidine blue are shown at (Cb) for the overexpression
plant SCI1OE 3.1.1; (Cc) wild-type SR-1 and (Cd) RNAi plant SCI1Ri 14.2, providing
evidence for the different areas occupied by each SSZ. Scale bar represents 500μm for all
photos. The blue and black arrows indicate, respectively, the deeper carpel junction in
30
SCI1OE 3.1.1 plant and the vascular bundle. (D) Longitudinal sections of stage 11
stigmas/styles at a higher magnification (200x), demonstrating the altered number of cells in
the stigmatic secretory zone (E) Histological sections of stage 11 styles. Longitudinal
sections of the SCI1OE3.1.1, SR-1 and SCI1Ri14.2 plants, showing the thickness differences
in the stylar transmitting tissue (Ea) and the parenchyma tissue (Eb), taken on the
longitudinal plane indicated by Ec2. Note the differences in the STT thickness of each plant,
indicating the alteration on the cell number in this tissue. (Ec) Transversal section of the
wild-type SR-1 style showing the two different longitudinal planes where the stigmatic
secretory zone area, cell layers and cell number as well as the transmitting tissue thickness
were determined (Ec1; Supplemental Table 1) and where the style thickness and parenchyma
cell number were determined (Ec2; Supplemental Table 1). The longitudinal plane 2
corresponds to the first cell layer behind the vascular bundle. Scale bar represents 50μm in
Ea/Eb and 500μm in Ec. p, parenchyma; stt, stylar transmitting tissue; ssz, stigmatic
secretory zone; vb, vascular bundle; black arrow, vascular bundle.
Figure 4 –Stigma/style tissue analyses in transgenic and wild-type plants.
Longitudinal sections of stigmas/styles from each transgenic and wild-type plants were
analyzed as described in Figure 3Ca for SSZ cell layers, SSZ area, SSZ cell number (in a
20,000 000μm2 area), style thickness (μm), STT thickness (μm) and parenchyma thickness
(n=3 for each plant shown). The above values represent percentages of the wild-type values,
considered as 100%. The raw data can be found on Supplemental Table 1.
* Significantly different from the wild-type by Student’s t-test (p<0.05).
Figure 5 – Very early upper pistil development and differentiation in the wild-type plant
(SR-1).
(A) Scanning electron micrographs of pistils at very early stages of flower development.
Flower buds at the indicated stages (stage -4 to stage 1, respectively Aa to Ae) had the sepals
and petals removed to allow visualization of the pistils. Stigmatic secretory zone formation
and development (Af to Aj), together with epidermis differentiation in papillar cells (Ak to
Ao). Scale bars indicated in each image are 500µm (Aa to Ae), 200µm (Af to Aj) and 50µm
(Ak to Ao). (B) A scheme to illustrate the upper pistil development and stigmatic secretory
zone formation, from stages -4 to 1 (Ba to Be, respectively) of flower development. yellow,
stigmatic tissue; blue, carpel epidermis. Scale bar corresponds to 500µm for all images. (C)
Expression level of the SCI1 gene determined by qRT-PCR in wild-type stigmas/styles from
31
stage -4 to 1 of flower development. Expression in arbitrary units (A.U.) as described in
Figure 1M.
Figure 6 – Scanning electron microscopy (SEM) of the transgenic (T1) and wild-type SR-1
plants.
(A) The early style elongation of the SCI1Ri14.2 RNAi plant (Aa, Ab and Ac), compared to
the wild-type plant (Ad, Ae and Af). Observe that at stage 1 (Ac), the SCI1Ri14.2 stigma is
already positioned above anthers. The style elongation is delayed in SCI1OE3.1.1
overexpression plant (Ag, Ah and Ai). At stage -3 of flower development, it is possible to
observe that the ectopic expression of SCI1 gene in anthers has delayed its development. This
organ has the same size and morphological markers (Koltunow et al., 1990) in both
SCI1Ri14.2 RNAi and the wild-type plants. (B) The premature enlargement of the upper pistil
part of the SCI1Ri14.2 transgenic RNAi plant (Ba, Bb and Bc), compared to the wild-type
plant (Bd, Be and Bf), in contrast to the developmental delay observed for the SCI1OE3.1.1
overexpression plant (Bg, Bh and Bi), at stages -4 (Ba, Bd and Bg), -3 (Bb, Be and Bh) and 2 (Bc, Bf and Bi) of flower development. (C) The upper pistil growth and differentiation of
the papillae cells that initiates in SCI1Ri14.2 transgenic RNAi plant and in wild-type plant
through stage -4 (Ca and Cd, respectively) and develops through stages -3 and -2 (Cb;Cc
and Ce;Cf, respectively) onward, in contrast to the SCI1OE3.1.1 overexpression plant that
initiates the papillae development through stage -3 (Ch) and develops through stage -2 (Ci)
onward. The scale bars are indicated in each image and represents 500μm (Aa to Ai), 100μm
(Ba to Bi), and 50µm (Ca to Ci).
Figure 7 – Interaction of SCI1 with A-type cyclins.
(A) Interactions were tested using the yeast 2-hybrid system. Baits (GUS, AtCYCA1;1,
AtCYCA2;2, AtCYCD5;1, AtCYCD6;1, AtCDKA;1, and AtCDKB1;1) and prey (AtSCI1,
and AtKRP2) constructs were combined by yeast mating. The GUS bait serves as negative
control. Interaction between prey and bait was assayed by growth on medium lacking
histidine (-His) and by β-galactosidase activity (Lac-Z). The asterisk indicates the use of an
N-terminal deleted version of AtCYC2;2 (the first 64 amino-acids deleted), removing the
destruction box to stabilize the protein. (B) Interactions were tested using the BiFC
technique. Leaf epidermal cell transformed with the NtSCI1 fused to the hGFP (amino acids
1 to 149) and the NtCYCA1;1 fused to the tGFP (amino acids 150 to 238). Upper and middle
32
panel - Restored GFP fluorescence, which is nuclear localized (different magnifications are
shown); bottom panel – middle panel merged with the bright field. Scale bars are 10µm.
Figure 8 – Transcript levels for each gene in stage 4 stigmas/styles of wild-type SR-1 (black
bars), SCI1Ri (dashed bars) and SCI1OE (chess-painted bars) transgenic plants, determined by
qRT-PCR.
(A) SCI1 gene, (B) NtARF8 gene, (C) NtAux/IAA13 gene and (D) NtAux/IAA19 gene.
Arbitrary units (A.U.) in A as described in Figure 2, except for the use of the polyubiquitin
gene as internal control (see Methods). The left axis shows the SCI1Ri plant values and the
right axis shows the SCI1OE plant values. Arbitrary units (A.U.) in B-D as described in Figure
1M, except for the use of the polyubiquitin gene as internal control. The statistically
significant differences between each plant vs. the wild-type by Student’s T-test are indicated
(*p<0.05; **p≤0.001).
Figure 9 - A proposed model for SCI1 action in upper pistil development. After the two
carpels are formed and fused, cells positioned at the upper pistil start dividing at the carpel
junction site. When an appropriate number of cells is reached, they start differentiation in
stigmatic secretory zone (SSZ) and stylar transmitting tissue (STT). In SR-1, the SCI1 level
would regulate cell proliferation and determine the correct developmental stage in which the
differentiation occurs. In RNAi plants (SCI1Ri), the reduced SCI1 level would allow cells to
start proliferating earlier or quicker, reaching the threshold for differentiation in an earlier
flower developmental stage. As these cells would stop dividing at the same developmental
moment as in SR-1, probably under the control of a yet unknown factor, they would
accumulate a greater number of cells. In overexpression plants (SCI1OE), the very high SCI1
level would reduce or delay cells divisions, which consequently would differentiate later.
Again, the unknown factor, that is independent on SCI1, would regulate the moment in which
these cells would stop proliferation, resulting in a reduced number of stigma/style specialized
cells (SSZ and STT).
33
Table 1: Primers used in the gene expression analyses by real time RT-PCR.
Gene Name
Forward (5’- 3’)
Reverse (5’- 3’)
β -actin
agatgatgctccaagggctgtgtt
ccatgccaaccatcacaccagt
polyubiquitin
tccgacacaatcgacaacgtaa
gacgtactgttgtaatcggctagggtacgtc
SCI1
gccacaaacataaggataaatcg
cagccacgtagcaaattcattgtt
NtARF8
ggcaggattgcagtgaaagctaca
ggtctgattctcttcttcccaagg
NtAux/IAA13
gctcacaactacctctgaagatcgca
acctcacgggtaacaaactggtca
NtAux/IAA19
gctgcgagctttaagcacattatcc
ggacgagtggcctctcatataatc
The primer sequences were determined based on the sequence of the following TOBEST
clusters: β-actin (TOBC110E12), polyubiquitin (TOBC018H09), NtARF8 (TOB044H02),
NtAux/IAA13 (TOB100E10) and NtAux/IAA19 (TOB056F02).
34
References
Altschul, S.F., Madden, T.L., Schaffer, A.A., Zhang, J., Zhang, Z., Miller, W., Lipman,
D.J. (1997). Gapped BLAST and PSI-BLAST: A new generation of protein database search
programs. Nucl. Acids. Res. 25: 3389–3402.
Beemster, G.T., Fiorani, F., Inzé, D. (2003). Cell cycle: the key to plant growth control?
Trends Plant Sci. 8: 154-158.
Benjamins, R., Scheres, B. (2008). Auxin: the looping star in plant development. Annu.
Rev. Plant Biol. 59: 443-465.
Boudolf V, Barrôco R, Engler Jde A, Verkest A, Beeckman T, Naudts M, Inzé D, De
Veylder L. (2004). B1-type cyclin-dependent kinases are essential for the formation of
stomatal complexes in Arabidopsis thaliana. Plant Cell. 16:945-955.
Brasileiro, A.C.M., Carneiro, V.T.C. (1998). Manual de Transformação Genética de
Plantas. Editora Embrapa Produção de Informação. Brasília.
Brown, J.W., Shaw, P.J., Shaw, P., Marshall, D.F. (2005). Arabidopsis nucleolar protein
database (AtNoPDB). Nucleic Acids Res. 1: D633-636.
Cheng Y, Dai X, Zhao Y. (2006). Auxin biosynthesis by the YUCCA flavin
monooxygenases controls the formation of floral organs and vascular tissues in Arabidopsis.
Genes Dev. 20: 1790-1799.
Churchman ML, Brown ML, Kato N, Kirik V, Hülskamp M, Inzé D, De Veylder L,
Walker JD, Zheng Z, Oppenheimer DG, Gwin T, Churchman J, Larkin JC. (2006)
SIAMESE, a plant-specific cell cycle regulator, controls endoreplication onset in Arabidopsis
thaliana. Plant Cell 18: 3145-3157.
Cockcroft, C.E., den Boer, B.G., Healy, J.M., Murray, J.A. (2000). Cyclin D control of
growth rate in plants. Nature 405: 575-579.
Cresti, M., Went, J.L. van, Pacini, E., Willemse, M.T.M. (1976). Ultrastructure of
transmitting tissue of Lycopersicon peruvianum style: development and histochemistry.
Planta 132: 305-312.
Dean C, Van der Elzen P, Tamaki S, Dunsmuir P, Bedbrook J (1985) Differential
expression of the eight genes of the petunia ribulose bisphosphate carboxylase small subunit
multi-gene family. EMBO J 4: 3055-3061.
de Graaf, B.H.J., Derksen, J.W.M., Mariani, C. (2001). Pollen and pistil in the progamic
phase. Sex. Plant Reprod. 14: 41-55.
35
del Pozo, J.C., Diaz-Trivino, S., Cisneros, N., and Gutierrez, C. (2006). The balance
between cell division and endoreplication depends on E2FC-DPB, transcription factors
regulated by the ubiquitin-SCFSKP2A pathway. Plant Cell. 18: 2224–2235.
De Veylder, L., Beeckman, T., Beemster, G.T., Krols, L., Terras, F., Landrieu, I., van
der Schueren, E., Maes, S., Naudts, M., Inzé, D. (2001). Functional analysis of cyclindependent kinase inhibitors of Arabidopsis. Plant Cell 13: 1653-1668.
De Veylder L, Beeckman T, Inzé D. (2007). The ins and outs of the plant cell cycle. Nat
Rev Mol Cell Biol. 8: 655-665.
Disch, S., Anastasiou, E., Sharma, V.K., Laux, T., Fletcher, J.C., Lenhard, M. (2006).
The E3 ubiquitin ligase BIG BROTHER controls Arabidopsis organ size in a dosagedependent manner. Curr. Biol. 16: 272–279.
Echeverría, O., Vázquez-Nin, G., Juárez-Chavero, S., Moreno Díaz de la Espina, S.
(2007). Firing of transcription and compartmentalization of splicing factors in tomato radicle
nuclei during germination. Biol. Cell. 99: 519-530.
Eloy NB, Coppens F, Beemster GT, Hemerly AS, Ferreira PC. (2006). The Arabidopsis
anaphase promoting complex (APC): regulation through subunit availability in plant tissues.
Cell Cycle 5: 1957-1965.
Endo M, Matsubara H, Kokubun T, Masuko H, Takahata Y, Tsuchiya T, Fukuda H,
Demura T, Watanabe M. (2002). The advantages of cDNA microarray as an effective tool
for identification of reproductive organ-specific genes in a model legume, Lotus japonicus.
FEBS Lett 514: 229-237.
Gietz D, St Jean A, Woods RA, Schiestl RH. (1992) Improved method for high efficiency
transformation of intact yeast cells. Nucleic Acids Res.20:1425.
Goldberg, R.B. (1988). Plants: Novel developmental processes. Science 240: 1460–1467.
Goldman, M.H.S., Pezzotti, M., Seurinck, J., Mariani, C. (1992). Developmental
expression of tobacco pistil-specific genes encoding novel extensin-like proteins. Plant Cell
4: 1041–1051.
Goldman, M.H., Goldberg, R.B., Mariani, C. (1994). Female sterile tobacco plants are
produced by stigma-specific cell ablation. EMBO J. 13: 2976-84.
Hase, Y., Tanaka, A., Baba, T., Watanabe, H. (2000). FRL1 is required for petal and sepal
development in Arabidopsis. Plant J. 24: 21-32.
Hemerly, A., Engler, J. A., Bergounioux, C., Van Montagu, M., Engler, G., Inzé, D.,
Ferreira, P. (1995). Dominant negative mutants of the Cdc2 kinase uncouple cell division
from iterative plant development. EMBO J. 14: 3925-3936.
36
Himanen, K., Boucheron, E., Vanneste, S., de Almeida Engler, J., Inzé, D., Beeckman,
T. (2002). Auxin-mediated cell cycle activation during early lateral root initiation. Plant Cell
14: 2339-2351.
Ingram, G.C., Goodrich, J., Wilkinson, M.D., Simon, R., Haughn, G.W., Coen, E.S.
(1995). Parallels between UNUSUAL FLORAL ORGANS and FIMBRIATA, genes
controlling flower development in Arabidopsis and Antirrhinum. Plant Cell 7: 1501-1510.
Inzé, D., Veylder, L.D. (2006). Cell Cycle Regulation in Plant Development. Annu. Rev.
Genet. 40: 77-105.
Jackson, D. (1991). In- situ hybridization in plants. Molecular Plant Pathology: A Practical
Approach. Eds. Bowles, D.J., S.J. Gurr and M. McPherson. Oxford University Press.
Jain, M., Kaur, N., Garg, R., Thakur, J.K., Tyagi, A.K., Khurana, J.P. (2006). Structure
and expression analysis of early auxin-responsive Aux/IAA gene family in rice (Oryza
sativa). Funct. Integr. Genomics. 6: 47-59.
Jasinski, S., Riou-Khamlichi, C., Roche, O., Perennes, C., Bergounioux, C., Glab, N.
(2002a). The CDK inhibitor NtKIS1a is involved in plant development, endoreduplication
and restores normal development of cyclin D3; 1-overexpressing plants. J. Cell. Sci. 115:
973-982.
Jasinski, S., Perennes, C., Bergounioux, C., Glab, N. (2002b). Comparative molecular and
functional analyses of the tobacco cyclin-dependent kinase inhibitor NtKIS1a and its spliced
variant NtKIS1b. Plant Physiol. 130: 1871-1882.
Jasinski, S., Leite, C.S., Domenichini, S., Stevens, R., Raynaud, C., Perennes, C.,
Bergounioux, C., Glab, N. (2003). NtKIS2, a novel tobacco cyclin-dependent kinase
inhibitor is differentially expressed during the cell cycle and plant development. Plant
Physiol. Biochem. 41: 667-676.
Joubès, J., Chevalier, C., Dudits, D., Heberle-Bors, E., Inzé, D., Umeda, M., Renaudi,
J.P. (2000). CDK-related protein kinases in plants. Plant Mol. Biol. 43: 607-620.
Jurado, S., Díaz-Triviño, S., Abraham, Z., Manzano, C., Gutierrez, C., del Pozo, C.
(2008). SKP2A, an F-box protein that regulates cell division, is degraded via the ubiquitin
pathway. Plant J. 53: 828-841.
Kandasamy, M.K., Kristen, U. (1987). Developmental aspects of ultrastructure,
histochemistry and receptivity of the stigma of Nicotiana sylvestris. Ann. Bot. 60: 427-437.
Karimi, M., Inzé, D., Depicker, A. (2002) GATEWAY vectors for Agrobacterium mediated
plant transformation. Trends Plant Sci 7: 193–195.
37
Koltunow, A.M., Truettner, J., Cox, K.H., Wallroth, M., Goldberg, R.B. (1990).
Different Temporal and Spatial Gene Expression Patterns Occur during Anther Development.
Plant Cell 2: 1201-1224.
Kondorosi, E., Roudier, F., Gendreau, E. (2000). Plant cell-size control: growing by
ploidy? Curr. Opin. Plant Biol. 3: 488-492.
Krizek, B.A. (2009). Making bigger plants: key regulators of final organ size. Curr Opin
Plant Biol. 12: 17-22.
Larson-Rabin, Z., Li, Z., Masson, P.H., Day, C.D. (2009). FZR2/CCS52A1 Expression Is a
Determinant of Endoreduplication and Cell Expansion In Arabidopsis. Plant Physiol. 149:
874-884.
Le Foll, M., Blanchet S., Millan, L., Mathieu, C., Bergounioux, C., Glab, N. (2008). The
plant CDK inhibitor NtKIS1a interferes with dedifferentiation, is specifically down regulated
during development and interacts with a JAB1 homolog. Plant Science. 175:513-523.
Li M, Xu W, Yang W, Kong Z, Xue Y. (2007). Genome-wide gene expression profiling
reveals conserved and novel molecular functions of the stigma in rice (Oryza sativa L.). Plant
Physiol 144: 1797-1812.
Li Y, Zheng L, Corke F, Smith C, Bevan MW. (2008). Control of final seed and organ size
by the DA1 gene family in Arabidopsis thaliana. Genes Dev. 22:1331-1336.
Masuda HP, Cabral LM, De Veylder L, Tanurdzic M, de Almeida Engler J, Geelen D,
Inzé D, Martienssen RA, Ferreira PC, Hemerly AS. (2008). ABAP1 is a novel plant
Armadillo BTB protein involved in DNA replication and transcription. EMBO J. 27: 27462756.
McClure, B., Mou. B., Canevascini, S., Bernatzky, R. (1999). A small asparagine-rich
protein required for S-allele-specific pollen rejection in Nicotiana. Proc. Natl. Acad. Sci. 96:
13548-13553.
Menges M, de Jager SM, Gruissem W, Murray JA.(2005). Global analysis of the core cell
cycle regulators of Arabidopsis identifies novel genes, reveals multiple and highly specific
profiles of expression and provides a coherent model for plant cell cycle control. Plant J.
41:546-566.
Mizukami, Y. (2001). A matter of size: developmental control of organ size in plants. Curr.
Opin. Plant Biol. 4: 533-539.
Mizukami, Y., Fischer, R.L. (2000). Plant organ size control: AINTEGUMENTA regulates
growth and cell numbers during organogenesis. Proc. Natl. Acad. Sci. 97: 942-947.
38
Murashige, T., and Skoog, F. (1962). A revised medium for rapid growth and bioassays
with tobacco tissue cultures. Physiol. Plant 15: 473-497.
Napoli, C., Lemieux, C., Jorgensen, R. (1990). Introduction of a chimeric chalcone
synthase gene into Petunia results in reversible co-suppression of homologous genes in trans.
Plant Cell. 2: 279-289.
Nemhauser, J.L., Feldman, L.J., Zambryski, P.C. (2000). Auxin and ETTIN in
Arabidopsis gynoecium morphogenesis. Development. 127: 3877-3888.
O'Brien, T.P., Feder, N., McCully, M.E. (1964). Polychromatic staining of plant cell walls
by toluidine blue. Protoplasma. 59: 368–373.
Peres A, Churchman ML, Hariharan S, Himanen K, Verkest A, Vandepoele K, Magyar
Z, Hatzfeld Y, Van Der Schueren E, Beemster GT, Frankard V, Larkin JC, Inzé D, De
Veylder L. (2007) Novel plant-specific cyclin-dependent kinase inhibitors induced by biotic
and abiotic stresses. J Biol Chem. 282:25588-25596.
Quiapim AC, Brito MS, Bernardes LA, Dasilva I, Malavazi I, DePaoli HC, MolfettaMachado JB, Giuliatti S, Goldman GH, Goldman MH. (2009). Analysis of the Nicotiana
tabacum stigma/style transcriptome reveals gene expression differences between wet and dry
stigma species. Plant Physiol. 149: 1211-1230.
Ramirez-Parra, E., Desvoyes, B., Gutierrez, C. (2005). Balance between cell division and
differentiation during plant development. Int. J. Dev. Biol. 49: 467-477.
Ren H, Santner A, del Pozo JC, Murray JA, Estelle M. (2008). Degradation of the cyclindependent kinase inhibitor KRP1 is regulated by two different ubiquitin E3 ligases. Plant J.
53:705-716
Sambrook J., Russell, D.W. (2001) Molecular Cloning. A Laboratory Manual. Third
Edition. Cold Spring Harbor Laboratory Press, New York.
Satina, S. (1944). Periclinal chimeras in Datuna in relation to development and structure of
the style and stigma of calyx and secretion. Am. J. Bot. 77: 1111-1124.
Sawers, R.J., Farmer, P.R., Moffett, P., Brutnell, T.P. (2006). In planta transient
expression as a system for genetic and biochemical analyses of chlorophyll biosynthesis.
Plant Methods 2:15.
Schruff MC, Spielman M, Tiwari S, Adams S, Fenby N, Scott RJ. (2006). The AUXIN
RESPONSE FACTOR 2 gene of Arabidopsis links auxin signalling, cell division, and the
size of seeds and other organs. Development. 133:251-261.
39
Semighini, C.P., Marins, M., Goldman, M.H., Goldman, G.H. (2002). Quantitative
analysis of the relative transcript levels of ABC transporter Atr genes in Aspergillus nidulans
by real-time reverse transcription-PCR assay. Appl. Environ. Microbiol. 68: 1351-1357.
Swanson, R., Clark, T., Preuss, D. (2005). Expression profiling of Arabidopsis stigma
tissue identifies stigma-specific genes. Sex. Plant Reprod. 18: 163-171.
Takeda, D.Y., Wohlschlegel, J.A., Dutta, A. (2001). A bipartite substrate recognition motif
for cyclin-dependent kinases. J. Biol. Chem. 276: 1993-1997.
Tatematsu, K., Kumagai, S., Muto, H., Sato, A., Watahiki, M.K., Harper, R.M., Liscum,
E., Yamamoto, KT. (2004). MASSUGU2 encodes aux/iaa19, an auxin-regulated protein that
functions together with the transcriptional activator nph4/arf7 to regulate differential growth
responses of hypocotyl and formation of lateral roots in Arabidopsis thaliana. Plant Cell 16:
379–393.
Taylor, C.B. (1997). Comprehending cosuppression. Plant Cell 9: 1245-1249.
Testillano, P.S., González-Melendi, P., Coronado, M.J., Seguí-Simarro, J.M., MorenoRisueño, M.A., Risueño, M.C. (2005). Differentiating plant cells switched to proliferation
remodel the functional organization of nuclear domains. Cytogenet. Genome Res. 109: 166174.
Tian, C.E., Muto, H., Higuchi, K., Matamura, T., Tatematsu, K., Koshiba, T.,
Yamamoto, K.T. (2004). Disruption and overexpression of auxin response factor 8 gene of
Arabidopsis affect hypocotyl elongation and root growth habit, indicating its possible
involvement in auxin homeostasis in light condition. Plant J. 40: 333-343.
Tsukaya, H. (2002). Interpretation of mutants in leaf morphology: genetic evidence for a
compensatory system in leaf morphogenesis that provides a new link between cell and
organismal theories. Int. Rev. Cytol. 217: 1-39.
Tung, C.W., Dwyer, K.G., Nasrallah, M.E., Nasrallah, J.B. (2005). Genome-wide
identification of genes expressed in Arabidopsis pistils specifically along the path of pollen
tube growth. Plant Physiol. 138: 977-89.
Verkest, A., Weinl, C., Inzé, D., De Veylder, L., Schnittger, A. (2005). Switching the cell
cycle. Kip-related proteins in plant cell cycle control. Plant Physiol. 139: 1099-1106.
Visintin, R., Amon, A. (2000). The nucleolus: the magician’s hat for cell cycle tricks.
Current Opinion in Cell Biology 12: 372-377.
Wang H., Wu H.M., Cheung A.Y. (1993). Development and pollination regulated
accumulation and glycosylation of a stylar transmitting tissue-specific proline-rich protein.
Plant Cell 5: 1639-1650.
40
Wang, H., Zhou, Y., Gilmer, S., Whitwill, S., Fowke, L.C. (2000). Expression of the plant
cyclin-dependent kinase inhibitor ICK1 affects cell division, plant growth and morphology.
Plant J. 24: 613-623.
Wang H., Zhou Y., Bird D.A., Fowke L.C. (2008). Functions, regulation and cellular
localization of plant cyclin-dependent kinase inhibitors. J Microsc. 231:234-246.
Weijers, D., Benkova, E., Jäger, K.E., Schlereth, A., Hamann, T., Kientz, M., Wilmoth,
J.C., Reed, J.W., Jürgens, G. (2005). Developmental specificity of auxin response by pairs
of ARF and Aux/IAA transcriptional regulators. EMBO J. 24: 1874-1885.
Wolters-Arts, W., Derksen, J., Kooijman, J.W., Mariani, C. (1996). Stigma development
in Nicotiana tabacum. Cell death in transgenic plants as a marker to follow cell fate at high
resolution. Sex. Plant Reprod. 9: 243-254.
Wu, M.F., Tian, Q., Reed, J.W. (2006). Arabidopsis microRNA167 controls patterns of
ARF6 and ARF8 expression, and regulates both female and male reproduction. Development.
133: 4211-4218.
Yamaguchi M, Kato H, Yoshida S, Yamamura S, Uchimiya H, Umeda M. (2003) Control
of in vitro organogenesis by cyclin-dependent kinase activities in plants. Proc Natl Acad Sci
U S A. 100:8019-8023.
Zimber, A., Nguyen, Q.D., Gespach, C. (2004). Nuclear bodies and compartments:
functional roles and cellular signalling in health and disease. Cell Signal. 16: 1085-1104.
Zhou, Y., Fowke, L., and Wang, H. (2002). Plant CDK inhibitors: studies of interactions
with cell cycle regulators in the yeast two-hybrid system and functional comparisons in
transgenic Arabidopsis plants. Plant Cell Rep. 20: 967-975.
Zhou Y, Niu H, Brandizzi F, Fowke LC, Wang H. (2006) Molecular control of nuclear and
subnuclear targeting of the plant CDK inhibitor ICK1 and ICK1-mediated nuclear transport
of CDKA. Plant Mol Biol. 62:261-278.
Zhou, Y., Wang, H., Gilmer, S., Whitwill, S., Fowke, L.C. (2003). Effects of co-expressing
the plant CDK inhibitor ICK1 and D-type cyclin genes on plant growth, cell size and ploidy
in Arabidopsis thaliana. Planta 216: 604-613.
41
Figure 1
cl
DAPI
C
D
DAPI
J
“3D” Merge
400
200
0
Organs
Stigmas/
Styles
Ovaries
n
600
Stamens
nu is
M 800
Petals
Merge
Sepals
I
Leaves
SCI1-GFP
Stems
B
Roots
SCI1-GFP
Expression Level (A.U.)
A
E
Merge
F
Merge
K
Merge
cl
cl
nu is
cl
G
nu
nu
n
n
n
“3D” Merge
H
“3D” Merge
L
“3D” Merge
Expression Level (A.U.)
N 1000
100
10
1
0,1
0,01
0,001
1 2
3 4 5 6 7 8 9 10 11 12
Stages
P
O
ssz
ssz
vb
p
p
vb
stt
stt
Figure 1- SCI1 subcellular localization and gene expression analyses
(A-L) SCI1-GFP is nuclear localized in planta (tobacco leaves), and is clearly visible in
the interchromatic region (A, C, E and G) and in the nucleolus (B, D, F and H). (A, B):
35S:SCI1-GFP expression; (C, D): DAPI staining; (E, F): superposition of upper and
middle upper panels plus the chloroplast (red) fluorescence (merge); (G, H):
superposition of 10 (G) and 12 (H) stacks of the GFP, DAPI and chloroplast signals
through a cell slice (“3D” merge). Images are presented as stacks of neighboring
sections. (I, J, K, L): representative image in grayscale of the painels E, G, F and H,
respectively. Scale bars are 4μm in all panels. cl, chloroplast; is, interchromatin space;
n, nucleus; nu, nucleolus. (M) SCI1 expression level in the different organs of N.
tabacum (SR-1) as determined by qRT-PCR. Expression in arbitrary units (A.U.)
corresponds to the ratio of the copy number of cDNAs of the SCI1 gene on the copy
number of the constitutive control β-actin gene in a defined amount of total RNA of each
sample (see Methods).
(N) Expression level in stigmas/styles during the 12 stages of tobacco flower
development (Koltunow et al., 1990) (A logarithmic scale was used to enable the
detection of the lower expression levels). (O) In situ hybridization on longitudinal
sections of stage 4 stigmas/styles, using a sense probe prepared from clone TOBSH1002E06 (negative control) and (P) Localization of SCI1 mRNA as revealed by the
TOBSH1-002E06 antisense probe. The magnification factor is 40x; arrow cap, papillar
cell; p, parenchyma; ssz, stigmatic secretory zone; stt, stylar transmitting tissue; vb,
vascular bundle.
Figure 2
B 10
80
70
60
50
40
30
20
10
0
102
Plants
10
SCI1OE 3.1.1
SCI1OE 6.4
SCI1OE 8.3
SCI1OE 10.1
SCI1OE 11.1
SCI1OE 14.9
SCI1OE 13.9
SCI1OE 12.9
0,1
W.T.
1
SCI1OE 1.1
Expression Level (A.U.)
SCI1Ri 20.9
SCI1Ri 7.1
SCI1Ri 8.4
SCI1Ri 19.9
SCI1Ri 16.1
SCI1Ri 18.9
SCI1Ri 6.4.1
SCI1Ri 13.3
SCI1Ri 9.1.3
SCI1Ri 14.2
SCI1Ri 11.3
SCI1Ri 4.4.1
W.T.
3
SCI1Ri 17.9
Expression Level (%)
A100
90
Plants
Figure 2 – Expression level of the SCI1 gene in stage 4
stigmas/styles of SCI1Ri and SCI1OE transgenic plants,
determined by qRT-PCR.
Expression level in arbitrary units (A.U.) corresponds to the ratio
of the copy number of cDNAs of the SCI1 gene in each
transgenic plant (normalized by the internal control β-actin gene)
on the copy number of cDNAs of the SCI1 gene in wild-type plant
SR-1 (W.T.) (black bars) (equally normalized by the β-actin
gene), in a defined amount of total RNA (Methods). The dashed
bars represent the transgenic plants that showed altered
phenotypes with elongated styles and larger stigma surface. The
chess-painted bars correspond to plants with a smaller stigma
surface, in contrast to empty bars that represent the wild-typelike phenotype. (A) Expression level in percentage (A.U. x100)
for each RNAi transgenic plant obtained. (B) Expression level in
A.U. for each overexpression (O.E.) transgenic plant obtained (A
logarithmic scale was used to allow the comparison of the very
different expression levels).
Figure 3
A
*
SCI1Ri 14.2 (8.4%)
W.T. (100%)
Expression Level (A.U.)
B
a
b
SCI1OE 11.1
10000
SCI1Ri 4.4.1 (8.0%)
SCI1Ri 16.1 (27.8%)
c
W.T.
SCI1Ri 14.2
1000
100
10
1
C
a
SCI1OE 3.1.1
b
D
c
W.T.
E
d
SCI1Ri 14.2
c
½
p
400µm
stt
a
ssz
ssz
p
p
2
b
ssz
SCI1OE
3.1.1
p
W.T.
vb
vb
SCI1OE
3.1.1
W.T.
SCI1Ri
14.2
1
SCI1Ri
14.2
Figure 3 – Analyses of the SCI1 transgenic plant phenotypes.
(A) Flowers of three independent RNAi transformants showing the increased style
length at stages 12, 10 and 9 (from left to right) of flower development. (*) Upper view of
wild-type plant (W.T.), at stage 10 of flower development, showing the stigma/style
completely enclosed by the corolla. Scale bars represent 10mm. The percentages
correspond to the transcript level of the SCI1 gene in comparison to the wild-type level
considered as 100%. (B) Upper view of the stigma at stage 12 of flower development
showing the decreased size in overexpression plants (SCI1OE 11.1; Ba) and the
increased size in RNAi plants (SCI1Ri14.2; Bc), compared to the wild-type plant (W.T.;
Bb). Expression level is in arbitrary units as described in Figure 2. Scale bar represent
1mm for all images. (C) Longitudinal section scheme (Ca) representing the region where
the SSZ cell layers were counted (≡) (3D; Figure 4). The SSZ/STT intersection was
defined by a straight line drawn at the abaxial curves of the stigmatic umbrella-like
structure (green dotted line in 3Ca). In light gray is shown the SSZ area evaluated and
measured in the longitudinal sections (Figure 4; longitudinal plane 3Ec1). The square
(□) indicates the region in which the SSZ cell numbers were counted in an area of
20,000µm2. The stylar transmitting tissue is represented in dark gray and its thickness
was measured at 400µm below the SSZ/STT intersection (3Ca green dotted line). The
rectangle represents the region shown in 3Ea/b for each plant and in which the STT
thickness and parenchyma cell number were measured. Longitudinal sections of stage
11 stigmas/styles stained with 0.05% toluidine blue are shown at (Cb) for the
overexpression plant SCI1OE 3.1.1; (Cc) wild-type SR-1 (W.T) and (Cd) RNAi plant
SCI1Ri 14.2, providing evidence for the different areas occupied by each SSZ. Scale bar
represents 500µm for all photos. The blue and black arrows indicate, respectively, the
deeper carpel junction in SCI1OE 3.1.1 plant and the vascular bundle. (D) Longitudinal
sections of stage 11 stigmas/styles at a higher magnification (200x), demonstrating the
altered number of cells in the stigmatic secretory zone (E) Histological sections of stage
11 styles. Longitudinal sections of the SCI1OE3.1.1, W.T. and SCI1Ri14.2 plants, showing
the thickness differences in the stylar transmitting tissue (Ea) and the parenchyma
tissue (Eb), taken on the longitudinal plane indicated by Ec2. Note the differences in the
STT thickness of each plant, indicating the alteration on the cell number in this tissue.
(Ec) Transversal section of the wild-type style showing the two different longitudinal
planes where the stigmatic secretory zone area, cell layers and cell number as well as
the transmitting tissue thickness were determined (Ec1; Figure 4) and where the style
thickness and parenchyma cell number were determined (Ec2; Figure 4). The
longitudinal plane 2 corresponds to the first cell layer behind the vascular bundle. Scale
bar represents 50µm in Ea/Eb and 500µm in Ec. p, parenchyma; stt, stylar transmitting
tissue; ssz, stigmatic secretory zone; vb, vascular bundle; black arrow, vascular
bundle.
Figure 4
160
140
120
100
80
60
40
20
0
*
*
*
*
*
*
*
*
SCI1OE
*
W.T.
SCI1Ri
Figure 4 –Stigma/style tissues analyses in
transgenic and wild-type plants.
Longitudinal sections of stigmas/styles
from each transgenic and wild-type plants
were analyzed as described in Figure 3Ca
for SSZ cell layers, SSZ area, SSZ cell
number (in a 20,000µm2 area), style
thickness (µm), STT thickness (µm) and
parenchyma thickness (n=3 for each plant
shown). The above values represent
percentages of the wild-type values,
considered as 100%. The raw data can be
found on Supplemental Table 1.
* Significantly different from the wild-type
by Student’s t-test (p<0.05).
Figure 5
Stage -4
b
f
g
k
B
a
l
Stage -4
Stage -2
Stage -3
c
h
m
b Stage -3 c Stage -2 d
Stage -1
Stage 1
d
e
i
j
n
Stage -1
o
e
C 1,6
Expression Level (A.U.)
A
a
1,2
0,8
0,4
0
-4
-3 -2
Stages
-1
1
Stage 1
500µm
Figure 5 – Very early upper pistil development and differentiation in the wild-type plant
(SR-1).
(A) Scanning electron micrographs of pistils at very early stages of flower development.
Flower buds at the indicated stages (stage -4
- to stage 1, respectively Aa to Ae) had the
sepals and petals removed to allow visualization of the pistils. Stigmatic secretory zone
formation and development (Af to Aj), together with epidermis differentiation in papillar
cells (Ak to Ao). Scale bars indicated in each image are 500µm (Aa to Ae), 200µm (Af
to Aj) and 50µm (Ak to Ao). (B) A scheme to illustrate the upper pistil development and
stigmatic secretory zone formation, from stages -4 to 1 (Ba to Be, respectively) of flower
development. yellow, stigmatic tissue; blue,, carpel epidermis. Scale bar corresponds to
500µm for all images. (C) Expression level of the SCI1 gene determined by qRT-PCR in
wild-type stigmas/styles from stage -4 to 1 of flower development. Expression in
arbitrary units (A.U.) as described in Figure 1M.
1
STAGE -3
b
STAGE -2
c
STAGE +1
SCI1Ri14.2
A
a
Figure 6
e
f
h
i
W.T.
d
SCI1OE3.1.1
g
STAGE -4
b
STAGE -3
c
STAGE -2
SCI1Ri14.2
B
a
e
f
h
i
W.T.
d
SCI1OE3.1.1
g
STAGE -4
b
STAGE -3
c
SCI1Ri14.2
C
a
e
f
h
i
W.T.
d
SCI1OE3.1.1
g
STAGE -2
Figure 6 – Scanning electron microscopy
(SEM) of the transgenic (T1) and wild-type
SR-1 plants (W.T.).
(A) The early style elongation of the
SCI1Ri14.2 RNAi plant (Aa, Ab and Ac),
compared to the wild-type
wild
plant (Ad, Ae
and Af). Observe that at stage 1 (Ac), the
SCI1Ri14.2 stigma is already positioned
above anthers. The style elongation is
delayed in SCI1OE3.1.1
3
overexpression
plant (Ag, Ah and Ai)). At stage -3 of flower
development, it is possible to observe that
the ectopic expression of SCI1 gene in
anthers has delayed its development. This
same
size
and
organ
has
the
morphological markers (Koltunow et al.,
1990) in both SCI1Ri14.2 RNAi and the
wild-type plants. (B) The premature
enlargement of the upper pistil part of the
SCI1Ri14.2 transgenic RNAi plant (Ba, Bb
and Bc), compared to the wild-type plant
(Bd, Be and Bf), in contrast to the
developmental delay observed for the
SCI1OE3.1.1 overexpression plant (Bg, Bh
and Bi), at stages -4 (Ba, Bd and Bg), -3
(Bb, Be and Bh) and -2 (Bc, Bf and Bi) of
flower development. (C) The upper pistil
growth and differentiation of the papillae
cells that initiates in SCI1
SCI Ri14.2 transgenic
RNAi plant and in wild-type
wild
plant through
stage -4 (Ca and Cd,
Cd respectively) and
develops through stages -3 and -2 (Cb;Cc
and Ce;Cf, respectively) onward, in
contrast to the SCI1OE3.1.1 overexpression
plant that initiates the papillae development
through stage -3 (Ch) and develops
through stage -2 (Ci
Ci) onward. The scale
bars are indicated in each image and
represents 500µm (Aa
Aa to Ai), 100µm (Ba
to Bi), and 50µm (Ca to Ci).
Figure 7
B
NtCYCA1;1-tGFP
NtSCI1-hGFP
A
GUS
AtCYCA1;1
AtCYCA2;2*
AtCYCD5;1
Restored GFP
AtCYCD6;1
AtCDKA;1
AtCDKB1;1
-His
Lac-Z
GFP/Bright field
Figure 7 – Interaction of SCI1 with A-type cyclins.
(A) Interactions were tested using the yeast 2-hybrid system.
Baits (GUS, AtCYCA1;1, AtCYCA2;2, AtCYCD5;1, AtCYCD6;1,
AtCDKA;1, and AtCDKB1;1) and prey (AtSCI1, and AtKRP2)
constructs were combined by yeast mating. The GUS bait
serves as negative control. Interaction between prey and bait
was assayed by growth on medium lacking histidine (-His) and
by β-galactosidase activity (Lac-Z). The asterisk indicates the
use of an N-terminal deleted version of AtCYC2;2 (the first 64
amino-acids deleted), removing the destruction box to stabilize
the protein. (B) Interactions were tested using the BiFC
technique. Leaf epidermal cell transformed with the NtSCI1
fused to the hGFP (amino acids 1 to 149) and the NtCYCA1;1
fused to the tGFP (amino acids 150 to 238). Upper and middle
panel - Restored GFP fluorescence, which is nuclear localized
(different magnifications are shown); bottom panel – middle
panel merged with the bright field. Scale bars are 10µm.
0,1
SCI1Ri 11.3
SCI1Ri 4.4.1
SCI1Ri 17.9
SCI1Ri 14.2
0
Plants
0,01
0,025
0,025
0,02
0,02
0,015
0,015
0,01
0,01
*
*
**
** **
*
0,06
0,05
0,04
0,005
0,01
0
0
0
Plants
*
0,02
0,005
Plants
*
0,03
**
** ** *
Plants
Figure 8 – Transcript levels for each indicated gene in stage 4 stigmas/styles of wildtype (W.T.) (black bars), SCI1Ri (dashed bars) and SCI1OE (chess-painted bars)
transgenic plants, determined by qRT-PCR.
(A) SCI1 gene, (B) NtARF8 gene, (C) NtAux/IAA13 gene and (D) NtAux/IAA19 gene.
Arbitrary units (A.U.) in A as described in Figure 2, except for the use of the
polyubiquitin gene as internal control (see Methods). The left axis shows the SCI1Ri
plant values and the right axis shows the SCI1OE plant values. Arbitrary units (A.U.) in
B-D as described in Figure 1M, except for the use of the polyubiquitin gene as internal
control. The statistically significant differences between each plant vs. the wild-type by
Student’s T-test are indicated (*p<0.05; **p≤0.001).
SCI1OE6.4
SCI1OE3.1.1
0,5
0,03
SCI1OE8.3
1
**
*
**
0,07
W.T.
SCI1OE11.1
1
0,035
**
0,035
0,03
**
SCI1Ri17.9
SCI1Ri11.3
SCI1Ri4.4.1
10
0,04
0,04
IAA19
SCI1Ri14.2
1,5
**
D0,08
IAA13
0,045
Expresion Level (A.U.)
100
0,045
C
SCI1Ri14.2
SCI1Ri17.9
SCI1Ri11.3
SCI1Ri4.4.1
W.T.
SCI1OE11.1
OE
SCI1 8.3
SCI1OE6.4
SCI1OE3.1.1
2
0,05
Expresion Level (A.U.)
1000
Figure 8
ARF8
SCI1Ri14.2
SCI1Ri17.9
SCI1Ri11.3
SCI1Ri4.4.1
W.T.
SCI1OE11.1
OE
SCI1 8.3
SCI1OE6.4
SCI1OE3.1.1
2,5
10000
Expresion Level (A.U.)
SCI1/Ubiquitin
W.T.
SCI1OE 11.1
OE
SCI1 8.3
SCI1OE 6.4
SCI1OE 3.1.1
SCI1Ri Expression Level (A.U.)
3
B
SCI1OE Expression Level (A.U.)
A
SCI1 level
HIGH
BASAL
W.T.
SCI1OE
MED
SCI1Ri
LOW
Cell number
Figure 9
SSZ/STT Formed
b
Differentiation
Threshold
a
Carpel Formed
Development
Figure 9 - A proposed model for SCI1
action in upper pistil development.
After the two carpels are formed and
fused, cells positioned at the upper pistil
start dividing at the carpel junction site.
When an appropriate number of cells is
reached, they start differentiation in
stigmatic secretory zone (SSZ) and stylar
transmitting tissue (STT). In W.T., the SCI1
level would regulate cell proliferation and
determine the correct developmental stage
in which the differentiation occurs. In RNAi
plants (SCI1Ri), the reduced SCI1 level
would allow cells to start proliferating
earlier or quicker, reaching the threshold
for differentiation in an earlier flower
developmental stage. As these cells would
stop dividing at the same developmental
moment as in W.T., probably under the
control of a yet unknown factor, they would
accumulate a greater number of cells. In
overexpression plants (SCI1OE), the very
high SCI1 level would reduce or delay cells
divisions, which consequently would
differentiate later. Again, the unknown
factor, that is independent on SCI1, would
regulate the moment in which these cells
would stop proliferation, resulting in a
reduced number of stigma/style specialized
cells (SSZ and STT).
A
TOBSH1-002E06
SCI1-ORF
TOB068F10
1
1
TCAGAGGTGC GAGTAAATGG GGAGCGATAA GAAGACGACG GAGGAGAAGA
---------- ------ATGG GGAGCGATAA GAAGACGCCG GAGGAGAAGA
---------- ---------- ---------- ---------- ----------
TOBSH1-002E06
SCI1-ORF
TOB068F10
51
35
GGAAGCATAA GAGAAATTCG CCTTCTTCTC CACGAGATGA AGTGAAAAGC
GGAAGCATAA GAGAAGTTCG CCTTCTTCTC CACGAGATGA AGTGAAAAGC
---------- ---------- ---------- ---------- ----------
TOBSH1-002E06
SCI1-ORF
TOB068F10
101
85
AAACGCCAGA ATATCAAAGG GGATGAAGAG CGAAGGAAAG AAAAG---GA
AAACGCCAGA ATATCAAAGG GGATGAAGAG CGAAGGAAAG AAAAGAAGGA
---------- ---------- ---------- ---------- ----------
TOBSH1-002E06
SCI1-ORF
TOB068F10
148
135
CAAATCCAAG AAGGAGAAGC ACAAATCCCA TAAATCCAAA TGTCATTCTA
CAAATCCAAG AAGGAGAAGC ACAAATCCCA T--------- ------TCTA
---------- ---------- ---------- ---------- ----------
TOBSH1-002E06
SCI1-ORF
TOB068F10
198
170
1
GTGAAGAGAA GAAGTCAGGG GAAAAGCACA AAACCAAGAG CCACAAACAT
GTGAAGAGAA GAAGTCAGGG GAAAAGCACA AAACCAAGAG CCACAAACAT
---------- ---------- ---------- ---------- ----------
TOBSH1-002E06
SCI1-ORF
TOB068F10
248
220
1
AAGGATAAAT CGAAAAACAA GTTTGAAGAG TTATCAAAAG ATGATTATTT
AAGGATAAAT CGAAAAACAA GTTTGAAGAG TTATCAAAAG ATGATTATTT
---------- CGAAAAACAA GTTTGAAGAG TTATCAAAAG ATGATTATTT
TOBSH1-002E06
SCI1-ORF
TOB068F10
298
270
41
CTCTAAGAAC AATGAATTTG CTTCTTGGCT GAAAGATAAG AAGAATTTGT
CTCTAAGAAC AATGAATTTG CTACTTGGCT GAAAGATAAG AAGAATTTGT
CTCTAAGAAC AATGAATTTG CTACTTGGCT GAAAGATAAG AAGAATTTGT
TOBSH1-002E06
SCI1-ORF
TOB068F10
348
320
91
TCTTCTCAGA TCTTTCGTCT GAGACTGCAC GCAATTTATT CTCTGACTTT
TCTTCTCAGA TCTTTCGTCT GAGACTGCAC GCGATTTATT CTCTGACTTT
TCTTCTCAGA TCTTTCGTCT GAGACTGCAC GCGATTTATT CTCTGACTTT
TOBSH1-002E06
SCI1-ORF
TOB068F10
398
370
141
GTCATACAAT GGAACAAGGG AAAGCTTGAC AGCCAATACT ATGAGGGAAT
GTCATACAAT GGAACAAAGG AAAGCTTGAC AGCCAATACT ATGAGGGAAT
GTCATACAAT GGAACAAAGG AAAGCTTGAC AGCCAATACT ATGAGGGAAT
TOBSH1-002E06
SCI1-ORF
TOB068F10
448
420
191
TGCAACCGG- ---------- ---------- ---------- ---------TGCAACCGGG CCTCGGTCAT CCCACGCTTG GAATATCAAA AAGTAA---TGCAACCGGG CCTCGGTCAT CCCACGCTTG GAATATCAAA AAGTAATAGA
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
241
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------ATTCCATGTT TCTTCTCTTT AAGGAGCAGT TGTATTGTCT TTTTTCCAGT
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
291
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------TGTCCGATGG TTATGTCTTA GGCTTCTGTA TTTTTGTGTT TTCTTTTTTC
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
341
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------AGCAGAACAT GCTTAGTCTT ATGAAGTTAC TTTCTTGGAT AATGGTAAAA
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
391
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------TCACCATCTC TGTTGTAAGC TATGTTGTCC GGACTCTCCA AAATCCTGCC
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
441
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------GCATCCCCGT CGGACCCTCC AAAAATGCAC ATACTTTTAG AGGATCCGAC
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
491
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------ACGCACCTGG TAACATTTTT AGTGAGTCTG GGCAACATAG GTTGTAAGAG
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
541
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------TTCTATGCTA GTCCTACCAC GTGACTGCAT TTATCATGGC CTTTGGCTGC
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
591
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------AGAGACGGAC AATACTTCTT ATGATTCACG TAAACTCAAT AGTTTTTATT
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
641
---------- ---------- ---------- ---------- ------------------- ---------- ---------- ---------- ---------CATACTTTCT ATATGTATTA AAAATTTATT GAATATTTAT AAATGTTAAA
TOBSH1-002E06
SCI1-ORF
TOB068F10
457
466
691
---------- ---------- ---------- ---------- ------------ ---------- ---------- ---------- --ACTATAAATT CCAATAAAAA AAAAAAAAAA AAAAAAAAAA AAA
B
atggggagcgataagaagacgccggaggagaagaggaagcataagagaagttcgccttct
M G S D K K T P E E K r k h k r s s p s
tctccacgagatgaagtgaaaagcaaacgccagaatatcaaaggggatgaagagcgaagg
s p r d e v k s k r q n i k g d e e r r
aaagaaaagaaggacaaatccaagaaggagaagcacaaatcccattctagtgaagagaag
k e k k d k s k k e k H K S H S S E E K
aagtcaggggaaaagcacaaaaccaagagccacaaacataaggataaatcgaaaaacaag
K S G E K H K T K S H K H K D K S K N K
tttgaagagttatcaaaagatgattatttctctaagaacaatgaatttgctacttggctg
F E E L S K D D Y F S K N N E F A T W L
aaagataagaagaatttgttcttctcagatctttcgtctgagactgcacgcgatttattc
K D K K N L F F S D L S S E T A R D L F
tctgactttgtcatacaatggaacaaaggaaagcttgacagccaatactatgagggaatt
S D F V I Q W N K G K L D S Q Y Y E G I
gcaaccgggcctcggtcatcccacgcttggaatatcaaaaagtaa
A T G P R S S H A W N I K K *
60
20
120
40
180
60
240
80
300
100
360
120
420
140
465
154
Supplemental Figure 1 – Sequences of the SCI1 cDNA clones and the deduced amino acid sequence showing
the functional domains identified by in silico analysis. (A) Nucleotide sequence of the clone TOBSH1-002E06,
representing three identical sequences identified by SSH (De-Paoli et al., manuscript in preparation), the clone
TOB068F10, identified by homology search against the TOBEST database (Quiapim et al., 2009), and the PCR
product of the putative open reading frame (“SCI1-ORF”), which was used in SCI1Ri and SCI1OE constructions,
aligned by Dialign 2.2.1 (Brudno et al., 2003). The PCR primer sequences are underlined, the bold letters
represent the 5’UTR sequence and the letters in italics represent the 3’UTR sequence. The initial ATG is
highlighted in light gray, the stop codon is highlighted in dark gray and the mismatches are highlighted in black.
The gaps (-) were introduced to better align the sequences, which showed codon deletions and/or insertions
(AAG, encoding the amino acid Lysine; and CATAAATCCAAATGT, encoding the amino acids Lysine,
Serine, Lysine, Cysteine and Histidine, respectively). (B) The “SCI1-ORF” nucleotide and deduced amino acid
sequences showing the putative lysine-rich domain (LRD) in light gray, the nuclear localization signal (NLS)
indicated in lowercase letters, the ser/thr/tyr phosphorylation sites (prediction ≥ 96%) are shown by bold
underlined amino acids and the two cyclin interaction domains (LIG_CYCLIN) are highlighted in dark gray.
The first amino acid “K” from the LIG_CYCLIN domain also belongs to the LRD domain. The bold underlined
nucleotide sequences represent the qRT-PCR primers. The symbol * refers to the stop codon. The nucleotide
and amino acid positions are indicated.
NtSCI1
SlSCI1
StSCI1
VvSCI1
HvSCI1
OsSCI1
ZmSCI1
AtSCI1
┌----------DOMAIN I----------┐┌------------------------DOMAIN II-----------------------------┐┌--MGSDKKTPEEKRKHKRSSPSSPR-DEVKS-KRQNIKGD-----------EERRK-E--KKDKS-KKEKHKSHSSEEKKSGEKHKTKSH-KHKDKSKNK
MGSDRKRKEEKMKHKRNSPSSPQADEGRS-KRQKIKGD-----------EETSK-DKSRKEKH-KSHKSKRRSNEDRESGEKHKSKSH-KHKDKLKIK
MGSDRKMKEEKIKHKRSSPSSPQADEGRS-KRQKIKGD-----------EEPSK-DKSRKDKH-KSHKSKRHSNEEKESGEKHKSKSH-KHKDKLKIK
MGS-----ESKEKKRRSSPSISS-DGGKG-KRRRSVED------------K-KE-K--RKDKK-SHRHSKHHSDKEKKSEGKHKDKRH-KRDRHLILE
MGGG----EAKSRKRRSSPSSGEEGERERKKRRDKKESRRSGR----GSDEEEE-KRRRKKKHGDRGKDKERDSKERRSKEKDKSKRKDKDVSSLKQD
MGG-----EGKSRKRRSSPSSGEEEERERKKRRDKKESRRSSRDEQAEEEEEEERRRRKKKKHGDRGKDKERDSKEKHSKEKEKSKRKHKD-----DD
MGGG----EGKSKKRRSSTSSAEEEGGER-KRRDKKESKRRSRDD----REDDDDRHKKKGKHIDRNKGKERDSKDRHSKEKT-SKRKDKD-----AA
MVSERSSKEKKRSRARSEDSSSSDYEEKVKRHRGTEKD-----------DERRSRRSDKKDK--KSHKHHKSSTSKKSKDDKPKKKHT-ESDHKLKEG
cons
* .
NtSCI1
SlSCI1
StSCI1
VvSCI1
HvSCI1
OsSCI1
ZmSCI1
AtSCI1
-------------------------------DOMAIN III---------------------------------┐
FEELSKDDYFSKNNEFATWLKDKKNLFFSDLSSETARDLFSDFVIQWNKGKLDSQYYEGIATGPRSSHAWNIK-K
FEELSKDDYFSKNNEFATWLKDKKKLFFSDLSSEAARDLFSDFVKEWNKGKLDIQYYEGITTAPRSSHAWNIK-K
FEELSKDDYFSKNNEFATWLKEKKKLFFSDLSSEAARDLFSNFVKDWNKGKLDIQYYEGITTAPRSSHAWNIK-K
FQELSNDDYFSKNNEFAAWLKEKRKIYFADLSTESARELFTEFVKDWNKQKLESKYYEGIVTGPRTSHKWNIK-Q
IKEISKDDYFAKNNEFATWLKEEKGKYFSDLSSESARDIFLKFVKQWNKGKLPSQYYEGITTGPRSAHNWNIKKA
FKEISKDDYFAKNNEFATWLKEEKGKFFSDLSSESARDLFEKFVKTWNKGKLPSQYYEGITRGPRSAHKWNIK-V
FKEISKDDYFAKNNEFATWLKEEKGKYFSDLSSESARDLFLKFVKQWNKGKLPSQYYEGITSGPRSAHNWNIK-A
IPELSMEDYFSKNNEFATWLKEEKRTYFNDLTTEAARGLFSRFVKRWNRGKLESRYYEGISTAPRTAHDWMIKRR
Cons
: *:* :***:******:***:::
* *
: *.. *
.
:::
.
:* **::*:** :*
:
**
**: **
.
:* *
:*****
.
: :
. ..: .
*
*
:
.**::* * **
BAD AVG GOOD
Supplemental Figure 2 – Amino acid sequence alignment of the SCI1 protein and the putative homologous
sequences found in other plant species. Multiple sequence alignment built on T-COFFEE program (version
5.53). The color scale represents the alignment score. The proposed Domains I, II and III are indicated. NtSCI1
(the N. tabacum sequence here described), SlSCI1 - tomato (AC212309), StSCI1 - potato (AC233353), VvSCI1
- grapevine (AM424297), HvSCI1 - barley (AK252156), OsSCI1 - rice (NM_001052562), ZmSCI1 - corn
(EU958997), AtSCI1 – Arabidopsis At1g79200 (NM_106571).
A
50 µm
C
E
B
50 µm
D
20 µm
20 µm
Supplemental Figure 3 – Analysis of anther morphological markers from floral buds at very early stages of
flower development. Anthers were collected from the same floral buds as the ones from which the
stigmas/styles were used for scanning electron microscopy (SEM) analysis and for RNA extraction for qRTPCR. Anthers were dissected from floral buds at the relevant developmental stages, fixed, embedded in
paraffin, transversely sliced into 8µm sections and stained with 0.05% toluidine blue. This analysis was
performed to confirm the correct evaluation of the flower developmental stage by comparison with the anther
morphological markers described by Koltunow et al (1990). (A) Transversal section of anther at stage -4,
stained with 0.05% toluidine blue, showing the microspore mother cell (white arrow), formed inside the pollen
sac, visibly distinguishable from the tapetum cells. At this stage, the tapetum is clearly divided in inner (red
arrow) and outer tapetum (yellow arrow). (B) Transversal section of anther at stage -3, with an evident callose
deposition in the microspore mother cell walls (brown arrow) (C) Anther stage -2, showing the larger and
multinucleated (black arrows) tapetum. (D) Anther stage -1, detailing the dyad (black and green arrows),
resultant from the meiosis I completion. (E) Anther stage 1, presenting the completed meiosis shown clearly by
tetrad formation (white arrow).
A
NtSCI1
AtSIM
101
89
KDKKNLFFSDLSSET
KD+
FFS + ++T
KDEIERFFSSVYNQT
**: : ***.: .:*
115
103
B
NtSCI1
AtSIM
NtaKIS2
NtKIS1a
NsKIS1
AtKRP1
AtKRP2
Consensus
Prim.cons.
KDKKNLFFSDLSSETAKDEIERFFSSVYNQTK-TDLEEFFAAAEKDILK
-KEIEEFFAARQKAILK
-KEIEDFFAVRQKAIFK
-SEIEDFFVEAEKQLKE
-AELEDFFQVAEKDLRN
ei# FF
k
K2EIEDFFA2AEK3I2K
C
Supplemental Figure 4 – Sequence analysis between SCI1 and the most closely related classes of cell cycle
regulators. (A) Similarity between NtSCI1 and AtSIM revealed by BLASTP two sequences. The bold
underlined region represents the conserved domain between AtSIM and CKI/KRPs classes as shown by
Churchman et al. (2006). This region includes one of the cyclin interaction domains recognized at the NtSCI1
protein sequence. (B) Alignment between NtSCI1, AtSIM and the most related CKI/KRPs at the conserved
region of the CDK inhibitors mentioned above. The alignment was produced by the Multalin program
(http://www.expasy.org/tools/#align). Note the two conserved phenylalanine (F) residues and the very charged
region before them present on all proteins aligned. (C) Phylogram tree generated by ClustalW2 alignment
performed using the sequences showed in B. The colors were inserted to facilitate the visualization of the three
different classes of cell cycle regulators (CKI/KRPs, SIM and SCI1).
Supplemental Table 1 –Stigma/style tissues analysis in transgenic and wild-type plants.
SCI1OE 3.1.1
Wild type
SCI1Ri 14.2
Number of SSZ cell
layersa
*8.3 ± 0.6
13.0 ± 1.0
*17.7 ± 2.1
SSZ area (µm2)a
*191,381.3 (77%) ± 10,834
248,272.0 (100%) ± 9,441
*367,355.3 (148%) ± 13,474
29.3 ± 4.0
36.0 ± 1.0
36.7 ± 1.5
*510.7 ± 16.8
559.9 ± 20.4
*657.0 ± 37.3
*58.5 ± 7.2
88.4 ± 7.1
*113.1 ± 5.5
SSZ cell number
b
Style thickness (µm)
STT thickness (µm)
c
d
e
Parenchyma cell layers 14.0 ± 1.0
14.3 ± 0.6
*17.3 ± 0.6
As shown in Figure 3Ca and 3D.
b
Counted in an area of 20,000µm2, Figure 3Ca and 3D.
c
Measured at 400µm below the intersection of the stigmatic secretory zone and transmitting tissue, as shown in Figure
3Ca/3Eb.
d
Measured at 400µm below the intersection of the stigmatic secretory zone and transmitting tissue (showed in Figure 3Ca and
3Ec1).
e
Counted just behind the vascular bundle, as shown in Figure 3Ec2.
The values represent the mean of three individual determinations with the standard deviation.
*Significantly different from the wild-type by Student’s t-test (p<0.05).
a
Supplemental Table 2 - Pairwise 2-hybrid interactions between Arabidopsis SCI1, and CDK
and cyclins.
Gene
CDKA;1
CDKB1;1
CDKB1;2
CDKB2;1
CDKB2;2
CDKC;1
CDKC;2
CDKD;1
CDKD;2
CDKD;3
CDKE;1
CYCA1;1
CYCA1;1c
CYCA2;1c
CYCA2;2
OD600 cells
grown in SD
-L/-T/+H
mediuma
0.565
0.564
0.447
0.596
0.586
0.562
0.625
0.552
0.515
0.305
0.570
0.663
0.578
0.559
0.524
OD600 cells
grown in SD
-L/-T/-H
mediuma
0.079
0.068
0.077
0.088
0.077
0.079
0.074
0.073
0.064
0.072
0.081
0.589
0.073
0.073
0.071
CYCA2;2c
CYCA2;3
CYCA3;1c
CYCA3;2
CYCB1;1
CYCB1;1c
CYCB1;3
CYCB1;3c
CYCB1;4c
CYCB2;1
CYCB2;1c
CYCB2;2
CYCB2;3
CYCB3;1
CYCC1;1
CYCD1;1
CYCD2;1
CYCD3;2
CYCD4;2
CYCD5;1
CYCD6;1
CYCH;1
CYCP3;1
CYCP3;2
CYCT1;3
CDKA;1
0.434
0.552
0.573
0.598
0.410
0.544
0.534
0.589
0.488
0.540
0.567
0.555
0.568
0.499
0.356
0.510
0.387
0.474
0.540
0.652
0.502
0.488
0.559
0.564
0.580
0.565
0.518
0.096
0.081
0.073
0.085
0.070
0.084
0.062
0.069
0.069
0.095
0.065
0.078
0.071
0.082
0.082
0.073
0.055
0.078
0.114
0.069
0.083
0.072
0.080
0.082
0.079
a
X-Gal positive
14%
12%
17%
15%
13%
14%
12%
13%
12%
24%
14%
89%
13%
13%
14%
+
-
119%
17%
14%
12%
21%
13%
16%
11%
14%
13%
17%
12%
14%
14%
23%
16%
19%
12%
14%
17%
14%
17%
13%
14%
14%
14%
+
-
OD600 of cells after two days of growth.
b
c
Ratiob
Ratio of the OD600 of cells grown in -L/-T/-H on -L/-T/+H.
Truncated version lacking the destruction box in order to stabilize the protein
Supplemental Table 3 - Pairwise 2-hybrid interactions between Arabidopsis KRP2, and CDK
and cyclins.
Gene
CDKA;1
CDKB1;1
CDKB1;2
CDKB2;1
CDKB2;2
CDKC;1
CDKC;2
CDKD;1
CDKD;2
CDKD;3
CDKE;1
CYCA1;1
CYCA1;1c
CYCA2;1c
CYCA2;2
CYCA2;2c
CYCA2;3
CYCA3;1c
CYCA3;2
CYCB1;1
CYCB1;1c
CYCB1;3
CYCB1;3c
CYCB1;4c
CYCB2;1
CYCB2;1c
CYCB2;2
CYCB2;3
CYCB3;1
CYCC1;1
CYCD1;1
CYCD2;1
CYCD3;2
CYCD4;2
CYCD5;1
CYCD6;1
CYCH;1
CYCP3;1
CYCP3;2
CYCT1;3
CDKA;1
a
OD600 cells
grown in SD
-L/-T/-H
mediuma
0.685
0.076
0.090
0.112
0.152
0.108
0.121
0.103
0.590
0.092
0.090
0.082
0.407
0.109
0.115
0.157
0.090
0.120
0.128
0.123
0.085
0.096
0.107
0.085
0.531
0.108
0.407
0.136
0.102
0.108
0.072
0.403
0.122
0.422
0.521
0.471
0.097
0.068
0.093
0.106
0.685
Ratiob
X-Gal positive
103%
11%
13%
16%
21%
17%
19%
15%
90%
19%
19%
11%
57%
16%
16%
22%
13%
28%
18%
27%
15%
19%
15%
22%
94%
15%
56%
19%
20%
15%
13%
93%
29%
59%
102%
98%
27%
10%
13%
15%
103%
+
+
+
+
+
+
OD600 of cells after two days of growth.
b
c
OD600 cells
grown in SD
-L/-T/+H
mediuma
0.664
0.676
0.668
0.698
0.716
0.645
0.635
0.681
0.652
0.489
0.466
0.718
0.714
0.688
0.723
0.727
0.702
0.422
0.709
0.46
0.586
0.512
0.715
0.395
0.562
0.701
0.725
0.721
0.517
0.708
0.552
0.433
0.419
0.718
0.513
0.483
0.356
0.710
0.720
0.696
0.664
Ratio of the OD600 of cells grown in -L/-T/-H on -L/-T/+H.
Truncated version lacking the destruction box in order to stabilize the protein
Supplemental Table 4 – Amino acid sequences belonging to each ELM (Eukaryotic Linear Motif) interacting
domain identified in NtSCI1 protein and CDK/cyclin complex inhibitors, described in N. tabacum and some
related organisms.
Genes
ELM Domains
MOD
LIG MAPK_1
ProDKin_1
LIG
CYCLIN_1
MOD_CDK
NtSCI1
LIG_APC
C D/KEN
box
-
KNLFF
RDLF
-
DKKTPEE
KRSSPSS
SPSSPRD
KDKKNLFF
SIM
-
RKLM
-
GCTTPTS
TATTPPP
PPSTPSS
-
SRM1
RKLLV
KLLV
NFPTPIK
RSCKRKLLV
SRM2
KDLRF
NFPTPIK
RCSTPTS
VVDSPPP
SLCTPTS
SRM3
RKLTP
GCKTPTS
RKLTPVN
RKRPREISL
KRPREISL
KKTRLSKDLRF
KTRLSKDLRF
KRKLTPVNV
RKLTPVNV
NtaKIS2
EREVLE
SRRRLT
PKENA
KSLDL
-
SLVTPTS
ELSSPKE
KSKGIGEMTV
NsKIS1
LKEND
-
RERTPEK
-
NtKIS1*
-
-
RQRTPEK
AtKRP1
-
KELI
RKLF
-
KLISPAL
VGDTPAG
NLVSPSG
RERTPEK
KLMSPPL
VGDTPAG
SLVSPSS
RQRTPEK
-
AtKRP2
SRIILS
SRRRLR
LRKSLH
LRNKLL
-
-
IILSPCV
RRDSPPV
RKCKGIEEVTI
KCKGIEEVTI
MOD
CK1_1
MOD CK2_1
SSPSSPR
SPSSPRD
SHSSEEK
SDLSSET
SSETARD
STPSSLG
SSTTTAI
STTTAIT
SFRSCSR
SKTSKTK
STPTSQE
SAPSATA
SLCTPTS
DKKTPEE
KDKSKKE
KSHSSEE
SSGTKRK
RLPTRPE
IPDSDHE
EDPTVNE
GEMTVME
CVRSHSE
ELSSPKE
PSETDLE
SLVTPTS
SELSSPK
SNGSSKS
SSKSLDL
SKESIKA
SCCSRNG
SAESKEA
TSLSKYE
STPTSQE
LLVSTCE
LLETLEE
KPRSQEE
QDSSKKE
RNGSTEE
KLSSERE
KMPSEKE
SLVSPSS
SPSSSVN
SAESKEA
EEVTIME
RNGSSEE
KMPSEKE
RSRRIVYV
SEKSSSV
RSEKSSSVSV
SSVSVVG
KKKELIHL
SSCSGSN
KKELIHL
KRRKMEEEVDL SETSVVI
RRKMEEEVDL
SSVSCCS
RKMEEEVDL
SCCSTSE
KSKRRIEFVDL SEESMNM
KRRIEFVDL
RRIEFVDL
GRKTMEE
ETVTAEE
EMPTESE
PTESEIE
SCCSTSE
CCSTSEE
LRKSLHE
LHETVKE
Nta, Nicotiana tabacum; Ns, Nicotiana sylvestris; Nt, Nicotiana tomentosiformis; At, Arabidopsis thaliana.
*The alternative spliced forms NtKIS1a and NtKIS1b have these same domains sequences. The symbol “-”
means the specified domain is either absent or has an e-value above the ELM threshold.
LIG_APCC Dbox_1/KENbox_2, motif containing an RXXL and KEN motif, respectively, that binds to the
Cdh1 and/or Cdc20 components of APC/C thereby targeting the protein for 26S proteasome mediated
degradation in a cell cycle dependent manner, Dbox .R..L. , KEN .KEN.;
LIG CYCLIN_1, Substrate recognition site that interacts with cyclin and thereby increases phosphorylation by
CDK/cyclin complexes, predicted protein should have the MOD_CDK site, also used by cyclin inhibitors
[RK].L.{0,1}[FYLIVMP];
MOD_CDK, Substrate motif for phosphorylation by CDK, ...([ST])P.[KR]
MOD ProDKin_1, Proline-Directed Kinase (e.g. MAPK, CDK-like kinases) phosphorylation site in higher
eukaryotes, ...([ST])P..;
LIG MAPK_1, MAPK interacting molecules (e.g. MAPKKs, substrates, phosphatases) carry docking motif
that help to regulate specific interaction in the MAPK cascade, the classic motif approximates (R/K)xxxx#x#
where # is a hydrophobic residue, [KR]{0,2}[KR].{0,2}[KR].{2,4}[ILVM].[ILVF];
MOD CK1_1 and MOD CK2_1, CK1 and CK2 phosphorylation sites, S..([ST])... and ...([ST])..E ,
respectively.
Accession numbers are: SIM (At5g04470); SMR1 (At3g10525); SMR2 (At1g08180); SMR3 (At5g02420); N.
tabacum NtaKIS2 (CAD56868); N. sylvestris NsKIS1 (CAC82732); N. tomentosiformis NtKIS1a (CAC82731);
N. tomentosiformis NtKIS1b (CAC82734); A. thaliana AtKRP1 (NP_179924); A. thaliana AtKRP2
(NP_190632).
Anexo 2
Curriculum vitae
CURRICULUM VITAE
HENRIQUE CESTARI DE PAOLI
May, 2010
1. Professional Address:
University of São Paulo
Faculty of Philosophy, Sciences and Languages of Ribeirão Preto
Department of Biology
14040-901, Ribeirão Preto, SP, Brazil
Phone: (+55) 16 3602 3702; (+1 858) 822 2673
Fax: (+55) 16 3602 4886
E-mail: [email protected]; [email protected]; [email protected]
2. Present Position
PhD Student at University of São Paulo (USP), Dr. Maria Helena de Souza
Goldman’s laboratory, Department of Genetics at Faculty of Medicine at
Ribeirão Preto/SP.
PhD Visiting Graduate Student at University of California San Diego (UCSD), Dr.
Yunde Zhao’s laboratory, Department of Cell & Developmental Biology.
Research Projects:
a) Auxin signaling;
b) Characterization of plant reproductive genes;
c) Studies and manipulation of flowering in plants.
3. Education
2006 – Nowadays PhD in Genetics. University of São Paulo, Faculty of Medicine of
Ribeirão Preto, Department of Genetics, with an internship at University
of California San Diego/USA.
Thesis Title: “SCI1, A NOVEL TISSUE-SPECIFIC INHIBITOR OF CELL
PROLIFERATION RELATED TO AUXIN SIGNALING”.
Thesis Advisor: Dr. Maria Helena de Souza Goldman
2004 – 2006 Ms. in Genetics. University of São Paulo, Faculty of Medicine of Ribeirão
Preto, Department of Genetics.
Dissertation Title: ANALISYS OF GENE EXPRESSION IN Nicotiana
tabacum PISTIL: IDENTIFICATION OF SPECIFIC GENES BY
SUBTRACTIVE HYBRIDIZATION AND CELLULAR AND TEMPORAL
CHARACTERIZATION.
Dissertation Advisor: Dr. Maria Helena de Souza Goldman
2000 – 2003 B.Sc. in Biological Sciences
Izabela Hendrix University, FAMIH, Belo Horizonte, Brazil.
Monograph
Title:
MOLECULAR
CHARACTERIZATION
OF
MICRORGANISMS IN THE SAVANES ECOSSYSTEM - Coletotrichum
graminicola.
Monograph Advisor: Dr. José Edson Fontes Figueiredo (EMBRAPA).
4. Academic experience
- Continuing Education
1 – International Course and Practical Training on Molecular Techniques in
Plant Science. Cold Spring Harbor Laboratory (CSHL). New York, USA.
2007 (210h)
2 – International Course and Practical Training on Functional Genomics of
Vegetal Reproduction. Federal University of Rio de Janeiro, UFRJ.
Financial aid from “National Council of Research and Development”
(CNPq), Brazil. 2006 (90h).
3 – International Course on Molecular Mechanisms Controlling Flower
Development. University of Campinas (UNICAMP), Campinas, Brazil.
2007 (24h).
4 – Practical Training on Bidimensional Electrophoresis. Brazilian Company of
Agricultural Research (EMBRAPA), CNPMS, Sete Lagoas, Brazil, 2005
(40h).
5 – Course on Differential expression of genes in fungi. University of São Paulo
(USP-RP), Ribeirão Preto. Financial aid from “National Council of
Research and Development” (CNPq), Brazil. 2004 (34h).
6 – Course on Cell cycle control, Federal University of Minas Gerais, Belo
Horizonte, Brazil. 2004 (45h).
- Advising
Marcella de Francisco Amorim. (Undergraduate program) Gene expression analysis of
the ABC transporters from WBC family using real time RT-PCR (2006). Principal
Advisor: Maria Helena S. Goldman; Supervisor: Ms. Henrique C. De Paoli (DEPAOLI, H.C.).
DE-PAOLI, H.C. Surveyor of the International Symposium of Undergraduate Science of
the University of São Paulo, 14ºSIICUSP. Ribeirão Preto, Brazil, 2006
Alexandre Palma Boer Martins. (Undergraduate program) Subcloning and sequencing
genomic clones of the pistil specific methyl transferase from Nicotiana tabacum L.
(2005). Principal Advisor: Maria Helena S. Goldman; Supervisor: Henrique C. De Paoli
(DE-PAOLI, H.C.).
- Oral Presentations
1 – “SCI1 is a component of the nuclear signal transduction pathway
engaging auxin signaling and cell division/differentiation control in
upper pistil”. 6th Cell Cycle Meeting – Salk Institute, San Diego, CA, USA.
2009.
2 – “Suppression Subtractive Hybridization (SSH) to identify genes
preferentially expressed in stigma/style of Nicotiana tabacum”. XXXIV
Congress of the Brazilian Society for Biochemistry and Molecular Biology –
SBBQ, Águas de Lindóia, SP, Brazil. 2005.
3 – “Assessment of the genetic diversity of Colletotrichum graminicola
isolated from sorghum in Brazil based on sequencing analysis of 18S
rDNA and 18S-28S intergenic spacer”. I Biological Sciences Week, Belo
Horizonte, Brazil, 2001.
- Lectures
1 – “SCI1 is a component of the nuclear signal transduction pathway
engaging auxin signaling and cell division/differentiation control in
upper pistil”.Richard B. Simches Research Center, MGH/Harvard Medical
School, Boston/MA, USA. 2009.
2 – “SCI1 is a component of the nuclear signal transduction pathway
engaging auxin signaling and cell division/differentiation control in
upper pistil”. Joint Lab Meeting UCSD/SALK Institute, San Diego/CA,
USA. 2009.
3 – “A Nicotiana tabacum pistil specific lysine-rich protein controls stigma
size and inhibits style elongation: a component of the auxin signal
transduction pathway?. International Course and Practical Trainning on
Molecular Techniques on Plant Science, New York/NY, USA. 2007.
4 - “Studies of gene expression in plants”. X Summer Course of Genetics.
Faculty of Medicine of Ribeirão Preto.. University of São Paulo Department of Genetics. Ribeirão Preto, SP, Brazil. 2005; 2010.
5 – Discipline on General Genetics. Izabela Hendrix University, Belo Horizonte,
Brazil. 2002 and 2003.
- Relevant Abstracts and/or Participation in Meetings
DE-PAOLI, H. C.; BRITO, M. S. ; QUIAPIM, A. C.; TEIXEIRA, S. P.; GOLDMAN, G. H.;
DORNELAS, M. C.; ZHAO, Y.; GOLDMAN, M. H. S. SCI1 is a component of the
nuclear signal transduction pathway engaging cell division/differentiation control
and auxin signaling in upper pistil. In: 6th Cell Cycle Meeting – Salk Institute, San
Diego, CA, USA. 2009.
DE-PAOLI, H. C.; BRITO, M. S. ; QUIAPIM, A. C. ; PRANCHEVICIUS, M. C. S. ;
DORNELAS, M. C. ; TEIXEIRA, S. P. ; GOLDMAN, G. H. ; GOLDMAN, M. H. S. A
novel stigma/style-specific gene, SCI1, encodes a lysine-rich protein that controls
cell division and differentiation. In: XX International Congress on Sexual Plant
Reproduction. Brasília/DF, Brazil, 2008.
DE-PAOLI, H. C. ; BRITO, M. S.; QUIAPIM, A. C. ; DORNELAS, M. C. ; GOLDMAN, G.
H. ; McClure, B. ; GOLDMAN, M. H. S. The self-incompatibility related HT-protein
remains functional in self-compatible Nicotiana tabacum: does it have general role
in pollination? In: XX International Congress on Sexual Plant Reproduction.
Brasilia/DF, Brazil, 2008.
DE-PAOLI, H. C. ; BRITO, M. S. ; QUIAPIM, A. C. ; DORNELAS, M. C. ; TEIXEIRA, S.
P. ; GOLDMAN, G. H. ; GOLDMAN, M. H. S. . A novel tobacco gene encodes a
pistil-specific lysine-rich protein that controls stigma size via cell cycle inhibition
and influences its developmental timing. In: III International Symposium of
Developmental Biology. Uberaba, Brazil, 2007.
DE-PAOLI, H. C. ; BRITO, M. S. ; QUIAPIM, A. C. ; RODRIGUES, RAO ; DORNELAS,
M. C. ; TEIXEIRA, S. P. ; GOLDMAN, G. H. ; GOLDMAN, M. H. S. . A tobacco
pistil-specific lysine-rich protein controls stigma size and inhibits style elongation.
In: 53º Brazilian Congress of Genetics. Águas de Lindóia, Brazil, 2007.
DE-PAOLI, H. C. ; BRITO, M. S. ; QUIAPIM, A. C. ; RODRIGUES, RAO ; DORNELAS,
M. C. ; TEIXEIRA, S. P. ; GOLDMAN, G. H. ; GOLDMAN, M. H. S. . A Tobacco
pistil-specific lysine-rich protein controls stigma size and inhibits style elongation.
In: Plant Biology & Botany 2007, Chicago, USA, 2007.
DE-PAOLI, H. C. ; BRITO, M.S.; QUIAPIM, A.C.; RODRIGUES, R.A.O.; DORNELAS,
M.C.; TEIXEIRA, S.P.; GOLDMAN, G.H.; GOLDMAN, M.H.S. A Nicotiana tabacum
pistil specific lysine-rich protein controls stigma size and inhibits style elongation.
In: I Brazilian Symposium of Molecular Genetics of Plants. Natal, Brazil, 2007.
DE-PAOLI, H. C. ; BRITO, M. S. ; QUIAPIM, A. C. ; GOLDMAN, G. H. ; GOLDMAN, M.
H. S. . Suppression subtractive hybridization reveals genes specifically expressed
in wet stigma and underlie plant reproduction mechanisms not present in dry
stigmas. In: XIXth International Congress on Sexual Plant Reproduction: from
gametes to genes. Budapest. Hungary, 2006.
DE-PAOLI, H. C. ; BRITO, M. S. ; QUIAPIM, A. C. ; GOLDMAN, G. H. ; GOLDMAN, M.
H. S. . Suppression Subtractive Hybridization reveals the reproductive organs
specific transcriptome of Nicotiana tabacum. In: International Symposium of
Graduation and Research. 2ºSINPOSPq. Ribeirão Preto, Brazil. 2006.
DE-PAOLI, H. C. ; BRITO, M. S. ; QUIAPIM, A. C. ; RIBEIRO, D. C. D. ; Marques, E.R. ;
MOLFETTA, J. B. ; GOLDMAN, G. H. ; DORNELAS, M. C. ; GOLDMAN, M. H. S.
Suppression subtractive hybridization to identify genes preferentially expressed in
stigma/style of Nicotiana tabacum. In: 51º Brazilian Congress of Genetics, Águas
de Lindóia SP, Brazil, 2005.
5. Publications
DE-PAOLI, H.C., ZHAO, Y., GOLDMAN, M.H.S. (2010). SCI1 is a component of the
nuclear signal transduction pathway engaging cell division/differentiation
control and auxin signaling in upper pistil. Manuscript in preparation.
DE-PAOLI, H.C.; BRITO, M. S. ; QUIAPIM, A. C. ; TEIXEIRA, S. P. ; DeVeylder, L. ;
STRINI, E.J.; GOLDMAN, G. H. ; DORNELAS, M. C. ; GOLDMAN, M. H. S.
(2010). A novel tobacco stigma/style gene, SCI1, encoding a lysine-rich
protein that controls cell division and differentiation. Plant Cell, submitted.
DE-PAOLI, H.C., BRITO, M.S., QUIAPIM, A.C., DORNELAS, M.C., GOLDMAN, G.H.,
McCLURE, B.A., GOLDMAN, M.H.S.(2010) The self-incompatibility related HTprotein remains functional in self-compatible Nicotiana tabacum: does it have
general role in pollination? Manuscript in preparation.
DE-PAOLI, H.C., BRITO, M.S., QUIAPIM, A.C., BERNARDES, L., GOLDMAN, G.H.,
GOLDMAN, M.H.S. (2010) Unraveling the specific pattern of gene expression
in the wet stigma specie N. tabacum by suppression subtractive
hybridization underlie unique plant reproduction mechanisms. Manuscript in
preparation.
AVANCI, N.C., ANGELO, P.C.S., CALIXTO, C.P.G., MOLFETTA-MACHADO, J.B., DEPAOLI, H.C., QUIAPIM, A.C., GOLDMAN, G.H., BARKMAN, T.J., MURAKAMI,
M.T., MORAES, L.A.B. & GOLDMAN, M.H.S.(2010) Is MeJa a hormone with a
new role in Solanaceae sexual reproduction? Characterization of a novel
tobacco pistil-specific methyltransferase gene. Manuscript in preparation.
QUIAPIM, A. C., BRITO, M.S., Malavazi, I., Bernardes, L.A.S., DE-PAOLI, H.C.,
Goldman, G.H., Goldman, M.H.S. (2009) Analysis of the Nicotiana tabacum
stigma/style transcriptome reveals gene expression differences between wet
and dry stigma species. Plant Physiol. 149:1211-1230.
FIGUEIREDO, José Edson Fontes ; COELHO, Vinicio Tadeu da Silva ; COELHO, E. A.
F. ; OLIVEIRA, J. S. ; SANTORO, M. M. ; ALMEIDA, P. L. B. ; DE-PAOLI, H. C. ;
CORREA, G. V. ; TEIXEIRA, M. A. (2005) Bioquímica molecular como
ferramenta para conservação e uso da biodiversidade agrícola tropical:
desenvolvimento de marcador imunoquímico para identificação de bactérias
endofíticas do milho. Boletim de Pesquisa e Desenvolvimento - Embrapa
CNPMS, v. 02, p. 1-12, 2005. ISSN/ISBN: 16790154
FIGUEIREDO, José Edson Fontes ; QUINTÃO, Pablo Lopes ; DE-PAOLI, H. C. ;
COELHO, Vinicio Tadeu da Silva ; BRESSAN, Wellington ; GUIMARÃES, Cláudia
Teixeira ; GOMES, Eliane Aparecida (2003). Caracterização Molecular de
Microrganismos Isolados do Ecossistema Agrícola do Cerrado: bactérias
endofíticas do milho. Comunicado Técnico - Embrapa CNPMS, v. 66, p. 1-10,
2003. ISSN/ISBN: 16790162.
FIGUEIREDO, José Edson Fontes ; COELHO, Vinicio Tadeu da Silva ; DE-PAOLI, H. C.
; BRESSAN, Wellington ; PURCINO, Antônio Álvaro Corcetti ; ROCHA, Tales
Lima ; PRATES, Maura Vianna (2003). Desenvolvimento de Marcador
Molecular para Bacillus subtilis Utilizando Proteomics. Comunicado Técnico Embrapa CNPMS, v. 67, p. 1-10, 2003. ISSN/ISBN: 16790162.
FIGUEIREDO, José Edson Fontes ; DE-PAOLI, H. C. ; QUINTÃO, Pablo Lopes ;
COELHO, Vinicio Tadeu da Silva ; CASELA, Carlos Roberto ; FERREIRA, A. S. ;
BRESSAN, Wellington ; GUIMARÃES, Cláudia Teixeira ; GOMES, Eliane
Aparecida (2003). Caracterização Molecular de Microrganismos do
Ecossistema Agrícola do Cerrado: Colletotrichum graminicola. Comunicado
Técnico - Embrapa CNPMS, v. 68, p. 1-10, 2003. ISSN/ISBN: 16790162.
FIGUEIREDO, José Edson Fontes ; DE-PAOLI, H. C. ; COELHO, Vinicio Tadeu da Silva
; PINTO, Nicésio F J de A ; FERNANDES, Fernando Tavares ; BRESSAN,
Wellington ; GUIMARÃES, Cláudia Teixeira ; GOMES, Eliane Aparecida (2003).
Caracterização Molecular de Microrganismos do Ecossistema Agrícola do
Cerrado: Acremonium strictum. Comunicado Técnico - Embrapa CNPMS, v. 69,
p. 1-10, 2003. ISSN/ISBN: 16790162.
6. Awards
1 – Honorable Mention. In: I Brazilian Symposium of Molecular Genetics of Plants.
Natal, Brazil, 2007.
2 – Prêmio Pós-graduação, Brazilian Society of Genetics. Águas de Lindóia.
Brazil, 2007.
7. Idioms
Idiom
Reading Skills
Portuguese*
English
Spanish
Chinese
*Mother tongue
+++++++
+++++
+++
Speaking
Skills
+++++++
+++++
++
+
Listening
Skills
+++++++
+++++
+
+
Writing Skills
+++++++
+++++
+












