UNIVERSIDADE FEDERAL DE SANTA CATARINA
CENTRO DE CIÊNCIAS AGRÁRIAS
DEPARTAMENTO DE AQUICULTURA
PROGRAMA DE PÓS-GRADUAÇÃO EM AQUICULTURA
DEFESA ANTIVIRAL EM Litopenaeus vannamei CONTRA O
VÍRUS DA SÍNDROME DA MANCHA BRANCA (WSSV),
INDUZIDA VIA RNA DE INTERFERÊNCIA, E SUA INFLUÊNCIA
NA EXPRESSÃO DE ALGUNS GENES IMUNOLÓGICOS
Dissertação de Mestrado apresentada ao
Programa de Pós-Graduação em
Aquicultura da Universidade Federal de
Santa Catarina, como parte dos
requisitos necessários à obtenção do
título de Mestre em Aquicultura.
Orientadora: Margherita Anna Antônia Maria Barracco
Co-orientadora: Luciane Maria Perazzolo
CRISTHIANE GUERTLER
Florianópolis/SC
2010
Catalogação na fonte pela Biblioteca Universitária
da
Universidade Federal de Santa Catarina
G935d
Guertler, Cristhiane
Defesa antiviral em Litopenaeus vannamei contra o
vírus da síndrome da mancha branca (WSSV), induzida
via RNA de interferência, e sua influência na
expressão de alguns genes imunológicos [dissertação]
/ Cristhiane Guertler ; orientadora, Margherita Anna
Antônia Maria Barracco. – Florianópolis, SC, 2010.
103 p.: 6 figs., 2 tabs.
Dissertação (mestrado) - Universidade Federal de
Santa Catarina, Centro de Ciências Agrárias.
Programa de Pós-Graduação em Aquicultura.
Inclui referências
1. Aquicultura. 2. Litopenaeus vannamei.
3. Crustáceos - Imunologia. 4. Vírus da síndrome da
mancha branca. 5. RNA de interferência. 6. Expressão
gênica. I. Barracco, Margherita Anna Antonia Maria.
II. Universidade Federal de Santa Catarina. Programa
de Pós-Graduação em Aquicultura. III. Título.
CDU 639.3
Defesa antiviral em Litopenaeus vannamei contra o vírus da
síndrome da mancha branca (WSSV), induzida via RNA de
interferência, e sua influência na expressão de alguns genes
imunológicos.
Por
CRISTHIANE GUERTLER
Esta dissertação foi julgada adequada para a obtenção do título de
MESTRE EM AQÜICULTURA
e aprovada em sua forma final pelo Programa de
Pós-Graduação em Aqüicultura.
_____________________________________
Prof. Cláudio Manoel Rodrigues de Melo, Dr.
Coordenador do Curso
Banca Examinadora:
__________________________________________________
Dra. Margherita Anna Antônia Maria Barracco – Orientadora
__________________________________________________
Dr. Cláudio Humberto Mejía Ruiz
__________________________________________________
Dr. Walter Quadros Seiffert
Aos meus pais, Henrique e Anajara, com gratidão e amor.
AGRADECIMENTOS
Inicialmente, quero agradecer a Deus por ter me dado a vida e a
oportunidade de desfrutá-la junto às pessoas especiais que conheci e que
ainda conhecerei.
Aos meus pais, Henrique e Anajara, pelo amor incondicional e
apoio absoluto durante toda minha vida. Vocês são para mim o exemplo
de amor, bondade e perseverança. Obrigada por terem criado as
condições para que eu pudesse sempre realizar meus sonhos. Amo
vocês.
À minha irmã Ágatha, que também foi responsável pela minha
conquista, já que além do incentivo também se privou de muita coisa
para que eu pudesse chegar até aqui.
Ao meu irmão Gustavo, e a mais nova componente da família,
minha sobrinha Marina, que já chegou ao mundo lutando e que com
certeza apareceu em novas vidas para torná-las mais leves e felizes.
Ao Clênio, meu amigo e amor, que aguentou bravamente minhas
inúmeras oscilações de humor durante o mestrado e ainda assim
continuou sendo meu maior incentivador. Obrigada pelo amor,
companheirismo e amizade durante todos esses anos. Você é muito
importante na minha vida.
Às minhas queridas amigas Bárbara, Manoela, Giselle e Liane.
Gurias, obrigada pela amizade verdadeira. Vocês são demais! Amo
muito todas vocês.
À professora Luciane, pela orientação, respeito e amizade. Com
certeza, seus esforços para a realização deste projeto serviram de
motivação para que eu fizesse um bom trabalho. Obrigada
principalmente, pela oportunidade inicial e por acreditar em minha
capacidade confiando a mim um projeto relativamente distante de
minhas atribuições profissionais e que encarei como um desafio
profissional.
À professora Margherita, por ajudar a moldar minha formação
profissional, já que é para mim um exemplo de dedicação e ética
profissional. Mais uma vez obrigada por responder àquele e-mail, pois
devido a isso fui apresentada ao mundo da pesquisa, que tanto me
encantou e também iludiu, mas que graças a isso pude encontrar meu
verdadeiro lugar dentro da minha profissão.
Aos colegas e amigos do Laboratório de Imunologia Aplicada à
Aquicultura: Paulinha (brava companheira de trabalho durante feriados
e finais de semana), Mirian, Pri, Liege, Dani, Pedro, Mirella, Gabi, Érik,
Tailin, Douglas e Scheila. Um agradecimento especial à Mirian pelo
apoio técnico que tanto facilitou meu trabalho, e à Priscila pela ajuda
durante a reta final dos experimentos.
Ao professor Maurício Lehmann, pela amizade e por todo apoio
durante a fase de infecção experimental em Araquari.
Ao professor Claudio Humberto Mejía-Ruíz, pelo auxílio no
preparo do dsRNA e por pacientemente sempre estar disposto a
esclarecer minhas dúvidas.
Ao sempre prestativo Felipe Vieira, pelo fornecimento dos
animais utilizados no experimento.
Ao Carlito e ao professor Cláudio Mello, pela dedicação intensa
aos alunos e ao programa de pós-graduação. Vocês deveriam servir de
exemplo para muitos funcionários públicos.
À Coordenação de Aperfeiçoamento de Pessoal de Nível
Superior (CAPES) pela concessão da bolsa de mestrado.
À Financiadora de Estudos e Projetos (FINEP), pela concessão
dos recursos destinados a aquisição de equipamentos e reagentes.
RESUMO
A proteção antiviral específica contra o vírus da síndrome da mancha
branca (WSSV) induzida via RNA de interferência (RNAi) e sua
influência na expressão de alguns genes imunológicos foram avaliados
em Litopenaeus vannamei. Os animais foram injetados com dsRNA de
sequência específica (vp28 do envelope viral), e posteriormente
desafiados com o WSSV. Camarões tratados com dsRNA vp28 tiveram
a infecção viral limitada, uma alta taxa de sobrevivência (73%) e
ausência do vírus em 80% dos sobreviventes, confirmando a ativação do
mecanismo antiviral específico via RNAi. Animais desafiados e não
tratados com dsRNA vp28 tiveram uma queda significativa no
hemograma (85%) e depleção gênica da argonauta, caspase-3, fator
antilipopolissacarídeo
(ALF),
pró-fenoloxidase
(proPO)
e
transglutaminase (TGase). Uma superexpressão gênica da proteína que
reconhece LPS e β-1,3-glicanas (LGBP) e do inibidor de proteases α2macroglobulina (α2M) foi verificada nesses animais, indicando o
envolvimento destas proteínas na fase aguda da infecção por WSSV. O
tratamento com dsRNA vp28 limitou a infecção e restabeleceu as
condições imunológicas gerais dos animais através do aumento do
hemograma e da modulação positiva dos genes argonauta, ALF, proPO
e TGase, sugerindo a participação destas proteínas na defesa do
organismo contra o WSSV. Os resultados deste estudo apontam para
uma potencial utilização do dsRNA vp28 como agente terapêutico antiWSSV e confirmam que a técnica de RNAi é uma abordagem
preventiva promissora e efetiva para o controle das viroses de
crustáceos.
Palavras chave: Litopenaeus vannamei, imunologia de crustáceos,
WSSV, RNA de interferência, dsRNA vp28, expressão gênica.
ABSTRACT
The main purpose of this study was to evaluate the specific antiviral
protection induced by the RNA-interference (RNAi) phenomenon in the
shrimp Litopenaeus vannamei during an experimental infection with the
white spot syndrome virus (WSSV) and the expression of several
immune genes through semi-quantitative RT-PCR. Shrimp were
injected with a dsRNA-specific sequence (vp28 protein from viral
envelope), and then challenged with the virus. In animals treated with
sequence-specific dsRNA the progress of viral infection was limited and
their survival (73%) and viral clearance (80%) were much higher than
in experimentally infected shrimp. These results confirmed that RNAi
mechanism is effectively capable to trigger a specific antiviral
protection in shrimp. Experimentally infected shrimps, not treated with
dsRNA exhibited a strong reduction on their haemocyte numbers (85%).
Also, the expression of different genes such as argonaute, caspase-3,
antilipopolysaccharide factor (ALF), prophenoloxidase (proPO) and
transglutaminase (TGase) was markedly down-regulated in these
animals. Interestingly, there was an overexpression of the
lipopolysaccharide and β-1,3-glucans binding protein (LGBP) and the
protease inhibitor α2-macroglobulin (α2M) genes in these infected
animals suggesting the involvement of these proteins in acute phase
WSSV-infection. The treatment with vp28-dsRNA was able to limit
viral infection and to restore the normal immunological conditions of
the animals. The treated animals maintained the number of their
hemocytes (immunocompetent cells) and the expression of several
immune protein genes (argonaute, ALF, proPO and TGase) at their
normal levels. These results suggest that vp28-dsRNA can serve as a
potential therapeutic tool to combat WSSV and that RNAi phenomenon
is effectively a promising and effective approach to control viral
diseases in crustaceans.
Key words: Litopenaeus vannamei, crustacean immune system, WSSV,
RNA interference, dsRNA vp28, gene expression.
LISTA DE FIGURAS
Figura 1. Esquema explicativo do desenho experimental. No
período de pré-infecção, os animais foram injetados com tampão
(G1 e G2) ou com dsRNA vp28 (G3) e, após 48h, infectados
(G2 e G3) ou não (G1) com WSSV. As coletas de hemolinfa
ocorreram 6h após injeção com dsRNA vp28 (G3); e 0, 3, 6, 24, 48
e 72h após a infecção com WSSV negativo (G1) ou positivo (G2)......46
Figura 2. Contagem total de hemócitos (CTH) em L. vannamei
imediatamente antes das injeções (0h); 6h após injeção com
dsRNA vp28 (G3); e 3, 6, 24, 48 e 72h pós-infecção com
inóculo negativo (G1) ou positivo (G2 e G3) para o vírus da
síndrome da mancha branca (WSSV). Letras maiúsculas
representam diferenças significativas (p<0,05) na CTH dentro de
um mesmo grupo nos diferentes períodos analisados. Letras
minúsculas representam diferenças significativas (p<0,05) na
CTH entre os grupos dentro de um mesmo período...... ...................52
Figura 3. Análise semiquantitativa por RT-PCR dos genes
argonauta, caspase-3, alfa 2-macroglobulina (α2M), fator
antilipopolissacarídeo (ALF), proteína que se liga a LPS e β-1,3glicanas (LGBP), pró-fenoloxidase (proPO) e transglutaminase
(TGase) nos hemócitos de L. vannamei. As análises foram feitas
antes do início das injeções (0h- expressão gênica basal); 6h
após injeção de dsRNA vp28 (G3) e 3, 6, 24, 48 e 72h após a
infecção com inóculo negativo (G1) ou positivo (G2 e G3) para
o vírus da síndrome da mancha branca (WSSV). Os valores
correspondem à razão entre a densidade de cada banda do gene
de interesse pela banda actina nos diferentes tempos. A
expressão relativa basal (0h) foi arbitrariamente estabelecida
com o valor 1 A densitometria das bandas foi realizada
utilizando-se o programa Scion Image®..................... ......................58
Figura 4. Expressão em relação ao controle dos genes: argonauta
(A), caspase-3 (B), alfa 2-macroglobulina (C), fator
antilipopolissacarídeo (D), proteína que se liga a LPS e β-1,3glicanas (E), pró-fenoloxidase (F) e transglutaminase (G) nos
hemócitos de L. vannamei antes do início das injeções (0h) e 3,
6, 24, 48 e 72h após a infecção com o vírus da síndrome da
mancha branca (WSSV) nos grupos G2 (tampão/WSSV+) e G3
(dsRNA/WSSV+)......................... ....................................................59
Figura 5. Porcentagem da mortalidade cumulativa dos animais em
cada tratamento durante 30 dias..... ..................................................60
Figura 6. Eletroforese em gel de agarose (1%) representando os
produtos amplificados por PCR em Tempo Real, com amostras
de DNA genômico de animais infectados com WSSV e
previamente tratados com dsRNA vp28 (linhas 1 a 18). A análise
foi feita 30 dias após a infecção, utilizando-se iniciadores VP19
(fragmento de 375 pb). M: marcador de peso molecular (100pb);
CN: Controle Negativo (sem adição de DNA); I: Inóculo
Positivo para WSSV. Linhas 7 a 10: animais WSSV
positivos............................................................................................61
LISTA DE TABELAS
Tabela 1. Pares de iniciadores utilizados para as análises de RTPCR de sete genes relacionados ao sistema imune em
Litopenaeus vannamei. Os iniciadores argonauta e proPO
referem-se à sequências de P. monodon e F. chinensis....... .......... 50
Tabela 2. Detecção de WSSV nos animais G1: tampão/WSSV-,
G2: tampão/WSSV+ e G3: dsRNA/WSSV+, por PCR
convencional, em um (+) ou dois passos (nested-PCR) (++). As
análises em cada período foram feitas com DNA genômico
extraído de um pool de 3 animais/grupo......................................... 61
LISTA DE ABREVIATURAS
α2M – α2- macroglobulina
A260/280nm – Razão da absorbância 260/280 nm
ALF – Fator antilipopolissacarídeo
CTH – Contagem total de hemócitos
dsRNA – double-stranded RNA (RNA dupla fita)
EDTA – Ácido diamino tetracético
HG – Hemócitos granulares
HGG – Hemócitos com grânulos grandes
HGP – Hemócitos com grânulos pequenos
HH – Hemócitos hialinos
IAP – Inhibitor of Apoptosis Proteins (Proteínas inibidoras de apoptose)
IFN – Interferon
IHHNV – Infectious Hypodermal and Hematopoietic Necrosis Virus
(Vírus da Necrose Hipodérmica e Hematopoietica)
IMNV – Infectious Myonecrosis Virus (Vírus da Mionecrose Infecciosa
Muscular)
LBP – Proteína que se liga a LPS
LGBP – Proteína que se liga a ambos β-glicanas e LPS
LPS – Lipopolissacarídeos
MAS – Solução anticoagulante Alsever modificada
NaCl – Cloreto de sódio
NaOH – Hidróxido de sódio
p – Nível de probabilidade
PAM – Peptídeos antimicrobianos
PAMPs – Padrões moleculares associados aos patógenos
pb – Pares de base
PCR - Reação em cadeia da polimerase
PG – Peptidoglicanas
proPO – Pró-fenoloxidase
PRPs – Proteínas de reconhecimento de padrão
RISC - RNA-induced silencing complex RNA (complexo multiproteico
de silenciamento induzido por RNA)
RNAi – RNA de interferência
RT-PCR – PCR em Tempo Real
SDS – Sodium dodecyl sulfate
TGase – Transglutaminase
TSV – Taura Syndrome Virus (Vírus da Síndrome de Taura)
WSS – White Spot Syndrome (Síndrome da Mancha Branca)
WSSV – White Spot Syndrome Virus (Vírus da Síndrome da Mancha
Branca)
YHV – Yellow Head Virus (Vírus da Cabeça Amarela)
SUMÁRIO
1. INTRODUÇÃO ................................................................................ 17
Sistema imune dos crustáceos ............................................................... 23
Respostas antivirais em crustáceos........................................................ 28
OBJETIVOS ......................................................................................... 37
Objetivo Geral....................................................................................... 37
Objetivos Específicos............................................................................ 37
1. INTRODUÇÃO ................................................................................ 39
2. MATERIAIS E MÉTODOS ............................................................. 43
2.1. Animais .......................................................................................... 43
2.2. Preparo do inóculo viral ................................................................. 43
2.3. Síntese do dsRNA vp28 ................................................................. 44
2.4. Ativação do sistema de RNA de interferência em L. vannamei injeção com dsRNA vp28 e infecção experimental com WSSV........... 44
2.5. Detecção da infecção dos camarões por WSSV............................. 46
2.6. Extração de hemolinfa.................................................................... 47
2.7. Contagem total de hemócitos (CTH).............................................. 48
2.8. Extração dos RNA totais e síntese do cDNA ................................. 48
2.9. Análise da expressão gênica semiquantitativa de algumas
proteínas imunológicas de L. vannamei ................................................ 48
3. ANÁLISE ESTATÍSTICA ............................................................... 51
4. RESULTADOS................................................................................. 51
4.1. Contagem Total de Hemócitos (CTH) ........................................... 51
4.2. Expressão gênica ............................................................................ 52
4.2.1. Argonauta: a proteína slicer do RISC do sistema RNAi.............. 53
4.2.2. Caspase-3: uma protease executora da via apoptótica ............... 53
4.2.3. Alfa 2-macroglobulina (α2M): um inibidor de proteases de
amplo espectro ...................................................................................... 54
4.2.4. Fator antilipopolissacarídeo (ALF): um peptídeo
antimicrobiano com atividade antiviral ................................................ 54
4.2.5. Proteína que se liga a LPS e β-1,3-glicanas (LGBP): uma
proteína de reconhecimento padrão...................................................... 55
4.2.6. Pró-fenoloxidase: um zimógeno do sistema de ativação da
proPO.................................................................................................... 56
4.2.7. Transglutaminase (TGase): a enzima-chave no processo de
coagulação da hemolinfa ...................................................................... 56
4.3. Mortalidade dos animais ................................................................ 60
4.4. Detecção do DNA viral (WSSV) nos tecidos dos animais............. 60
5. DISCUSSÃO..................................................................................... 62
6. REFERÊNCIAS................................................................................ 74
REFERÊNCIAS BIBLIOGRÁFICAS DA INTRODUÇÃO ................ 86
16
17
1. INTRODUÇÃO
Atualmente, devido à crescente estagnação da pesca e à procura
comercial na indústria alimentícia, a aquicultura vem constituindo uma
grande alternativa para o suprimento da demanda por proteína de origem
animal, apresentando elevado crescimento dentre os setores produtores
de alimento. Segundo a FAO (2006), em 1996 a pesca de captura
representava 73% da produção mundial de pescados, enquanto a
aquicultura participava com a pequena parcela de 27%. No entanto,
apenas 10 anos depois, em 2006, a pesca participava com 58% da
produção mundial de pescados, enquanto a aquicultura aumentou sua
contribuição em 42%.
A exploração indiscriminada dos estoques pesqueiros juntamente
com a crescente diferença entre a oferta de pescado capturado e a
demanda de consumo acabaram por tornar a aquicultura a alternativa
mais viável no mundo para produção de alimento de elevado valor
proteico (CAMARGO e POUEY, 2005). Dentro da maricultura, o
cultivo de camarões marinhos, tem se expandido de forma bastante
acelerada em diversos países ao redor do mundo, sendo responsável pela
maior parte do volume financeiro envolvido no comércio internacional
de frutos do mar (SEIFFERT et al., 2006).
A carcinicultura é uma atividade fundamental na pauta de
exportações de países em desenvolvimento, sendo responsável pela
geração de milhões de empregos. Atualmente, a China e a Tailândia são
os principais produtores mundiais, e, na América Latina, os principais
produtores são México, Equador e Brasil, sendo que as espécies mais
cultivadas no mundo atualmente são Litopenaeus vannamei e Penaeus
monodon (FAO, 2006).
O cultivo de camarões marinhos teve sua origem no Sudeste da
Ásia, onde por séculos, pescadores da região despescavam viveiros
abastecidos por marés. No entanto, a carcinicultura em seus moldes
atuais surgiu na década de 30 do século XX, quando cientistas japoneses
iniciaram os trabalhos de larvicultura com Marsupenaeus japonicus
(ROSEMBERRY, 1992).
No Brasil, a atividade teve início durante a década de 80 com a
introdução do peneídeo M. japonicus no Nordeste. Entretanto, apesar
desta espécie, na ocasião, ser a mais cultivada na Ásia, ela não se
adaptou bem às condições brasileiras, principalmente em função das
baixas salinidades nas zonas de produção (BARBIERI e OSTRENSKY,
2002). Seguiram-se então tentativas para a domesticação das espécies
nativas Litopenaeus schmitti (OSTRENSKY, 1997) e Farfantepenaeus
18
paulensis (WASIELESKY, 2000) nas regiões Sul e Sudeste, e
Farfantepenaeus subtilis no Norte e Nordeste (TAVARES e SANTOS,
2006). No entanto, a baixa produtividade e lucratividade destas espécies
levaram à desativação das fazendas.
Ainda na década de 80, a espécie L. vannamei foi introduzida no
país primeiramente na região Nordeste. Em Santa Catarina a espécie foi
inicialmente utilizada em 1998 pela UFSC (Universidade Federal de
Santa Catarina) e EPAGRI (Empresa de Pesquisa Agropecuária e
Extensão Rural do Estado de Santa Catarina) com o intuito de viabilizar
a atividade no Estado.
Conhecido como camarão branco do Pacífico, L. vannamei possui
uma distribuição natural que vai desde as águas do Oceano Pacífico, na
província de Sonora (México), até o sul de Tumbes, no Peru (PÉREZFARFANTE e KENSLE, 1997). Esta espécie possui excelentes
características zootécnicas, como rusticidade no manejo, boa conversão
alimentar, rápido crescimento, facilidades na reprodução em cativeiro,
além de poder ser cultivada em águas oligohalinas crescendo em
ambientes com salinidade variando de 0,5 a 40 ppt (NUNES, 2001). O
seu cultivo em águas interiores, sem influência de águas marinhas, já foi
implantado com sucesso em países como o Equador, México, Panamá,
Tailândia e Estados Unidos, sendo que no Brasil é encontrado também
nos estados do Ceará, Paraíba e Piauí (FIGUEIREDO et al., 2004).
Além disso, em função de sua típica coloração esbranquiçada esta
espécie apresenta uma alta aceitação no mercado americano (NUNES,
2001).
No Brasil, este peneídeo se adaptou às condições climáticas e
ambientais encontradas no Nordeste brasileiro, sendo que nesta região
são obtidas até 2,5 safras por ano. Em 2005, a produção brasileira de L.
vannamei chegou a 65 mil toneladas cultivadas em 15 mil hectares de
viveiros, sendo o Nordeste brasileiro a região que contribui com 95% do
total de cultivo (IDEMA, 2006). Com relação à Santa Catarina os dados
do setor mostraram um crescimento vigoroso da produção, com um
volume triplicado no período de 1999 e 2002, sendo que no último ano
desse período, houve um incremento de 97% no setor. A produtividade
cresceu de forma bastante acentuada, como resultado de uma melhora
no desempenho dos recursos tecnológicos em uso nos cultivos, e
também do desenvolvimento da indústria de alimento animal no país,
corroborando para o incremento do segmento de camarão cultivado
(BUGLIONE NETO, 2009).
Desta forma, a atividade veio nos últimos anos experimentando
uma rápida evolução, no sentido de intensificação dos sistemas e das
19
técnicas de produção, visando um aumento da lucratividade e eficiência
nos cultivos. No entanto, a intensificação dos cultivos na maioria das
vezes não leva em conta os aspectos ecológicos e fisiológicos dos
animais (BACHÈRE, 2000), tendo como consequência o surgimento de
enfermidades entre os animais e a degradação ambiental (BORGHETTI;
OSTRENSKY; BORGHETTI, 2003). Sabe-se também que, fatores
ambientais potencialmente estressores, como alterações nos parâmetros
físico-químicos da água, a presença de metais pesados, pesticidas
agrícolas e poluentes no ambiente, podem alterar o sistema imune dos
camarões, debilitando-os e propiciando a instalação de doenças (LE
MOULLAC e HAFFNER, 2000; CHANG; LEE; LIU, 2006). Além
disso, a água dos cultivos abriga naturalmente inúmeros microorganismos potencialmente patogênicos, e uma vez instalada a doença, a
transmissão entre os animais é rapidamente veiculada (BOYD, 1979).
Os camarões podem ser suscetíveis a diferentes patógenos
(parasitas, fungos, bactérias e vírus) que geram enfermidades, muitas
delas endêmicas e/ou pandêmicas, que causam sérias perdas econômicas
advindas da diminuição na produção. No entanto, atualmente as
infecções virais são as principais responsáveis por perdas econômicas
catastróficas na carcinicultura a nível mundial, especialmente no cultivo
de L. vannamei. Dentre elas destacam-se àquelas causadas pelos vírus da
necrose hipodérmica e hematopoiética (IHHNV), vírus da síndrome de
Taura (TSV), o vírus da cabeça amarela (YHV) e o vírus da síndrome da
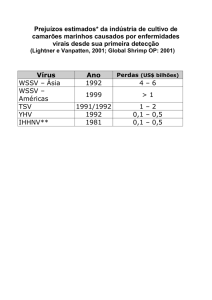
mancha branca (WSSV) (LIGHTNER, 2003).
Assim como em outros países, a carcinicultura no Brasil também
encontrou limitações ao seu desenvolvimento. Em 2004, com o advento
da ação antidumping imposta pelos Estados Unidos, associado a fatores
como a desvalorização do dólar, o setor camaroneiro perdeu
competitividade nas exportações (ROCHA, 2007). Somado a isso, duas
viroses causaram sérios prejuízos à carcinicultura nacional: a
Mionecrose Infecciosa (NIM) nos cultivos da Região Nordeste, causada
pelo vírus da mionecrose infecciosa (IMNV), e a Síndrome da Mancha
Branca (WSS) em Santa Catarina, causada pelo vírus da síndrome da
mancha branca (WSSV). Juntas, estas duas viroses rebaixaram o posto
brasileiro de líder americano na produção de camarão, obtido em 2003
graças as suas 94.000 toneladas de produção, para um pouco atrativo
terceiro lugar, precedido pelo México e Equador. Além disso, o Brasil
perdeu a liderança mundial em termos de produtividade com perdas na
produção na ordem de 40 mil toneladas entre o período de 2003 a 2005
(SEIFFERT et al., 2006).
20
A síndrome da mancha branca é considerada a enfermidade viral
mais devastadora para a indústria camaroneira registrada até o momento,
sendo que o WSSV vem dizimando populações inteiras de cultivo
(LIGTHNER, 2003; FLEGEL, 2006). Segundo a OIE (2006) as perdas
na produção devido ao WSSV passam de 10 bilhões de dólares desde
1993 em todo o mundo. Mortalidades massivas podem ocorrer em um
período muito curto, sendo que em casos mais severos podem chegar a
100% entre 2 a 7 dias após a detecção dos sinais clínicos (CHOU et al.,
1995; CHANG et al., 1999). Este vírus foi detectado pela primeira vez
em Taiwan, em 1992, de onde se espalhou rapidamente para a maioria
dos países produtores (CAI et al., 1995; FLEGEL, 1997;
ROSEMBERRY, 2002). Muito recentemente, a ocorrência deste vírus
foi também detectada de forma natural em espécies nativas de
crustáceos
na
Argentina
(MARTORELLI;
OVERSTREET;
JOVONOVICH, 2010).
No caso do Brasil, os primeiros surtos da WSS foram detectados
em cultivos de Santa Catarina, em 2004, onde apenas 200 dos 1.600
hectares de viveiros em produção não foram afetados pela enfermidade,
resultando assim em prejuízos na ordem de R$ 6 milhões (SEIFFERT et
al., 2006). Em termos de produção, em 2004 a carcicicultura catarinense
estava em franca expansão comercializando 4.189 t do crustáceo,
enquanto que em 2007 essa cifra caiu para apenas 300 t
(EPAGRI/CEDAP, 2008).
A WSS geralmente se manifesta quando ocorre um desequilíbrio
no cultivo, advindo, por exemplo, de altas densidades populacionais, má
preparação do solo, baixa qualidade de água, excesso de matéria
orgânica no solo e mudanças bruscas na temperatura da água
(PEREIRA; LEGAT; LEGAT, 2006; BUCHELI e GARCIA, 2005;
SEIFFERT; WINCLER; MAGGIONI, 2005). Os principais sinais
clínicos da enfermidade são o surgimento de calcificações brancas
arredondadas na parte interna do exoesqueleto do rostro, atividade
errática do camarão na periferia do viveiro, redução rápida no consumo
de alimento, atividade metabólica reduzida dos animais e coloração
avermelhada no corpo (BUCHELI e GARCIA, 2005).
As manchas brancas, cuja presença confere denominação à
enfermidade, são decorrentes de depósitos de sais de cálcio na epiderme
cuticular dos camarões infectados. No entanto, o mecanismo de
formação destas manchas ainda é pouco conhecido, mas sabe-se que a
infecção por WSSV pode provocar uma disfunção do epitélio
subcuticular do animal, resultando assim no acúmulo dos sais de cálcio
(WANG et al., 1999). Em L. vannamei estas manchas nem sempre são
21
observadas, ocorrendo apenas em estágios mais avançados da
enfermidade (NUNES e MARTINS, 2002). Além disso, os pontos
brancos na cutícula, nem sempre constituem um sinal clínico desta
síndrome, podendo ser causada por doenças de origem bacteriana, ou até
por condições ambientais, como alta alcalinidade da água (WANG et al.,
2002).
O WSSV é um vírus envelopado, baciliforme e pertence ao
gênero Whispovirus e à família Nimaviridae (vide revisões de
ESCOBEDO-BONILLA et al., 2008; BONAMI, 2008). Seu material
genético é composto por uma molécula de DNA dupla fita, circular e
longa, com tamanho variável entre 293 kpb a 307 kbp (van HULTEN et
al., 2001; YANG et al., 2001; CHEN et al., 2002), sendo encontradas
várias linhagens de vírus que são extremamente virulentas e patogênicas
para várias espécies de peneídeos e outros crustáceos (DURAND et al.,
1997). O WSSV infecta especialmente as células de origem ectodermal
e mesodermal, incluindo células da epiderme, brânquias, intestino
(CHANG et al., 1996), órgão linfoide (DURAND et al, 1996; CHANG
et al, 1998), músculo, coração (KOU et al., 1998), gônadas (LO et al.,
1997), hemócitos e células associadas ao sistema nervoso central
(RAJENDRAN et al., 1999; WANG et al., 1999).
Atualmente, muitos estudos têm sido realizados com o objetivo
de caracterizar e analisar a função de proteínas do WSSV, especialmente
àquelas do envelope viral que estão diretamente associadas à adesão e
entrada do vírus na célula hospedeira, sendo elas: vp19, vp26, vp28,
vp68, vp281 e vp466 (van HULTEN; GOLDBACH; VLAK, 2000; YI
et al., 2004; WU et al., 2005).
A confecção de vacinas, no termo clássico da palavra, é inviável
para invertebrados, como os crustáceos, devido ao fato do organismo
destes animais não estar apto a produzir anticorpos antígeno-específicos
e células de memória, não produzindo, portanto, uma memória
imunológica a longo prazo (vide revisão BARRACCO; PERAZZOLO;
ROSA, 2008). Desta forma, camarões não podem ser imunizados da
mesma forma que os peixes. No entanto, atualmente, muitos estudos têm
relatado uma proteção a médio prazo contra mortalidades ocasionadas
por vírus, como o WSSV, por meio de técnicas de “vacinação” que
utilizam como agentes terapêuticos proteínas virais purificadas ou
recombinantes, vírus inativados ou “vacinas” de DNA (YI et al., 2004;
WU et al., 2005; WITTEVELDT; VLAK; van HULTEN, 2004; SYEDMUSTHAQ et al., 2009).
As proteínas do envelope viral, especialmente aquelas associadas
à adesão e entrada do vírus na célula hospedeira, são as mais visadas
22
para produção destas “vacinas”. Dentre elas, a vp28 é considerada uma
das proteínas mais eficazes, sendo capaz de conferir os melhores níveis
de proteção em crustáceos contra o WSSV (vide revisão de JOHNSON;
van HULTEN; BARNES, 2008; KUMAR et al., 2008). Esta proteína é a
mais abundante no envelope viral e além de estar envolvida na adesão
do WSSV às células do hospedeiro (YI et al., 2004), a vp28 interage
com proteínas intracelulares do hospedeiro, tais como a Rab7GTPase
(SRITUNYALUCKSANA et al., 2006), a proteína de choque térmico
HSP70 (XU et al., 2009) e o transdutor de sinal e ativador de transcrição
STAT (LIU et al., 2007). Quando utilizada como “vacina”, o
mecanismo de ação da vp28 é o de funcionar como um “bloqueador
físico” dos receptores celulares para o vírus, impedindo a ligação
posterior e entrada do mesmo na célula hospedeira e limitando, por
consequência, o processo infeccioso. (JOHNSON; van HULTEN;
BARNES, 2008). No entanto, na ausência de uma expressão continuada
das proteínas virais no organismo, como ocorre habitualmente, a
reciclagem dos receptores/proteínas ligantes nas células do hospedeiro
leva ao retorno da “suscetibilidade” do animal ao vírus em um curto
período de tempo (PERAZZOLO; BARRACCO; ROSA, 2010). Sendo
assim os métodos de “vacinação” disponíveis para crustáceos conferem,
no máximo, uma imunoproteção a médio prazo (vide revisão de
PERAZZOLO; BARRACCO; ROSA, 2010).
O estudo do mecanismo de resistência de camarões frente a
infecções causadas por vírus é ainda pouco compreendido, sendo assim,
como descrito anteriormente, as infecções virais o principal fator
realmente limitante para o sucesso da carcinicultura, na atualidade. No
entanto, é importante salientar que a presença de enfermidades não
inviabiliza a atividade, exigindo, porém adaptações à nova realidade.
Todos os países produtores de camarão já tiveram problemas com
enfermidades, e ainda assim a carcinicultura é considerada uma
atividade lucrativa (PEREIRA; LEGAT; LEGAT, 2006). Porém, no que
diz respeito ao controle e prevenção de doenças nos cultivos, métodos
convencionais de tratamento, como o uso de drogas químicas, não
apresentam eficácia contra viroses. Atualmente, a profilaxia e o controle
de enfermidades em cultivos estão basicamente relacionados à tentativa
de reduzir as condições de estresse dos animais através do uso de
práticas adequadas de manejo.
Convém lembrar que, além de infectar diferentes tecidos do
camarão, o WSSV compromete seriamente o sistema imunológico dos
animais, uma vez que as suas células imunocompetentes ou hemócitos
são as principais células-alvo para a sua replicação (WANG et al.,
23
2002). Neste contexto, a mais ansiada e urgente contribuição para a
carcinicultura na atualidade é indiscutivelmente o controle das infecções
virais (PERAZZOLO; BARRACCO; ROSA, 2010). Desta forma, tornase cada vez mais importante a busca de terapias eficazes embasadas no
conhecimento prévio do sistema imunológico desses animais, uma vez
que estudos dessa natureza poderão auxiliar na compreensão dos
mecanismos envolvidos nas respostas antivirais, na interação entre
patógeno e hospedeiro e na relação suscetibilidade/resistência desses
animais frente a esses parasitas.
Sistema imune dos crustáceos
Assim como mencionado anteriormente, os crustáceos possuem
apenas um sistema imune inato ou natural, portanto, não possuem a
imensa gama de anticorpos específicos e células de memória, que
compõem o sistema imune adaptativo dos vertebrados. Devido a esta
característica, é importante ressaltar, mais uma vez, que não é possível o
desenvolvimento de vacinas que os proteja a longo prazo contra
infecções reincidentes (vide revisão de PERAZZOLO; BARRACCO;
ROSA, 2010). No entanto, devemos salientar que a ideia de que os
crustáceos, assim como outros invertebrados, contam apenas com um
sistema de defesa desprovido de especificidade e memória imunológica
vem sendo recentemente contestada, frente às novas descobertas que
apontam para a potencial presença de uma “imunidade adaptativa
alternativa” (vide revisões de KURTZ e FRANZ, 2003; KURTZ e
ARMITAGE, 2006; JOHNSON; van HULTEN; BARNES, 2008).
O sistema circulatório dos crustáceos é do tipo aberto ou
semiaberto, por onde transita um tecido fluido denominado hemolinfa,
que corresponde ao sangue dos vertebrados. Sendo assim, o sistema
imune dos crustáceos está relacionado à sua hemolinfa, que é composta
por uma fração celular (hemócitos) e uma fração líquida (plasma), onde
estão dissolvidos os fatores humorais. Os hemócitos e os fatores
humorais atuam de maneira integrada na proteção do organismo contra
invasões e infecções por patógenos, garantindo assim sua sobrevivência
(vide revisão de BARRACCO; PERAZZOLO; ROSA, 2008).
Os hemócitos são as células imunocompetentes, responsáveis
pelas respostas celulares de defesa, e respondem à invasão de microorganismos e parasitas destruindo-os por fagocitose ou isolando-os em
agregados hemocíticos através da formação de nódulos e cápsulas
24
(MILLAR e RATCLIFFE, 1994). Os mecanismos de lise e morte dos
patógenos são ainda auxiliados pela atuação de moléculas microbicidas
e/ou citotóxicas, exocitadas pelos hemócitos no sítio de infecção, ou
resultantes da clivagem proteolítica de proteínas de sistemas de defesa
auxiliares (LEE e SÖDERHÄLL, 2002). Segundo suas características
morfofisiológicas, os hemócitos são classificados em: hemócitos
hialinos (HH), hemócitos com grânulos pequenos (HGP) e hemócitos
com grânulos grandes (HGG) (SÖDERHÄLL e SMITH, 1983), sendo
que a proporção na hemolinfa de cada tipo celular em animais sadios
pode variar drasticamente conforme a espécie (GARGIONI e
BARRACCO, 1998; HOLMBLAD e SÖDERHÄLL, 1999).
De uma maneira geral, os HHs de crustáceos são descritos como
os menores hemócitos, com uma alta relação núcleo citoplasmática e
contendo de nenhum a poucos grânulos (vide revisão de BARRACCO;
PERAZZOLO; ROSA, 2008). Para alguns autores, esse tipo de
hemócito pertence a uma linhagem celular distinta daquela dos
hemócitos granulares e está essencialmente relacionado aos mecanismos
de coagulação (HOSE; MARTIN; GERARD, 1990). Outros autores
afirmam que os HHs são as principais células fagocíticas
(JOHANSSON et al., 2000) e correspondem a uma forma intermediária
da linhagem de hemócitos granulares (van de BRAAK et al., 2002).
Os hemócitos granulares (HGPs e HGGs) são descritos como
células de citoplasma mais abundantes e ricas em grânulos de diversos
tamanhos e conteúdos (BARRACCO; PERAZZOLO; ROSA, 2008),
estando especialmente envolvidos na fagocitose de micro-organismos,
na formação de cápsulas e nódulos, na produção de moléculas
citotóxicas e microbicidas capazes de lisar e/ou degradar os patógenos
invasores e a maioria das proteínas envolvidas, direta ou indiretamente,
com o sistema imune dos crustáceos (HOSE; MARTIN; GERARD,
1990; GARGIONI e BARRACCO, 1998; DESTOUMIEUX et al., 2000;
van de BRAAK et al., 2002).
A primeira etapa de defesa dos crustáceos envolve o
reconhecimento dos agentes infecciosos por proteínas/receptores de
reconhecimento de padrões (PRPs, do inglês pattern recognition
proteins), situados na membrana dos hemócitos ou dissolvidos no
plasma e que reconhecem padrões moleculares expressos
especificamente nos patógenos (PAMPs, do inglês pathogen-associated
molecular patterns), e que estão ausentes no hospedeiro. As principais
PRPs caracterizadas em crustáceos reconhecem os seguintes PAMPs:
lipopolissacarídeos (LPS) da superfície de bactérias Gram-negativas,
peptidoglicanas de parede de bactérias Gram-positivas, β-glicanas da
25
parede de fungos e o RNA dupla fita (dsRNA, do inglês doublestranded RNA), produzido por vários vírus durante a sua replicação no
hospedeiro (LEE e SÖDERHÄLL, 2002).
Os vírus são endoparasitas intracelulares extremamente simples
formados por um capsídeo proteico que abriga um ácido nucleico (DNA
ou RNA, na forma de fita simples ou dupla) e que em alguns casos pode
estar envolvido por uma capa lipídica, derivada da membrana celular do
hospedeiro. A estrutura viral é desprovida dos PAMPs comumente
encontrados
em
bactérias
e
fungos
(carboidratos:
LPS/peptidoglicanas/β-1,3-glicanas).
Dentre as PRPs encontradas nos crustáceos destacam-se as
lectinas, que reconhecem açúcares específicos da superfície de
diferentes micro-organismos, as LBP e Mas-like que reconhecem
lipopolissacarídeos (LPS) da parede de bactérias Gram-negativas, as
βGBP e GBP que reconhecem β-1,3-glicanas da parede de fungos e a
LGBP que reconhece ambos LPS e β-1,3-glicanas (vide revisão de
JIRAVANICHPAISAL; LEE; SÖDERHÄLL, 2006). Uma vez dentro
do hospedeiro, os padrões moleculares do patógeno são reconhecidos e
ligados às suas respectivas PRPs e, no caso dos crustáceos, iniciam a
ativação dos hemócitos, desencadeando uma resposta imune celular
através da produção/liberação de uma série de moléculas
imunoefetoras/imunoreguladoras, bem como a modulação da expressão
de genes imunológicos específicos (vide revisão de BARRACCO;
PERAZZOLO; ROSA, 2008). No caso da LGBP, esta proteína já foi
identificada em crustáceos e insetos, sendo que em crustáceos são
proteínas pequenas (36-46 kDa) produzidas pelos hemócitos e/ou no
hepatopâncreas (vide revisão de BARRACCO; PERAZZOLO; ROSA,
2008). Em peneídeos, existem três relatos sobre a clonagem do gene da
LGBP: Litopenaeus stylirostris (ROUX et al., 2002), L. vannamei
(CHENG et al., 2005) e Fenneropenaeus chinensis (DU; ZHAO;
WANG, 2007), sendo que análises da expressão diferencial
demonstraram que ocorre uma modulação evidente deste gene nos
animais durante a fase aguda de infecção pelo WSSV (ROUX et al.,
2002; DHAR et al., 2003; YEH et al., 2009).
O reconhecimento do agente invasor desencadeia a ativação dos
hemócitos que podem migrar para os sítios de infecção, onde realizam a
fagocitose e/ou a formação de agregados celulares densos em torno das
partículas invasoras (nódulos e/ou cápsulas). Os patógenos podem,
então, ser neutralizados ou destruídos através de diferentes mecanismos,
incluindo: a liberação de enzimas degradativas, a ativação dos
componentes do sistema pró-fenoloxidase (proPO) e a produção de
26
compostos citotóxicos, como as espécies reativas de oxigênio (ROIs) e
nitrogênio (RNIs).
Além disso, os hemócitos de crustáceos produzem diferentes
proteínas e/ou peptídeos antimicrobianos (PAM), que apresentam uma
atividade microbicida rápida e potente contra um amplo espectro de
micro-organismos, incluindo bactérias, fungos filamentosos, leveduras,
e em alguns casos também contra vírus e protozoários (BACHÈRE et
al., 2004; REDDY; YEDERY; ARANHA, 2004).
Em camarões, os PAMs são produzidos constitutivamente pelos
hemócitos, sendo armazenados em seus grânulos, e seu mecanismo de
ação normalmente se dá a nível da membrana do micro-organismo. Os
PAMs podem apresentar uma atividade detergente sobre a membrana do
micro-organismo, uma vez que sendo eles na maioria moléculas
catiônicas interagem por atração eletrostática com os fosfolipídeos
aniônicos da membrana das bactérias, por exemplo, formando grandes
poros e gerando um influxo descontrolado de solutos e um
extravasamento do conteúdo citoplasmático, resultando na morte do
micro-organismo (BACHÈRE et al., 2004; BULET; STÖCKLIN;
MENIN, 2004).
Os principais peptídeos antimicrobianos de crustáceos são as
peneidinas (DESTOUMIEUX et al., 1997), as crustinas (GROSS et al.,
2001; BARTLETT et al., 2002) e os fatores antilipopolissacarídeos
(ALFs) (GROSS et al., 2001; SUPUNGUL et al., 2002). Os ALFs já
foram encontrados em vários peneídeos, dentre os quais Litopenaeus
setiferus (GROSS et al., 2001), P. monodon (SUPUNGUL et al., 2002),
F. chinensis (LIU et al., 2005), M. japonicus (NAGOSHI et al., 2006)
L. vannamei (de la VEGA et al., 2008), F. paulensis e L. schmitti
(ROSA; STOCO; BARRACCO, 2008) sendo ativos contra bactérias
Gram-positivas e Gram-negativas, e contra fungos filamentosos
(SOMBOONWIWAT et al., 2005; NAGOSHI et al., 2006). Além disso,
estudos recentes têm apontado para a relevância deste PAM na limitação
das infecções por WSSV em crustáceos (LIU et al., 2006;
THARNTADA et al., 2009).
Outra importante molécula, que é exocitada durante a ativação do
hemócito, é a enzima transglutaminase (TGase) que induz o processo de
coagulação da hemolinfa nos crustáceos. TGases são enzimas Ca2+
dependentes, usualmente compartimentalizadas dentro dos hemócitos
(LORAND et al., 1979; GREENBERG; BRICKBICHLER; RICE,
1991), e que promovem ligações covalentes γ-glutamil-ε-lisina entre
moléculas da proteína de coagulação (CP) (LORAND e CONRAD,
1984), formando assim, coágulos estáveis no local do ferimento. A
27
coagulação da hemolinfa é um mecanismo imunológico essencial para a
sobrevivência de invertebrados que possuem um sistema circulatório
aberto, como é o caso dos crustáceos, pois previne a perda da hemolinfa
e a disseminação dos patógenos pela cavidade corpórea do animal, por
ocasião de uma injúria no organismo (vide revisão BARRACCO;
PERAZZOLO; ROSA, 2008). Além disso, a coagulação é muito
importante durante o processo de muda, fase esta em que o animal
torna-se potencialmente mais exposto às injúrias e ataques microbianos.
Conforme dito anteriormente, durante as infecções, LPS e
peptidoglicanas de bactérias, e β-1,3-glicanas de fungos se ligam
diretamente a receptores hemocitários ou através de PRPs plasmáticas
ligadas aos respectivos PAMPs, induzindo uma degranulação ou
exocitose regulada, com liberação de várias moléculas imunoefetoras,
dentre as quais as moléculas do sistema de ativação da pró-fenoloxidase
(proPO). O sistema proPO consiste em uma cascata proteolítica
composta por vários zimógenos de proteases, pela proPO, além das
PRPs e moléculas associadas (vide revisões de LEE e SÖDERHÄLL,
2002; JIRAVANICHPAISAL; LEE; SÖDERHÄLL, 2006), e cujo
produto final é a melanina.
Em relação ao papel imunológico do sistema proPO, sabe-se que
esta via gera transitoriamente moléculas altamente citotóxicas, como as
quinonas, hemiquinonas e radicais livres como as ROIs, uma vez que
ocorre o consumo de oxigênio molecular que leva à destruição dos
patógenos invasores, sendo que alguns estudos relatam uma modulação
na expressão gênica da proPO, em camarões infectados por WSSV
(ROUX et al., 2002; AI et al., 2008; YEH et al., 2009). Contudo, a
ativação do sistema proPO deve ser estritamente regulada para evitar
uma ativação indesejada ou generalizada no organismo do crustáceo.
Para isso, os animais possuem inibidores plasmáticos de proteases,
como a pacifastina (LIANG; SOTTRUP-JENSEN; SÖDERHÄLL,
1997), os inibidores da família Kazal (JOHANSSON; KEYSER;
SÖDERHÄLL, 1994; JIMÉNEZ-VEGA e VARGAS-ALBORES,
2005), a serpina (LIANG e SÖDERHÄLL, 1995) e a α2-macroglobulina
(α2M) (HERGENHAHN; HALL; SÖDERHÄLL, 1988).
A α2M é uma glicoproteína plasmática constitutivamente
sintetizada pelos hemócitos (GROSS et al., 2001; RATTANACHAI et
al., 2004) que atua como um inibidor de proteases de amplo espectro
(serino, aspartato, cisteína e metalo-proteases), inibindo tanto as
proteases endógenas do hospedeiro, como aquelas exógenas produzidas
pelo patógeno durante o processo infeccioso (vide revisão de
ARMSTRONG, 2006). O papel imunomodulador da α2M nos
28
crustáceos ainda não está bem estabelecido. No entanto, sabe-se que ela
inibe parcialmente serino-proteases ativadoras do sistema proPO do
lagostim Pacifastacus leniusculus (HERGENHAHN; HALL;
SÖDERHÄLL, 1988; ASPÁN; HALL; SÖDERHÄLL, 1990) e
proteases produzidas pelo fungo Aphanomyces spp, um patógeno
conhecido para lagostins (DIEGUEZ-URIBEONDO e CERENIUS,
1998). Além disso, foi relatado que ocorre um aumento na expressão da
α2M em P. monodon infectados pelo WSSV (TONGANUNT et al.,
2005).
Respostas antivirais em crustáceos
Além dos processos acima descritos, os crustáceos possuem
ainda respostas antivirais, que, comparadas às respostas
antimicrobianas, são ainda muito pouco conhecidas (vide revisão de
LIU; SÖDERHÄLL; JIRAVANICHPAISAL, 2009). Atualmente, são
reconhecidos pelo menos três mecanismos antivirais nos crustáceos: a
apoptose celular, o sistema de RNA de interferência (RNAi) e um
mecanismo semelhante ao sistema interferon (IFN) dos vertebrados
(vide revisões de ROBALINO et al., 2007; LIU; SÖDERHÄLL;
JIRAVANICHPAISAL, 2009; PERAZZOLO; BARRACCO; ROSA,
2010).
A apoptose ou morte celular programada é um processo
fisiológico controlado geneticamente e que está envolvido na regulação
da homeostase do organismo, atuando tanto no desenvolvimento
embrionário, como no controle dos tecidos e também nas respostas
imunológicas contra vírus. A indução do processo apoptótico inicia-se
em resposta a uma variedade de estímulos, incluindo infecções virais, e
envolve várias alterações morfológicas celulares, como a condensação
da cromatina, a fragmentação do núcleo, o encolhimento celular e a
lobulação da membrana (vide revisões de ROULSTON; MARCELLUS;
BRANTON, 1999; CHOWDHURY; THARAKAN; BHAT, 2008). Por
fim, ocorre a fragmentação da célula em pequenas vesículas revestidas
por membrana, denominadas de corpos apoptóticos, que em geral são
rapidamente fagocitados pelas células vizinhas ou por macrófagos, sem
haver qualquer liberação intracelular de seu conteúdo, como no caso de
uma necrose, que desencadeia um processo inflamatório. Uma célula
que sofre apoptose morre de forma “silenciosa”, sem alertar as células
circunvizinhas e sem gerar um processo inflamatório (vide revisão de
29
ROULSTON; MARCELLUS; BRANTON, 1999; CHOWDHURY;
THARAKAN; BHAT, 2008).
Muitas proteínas e componentes virais perturbam a fisiologia
celular e fornecem sinais celulares que induzem a apoptose. Em
mamíferos, alguns componentes das vias pró-apoptóticas (a proteína
quinase R, a RNase L, o fator regulatório 3 do IFN e a quinase Nterminal c-Jun), podem ser ativados pelo dsRNA viral (GANTIER e
WILLIAMS, 2007). Logo, durante uma infecção viral, o dsRNA estaria
envolvido tanto na sinalização intracelular ativando os sistemas de
RNAi e interferon, como também induzindo a apoptose celular.
A maquinaria intracelular responsável pela apoptose parece ser
semelhante em todas as células animais e é mediada por enzimas
nucleases e proteases da família das caspases, que desencadeiam a morte
celular através da clivagem de proteínas específicas do núcleo e
citoplasma (vide revisão de CHOWDHURY; THARAKAN; BHAT,
2008). As caspases existem nas células como precursores inativos (prócaspases) que são normalmente ativados por clivagem de outras
caspases, resultando em uma cascata de amplificação proteolítica. Estas
proteases podem ser classificadas de acordo com seu pró-domínio e seu
papel na apoptose: caspases iniciadoras possuem pró-domínios longos e
estão envolvidas na iniciação da cascata proteolítica, enquanto as
caspases efetoras apresentam pró-domínios curtos ou inexistentes, e são
responsáveis pela clivagem de substratos celulares (RUPNARAIN et
al., 2004). Desta forma, a ativação inicial de um pequeno número de
caspases iniciadoras pode levar, através de uma cascata, à ativação de
um grande número de caspases executoras, que por sua vez, clivam
proteínas-chave na célula, incluindo proteínas citosólicas e das lâminas
nucleares (vide revisão de CHOWDHURY; THARAKAN; BHAT,
2008).
Até agora, 14 diferentes caspases já foram identificadas em
mamíferos, sendo que as caspases 2, 8, 9 e 10 são classificadas como
iniciadoras e as caspases 3, 6 e 7 como efetoras ou executoras (vide
revisão de GRIVICICH; REGNER; ROCHA, 2007; HUNTER;
LACASSE; KORNELUK, 2007). Em camarões, genes codificantes
tanto para caspases efetoras em Penaeus merguiensis (PHONGDARA;
WANNA; CHOTIGEAT, 2006), P. monodon (WONGPRASERT;
SANGSURIYA;
PHONGDARA,
2007)
e
L.
vannamei
(RIJIRAVANICH; BROWDY; WITHYACHUMNARNKUL, 2008),
quanto para uma caspase iniciadora em M. japonicus (WANG et al.,
2008) já foram identificados e clonados, sendo que em todos esses
peneídeos as caspases são induzidas pelas infecções com WSSV.
30
Um fato relevante a ser considerado é que a apoptose mostra-se
apenas efetiva como resposta antiviral quando acionada em estágio
precoce da infecção. Neste caso, ela pode limitar a produção de
partículas virais e reduzir ou eliminar a propagação da progênie viral
para outros tecidos. No entanto, muitos vírus conseguem retardar o
processo apoptótico, matando as células apenas no final do ciclo
infeccioso (vide revisão de ROULSTON; MARCELLUS; BRANTON,
1999). Este processo tardio é altamente indesejável, uma vez que vírus
empacotados em corpos apoptóticos não são detectados pelo sistema
imune do hospedeiro e acabam sendo fagocitados pelas células vizinhas,
propagando assim a infecção viral em seu organismo (BARRACCO;
PERAZZOLO; ROSA, 2008).
Assim, muitos vírus desenvolveram este mecanismo, ao longo da
evolução como forma de se evadir imunologicamente e garantir a sua
replicação e patogenicidade no hospedeiro. Como exemplo desse fato, é
comum os vírus produzirem proteínas antiapoptóticas de diferentes
classes. No caso do WSSV, um gene codificante para uma proteína
antiapoptótica denominada WSSV449 (ou ORF 390) já foi identificado
em seu genoma, sendo considerada uma nova classe de genes de
inibidores de apoptose (WANG et al., 2004). Em estudo posterior, LEU
et al. (2008) mostraram que a WSSV449 recombinante era capaz de se
ligar in vitro a uma caspase executora de P. monodon, inibindo a sua
atividade pró-apoptótica e confirmando, assim, a atividade
antiapoptótica dessa proteína viral. Sendo assim, estas proteínas virais
antiapoptóticas conseguem retardar o processo apoptótico na célula
infectada, em tempo hábil para impedir a progressão da infecção viral no
hospedeiro.
Por outro lado, o próprio hospedeiro possui reguladores
intracelulares de apoptose, como os da família de proteínas IAP (do
inglês, inhibitor of apoptosis proteins), em um processo natural para a
regulação da apoptose celular. As IAPs são as únicas proteínas
endógenas conhecidas capazes de regular tanto caspases ativas
iniciadoras (caspase 9), quanto efetoras (caspases 3 e 7), inibindo assim
a apoptose (LISTON; FONG; KORNELUK, 2003; HUNTER;
LACASSE; KORNELUK, 2007). As primeiras IAPs foram
originalmente descobertas como proteínas produzidas por alguns vírus
de insetos, sendo hoje detectadas em diversas espécies de invertebrados
e vertebrados (LISTON; FONG; KORNELUK, 2003). No caso dos
crustáceos, muito recentemente foi identificado e clonado o gene
codificante para uma IAP no camarão P. monodon (LEU et al., 2008).
31
Outro mecanismo antiviral importante dos vertebrados é o
sistema interferon (IFN). Os interferons (IFNs) são citocinas que
interferem na replicação viral e induzem um estado antiviral
inespecífico no hospedeiro. Em mamíferos, as infecções virais induzem
à produção de interferons que estimula a expressão de diversos genes
implicados nas respostas antivirais inespecíficas. Um potente indutor do
sistema IFN de vertebrados é o dsRNA, que como dito anteriormente, é
produzido pela maioria dos vírus durante seu ciclo de replicação dentro
do hospedeiro sendo, portanto, uma molécula diretamente associada às
infecções virais (JACOBS e LANGLAND, 1996). Nos invertebrados,
mais especificamente nos crustáceos, nenhuma molécula ou gene com
homologia estrutural aos IFNs de mamíferos foi, até o momento,
encontrada (ROSA e BARRACCO, 2008). Apesar disso, relatos
recentes indicam a presença nestes animais de moléculas análogas,
porém não homólogas aos IFNs de vertebrados, participando da defesa
antiviral inespecífica dos crustáceos (vide revisões ROBALINO et al.,
2007; LIU; SÖDERHÄLL; JIRAVANICHPAISAL, 2009).
Robalino et al. (2004) demonstraram um estado antiviral
inespecífico em L. vannamei injetados com um dsRNA de sequência
inespecífica, sendo observado um aumento na sua resistência contra
infecções posteriores por dois vírus não relacionados, o TSV e o WSSV,
ou seja, muito semelhante ao estado antiviral induzido pelos IFNs dos
mamíferos. Nesse estudo, camarões foram injetados com um
oligonucleotídeo sintético denominado poly (C:G) (polycytidylicpolyguanylic acid), que mimetiza um dsRNA viral e desencadeia
respostas antivirais inespecíficas. Os animais tratados tiveram um
aumento significativo na sua resistência a infecções posteriores tanto por
TSV, como por WSSV.
Durante as infecções por vírus, as células dos mamíferos
reconhecem dsRNA viral através de receptores celulares específicos do
tipo TLRs (do inglês, Toll-like proteins), como o Toll-like 3 (TLR3)
(ALEXOPOULOU et al., 2001) e os RLRs (do inglês, Retinoic acid –
inducible gene (RIG)-I- like receptors) (SAITO e GALE Jr., 2008).
A presença de receptores Toll em peneídeos já foi verificada nos
hemócitos de L. vannamei (YANG et al., 2007), P. monodon (ARTS et
al., 2007), M. japonicus (MEKATA et al., 2008), e F. chinensis (YANG
et al., 2008). Aparentemente, os receptores Toll até agora identificados
em camarões estão relacionados a respostas antibacterianas, uma vez
que alguns autores observaram um aumento na expressão gênica destes
receptores após injeção dos animais com peptidoglicanas (MEKATA et
al., 2008), Vibrio anguillarum (YANG et al., 2008) e Vibrio harveyi
32
(WANG et al., 2010). Sendo assim, até o momento ainda não foi
demonstrado um envolvimento de TLRs na resposta antiviral de
camarões (ARTS et al., 2007; MEKATA et al., 2008; LABREUCHE et
al., 2009; WANG et al., 2010). Apesar disto, pode-se supor que devam
existir outras famílias de receptores Toll ainda não detectadas nos
peneídeos (LABREUCHE et al., 2009) e que se relacionem às respostas
antivirais.
Além de induzir o sistema IFN e a apoptose em vertebrados, o
padrão molecular dsRNA viral é também capaz de ativar o mecanismo
de silenciamento pós-transcricional de genes, conhecido por RNA de
interferência (RNAi) (HANNON, 2002; ROBALINO et al., 2007).
Neste processo, o dsRNA desencadeia a destruição do RNA mensageiro
(RNAm) homólogo à sua própria sequência. O RNAi representa um
mecanismo de defesa natural contra vírus e transposons presentes nos
mais diferentes seres vivos, desde plantas até mamíferos (vide revisões
de HANNON, 2002; ALIYARI e DING, 2009). Muitos transposons e
vírus produzem RNA de dupla fita (dsRNA), pelo menos
transitoriamente em seus ciclos celulares, e o RNAi auxilia a manter sob
controle esses invasores potencialmente perigosos. Este sistema foi
inicialmente observado em plantas (NAPOLI; LEMIEUX;
JORGENSEN, 1990; VAN DER KROL et al., 1990) e a primeira
descrição em animais ocorreu em 1998 no nematódeo Caenorhabditis
elegans (FIRE et al., 1998), sendo posteriormente identificado em uma
ampla variedade de organismos.
A ativação deste sistema inicia-se pelo processamento de
precursores longos de dsRNA em RNAs pequenos (21-25 pb),
chamados small interference RNAs ou siRNAs, pela ação de um
complexo proteico contendo a enzima DICER, que são
endoribonucleases do tipo II que clivam especificamente dsRNA ou
regiões de grampo de RNAs de fita simples (vide revisão de ALIYARI e
DING, 2009). Os siRNAs resultantes são então incorporados a um
complexo multiproteico de silenciamento induzido por RNA (RISC do
inglês, RNA-induced silencing complex), que conduzirá à degradação
específica ou à repressão da tradução de RNAm com regiões
complementares à sequência do dsRNA desencadeante (vide revisão de
ROBALINO et al., 2007). De forma resumida, após a associação dos
siRNAs ao RISC, as fitas de siRNA são separadas, e a fita antisenso é
então utilizada como molde pelo RISC para encontrar e degradar os
RNAms alvos, que possuem sequências de nucleotídeos
complementares. Em última análise, a degradação do RNAm
impossibilita que a proteína correspondente seja traduzida (GELEY e
33
MULLER, 2004; WADHWA et al., 2004). Desta forma, o mecanismo
RNAi é atualmente utilizado como ferramenta em estudos que visem
desativar a expressão pós-transcricional de genes específicos, como
àqueles codificantes para proteínas virais em camarões, limitando ou
impedindo assim o processo infeccioso (ROBALINO et al., 2005;
TIRASOPHON; ROSHORM; PANYIM, 2005; YODMUANG et al.,
2006; KIM et al., 2007; TIRASOPHON et al., 2007;
ASSAVALAPSAKUL; CHINNIRUNVONG; PANYIM, 2009).
Diferentemente do sistema IFN que é induzido por qualquer
sequência de dsRNA, ou seja, de maneira sequência-inespecífica, a
proteção antiviral baseada no sistema RNAi é sequência-específica.
Interessantemente, destruindo grande parte do RNA alvo contido na
célula e, interrompendo temporariamente a síntese proteica
correspondente, a célula do hospedeiro inibe a replicação viral sem
matar a si mesma, o que é altamente vantajoso para o hospedeiro.
Dentre as proteínas-chave do sistema do RNAi destaca-se a
família das argonautas que são componentes fundamentais do complexo
de silenciamento multiproteico induzido por RNA ou RISC
(HAMMOND et al., 2001). A estrutura destas proteínas é constituída
por um domínio N-terminal PAZ, que participa do reconhecimento dos
siRNA, e um domínio C-terminal PIWI, com atividade de RNase e que
a caracteriza como uma proteína slicer do RISC (vide revisão PARKER
e BARFORD, 2006). Todos os complexos RISC caracterizados até o
momento possuem pelo menos uma proteína da família argonauta, como
componente (HAMMOND et al., 2001; HUTVAGNER e ZAMORE,
2002; MARTINEZ et al., 2002; MOURELATOS et al., 2002; VERDEL
et al., 2004). Em Drosophila, cinco genes codificantes para argonauta
foram identificados e classificados em duas subfamílas: AGO e PIWI
(vide revisão KIM; HAN; SIOMI, 2009).
Em camarões, as principais proteínas envolvidas na via de RNAi,
como a DICER (SU et al., 2008) e a argonauta, já foram identificadas
em P. monodon (UNAJAK; BOONSAENG; JITRAPAKDEE, 2006;
DECHKLAR; UDOMKIT; PANYIM, 2008). No caso da argonauta,
devido às características altamente conservadas dos domínios PAZ e
PIWI, os autores sugerem que esta proteína seja da subfamília AGO
(AGO1), estando envolvida no mecanismo de RNAi em camarões
(UNAJAK; BOONSAENG; JITRAPAKDEE, 2006; DECHKLAR;
UDOMKIT; PANYIM, 2008).
Um mecanismo antiviral específico induzido via RNAi foi
demonstrado em L. vannamei, a partir de injeções prévias nos animais
com sequências de dsRNA correspondentes a proteínas estruturais de
34
WSSV ou TSV (ROBALINO et al., 2005), onde a infecção subsequente
dos animais pelos respectivos vírus foi grandemente limitada. Após esse
primeiro relato, outros trabalhos se seguiram utilizando a técnica de
RNAi como uma terapia alternativa contra infecções virais em
camarões. Em P. monodon, sequências específicas de dsRNA foram
também capazes de suprimir a replicação in vivo do YHV
(YODMUANG et al., 2006), como in vitro (cultura primária de células)
(TIRASOPHON; ROSHORM; PANYIM, 2005). Ainda no caso do
WSSV, a replicação pôde ser eficientemente limitada em F. chinensis
por injeções com dsRNA correspondentes aos genes das proteínas virais
vp28, vp281 e proteína quinase (KIM et al., 2007).
Alguns estudos também indicam que pequenos RNAi (siRNA)
também sejam capazes de induzir a proteção antiviral contra o WSSV
em P. monodon (WESTENBERG et al., 2005), L. vannamei (WU et al.,
2007) e M. japonicus (XU; HAN; ZHANG, 2007). No entanto, os
resultados obtidos em camarões utilizando siRNA de genes virais
parecem não ser tão efetivos quanto aqueles com dsRNA, que possui
uma sequência longa. Aparentemente, esse fato está associado a
diferenças nos genes virais e aos fragmentos gênicos selecionados para a
sua síntese (vide revisão de SHEKHAR e LU, 2009).
No caso do dsRNA de sequência específica, além de atuar na
prevenção contra a infecção por vírus, um efeito terapêutico do mesmo
já
foi
demonstrado
com
sucesso
em
L.
vannamei
(ASSAVALAPSAKUL; CHINNIRUNVONG; PANYIM, 2009), e P.
monodon (TIRASOPHON et al., 2007). Em ambos os trabalhos, os
animais primeiramente desafiados com YHV e posteriormente injetados
com um dsRNA sequencia específica, mostraram uma forte inibição da
replicação viral, e consequentemente, elevadas taxas de sobrevivência.
Estes trabalhos indicam que a técnica do RNAi pode ser utilizada tanto
para fins profiláticos, prevenindo uma infecção viral, quanto para fins
terapêuticos, reduzindo/eliminando a infecção em animais já infectados.
Dessa forma, a utilização da técnica de RNAi vem despontando
no cenário científico internacional como uma ferramenta promissora e
efetiva para o controle de viroses em camarões cultivados (LIU;
SÖDERHÄLL; JIRAVANICHPAISAL, 2009), e constitui atualmente
uma das áreas de estudo mais ativas da biologia (ZERBINI, ALFENAS;
ANDRADE, 2005). No caso de humanos, o uso da técnica do RNAi
para inibir a replicação de alguns vírus patogênicos já está bem
documentada, como para o vírus da pólio (GITLIN; KARELSKY;
ANDINO, 2002), vírus da imunodeficiência adquirida HIV-1 (NOVINA
et al., 2002), vírus da hepatite C (RANDALL; GRAKOUI; RICE, 2003)
35
e hepatite B (GILADI et al., 2003). No entanto, os estudos sobre uma
possível utilização dessa técnica dentro da carcinicultura são ainda
muito recentes, sendo necessários ainda muitos estudos para viabilizar a
sua utilização prática como terapia antiviral em camarões.
Para isso, o conhecimento sobre os mecanismos moleculares
envolvendo respostas antivirais dos crustáceos é fundamental e estudos
que abordem a modulação da expressão gênica de proteínas
imunológicas durante o processo infeccioso são extremamente
relevantes. Diferentes estudos têm sido publicados na última década,
referentes à expressão gênica de PRPs (ROUX et al., 2002; CHENG et
al., 2005; WANG; CHANG; CHEN, 2007; YEH et al., 2009), de PAMs
(DESTOUMIEUX et al., 2000; SOMBOONWIWAT et al., 2006 ; de
LORGERIL et al., 2008), e de proteínas imunoefetoras e/ou
imunoreguladoras nos crustáceos (TONGANUNT et al., 2005; LIN et
al., 2007a,b; OKUMURA, 2007; VASEEHARAN et al., 2007; ZHAO et
al., 2007; AI et al., 2008). Estes estudos utilizam técnicas que envolvem
basicamente a identificação das sequências gênicas e a quantificação da
expressão gênica de diferentes moléculas imunológicas e que incluem,
basicamente, a técnica da reação em cadeia da polimerase (análises
semiquantitativas por PCR convencional e quantitativas por PCR em
Tempo Real).
Dentre as metodologias mais recentemente desenvolvidas,
destaca-se a técnica da PCR em Tempo Real ou PCR quantitativo
(qPCR), que detecta e quantifica, em tempo real, a amplificação de
ácidos nucleicos através da leitura do aumento da fluorescência do
fluoróforo utilizado. Por outro lado, na PCR tradicional (RT-PCR) os
produtos precisam ser posteriormente analisados por eletroforese
(DURAND e LIGHTNER, 2002). Apesar de sensível e confiável, o uso
da PCR em Tempo Real ainda é cara e requer equipamentos específicos,
limitando o seu uso rotineiro em muitos laboratórios (MARIN et al.,
2002).
Nesse sentido, metodologias alternativas menos onerosas podem,
de maneira semelhante, fornecer informações sobre a quantificação
relativa da expressão de genes, como é o caso da PCR semiquantitativa
(MARIN et al., 2002). Embora a PCR semiquantitativa não permita uma
quantificação absoluta, é possível inferir o número de moléculas
amplificadas em cada genoma, se o seu produto for avaliado na fase
exponencial da amplificação, onde o número de cópias de um fragmento
está em função da sua quantidade inicial. Assim, ao contrário da PCR
em Tempo Real, a PCR semiquantitativa possui a necessidade do
conhecimento prévio da cinética da reação, uma vez que é preciso
36
determinar o tempo ideal de reação no qual os produtos da PCR
correspondem à fase exponencial, em que as estimativas podem ser mais
acuradas (MARTINS, 2006).
Recentemente, diversos estudos envolvendo análises de
expressão de diferentes genes imunológicos em camarões têm sido
realizados utilizando a técnica da PCR em Tempo Real (ROUX et al.,
2002; TONGANUNT et al., 2005; BURGE et al., 2007; OKUMURA,
2007), e da PCR semiquantitativa (JIRAVANICHPAISAL et al., 2007;
ZHAO et al., 2007; WANG; CHANG; CHEN, 2007) para avaliar os
mecanismos de suscetibilidade e resistência de camarões frente às
infecções. Além disso, essas técnicas podem fornecer informações sobre
o estado de saúde dos crustáceos cultivados bem como dos mecanismos
envolvidos na interação patógeno-hospedeiro.
Embora nos crustáceos existam vários relatos de expressão
gênica em animais submetidos à infecção viral, não há na literatura, que
seja do nosso conhecimento, estudos sobre a modulação gênica em
camarões infectados por WSSV, previamente induzidos a uma proteção
antiviral específica, através do sistema de RNAi. Neste sentido, o
presente trabalho avaliou a expressão gênica de algumas proteínas
associadas ao sistema imune de L. vannamei cultivados em Santa
Catarina, previamente induzidos a uma proteção antiviral via RNAi e,
posteriormente, desafiados com o WSSV, através da técnica da PCR
semiquantitativa.
O artigo resultante deste trabalho será submetido à publicação na
revista Fish and Shellfish Immunology.
37
OBJETIVOS
Objetivo Geral
Avaliar a expressão gênica de algumas proteínas associadas ao
sistema imune de camarões Litopenaeus vannamei induzidos a uma
proteção antiviral específica (sistema RNAi) e, infectados
experimentalmente com o vírus da síndrome da mancha branca
(WSSV).
Objetivos Específicos
Avaliar a expressão semiquantitativa das proteínas: argonauta,
caspase-3, α2M, ALF, LGBP, proPO e TGase em camarões
tratados com dsRNA da proteína vp28 WSSV e posteriormente
desafiados com o WSSV;
Determinar a quantidade de hemócitos circulantes na hemolinfa
de camarões infectados ou não com o WSSV após ativação do
sistema de RNAi;
Avaliar a proteção antiviral conferida pelo tratamento com
dsRNA vp28, a partir da avaliação da taxa de mortalidade dos
animais.
38
Técnica de RNAi induzindo uma defesa antiviral em Litopenaeus
vannamei contra o WSSV e a modulação da expressão de alguns
genes imunológicos
Cristhiane Guertlera, Claudio Humberto Mejía-Ruízb, Margherita A.
Barraccoa, Luciane Maria Perazzoloa*
a
Laboratório de Imunologia Aplicada à Aquicultura, Departamento de Biologia
Celular, Embriologia e Genética, Universidade Federal de Santa Catarina, SC,
88.040-900, Brasil;bCentro de Investigaciones Biológicas del Noroeste, S.C. Mar
Bermejo no. 195, Colonia Playa Palo de Santa Rita. Código Postal 23090, La Paz,
México
A proteção antiviral específica contra o vírus da síndrome da mancha
branca (WSSV) induzida via RNA de interferência (RNAi) e sua
influência na expressão de alguns genes imunológicos foram avaliados
em Litopenaeus vannamei. Os animais foram injetados com dsRNA de
sequência específica (vp28 do envelope viral), e posteriormente
desafiados com o WSSV. Camarões tratados com dsRNA vp28 tiveram
a infecção viral limitada, uma alta taxa de sobrevivência (73%) e
ausência do vírus em 80% dos sobreviventes, confirmando a ativação
do mecanismo antiviral específico via RNAi. Animais desafiados e não
tratados com dsRNA vp28 tiveram uma queda significativa no
hemograma (85%) e depleção gênica da argonauta, caspase-3, fator
antilipopolissacarídeo
(ALF),
pró-fenoloxidase
(proPO)
e
transglutaminase (TGase). Uma superexpressão gênica da proteína que
reconhece LPS e β-1,3-glicanas (LGBP) e do inibidor de proteases α2macroglobulina (α2M) foi verificada nesses animais, indicando o
envolvimento destas proteínas na fase aguda da infecção por WSSV. O
tratamento com dsRNA vp28 limitou a infecção e restabeleceu as
condições imunológicas gerais dos animais através do aumento do
hemograma e da modulação positiva dos genes argonauta, ALF, proPO
e TGase, sugerindo a participação destas proteínas na defesa do
organismo contra o WSSV. Os resultados deste estudo apontam para
uma potencial utilização do dsRNA vp28 como agente terapêutico antiWSSV e confirmam que a técnica de RNAi é uma abordagem
preventiva promissora e efetiva para o controle das viroses de
crustáceos.
Palavras-chave: Litopenaeus vannamei, imunologia de crustáceos,
WSSV, RNA de interferência, dsRNA vp28, expressão gênica.
39
1. INTRODUÇÃO
As infecções virais representam, na atualidade, a maior ameaça
para o sucesso e sustentabilidade do cultivo de camarões a nível
mundial. O vírus da síndrome da mancha branca (WSSV) é o agente
etiológico da virose mais avassaladora da história da carcinicultura em
todo o mundo [1-3], sendo responsável por mortalidades que podem
chegar a 100% poucos dias depois de detectados os sinais clínicos nos
animais [4,5]. As perdas mundiais na produção advindas dessa virose
contabilizam cifras superiores a 10 bilhões de dólares, desde a sua
detecção em 1992 [6], o que tornou o controle das viroses em camarões
uma prioridade a nível mundial. No Brasil, a doença foi diagnosticada
em 2005, em cultivos de Litopenaeus vannamei no Sul do país, onde
causou perdas econômicas importantes ao setor, na ordem de mais de 6
milhões de reais [7].
Os crustáceos, como todos os invertebrados, possuem apenas um
sistema imune inato capaz de protegê-los contra patógenos invasores [8]
e a ausência de um sistema imune adaptativo impossibilita o
desenvolvimento de vacinas, capazes de proteger especificamente e a
longo prazo, limitando assim de maneira significativa os meios de
prevenção e controle das doenças infecciosas [9]. Torna-se, portanto
imperativo a busca de estratégias alternativas para limitar e/ou impedir
as infecções virais, visando minimizar as enormes perdas na atividade.
As respostas inatas dos crustáceos estão intimamente ligadas à
sua hemolinfa, por onde circulam as suas células imunocompetentes ou
hemócitos, e no qual estão dissolvidos os fatores humorais. Os seus
principais mecanismos de defesa incluem a coagulação da hemolinfa; a
melanização mediada pelo sistema de ativação da pró-fenoloxidase
(proPO); o reconhecimento e aglutinação celular mediada por lectinas,
os sistemas antibacterianos, antifúngicos e antivirais mediados por
peptídeos, RNA de interferência e por proteínas de reconhecimento
padrão; a produção de espécies reativas de oxigênio e de nitrogênio; e o
sistema fagocítico e de encapsulamento [10].
Um primeiro e essencial passo no processo de defesa desses
animais é o reconhecimento dos patógenos invasores pelas proteínas de
reconhecimento padrão (PRPs). As PRPs são produzidas pelo
hospedeiro que reconhecem e se ligam a padrões moleculares exclusivos
dos patógenos (PAMPs), tais como os lipopolissacarídeos (LPS) da
parede das bactérias Gram-negativas, as peptidoglicanas (PGs) das
bactérias Gram-positivas, as β-1,3-glicanas da parede de fungos e o
40
dsRNA (do inglês, double-stranded RNA) produzido durante a
replicação de vários vírus de DNA e RNA [11].
Uma vez que os PAMPS sejam reconhecidos por suas respectivas
PRPs, essas se ligam aos hemócitos ativando-os e promovendo uma
degranulação e/ou exocitose reguladas, liberando assim várias moléculas
imunoefetoras e imunoreguladoras, além de ativar cascatas de
sinalização intracelular que podem modular a expressão de vários genes
[12]. Uma importante PRP em crustáceos é a proteina que reconhece
ambos LPS e β-1,3-glicanas (LGBP), produzida pelos hemócitos [1315] e/ou hepatopâncreas [16,14], e sendo considerada uma proteína
induzível de fase aguda de infecções bacterianas e virais [16]. Em
peneídeos, o gene da LGBP foi identificado em Litopenaeus stylirostris
[16], L. vannamei [14] e Fenneropenaeus chinensis [15] sendo que
análises de expressão diferencial demonstraram a modulação de seu
gene codificante em animais infectados pelo WSSV [16-18].
O mecanismo de reconhecimento do não-próprio mais importante
dos crustáceos é o sistema de ativação da pró-fenoloxidase (proPO), que
consiste em uma cascata proteolítica composta por vários zimógenos de
proteases, pela proPO, além das PRPs e moléculas associadas e cujo
produto final é a melanina [11]. Esse sistema é ativado por componentes
da superfície de micro-organismos (PAMPs), que induzem a
degranulação ou exocitose regulada dos hemócitos, com liberação de
várias moléculas imunoefetoras, entre as quais as moléculas do sistema
proPO. Uma vez liberadas, ocorre a ativação deste sistema pelos
próprios componentes dos micro-organismos presentes na hemocele [8].
A enzima chave deste processo é a fenoloxidase (PO), que se encontra
armazenada nos grânulos dos hemócitos sob a forma inativa proPO [8],
sendo que sua a expressão gênica pode ser modulada negativamente
durante infecções por WSSV em peneídeos [16,18,19].
Para evitar uma ativação descontrolada e generalizada do sistema
proPO, os crustáceos contam ainda com uma variedade de inibidores de
proteases dissolvidos no seu plasma, que contribuem para a manutenção
da homeostase, minimizando a citotoxidade dos produtos intermediários
gerados durante o processo de melanização, tais como as
quinonas/hemiquinonas e os radicais livres. Dentre esses inibidores,
destacam-se a pacifastina [20], os inibidores da família Kazal [21,22], a
serpina [23] e a α2-macroglobulina (α2M) [24]. A α2M é uma
glicoproteína plasmática sintetizada pelos hemócitos [25,26] que atua
como um inibidor de proteases de amplo espectro (serino, aspartato,
cisteína e metalo-proteases), inibindo tanto proteases endógenas do
41
hospedeiro, como aquelas exógenas, produzidas pelo patógeno durante o
processo infeccioso [27].
Os hemócitos de crustáceos produzem ainda diferentes proteínas
e/ou peptídeos antimicrobianos (PAMs), como as peneidinas [28], as
crustinas [25,29] e os fatores antilipopolissacarídeos (ALFs) [25,30]. Os
ALFs incluem várias isoformas e possuem atividade contra bactérias
Gram-positivas, Gram-negativas e/ou fungos filamentosos [31,32],
participando também na limitação de infecções por WSSV no lagostim
Pacifastacus leniusculus [33,34].
Além disso, a disseminação dos patógenos pela cavidade
corpórea do crustáceo e a perda da hemolinfa durante injúrias podem ser
prevenidas pela coagulação da hemolinfa, mecanismo esse mediado pela
enzima transglutaminase (TGase) [35]. TGases são Ca2+ dependentes
que promovem ligações covalentes γ-glutamil-ε-lisina entre as proteínas
de coagulação da hemolinfa, formando coágulos estáveis [35,36] que
retém os micro-organismos e/ou vedam tecidos injuriados dos camarões.
Alguns estudos apontam para uma diminuição da expressão da TGase
[37], bem como um aumento no tempo de coagulação da hemolinfa [3840] em camarões infectados pelo WSSV.
Em relação aos mecanismos antivirais atualmente conhecidos nos
crustáceos podemos citar a apoptose celular, o sistema de RNA de
interferência (RNAi) e um sistema semelhante ao interferon (IFN) dos
vertebrados [41,42]. A apoptose é um processo fisiológico
geneticamente controlado que leva as células à autodestruição
silenciosa, sendo estimulada por vários agentes, dentre eles uma
infecção viral [43]. Em crustáceos, os mecanismos de regulação das vias
apoptóticas, bem como os indutores e os inibidores de tais vias, não são
ainda completamente conhecidos e compreendidos [44].
A apoptose, de maneira geral, é mediada por proteases da família
das caspases, que desencadeiam a morte celular através da clivagem de
proteínas específicas do núcleo e citoplasma, sendo classificadas como
iniciadoras (caspases 2, 8, 9 e 10) ou efetoras (caspases 3, 6 e 7) [45].
Em camarões, caspases executoras (caspase-3) foram identificadas e
clonadas em Penaeus merguiensis [46], Penaeus monodon [47] e L.
vannamei [48], assim como uma caspase iniciadora em Marsupenaeus
japonicus [49]. Foi mostrado que a infecção por WSSV pode levar a um
aumento da expressão da caspase-3 em peneídeos, sugerindo que a
apoptose seja um importante mecanismo antiviral nesses animais,
embora alguns estudos contestem essa hipótese [50,51].
Outro mecanismo antiviral recentemente demonstrado em
crustáceos, é o sistema de RNAi [41]. O RNAi é um mecanismo ligado
42
ao sistema imune inato de diferentes seres vivos, desde plantas até
mamíferos, que atua de maneira específica contra vírus e transposons
[52]. A ativação deste mecanismo inicia-se pelo processamento de
precursores longos de dsRNA em RNAs pequenos de 21-25 pb (small
interference RNAs ou siRNAs) pelo complexo enzimático DICER [53].
Estes pequenos RNAs fornecem as sequências específicas para um
complexo de silenciamento multiproteico induzido por RNA (RISC),
que dirige uma degradação específica, ou a repressão da tradução do
RNAm com regiões complementares à sequência do dsRNA
desencadeante [41]. Dentre as proteínas do sistema de RNAi destaca-se
a família das argonautas, que devido a sua estrutura e função são
conhecidas como a proteína slicer do RISC [54,55]. Em camarões, uma
argonauta (AGO 1) já foi identificada em P. monodon [56,57].
Desde o primeiro relato de indução do sistema RNAi em
camarões utilizado para bloquear especificamente a replicação do
WSSV [58], diversos estudos se seguiram utilizando esta técnica para
limitar diferentes infecções virais, a partir de injeções de sequências de
dsRNA específicas a proteínas estruturais dos vírus [59]. No caso do
WSSV, a replicação pode ser eficientemente suprimida por injeções com
sequências de dsRNA ou siRNA correspondentes aos genes das
proteínas virais vp19, vp28, vp281 e proteína quinase [58,60-62]. Dentre
essas, a vp28 é a proteína mais abundante do envelope viral, estando
particularmente envolvida na adesão do WSSV às células do hospedeiro
[63], sendo, portanto, fundamental ao sucesso do processo infeccioso.
Este fato faz da sequência da vp28 uma das melhores candidatas para a
produção de dsRNAs visando ativar via RNAi o sistema antiviral em
crustáceos, contra o WSSV.
Embora a aplicação prática da tecnologia de RNAi ainda seja
economicamente inviável na carcinicultura, o uso desta técnica
representa atualmente uma ferramenta promissora e efetiva para o
controle de viroses em crustáceos cultivados [42]. Sendo assim, o
presente trabalho avaliou a indução da defesa antiviral contra o WSSV
em L. vannamei, após a injeção de um dsRNA vp28 e relata, pela
primeira vez, a modulação da expressão de genes imunológicos
(argonauta, caspase-3, ALF, α2M, LGBP, proPO e TGase) nos
camarões onde a proteção antiviral foi induzida via RNAi.
43
2. MATERIAIS E MÉTODOS
2.1. Animais
Foram utilizados camarões juvenis (n=145, 12 ±1,5g), da espécie
Litopenaeus vannamei, em período de intermuda adquiridos da fazenda
experimental Yakult/UFSC (Balneário Barra do Sul, Santa Catarina). Os
animais foram transportados para o Instituto Federal Catarinense
(Campus Araquari, Santa Catarina) e mantidos em tanques de 50L
contendo água salgada filtrada e aeração constante, a uma taxa de
estocagem de 15 animais por tanque e alimentados uma vez ao dia com
ração comercial. A qualidade da água dos aquários foi monitorada
diariamente, sendo que foram analisados salinidade, temperatura, pH e
oxigênio dissolvido. Aproximadamente 50% da água dos aquários foi
renovada diariamente
2.2. Preparo do inóculo viral
A solução de inóculo de WSSV foi preparada segundo
metodologia adaptada de Prior et al. [64]. Brevemente, o tecido
muscular de camarões mortos e congelados e diagnosticados com
WSSV por amplificação viral (item 2.5) foi homogeneizado em tampão
salina gelado (NaCl 330mM, Tris 10 mM, pH 7,4) (1:10 p/v) e em
seguida centrifugado a 2.000 xg, por 20 min a 4°C. O sobrenadante foi
recuperado e centrifugado a 9.000 xg, por 10 min a 4°C, para em
seguida ser filtrado (0,45 µm) e armazenado em nitrogênio líquido.
Simultaneamente, um inóculo livre de WSSV foi preparado seguindo a
mesma metodologia, utilizando músculo de animais diagnosticados
como WSSV negativo.
Em seguida, o poder infectante do inóculo viral foi verificado por
meio da injeção intramuscular do inóculo puro (não diluído) (50 µl entre
o 1° e 2° segmentos dorsais) em 20 camarões diagnosticados negativos
para o WSSV, por PCR (item 2.5). Durante todo o período o
comportamento dos animais foi observado, e a mortalidade de 100% foi
registrada cinco dias após a injeção do inóculo. Tecidos de todos os
animais mortos foram coletados para verificar a presença do WSSV
(item 2.5.). O inóculo foi então mantido em nitrogênio líquido, até ser
utilizado no experimento definitivo (item 2.4).
44
2.3. Síntese do dsRNA vp28
O RNA dupla fita correspondente ao gene codificante da proteína
vp28 do WSSV foi sintetizado conforme protocolo preconizado pelo kit
BLOCK-It RNAi TOPO (Invitrogen, USA). Brevemente, a sequência
gênica correspondente à proteína vp28 foi amplificada por PCR
(fragmento
610pb)
utilizando-se
os
iniciadores
VP28F3:
5’ATGGATCTTTCTTTC3’ e VP28R3: 5’TTACTCGGTCTCAG3’
sob condições de 94°C por 4 min, seguido por 33 ciclos (94°C por 1
min, 58°C por 1 min e 72°C por 1 min), e 72°C por 7 min. Em seguida,
o produto amplificado foi ligado ao promotor T7 em ambos extremos
durante 15 min a 37 ºC, e após, foi realizada a amplificação deste
produto para produzir separadamente as fitas senso e antisenso do DNA
complementar. Essas amostras foram, então, submetidas a uma reação
de transcrição para produção das sequências do RNA senso e antisenso
correspondente, utilizando NTPs (15 mM) e a enzima BLOCK-It Mix.
Após tratamento das amostras com DNase (1U/µl), foi realizada a
purificação em colunas de membrana de sílica e quantificação a A260nm
(Biowave II – WPA) das amostras.
Em seguida, quantidades
equimolares de RNA senso e antisenso foram hibridizadas (2h a uma
temperatura decrescente de 100°C a 20°C) para formar o RNA dupla fita
denominado aqui de dsRNA vp28. A qualidade do dsRNA vp28
formado foi então avaliada por análise em gel de agarose (1%) e a
concentração mensurada espectrofotometricamente (A260nm) (Biowave II
– WPA).
2.4. Ativação do sistema de RNA de interferência em L. vannamei injeção com dsRNA vp28 e infecção experimental com WSSV
Antes do início do experimento a hemolinfa de 15% dos animais
foi coletada para verificar a presença do WSSV. Somente animais
diagnosticados como negativos para WSSV foram utilizados no
experimento. Os animais foram então aleatoriamente separados em três
grupos experimentais, em triplicata: G1: injetados com tampão (NaCl
330 mM, Tris 10 mM, pH 7,4) e posteriormente injetados com inóculo
puro de tecido de camarão livre de WSSV (controle negativo); G2:
injetados com tampão e infectados com inóculo positivo puro de WSSV
(controle WSSV+) e G3: injetados com dsRNA vp28 e posteriormente
infectados com inóculo positivo puro de WSSV.
45
Para induzir à proteção antiviral específica, ou seja, ativar o
sistema RNAi, os animais do grupo G3 foram previamente injetados
(50µl) intramuscularmente, com 1µg dsRNA vp28 a cada 3g de peso
vivo, entre o primeiro segmento abdominal e o cefalotórax, na região
dorsal. Os animais dos grupos G1 e G2 foram injetados apenas com
tampão. Após 48 horas dessa injeção, os animais G2 e G3 foram então
infectados com inóculo positivo WSSV (50µl) entre o primeiro
segmento abdominal e o cefalotórax, na região dorsal do animal. Da
mesma forma, os camarões do grupo controle, G1, foram injetados com
o mesmo volume do inóculo livre de WSSV. O esquema explicativo do
desenho experimental está representado na Figura 1.
Os camarões foram mantidos no sistema por 30 dias, sendo que a
coleta de hemolinfa foi feita 6h após injeção do dsRNA vp28 (coleta
apenas para o grupo G3) (1ª coleta), 0h (momento antes da injeção do
inóculo viral- 2ª coleta) e 3h, 6h, 24h, 48h e 72h após a injeção do
inóculo viral (3ª a 7ª coletas em todos os grupos) (Figura 1).
A primeira coleta (6h após injeção do dsRNA vp28) teve por
objetivo avaliar uma potencial resposta antiviral antes do inóculo com o
WSSV (que ocorreu 48h após), através da avaliação dos hemogramas e
da modulação na expressão dos genes imunológicos em questão. As
coletas posteriores (3, 6, 24, 48 e 72 h após o inóculo viral), tiveram por
objetivo avaliar uma proteção antiviral específica nos animais injetados
com dsRNA vp28 e inoculados com WSSV, igualmente através de
hemogramas e da expressão de genes imunológicos.
A porcentagem da mortalidade dos animais sobreviventes foi
monitorada diariamente, durante os 30 dias que se seguiram a partir do
desafio com WSSV, a fim de avaliar a proteção antiviral específica dos
camarões pelo dsRNA vp28.
46
d sRNA
VP28
Inoculação
WSSV -/+
48h
6h
0h
1ª
coleta
2ª
coleta
Pré-infecção com WSSV
3h
6h
3ª
coleta
4ª
coleta
24h
5ª
coleta
48h
6ª
coleta
72h
7ª
co leta
Pós-infecção com WSSV
Figura 1: Esquema explicativo do desenho experimental. No período de
pré-infecção, os animais foram injetados com tampão salina (G1 e G2)
ou com dsRNA vp28 (G3) e, após 48h, infectados (G2 e G3) ou não
(G1) com WSSV. As coletas de hemolinfa ocorreram 6h após injeção
com dsRNA vp28 (G3); e 0, 3, 6, 24, 48 e 72h após a infecção com
WSSV negativo (G1) ou positivo (G2).
2.5. Detecção da infecção dos camarões por WSSV
A extração do DNA genômico de pléopodes e/ou hemócitos
fixados em etanol 70% foi realizada em DNAzol (Invitrogen).
Brevemente, tecido de pléopodes lavados em água livre de RNAse
foram submetidos a digestão com proteinase K (concentração final 100
µg/ml) diluída em tampão de lise (100 mM Tris-HCl, 10 mM NaCl, 50
mM EDTA), e em presença de SDS (concentração final a 1%). A
mistura foi então incubada overnigth a 65°C, e o DNA extraído com
DNAzol (1ml/100mg tecido). Após centrifugação (10.000 xg por 10 min
a 4°C), o sobrenadante (DNA) foi precipitado com etanol 100%. No
caso dos hemócitos, após lavagens com Tris 10 mM (pH 7,5), os
mesmos foram submetidos à extração por DNAzol (1ml/pellet) e
precipitação do DNA por isopropanol (400µl). Finalmente, ambos os
DNAs dos pleópodes e hemócitos foram lavados com etanol 75%, secos
a temperatura ambiente e solubilizados em NaOH 8 mM (200µl). A
pureza das amostras foi avaliada em espectrofotômetro (A260/280nm)
(Biowave II - WPA) e a integridade do DNA foi verificada através da
amplificação do gene da proteína constitutiva actina (fragmento de 800
pb),
utilizando
os
iniciadores
AV1Fw:
5'TAATCCACATCTGCTGGAAGGTGG3'
e
AV2Rw:
5'TCACCAACTGGGATGACATGG3' sob as condições de PCR de
47
94°C por 5 min, seguido por 30 ciclos (94°C por 1 min, 55°C por 1 min
e 72°C por 1 min), e 72°C durante 10 min.
A amplificação das sequências de DNA correspondentes ao
WSSV foi realizada através da Reação em Cadeia da Polimerase em
dois passos (nested PCR), utilizando-se os iniciadores recomendados
pela OIE: WSSV146Fw1: 5’ACTACTAACTTCAGCCTATCTAG3’;
WSSV146Rv1:5’TAATGCGGGTGTAATGTTCTTACG3’ (fragmento
1447pb); e WSSV146Fw2: 5’GTAACTGCCCCTTCCATCTCCA3’;
WSSV146Rv2: 5’TACGGCAGCTGCTGCACCTTGT3’ (fragmento
941pb). As condições de PCR para WSSV146 foram 94°C por 4 min,
55°C por 1 min, 72°C por 2 min, seguido por 39 ciclos (94°C por 1 min,
55°C por 1 min e 72°C por 2 min), e 72°C durante 5 min. A
confirmação ou não da presença de sequências gênicas referentes ao
vírus nos animais analisados foi confirmada através da visualização em
fotodocumentador (UV) dos fragmentos de tamanho esperado em gel de
agarose (1%) corado com brometo de etídeo (0,5µg/ml).
Além da análise por PCR convencional, a presença do vírus
também foi investigada nos animais do grupo G3 por uma PCR em
Tempo Real (GE HealthcareTM). Para tanto, utilizou-se o kit
DyNAmoTM SYBR® Green qPCR (Finnzymes), a partir de 100ng (1µl)
de DNA genômico em um volume final de reação de 20 µl. Os
iniciadores utilizados referem-se a uma sequência da proteína do
envelope viral vp19: VP19Fw: 5’ATGGCCACCACGACTAACACT3’;
VP19Rv: 5’CTGCCTCCTCTTGGGGTA3’ (fragmento 375 pb). As
condições da reação para esses iniciadores foram 95°C durante 10 min,
seguidos por 40 ciclos de 95°C por 20s, 58°C por 20s e 72°C por 20s.
Todas as amostras foram analisadas em duplicatas.
2.6. Extração de hemolinfa
A hemolinfa foi coletada diretamente dentro de uma solução
anticoagulante MAS (Solução de Alsever Modificada) (336 mM NaCl,
115 mM glicose, 27 mM citrato de sódio, 9 mM EDTA, pH 7,2) (1:2),
na região ventral do abdômen, utilizando-se seringa (1ml) com agulha
(13 x 0,4 mm) que foi inserida entre o último esternito cefalotorácico e o
primeiro abdominal. Uma alíquota de hemolinfa total foi separada e
armazenada em formol MAS 4% (1:3) para posterior contagem total de
hemócitos. O restante da mistura foi centrifugada a 800 xg por 10 min a
4°C, o sobrenadante descartado e o pellet celular ressuspendido e
48
armazenado em Trizol® (Invitrogen®) até a posterior extração dos RNA
totais.
Conforme descrito anteriormente, a extração de hemolinfa foi
feita em diferentes tempos: 6h após a injeção do dsRNA vp28 (G3), e
nos tempos 0h, 3h, 6h, 24h, 48h e 72h (G1, G2 e G3). Cada coleta
representou 3 pools de 3 animais para cada grupo experimental.
2.7. Contagem total de hemócitos (CTH)
A hemolinfa de cada pool/grupo/coleta foi extraída e armazenada
em formol MAS 4% a uma diluição conhecida, sendo a contagem total
de hemócitos (CTH) por mL estimada em câmara de Neubauer, de modo
semelhante à realizada para glóbulos brancos [65].
2.8. Extração dos RNA totais e síntese do cDNA
Os RNAs totais foram extraídos dos hemócitos em Trizol
(Invitrogen®) de acordo com instruções do fabricante: os hemócitos
armazenados no reagente foram homogeneizados e submetidos à
extração por clorofórmio (5:1 Trizol/clorofórmio), e após centifugação
(12.000 xg 15min 4ºC), o RNA obtido foi precipitado em isopropanol
(2:1 Trizol/isopropanol) e lavado com etanol 75%. O pellet de RNA
precipitado foi então ressuspendido em água livre de RNase e as
amostras tratadas com Dnase I (Fermentas 1U/µl) e a mistura incubada
por 15 min a 20°C. Após a inativação da DNase (15 min a 65°C), as
amostras foram quantificadas espectrofotometricamente (GE Healthcare
NanoVueTM). Apenas soluções de RNA com razão de absorbância
(A260:A280) superior a 1.8 foram utilizados nos experimentos. Para a
síntese de cDNA utilizou-se o kit ImPromII-RT (Promega), onde 1 µg
de RNA foi reversamente transcrito utilizando-se a enzima ImPromII na
presença de 0,5 mM de cada dNTP, 20 µM de iniciador oligo dT, e
inibidor de RNAse (1U/µL), sendo que as amostras foram incubadas a
25ºC por 5min, 42ºC por 60min e 70ºC durante 15min.
2.9. Análise da expressão gênica semiquantitativa de algumas
proteínas imunológicas de L. vannamei
Os cDNAs obtidos foram utilizados para a reação de
amplificação das sequências das proteínas imunológicas de interesse:
49
argonauta, caspase-3, α2M, ALF, LGBP, proPO e TGase, utilizando-se
os iniciadores listados na Tabela 1. Todos os iniciadores correspondem à
sequências de L. vannamei, exceto aqueles do gene argonauta (P.
monodon) e proPO (F. chinensis).
Com o objetivo de determinar o número de ciclos para
amplificação de cada gene de interesse, para a análise semiquantitativa,
realizou-se preliminarmente várias PCRs para cada gene. Desta forma, o
número de ciclos para cada par de iniciadores (Tabela 1) foi escolhido
durante a fase exponencial da reação, onde não ocorreu saturação da
expressão dos genes de interesse. O mesmo ensaio foi realizado com o
gene que codifica a proteína constitutiva actina e que foi utilizado para
normalizar os níveis de cDNA presentes nas amostras. Conforme os
padrões encontrados para cada gene, os ciclos escolhidos foram 28
(argonauta), 24 (caspase-3), 24 (α2M), 17 (ALF), 30 (LGBP), 28
(proPO), 20 (TGase) e 20 (actina). As condições de PCR foram 94°C
por 5 min, seguido pelo número de ciclos citado acima para cada gene
sob as seguintes condições (94°C por 1 min, 55°C por 1 min e 72°C por
1 min), e 72°C durante 10 min, sendo que para a amplificação do gene
ALF a temperatura de anelamento utilizada foi de 60°C.
A confirmação da presença de sequências gênicas foi realizada
através da análise de bandas de tamanho esperado em gel de agarose
(1%). Após a visualização dos géis, estes foram fotodocumentados e
digitalizados a partir do programa DigiDoc-ItLS. A densitometria das
®
bandas foi realizada utilizando-se o programa Scion Image for
Windows, sendo que a expressão do gene de interesse (quantidade
relativa de RNAm) foi determinada através da razão entre gene
interesse/gene controle (actina), a partir dos valores de densitometria das
bandas amplificadas. Foram realizadas de 3 a 5 PCRs para cada gene
utilizado, e os valores apresentados correspondem a uma média dos
valores obtidos na análise de densitometria das bandas dos géis destas
PCRs.
Gene
GenBank
Iniciadores
Forward/Reverse
β-actina
AF300705
Argonauta
DQ343133.1
Alfa 2M
AB108542
ALF
AY723296
Caspase-3
EU421939
5’-CGAAGTCAAAGCCAGAAACA-3’
5’-ACTGCTACTTCCCTGGTGAC-3’
420 pb
LGBP
AY723297
5’-CTGGCTCTAGCAGCAACCTC-3’
5’-CCTCGTCCACGTAGAACTCC-3’
399 pb
proPO
AY723296
5'-AGGTGGCTCTGTCCTCTTCA-3'
476 pb
5'-TAATCCACATCTGCTGGAAGGTG-3’
5'-TCACCAACTGGGATGACATGG-3'
5’-GTATCCAGGGCACAAGTCGT-3’
5’-GTGTGACCCCTCTCCACTGT-3’
5'-CCGTSAGCCAAGTRGAGCC-3'
5'-CGAGTGGGTKGGCAAAGC-3'
5´-CTGTGGAGGAACGAGGAGAC-3´
5’-CCACCGCTTAGCATCTTGTT-3´
Fragmento
amplificado
850 pb
224 pb
850 pb
240 pb
5-'ACTCCGGTCCGTGTTCTATG-3'
Transglutaminase
EU164849.1
5'-ATACAGAAGCCGGATATCAAG-3'
5’-GTTTCTTTGGGAAAACCTTC-3'
243 pb
50
Tabela 1: Pares de iniciadores utilizados para as análises de RT-PCR de sete genes relacionados ao sistema imune em
Litopenaeus vannamei. Os iniciadores argonauta e proPO referem-se à sequências de P.monodon e F. chinensis respectivamente.
51
3. ANÁLISE ESTATÍSTICA
Para as análises da CTH nos diferentes grupos foi utilizado o
teste ANOVA (α<0,05), seguido pelo teste de comparação de médias de
Tuckey.
4. RESULTADOS
4.1. Contagem Total de Hemócitos (CTH)
A contagem total de hemócitos em L. vannamei não tratados com
dsRNA vp28 nem desafiados com o WSSV (grupo controle, G1)
mostrou uma diminuição no número de hemócitos circulantes a partir
das 3h após a injeção com o inóculo WSSV negativo (Figura 2), onde
registrou-se uma queda ainda mais acentuada nas 6h e permanecendo
ainda baixa na análise de 24h pós-injeção (p<0,05). Contudo, a partir
das 48h observou-se um retorno do número de células circulantes aos
índices normais, encontrados no início do experimento (Figura 2).
Nos camarões desafiados com WSSV e que não foram tratados
com dsRNA vp28 (G2), ocorreu uma diminuição significativa (2x
menor que no tempo zero) na quantidade total de hemócitos circulantes
a partir de 3h da infecção viral, sendo que uma queda ainda mais
proeminente (4x) foi verificada 6h após a infecção (Figura 2). De
maneira interessante, após 24h pós-desafio viral, houve um aumento de
(2,5x) no número de hemócitos desses animais, em relação ao tempo
imediatamente anterior. No entanto, uma nova queda na CTH foi
observada no segundo dia pós-infecção (48h), alcançando valores
extremamente baixos no terceiro dia (72h; 5,3x106 cél/ml), ou seja, 6x
inferior àquele da pré-infecção (31x106 cél/ml) (Figura 2).
Já, nos animais tratados com dsRNA vp28 e posteriormente
desafiados com WSSV (G3), 6h após a injeção com dsRNA vp28 já se
observou uma redução de cerca de 50% no número de hemócitos
circulantes (Figura 2). Essa dimimuição continuou sendo significativa
até as 48 horas pós-infecção com WSSV (2,3x inferior ao valor da préinfecção), sendo que, no entanto, uma recuperação na CTH foi
encontrada nas 72h pós-infecção e cujo valor (27x106 cél/ml) ficou
muito próximo àquele encontrado no início do experimento (31x106
cél/ml).
52
(G1)salina/WSSV-
Número de hemócitos x 106 células.ml -1
(G2)salina/WSSV+
45
(G3)dsRNA/WSSV+
a
40
35
a
a a a
30
a
a
25
a
a
20
a b
b
b b
15
c
10
b b
b
5
0
A A A
B
B B C
B C C
B D D
A E B
A C E
0h
6h
3h
6h
24h
48h
72h
Pré-infecção
Pós-infecção
Figura 2: Contagem total de hemócitos (CTH) em L. vannamei imediatamente
antes das injeções (0h); 6h após injeção com dsRNA vp28 (G3); e 3, 6, 24, 48 e
72h pós-infecção com inóculo negativo (G1) ou positivo (G2 e G3) para o vírus
da síndrome da mancha branca (WSSV). Letras maiúsculas representam
diferenças significativas (p<0,05) na CTH dentro de um mesmo grupo nos
diferentes períodos analisados. Letras minúsculas representam diferenças
significativas (p<0,05) na CTH entre os grupos dentro de um mesmo período.
4.2. Expressão gênica
A quantificação dos transcritos correspondentes aos genes
codificantes para sete proteínas imunológicas (argonauta, caspase-3,
ALF, LGBP, α2M, proPO e TGase) foi realizada através da técnica da
PCR semiquantitativa (item 2.9). Nessa análise, a expressão relativa de
RNAm foi determinada através da razão entre a expressão de cada gene
alvo pela expressão do gene da proteína constitutiva actina, a partir dos
valores de densitometria encontrados na análise das bandas
correspondentes aos fragmentos amplificados por PCR (Figura 3). As
análises dos níveis de expressão gênica foram feitas antes das injeções
(0h= expressão basal), 6h após a injeção do dsRNA vp28 (análise feita
somente em G3) e nas 3, 6, 24, 48 e 72h após injeção com o inóculo
negativo (G2) ou positivo (G3) para WSSV.
53
4.2.1. Argonauta: a proteína slicer do RISC do sistema RNAi
Nos animais do grupo controle, não tratados com dsRNA vp28
nem desafiados com o vírus, (G1) observou-se uma alteração nos níveis
de expressão do gene argonauta apenas a partir das 6h após injeção com
inóculo negativo (Figura 3). Um aumento de cerca de 2x na expressão,
em relação ao valor basal, foi encontrado nas 6 e 24h após inóculo
negativo, enquanto que um aumento menos pronunciado ocorreu nas 48
e 72h seguintes (20% e 60%, respectivamente) (Figura 3). Já,
diferentemente do grupo controle, nos animais G2 (não tratados com
dsRNA vp28, mas infectados com WSSV) registrou-se um aumento de
80% no nível de expressão do gene argonauta logo nas primeiras 3h
após a infecção viral (Figuras 3 e 4A). No entanto, essa expressão
diminuiu nas análises posteriores, sendo na maioria delas inferior aos do
grupo controle, não infectado (Figura 3).
Nos animais G3 (tratados com dsRNA vp28 e 48 horas após
infectados com WSSV) observou-se um aumento de 80% nos níveis de
expressão da argonauta ainda no período pré-infecção, ou seja, 6h após a
injeção do dsRNA vp28 (Figura 3). No entanto, após o desafio dos
animais com WSSV, dois picos de expressão da argonauta foram
encontrados nas 3 e 48h, sendo que nas outras análises (6, 24 e 72h) os
valores de expressão dessa proteína slicer se mantiveram levemente
superiores ao controle (Figura 3) e superiores ao grupo infectado pelo
vírus e que não recebeu o tratamento com dsRNA (Figura 4A).
4.2.2. Caspase-3: uma protease executora da via apoptótica
Após a injeção do inóculo negativo, o grupo controle (G1)
apresentou elevados níveis de expressão gênica referentes à caspase
durante a maioria das análises, de cerca de 3x em relação à expressão
basal (Figura 3). A exceção ocorreu na análise de 48h onde a expressão
foi superior apenas de 1,7x. Já o grupo desafiado com WSSV, mas não
tratado com dsRNA vp28 (G2) não apresentou diferenças nos níveis de
expressão 3h após inoculação, com relação ao período correspondente
do G1 controle (Figuras 3 e 4B). Essa expressão foi, contudo,
diminuindo nas análises posteriores, ao ponto de 72h pós-desafio
registrar-se uma queda de 94% no nivel de expressão dessa caspase
efetora, em relação ao respectivo controle (Figura 4B).
Em relação aos animais desafiados com vírus e tratados com
dsRNA vp28 (G3), houve um aumento de 50% na expressão da caspase
já nas primeiras 6h após a injeção com dsRNA (Figura 3).
54
Curiosamente, os níveis de expressão dessa proteína apoptótica foram
menores que àqueles do grupo controle em todas as análises seguintes,
com exceção do período de 48h, onde foi registrado um aumento de
50% (Figura 3). No entanto, em todos os períodos a partir das 6h, os
níveis de expressão da caspase foram superiores neste grupo com
relação aquele apenas infectado pelo WSSV (Figuras 3 e 4B).
4.2.3. Alfa 2-macroglobulina (α2M): um inibidor de proteases de
amplo espectro
Os animais do grupo controle (G1) apresentaram uma queda de
~40% nos níveis de expressão da α2M até as 6h após a injeção dos
animais com inóculo negativo (Figura 3). No entanto, um aumento na
expressão deste gene foi observado nas 24h (50% a mais que o basal),
sendo que uma ligeira queda ocorreu nas 48 e 72h, onde a expressão
ficou próxima à basal (Figura 3).
Já, nos animais G2, houve um aumento importante da expressão
gênica da α2M até 48h após a infecção com WSSV, especialmente nas 6
e 48h cujos valores foram respectivamente 90% e 60% superiores aos
controles (Figuras 3 e 4C). De maneira interessante, uma queda muito
proeminente (70%) foi registrada na expressão, 72h após o desafio viral
(Figuras 3 e 4C).
No grupo G3, 6h após o tratamento prévio com dsRNA vp28, não
houve alteração no nível de expressão da α2M (Figura 3). Após o
desafio viral, os níveis de expressão deste inibidor de proteases foram
muitos próximos ao controle durante todos os períodos, exceto 6h após a
infecção quando um aumento de 65% na expressão foi observado
(Figuras 3 e 4C). Interessantemente, em relação ao G2, o grupo G3
apresentou níveis de expressão da α2M mais baixos até 48h pósinfecção, sendo que apenas no período seguinte (72h) a expressão deste
inibidor de proteases foi superior no grupo tratado com dsRNA vp28
(Figura 4C).
4.2.4. Fator antilipopolissacarídeo
antimicrobiano com atividade antiviral
(ALF):
um
peptídeo
Os níveis de expressão do ALF em L. vannamei não tratados com
dsRNA vp28 nem desafiados com o vírus (G1) foram elevados durante
todo o período analisado, apresentando os maiores picos de expressão
nas 6 e 72h após a injeção com o inóculo negativo, e cujos valores
foram de 2,2x e 2,5x superiores ao basal, respectivamente (Figura 3). Já,
55
nos animais não tratados com dsRNA vp28 e desafiados com o WSSV
(G2), os níveis de expressão gênica desse PAM diminuíram
acentuadamente ao longo da infecção (Figuras 3 e 4D), estando 90%
inferiores ao controle no período de 72h (Figura 3).
Em G3, a expressão desse PAM praticamente não se alterou nas
6h após injeção com dsRNA, apenas um aumento discreto (1,2x) foi
observado em relação ao valor basal (Figura 3). No entanto, após a
infecção viral, diferentemente do grupo anterior, os animais de G3
tiveram a expressão do ALF aumentada de maneira importante 3h e 24h
após a infecção viral (até 60% superior ao respectivo controle) (Figuras
3 e 4D). Curiosamente, 72h após a infecção o nível de expressão deste
gene decaiu (30%) (Figuras 3 e 4D), contudo os níveis de expressão do
ALF foram bastante superiores em todos os períodos com relação ao
grupo infectado com WSSV mas não tratado com dsRNAvp28 (Figura
4D).
4.2.5. Proteína que se liga a LPS e β-1,3-glicanas (LGBP): uma
proteína de reconhecimento padrão
A expressão do gene da LGBP nos hemócitos dos camarões não
tratados com dsRNA vp28, nem desafiados com o vírus (G1) aumentou
em todos os períodos analisados, especialmente nas 3 e 24h após o
inóculo negativo (1,9x e 2,2x, respectivamente) (Figura 3). No entanto,
nas 72h houve uma tendência ao retorno dos níveis de expressão deste
gene (1,2x) ao basal (Figura 3). Já, no grupo apenas desafiado com
WSSV (G2), a expressão dessa PRP foi drasticamente diminuída (80%)
nas 3h após a infecção viral, seguida por um importante aumento nos
períodos subsequentes, especialmente nas 48h (90%) e 72h (60%) após
o desafio (Figuras 3 e 4E).
Nos animais do G3 observou-se uma redução de 50% no nível da
expressão do gene da LGBP nas 6h após tratamento com dsRNA vp28,
antes do desafio viral (Figura 3). No entanto, 3h após o desafio dos
animais com WSSV os níveis de expressão da LGBP estavam
semelhantes ao controle. De maneira interessante, um único aumento
(30%) na expressão foi verificado nas 6h pós- desafio viral, sendo que
os valores subsequentes sempre foram próximos ao controle, e inferiores
àqueles do grupo desafiado com WSSV, mas não tratado com dsRNA
(Figura 4E).
56
4.2.6. Pró-fenoloxidase: um zimógeno do sistema de ativação da
proPO
Um aumento nos níveis de expressão gênica da proPO nos
animais controle (G1) foi observado nas primeiras 6h após a injeção do
inóculo negativo, seguido por um retorno ao nível basal, na análise de
24h (Figura 3). Interessantemente, um aumento de até 2x na expressão
ocorreu no segundo e terceiro dias após a injeção. Já nos animais
desafiados G2, uma queda de 50% nos níveis de expressão da proPO já
foi observada logo após a injeção com WSSV, chegando a ser 70%
inferior ao controle 72h pós-infecção. Curiosamente, um único pico de
expressão foi encontrado às 24h, sendo ~100% superior ao controle
(Figuras 3 e 4F).
No caso do G3, 6h após a injeção dos camarões com dsRNA
vp28, a expressão da proPO caiu pela metade (Figura 3). No entanto,
após o desafio dos animais com WSSV um aumento na expressão deste
zimógeno foi registrado até as 24h, quando os níveis de expressão
chegaram a ser 3,5 vezes superiores a dos animais controle e 40% dos
animais não tratados com dsRNA vp28 (Figura 4F). Porém, nas análises
subsequentes (48 e 72h) essa expressão diminuiu (Figura 4F), ficando os
valores mais próximos aos dos animais controle, no mesmo período
(Figura 3). Comparando a expressão da proPO nos animais desafiados
com WSSV, observa-se que a expressão deste gene foi sempre superior
nos camarões tratados com dsRNA vp28 (G3), em relação aos não
tratados (G2) (Figura 4F).
4.2.7. Transglutaminase (TGase): a enzima-chave no processo de
coagulação da hemolinfa
A análise da expressão gênica da TGase nos camarões controle
(G) mostrou-se inalterada 3 e 48h após a injeção com inóculo negativo.
No entanto, aumentos de cerca de 70% nos níveis de expressão foram
observados nas outras análises (6, 24h e 72h) (Figura 3). Já a expressão
dessa enzima nos animais infectados com o WSSV, mas não tratados
com dsRNA vp28 (G2) foi depletada na maioria das análises, chegando
a ser 50% menor que o controle nas 72h pós-infecção. Apenas na análise
de 48h, houve um aumento (50%) na expressão, em relação ao controle
(Figuras 3 e 4G). No entanto, o tratamento com dsRNA vp28 não foi
suficiente para estimular a expressão gênica da TGase, uma vez que um
aumento de apenas 10% foi registrado 6h após a injeção dos animais
com esse oligonucleotídeo (Figura 3). Após a infecção dos animais com
57
WSSV a expressão da TGase foi aumentada, sendo sempre superior ao
G2 (Figuras 3 e 4G). Novamente, um pico de expressão (60% superior
ao controle) foi encontrado nesses animais no período de 48h, a
exemplo do G2 (Figuras 3 e 4G). De modo semelhante a G2, uma queda
na expressão (30%) foi observada nas 72h pós-desafio (Figura 4G).
M
(G1) salina/WSSV-
(G2) salina/WSSV+
0h
3h
6h
24h 48h 72h
1.0
0.9
2.1
1.9
dsRNA VP28
58
basal
(G3) dsRNA/WSSV+
3h
6h
24h
48h
72h
6h
3h
1.2
1.6
1.7
1.2
1.1
1.3
0.9
1.8
1.8
1.7
2.9
2.8
1.9
1.3
0.2
6h
2.3
24h 48h
2.4
2 .0
72h
2.1
Argonauta
224pb
1.0
2.9
3.2
3.0
0.7
1.4
1.5
2.2
1.7
2.6
2.2
Caspase-3
420pb
1.0
0.7
0.6
1.5
1.1
1.2
1.0
1.4
2.2
1.7
1.9
2.5
1.0
1.1
1.3
1.8
0.4
1.0
0.3
1.2
0.8
0.9
1.2
1.4
1.3
α2M
850pb
0.9
1.3
1.1
0.6
2.8
2.3
2.4
2.5
2.1
1.7
1.6
0.9
ALF
240pb
1.0
1.9
1.7
2.2
1.5
1.2
0.4
2.4
2.4
1.9
2.3
1.1
2.7
2.9
1.0
0.9
2.4
2.0
0.5
1.7
2.3
0.5
2.6
3,3
1.8
LGBP
399pb
1.0
1.0
0.9
3.5
2.2
2.2
proPO
476pb
1.0
1.0
1.7
1.7
1.1
1.6
1.1
1.2
1.3
1.7
0.7
1.1
1.3
2.1
1.8
1.8
1.2
TGase
243pb
Actina
800pb
Figura 3: Análise semiquantitativa por RT-PCR dos genes argonauta, caspase-3, alfa 2-macroglobulina (α2M), fator
antilipopolissacarídeo (ALF), proteína que se liga a LPS e β-1,3-glicanas (LGBP), pró-fenoloxidase (proPO) e transglutaminase
(TGase) nos hemócitos de L. vannamei. As análises foram feitas antes do início das injeções (0h- expressão gênica basal); 6h após
injeção de dsRNA vp28 (G3) e 3, 6, 24, 48 e 72h após a infecção com inóculo negativo (G1) ou positivo (G2 e G3) para o vírus
da síndrome da mancha branca (WSSV). Os valores correspondem à razão entre a densidade de cada banda de cada gene de
interesse pela banda actina nos diferentes tempos. A expressão relativa basal (0h) foi arbitrariamente estabelecida com o valor 1.
A densitometria das bandas foi realizada utilizando-se o programa Scion Image®.
59
2,5
(A) Argonauta
2
(G2) salina/WSSV+
1,5
(G3) dsRNA/WSSV+
1
0,5
Expressão relativa RNAm (unidades arbitrárias)
0
0h
2
3h
6h
24h
48h
72h
2,5
(B) Caspase-3
(C) Alfa 2M
2
1,5
1,5
1
1
0,5
0,5
0
0
0h
3h
6h
24h
48h
72h
2
0h
2
(D) ALF
1,5
1,5
1
1
0,5
0,5
0
6h
24h
48h
72h
6h
24h
48h
72h
24h
48h
72h
(E) LGBP
0
0h
3h
6h
24h
48h
72h
3,5
3
3h
0h
3h
2
(G) TGase
(F) proPO
1,5
2,5
2
1
1,5
1
0,5
0,5
0
0
0h
3h
Pré-infecção
6h
24h
48h
Pós-infecção
72h
0h
Pré-infecção
3h
6h
Pós-infecção
Figura 4: Expressão em relação ao controle dos genes: argonauta (A), caspase3 (B), alfa 2-macroglobulina (C), fator antilipopolissacarídeo (D), proteína que
se liga a LPS e β-1,3-glicanas (E), pró-fenoloxidase (F) e transglutaminase (G)
nos hemócitos de L. vannamei antes do início das injeções (0h) e 3, 6, 24, 48 e
72h após a infecção com o vírus da síndrome da mancha branca (WSSV) nos
grupos G2 (tampão/WSSV+) e G3 (dsRNA/WSSV+).
60
4.3. Mortalidade dos animais
Os animais não tratados com dsRNA vp28 e infectados com
WSSV (G2), apresentaram uma mortalidade total (100%) em 5 dias
(Figura 5). Já, no grupo tratado com dsRNA e posteriormente infectado
com WSSV (G3) observou-se uma linha ascendente de mortalidade
apenas até o quinto dia pós-infecção (27%), não registrando-se mais
nenhuma mortalidade nos 25 dias posteriores, cuja sobrevivência no
grupo tratado com dsRNAvp28 foi de 73% ao final de 30 dias de
experimento (Figura 5). A mortalidade dos camarões controle (G1) foi
relativamente baixa durante o período experimental (7%) (dados não
apresentados).
100
Mortalidade cumulativa (%)
90
80
70
60
50
salina/WSSV+
40
dsRNA/WSSV+
30
20
10
0
1 2 3 4 5
10
15
20
25
30
Dias pós-infecção
Figura 5: Porcentagem da mortalidade cumulativa dos animais em cada
tratamento durante 30 dias.
4.4. Detecção do DNA viral (WSSV) nos tecidos dos animais
A presença de WSSV nos animais de cada grupo experimental foi
avaliada simultaneamente à expressão gênica das proteínas
imunológicas, nas 3, 6, 24, 48 e 72h, por PCR convencional (um passo e
dois passos-nested PCR). Os animais sobreviventes do G3 foram
61
avaliados individualmente quanto à presença do vírus, no 30º dia, por
PCR em Tempo Real.
Nos animais do grupo controle (G1), o WSSV não foi detectado
em nenhum dos períodos analisados (Tabela 2). No entanto, nos
camarões injetados com WSSV e que não receberam um tratamento com
dsRNA vp28 (G2), a presença do vírus foi detectada nas 6h após o
desafio e somente por nested PCR (++), sendo que nos períodos
seguintes o vírus pôde já ser detectado na primeira etapa da PCR (+,
Tabela 2). Nos animais G3, que receberam um tratamento prévio com
dsRNA vp28, o WSSV pôde ser detectado somente por nested-PCR, e a
partir de 48h após o desafio (Tabela 2).
Tabela 2: Detecção de WSSV nos animais G1: tampão/WSSV-, G2:
tampão/WSSV+ e G3: dsRNA/WSSV+, por PCR convencional, em um (+) ou
dois passos (nested-PCR) (++). As análises em cada período foram feitas com
DNA genômico extraído de um pool de 3 animais/grupo.
Horas pós-injeção
Tratamento
3h
6h
24h
48h
72h
(G1)
salina/WSSV-
-
-
-
-
-
(G2)
salina/WSSV+
-
++
+
+
+
(G3)
dsRNA/WSSV+
-
-
-
++
++
A presença do WSSV nos animais G3 que sobreviveram 30 dias
após o desafio com WSSV foi encontrada em apenas 20% dos camarões
(Figura 6, linhas 7 a 10), apesar de nenhum sinal clínico ser visualizado
nesses animais (Tabela 2).
M
CN
I
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
Figura 6: Eletroforese em gel de agarose (1%) representando os produtos
amplificados por PCR em Tempo Real, com amostras de DNA genômico de
animais infectados com WSSV e previamente tratados com dsRNA vp28 (linhas
1 a 18). A análise foi feita 30 dias após a infecção, utilizando-se iniciadores
vp19 (fragmento de 375 pb). M: marcador de peso molecular (100pb); CN:
Controle Negativo (sem adição de DNA); I: Inóculo Positivo para WSSV.
Linhas 7 a 10: animais WSSV positivos.
62
5. DISCUSSÃO
A síndrome da mancha branca (WSS) é a enfermidade mais
importante no que se refere às perdas produtivas e econômicas de todos
os tempos na carcinicultura mundial. Desde o seu surgimento em 1992,
vários estudos vêm sendo conduzidos com o intuito de minimizar o
impacto causado por esta virose nos cultivos. A compreensão dos
mecanismos de defesa antivirais desencadeados pelos crustáceos se
torna cada vez mais imprescindível para um amplo entendimento dos
processos envolvidos na interação patógeno/hospedeiro, como também
para o desenvolvimento de métodos mais eficazes de prevenção e
controle da WSS. Nesse contexto, a utilização da tecnologia do RNAi se
destaca como uma das mais efetivas e potenciais terapias antivirais para
as viroses de crustáceos de interesse econômico [42,50].
O uso potencial de dsRNA para bloquear especificamente a
progressão de infeções virais em peneídeos tem sido utilizado contra, no
mínimo, três vírus não relacionados: WSSV [58,60-62], TSV (Taura
Syndrome Virus) [58] e YHV (Yellow Head Virus) [66-68]. Nesses
estudos, sequências específicas de dsRNA correspondentes à proteínas
virais foram utilizadas como agente terapêutico, e uma vez que o animal
tenha entrado em contato com o vírus correspondente, uma resposta
antiviral específica foi desencadeada levando a uma redução nos índices
de mortalidade e a uma importante limitação na replicação viral. No
caso do WSSV, a replicação pôde ser eficientemente suprimida por
injeções com dsRNA ou siRNA correspondentes aos genes das proteínas
virais vp19, vp28, vp281 e proteína quinase [58,60-62].
No presente estudo, foram avaliados os hemogramas e a
expressão gênica de algumas proteínas associadas ao sistema imune em
L. vannamei induzidos a uma proteção antiviral específica por injeções
com dsRNA de sequência homóloga ao gene codificante para a proteína
vp28 do envelope do WSSV. Os animais foram primeiramente tratados
com dsRNA vp28 e 48h após, desafiados com inóculo puro de WSSV.
O tratamento dos camarões com dsRNA vp28 limitou de maneira
importante a infecção por WSSV, onde elevadas taxas de sobrevivência
(73%) foram registradas após 30 dias da infecção, além de se observar a
ausência do vírus em 80% dos animais sobreviventes. Em contrapartida,
animais infectados com WSSV, mas não tratados com dsRNA vp28,
tiveram uma mortalidade de 100% 5 dias após o desafio. A eficiência da
sequência dsRNA vp28 utilizada nesse estudo corrobora os resultados
anteriormente encontrados por Méjia-Ruiz et al. [62]. Nesse estudo, uma
única dose de dsRNA vp28 (4µg/12g peso vivo) foi suficiente para
conferir uma atividade antiviral em L. vannamei por 30 dias, em 50%
63
dos animais que foram submetidos a infecções reincidentes por WSSV.
Além disso, os sobreviventes tratados com dsRNA vp28 não
apresentavam mais o vírus no organismo [62].
Sabe-se que o grau de proteção conferido por dsRNA específico
varia entre os diferentes genes virais-alvo, indicando que a escolha do
gene alvo é um fator limitante no sucesso da supressão da infecção viral
[58]. Por exemplo, camarões injetados com dsRNA correspondentes aos
genes da vp28 e da proteína quinase (proteína não estrutural do vírus)
apresentam uma maior taxa de sobrevivência, do que aqueles injetados
com dsRNA da vp281, outra proteína do envelope do WSSV [60].
A vp28 é uma das principais proteínas do envelope do WSSV,
sendo responsável pela entrada do vírus nas células do hospedeiro e,
necessitando, contudo, ser sintetizada junto ao virion para que o
processo de infecção se efetive. Uma vez que a síntese da vp28 for
bloqueda via RNAi, a ocorrência dos processos de infecção e replicação
virais é limitada ou interrompida, como demonstrado em nosso estudo e
por outros precedentes [58,60-62,69]. Kim et al. [60] relataram uma
sobrevivência de 100% em P. chinensis infectados com altas doses de
WSSV e previamente injetados com dsRNA vp28 (6µg/6g peso vivo).
Já, Robalino et al. [58] relataram uma sobrevivência de 85% em L.
vannamei injetados com o WSSV e que receberam uma injeção prévia
de dsRNA vp28.
Em nosso trabalho, o fato do tratamento com dsRNA vp28 não
ter causado uma proteção e uma ausência do vírus em 100% dos
animais, deve-se provavelmente ao elevado poder infectante do inóculo
viral utilizado, ou ainda referente à dose utilizada de dsRNA (4µg/12g
peso vivo). Alguns autores sugerem que o efeito inibitório do dsRNA
sob a replicação viral seja dose-dependente. Robalino et al. [58]
observaram uma maior sobrevivência (81%) em L. vannamei (1-2g)
infectados com o WSSV quando injetados com 10µg de dsRNA vp19,
em contrapartida aos 29% de sobrevivência observados em animais
injetados com apenas 0,1µg dsRNA vp19. Em outro estudo, avaliando a
infecção do YHV em P. monodon [67] foi relatada uma completa
proteção dos animais (100% de sobrevivência) somente quando altas
doses de dsRNA (25µg/10g de peso vivo) foram injetadas. No entanto, a
maioria dos estudos com camarões preconiza que doses entre 1-5 µg de
dsRNA específico seja suficiente para limitar a replicação viral e
diminuir significativamente a mortalidade dos animais, posteriormente
infectados pelo vírus a que corresponde [59].
No presente estudo, contudo, um fato a ser considerado é que
todos os animais sobreviventes à infecção por WSSV permaneceram
vivos por 30 dias, mesmo que 20% deles ainda apresentavam o vírus em
64
seu organismo. Esses achados são extremamente relevantes, uma vez
que demonstram que o tratamento prévio de L. vannamei com dsRNA
vp28, na concentração utilizada, foi capaz de induzir o sistema RNAi,
levando ao controle da infecção viral em sua fase inicial, além de
promover a ausência do vírus na maioria dos animais. Dessa maneira, a
utilização de dsRNA vp28 poderia representar uma potencial terapia
antiviral de caráter preventivo contra o WSSV, em camarões desafiados
com altas doses virais.
Em relação ao número de hemócitos circulantes foi observada
uma redução na contagem de hemócitos totais (CTH) em todos os
animais, nas primeiras 6 horas pós-inoculação. Contudo, a CTH nos
animais controle e naqueles tratados com dsRNA vp28/desafiados com
WSSV houve uma recuperação nos valores a partir de 48 e 72 horas da
injeção, diferentemente do grupo apenas desafiado com vírus, que
continuou com sua CTH diminuída. Relatos de diminuições na CTH em
camarões que sofreram estresse por injeções de qualquer natureza são
comuns e podem, em parte, estar associados à infiltração dos hemócitos
no local da injúria para auxiliar no reparo dos tecidos lesados pela
injeção [40]. Por outro lado, a recuperação na CTH dos animais tratados
com dsRNA vp28 e desafiados com vírus sugere que, a ativação do
sistema RNAi, além de limitar a progressão viral, propiciou uma
recuperação nas condições gerais de saúde dos animais, possibilitando
que novas células imunocompetentes fossem produzidas pelo tecido
hematopoiético. Em contrapartida, a queda brusca e crescente da CTH
nos animais apenas desafiados com WSSV, indica que a depleção dos
hemócitos circulantes esteja diretamente associada à replicação viral,
uma vez que ocorreu em estágio mais agudo da infecção.
Sabe-se que os hemócitos são um dos principais sítios de
replicação do WSSV [70] e diversos trabalhos já demonstraram que há
uma diminuição significativa na quantidade de hemócitos em camarões
infectados [16,18,38,40,71-74]. Esse declínio na CTH nos animais
infectados deve particularmente ter ocorrido devido à lise celular intensa
advinda da expressiva replicação viral nos hemócitos. Efetivamente,
Sarathi et al. [40] observaram uma queda na CTH de Fenneropenaeus
indicus após infecção severa com WSSV associada tanto à lise, quanto à
apoptose celulares. Embora os fenômenos de lise e apoptose celulares
não terem sido diretamente verificados em nosso estudo, esses
fenômenos usuais em infecções virais deve ter também ocorrido em
nossos animais infectados. Apesar da apoptose celular não ter sido
sugerida nos animais pelos nossos resultados das análises de expressão
gênica da caspase-3, que foi modulada negativamente pelo WSSV
(dados discutidos adiante), a hipótese de que a diminuição na CTH
65
ocorreu em parte pela apoptose celular não deve ser completamente
descartada. No mínimo duas classes de caspases foram até o momento
identificadas em camarões: uma caspase-3 efetora [46-48] e uma
caspase 8 ou 10 iniciadora [49]. No entanto, é lógicamente esperado que
existam outras classes de capases, a exemplo do que ocorre em outros
animais. Essa hipótese é corroborada pelos recentes achados de Chang
et al. [75], onde L. vannamei infectados com Vibrio alginolyticus
apresentaram hemócitos apoptóticos em período anterior ao aumento da
atividade enzimática e expressão da caspase-3, indicando assim que a
apoptose estaria sendo ativada por outras caspases efetoras,
possivelmente a caspase-7, que em aves participa dos estágios iniciais
do processo apoptótico [76]. Por outro lado, a redução significativa na
CTH observada em L. vannamei em estágios mais avançados da
infecção (72h pós-desafio) pode ainda estar relacionada a uma
diminuição na produção dos hemócitos pelos órgãos hematopoiéticos,
uma vez que já foi demonstrado em outro crustáceo, no lagostim
Pacifastacus leniusculus, que o WSSV infecta as células do tecido
hematopoiético, prejudicando seu funcionamento e, consequentemente,
a produção de novos hemócitos [77].
Em relação à análise de expressão gênica de proteínas
imunológicas nos diferentes grupos de L. vannamei avaliados nesse
estudo, observou-se um comportamento bastante diferenciado para cada
gene no que se refere ao grau de expressão entre os animais desafiados
com WSSV e tratados ou não com dsRNA vp28. Nos animais controle,
não injetados nem com dsRNA vp28, nem com o vírus, houve uma
alteração nos níveis de expressão de alguns genes (caspase-3, ALF e
proPO) nos períodos analisados, estando isso muito provavelmente
relacionado à manipulação dos animais e à injeção com inóculo
negativo. Alterações na expressão de alguns genes imunológicos, tais
como PAMs, já foram relatadas em outros trabalhos onde os camarões
foram injetados apenas com solução salina [78,79]. Além disso, convém
ressaltar que o inóculo negativo utilizado em nosso estudo continha um
pool de proteínas exógenas (de outro animal), e que isso parece ter
alterado, ao menos transitoriamente, a expressão de alguns genes
imunológicos.
Dentre as proteínas do sistema RNAi, a família das argonautas
são as proteínas-chave para o silenciamento gênico pós-transcricional,
uma vez que são componentes fundamentais (proteína slicer) do
complexo multiproteico de silenciamento induzido (RISC)
[54,55,80,81]. Em camarões, duas isoformas da proteína argonauta já
foram clonadas em P. monodon, a PmAgo [56] e a PemAgo [57]. Em
nosso estudo, a análise da expressão gênica da argonauta em L.
66
vannamei infectados com WSSV, mas não tratados com dsRNA vp28,
aumentou somente nas primeiras 3 horas após o desafio, sugerindo uma
estimulação do sistema RNAi restrita ao inicio do processo infeccioso.
Sabe-se que infecções virais severas, como a utilizada em nosso estudo,
levam a uma rápida replicação e evolução do processo infeccioso o que
gera nos animais uma condição de debilidade imunológica generalizada
[70]. À medida que a infecção viral progrediu, uma depleção do sistema
RNAi parece ter ocorrido nos animais, uma vez que a partir das 6h a
expressão da argonauta diminuiu consideravelmente. Em outro estudo
com P. monodom infectados com YHV ou com WSSV, a expressão da
argonauta foi aumentada apenas nas primeiras 24h pós-infeccão [56],
seguido por um declínio rápido na expressão 30h pós-infeccão e sendo
praticamente indetectável nos animais moribundos. A expressão da
argonauta não foi aqui verificada em animais moribundos, no entanto, a
mortalidade total dos animais em 5 dias reforça a hipótese de Unajak et
al. [56], de que a diminuição da expressão dessa proteína slicer, em
estágios mais avançados da infecção, reflete a ausência da atuação do
sistema RNAi inibindo a replicação viral no hospedeiro. Por outro lado,
em nosso estudo, em L. vannamei tratados com dsRNA vp28 observouse uma superexpressão da argonauta (80%) ainda 48h antes do contato
dos animais com o vírus, ou seja, na ausência da forma viral infectante,
expressão essa se mantendo alta até o final das análises (72h). O fato de
estes animais apresentarem uma superexpressão da argonauta ao longo
do experimento, aliado aos altos índices de sobrevivência e a ausência
do vírus em 80% dos sobreviventes, indicam claramente que houve a
ativação do sistema de RNAi nos animais e que este promoveu uma
defesa antiviral contra o WSSV.
Outro mecanismo antiviral desencadeado nos crustáceos é a
apoptose celular, processo este mediado pelas caspases [44,45]. Vários
estudos relatam a indução de apoptose ao longo de infecções virais em
camarões [50,51,73,82]. Em nosso estudo, uma progressiva diminuição
na expressão da caspase-3 foi encontrada nos camarões desafiados com
WSSV (não tratados com dsRNA vp28), chegando a uma depleção
quase total na sua expressão, 72h pós-infecção. O desafio de L.
vannamei com WSSV promoveu uma rápida evolução do processo
infeccioso, debilitando os animais e depletando a expressão dessa
caspase efetora. Por outro lado, apesar de a expressão da caspase nos
camarões tratados com dsRNA vp28 ter sido, em geral, maior do que
nos animais apenas desafiados com WSSV, os níveis de transcritos
foram em geral menores do que os dos animais controle, sugerindo que
a presença do WSSV modula negativamente a expressão gênica da
caspase-3.
67
Embora a apoptose celular não tenha sido diretamente investigada
em nosso estudo, sabe-se que ao longo da evolução os vírus
desenvolveram estratégias para evitar ou retardar este processo e
garantir assim sua replicação no hospedeiro. Vírus de insetos, como
certos baculovírus, podem se beneficiar com a expressão de genes
antiapoptóticos [83,84], agindo diretamente na via de sinalização de
apoptose ou inibindo a atividade de caspases [85]. No caso do WSSV,
um gene codificante para uma nova classe de proteína antiapoptótica,
denominada WSSV449 (ou ORF 390), já foi identificada em seu
genoma [86], sendo que a sua forma recombinante é capaz de inibir in
vitro uma caspase efetora de P. monodon [87].
Além disso, outro fato a ser considerado é de que os animais
severamente infectados estavam imunodeprimidos e impedidos assim de
acionar mecanismos antivirais para conter a infecção brutal, que levou a
morte 100% dos animais em 5 dias. Curiosamente, há relatos na
literatura que descrevem a indução da caspase-3 em P. merguiensis
moribundos infectados pelo WSSV [46] e em P. monodon apenas 48h
após a infecção pelo mesmo vírus [47]. Cabe, contudo salientar que os
estudos mencionados acima foram com outras espécies de peneídeos e
com cepas virais diferentes, além de as análises serem pontuais durante
a infecção, não permitindo dessa forma estabelecer comparações diretas
com nossos resultados. Além disso, como mencionado anteriormente, os
mecanismos de regulação das vias apoptóticas em crustáceos, dos
indutores e inibidores, não são ainda completamente conhecidos [44].
Consequentemente, as divergências encontradas entre os trabalhos
revelam que maiores estudos devem ser coduzidos para elucidar o papel
antiviral da apoptose contra infecções pelo WSSV em crustáceos, bem
como sobre a atuação de proteínas virais antiapoptóticas beneficiando o
processo infeccioso.
Outra proteína recentemente descrita em crustáceos estando
envolvida na limitação de infecções por WSSV é o peptídeo
antimicrobiano ALF [33,34]. No presente estudo, uma depleção na
expressão gênica desse PAM foi encontrada nos animais apenas
desafiados com WSSV, ao longo de todo o experimento, indicando uma
modulação negativa desse gene associada à progressão da replicação por
WSSV em L. vannamei. Por outro lado, a superexpressão do ALF
encontrada nos camarões tratados com dsRNA vp28, nas 3 e 24h após o
desafio com WSSV, sugere que esse PAM deva atuar na fase inicial do
processo infeccioso, embora o seu papel antiviral não tenha sido aqui
investigado. Interessantemente, parece que a expressão do ALF não é
modulada em L. vannamei (1,5g) submetidos a infecções brandas por
WSSV [88]. Já, no lagostim P. leniusculus uma superexpressão do ALF
68
foi registrada nos animais severamente infectados por WSSV [33].
Diferenças entre os nossos resultados e aqueles obtidos por Liu et al.
[33] podem ser em parte devido ao fato de que os crustáceos sejam um
grupo taxonômico diverso, cujas respostas às infecções podem ser
bastante diversificadas, especialmente considerando-se animais de
diferentes habitats, como é o caso de peneídeos e lagostinsde água doce.
Devemos, contudo considerar que existem diferentes isoformas de ALF,
cuja expressão também pode ser diferenciada, além de existir diferentes
cepas de WSSV, com graus diferenciados de virulência.
Se por um lado o papel antiviral do ALF não tenha ainda sido
comprovado, por outro, vários estudos indicam que esse PAM tenha um
efeito inibitório sobre a infecção por diferentes vírus, tais como o
WSSV [34], adenovirus e o vírus da herpes [89]. Liu et al. [34]
demonstraram que o silenciamento gênico pós-transcricional do ALF
leva a um aumento na taxa de replicação do WSSV em P. leniusculus,
sugerindo que esse PAM possua um papel antiviral nos crustáceos. Mais
recentemente, Tharntada et al. [34] demonstraram que uma forma
recombinante de ALF era capaz de inibir in vitro a replicação do WSSV,
em cultura de células hematopoiéticas de P. leniusculus. Nesse mesmo
estudo, ficou ainda demonstrado que camarões P. monodon previamente
injetados com o ALF recombinante tinham a infecção por WSSV
limitada e sua sobrevivência aumentada. Embora não haja, ao nosso
conhecimento, relatos sobre a expressão do ALF em crustáceos
previamente induzidos ao sistema RNAi e desafiados posteriormente
com vírus, nossos resultados demonstram que o tratamento com dsRNA
vp28 levou ao restabelecimento das condições de saúde dos camarões,
limitando a replicação viral através da atuação do sistema RNAi, e
permitindo que PAMs importantes, como o ALF, continuassem sendo
expressos. Em contrapartida, animais desafiados com altas doses de
WSSV e não tratados com dsRNA vp28 tiveram a expressão do ALF
depletada, inviabilizando assim uma potencial atividade antiviral desse
PAM contra o WSSV.
A expressão da proteína que reconhece tanto LPS quanto β-1,3glicanas (LGBP) também foi investigada neste estudo. De maneira
muito interessante, a depleção dos transcritos da LGBP nas 3h após o
desafio viral dos animais (não tratados com dsRNA vp28) foi
semelhante àquela dos animais tratados com dsRNAvp28 após 6h, ainda
antes do desafio com WSSV. Poder-se-ia então especular que, algo na
sequência ou no tamanho do dsRNA vp28 promova a inibição da
expressão gênica da LGBP nessa fase inicial, uma vez que essa
sequência de oligonucleotideo é virtualmente encontrada também nos
animais que foram infectados com WSSV. Os mecanismos que levam a
69
essa depleção gênica são ainda desconhecidos, porém poderiam estar
relacionados à sequência e ao tamanho do oligonucleotídeo dsRNA
vp28 e merecem ser melhor investigados no futuro. Por outro lado, os
altos níveis de expressão da LGBP nos animais apenas infectados pelo
WSSV, 6h após o desafio indicam que a LGBP seja uma proteína
induzível de fase aguda da infecção por WSSV. Esta hipótese foi
reforçada pela análise de expressão com os camarões tratados com
dsRNA vp28 que foi muito semelhante àquela do grupo controle. Assim
como em nosso estudo, um aumento acentuado na expressão da LGBP
foi relatado em L. stylirostris [16,17], e L. vannamei [18] na fase aguda
de uma infecção por WSSV.
Estes resultados conjuntos sugerem um possível envolvimento da
LGBP em infecções virais e não somente em infecções bacterianas e
fúngicas, como comumente preconizado para essa PRP. Yeh et al. [90]
hipotetizaram que esta PRP poderia reconhecer componentes virais, e
por isso sua expressão aumentaria mediante a fase aguda de uma
infecção viral. Estes autores observaram que 12h após a injeção de
Macrobrachium rosembergii com o oligonucleotídeo sintético poly I:C,
que mimetiza um dsRNA viral e ativa o sistema interferon em
mamíferos [91], houve um aumento da expressão da LGBP no
hepatopâncreas dos animais. O fato de a LGBP ter sido superexpressa
em L. vannamei severamente infectados por WSSV, não credencia uma
hipótese definitiva de que essa PRP tenha um papel antiviral,
considerando que se assim fosse os animais não teriam morrido em 5
dias. Ao contrário, poderíamos hipotetizar que sendo as LGBPs
proteínas clássicas de reconhecimento-padrão, produzidas tanto pelos
hemócitos [13-15] quanto pelo hepatopâncreas dos crustáceos [14,16],
essa PRP poderia estar reconhecendo algum componente viral e
facilitando de algum modo a infecção e/ou replicação do WSSV no
hospedeiro, e por isso uma superexpressão seria observada na fase
aguda da infecção. No entanto, essas hipóteses antagônicas de um papel
pró ou antiviral para a LGBP necessitam ser investigadas, para melhor
compreender essa questão.
Outra análise feita em nosso estudo foi avaliar a expressão gênica
do inibidor de proteases α2-macroglobulina (α2M). Essa glicoproteína
plasmática inibe tanto proteases endógenas, como as serino-proteases
ativadoras do sistema proPO [24,92], quanto exógenas produzidas pelo
patógeno, como os vírus, e que potencializam o processo infeccioso
[93]. A expressão da α2M em L. vannamei infectados severamente com
WSSV foi modulada positivamente até 48h pós-desafio. Esse aumento
na produção de transcritos da α2M indica um envolvimento desse
inibidor de proteases na fase aguda da infecção viral, como ocorre com a
70
LGBP. De modo semelhante, um aumento nos transcritos da α2M foi
também encontrado em P. monodon após 48h de infecção por WSSV
[94] e em M. japonicus infectados pelo WSSV [95].
A superexpressão da α2M na fase aguda do desafio viral pode ser
devido ao fato que esse inibidor de proteases participe limitando a
infecção, inibindo proteases virais. Uma endonuclease já foi identificada
no WSSV (vp38B) [96], porém a sua participação no processo
infeccioso não foi ainda demonstrada. Por outro lado, e mais provável
ainda, poder-se-ia aventar que a α2M estaria sendo superexpressa nos
camarões severamente infectados pelo WSSV para “fechar” as proteases
endógenas do hospedeiro, intensamente liberadas durante a lise celular
advinda da intensa replicação viral nas células. De qualquer modo,
podemos supor que esse inibidor de proteases atue no sentido de
minimizar a agressão do patógeno aos tecidos do hospedeiro,
contribuindo para a homeostasia do organismo do animal, sendo esse o
motivo de ser superexpresso durante a fase aguda da infecção.
A participação da α2M em processos infecciosos em crustáceos já
foi sugerida em outros estudos, através do aumento não somente da sua
expressão em camarões infectados por vírus, como acima relatado, mas
também em animais tratados com componentes bacterianos [97-101] ou
submetidos a uma alimentação contendo peptidoglicanas [102]. Além
disso, a α2M é capaz de inibir in vitro proteases do fungo Aphanomyces
spp, um patógeno conhecido de lagostins [103]. Um fato relevante
encontrado em nosso estudo é que o aumento dos RNAm para α2M nas
primeiras 48h da infecção por WSSV é consoante com os nossos
achados anteriores de aumento da atividade biológica da α2M, após 72h
do desafio viral [74]. Em outras palavras, a superexpressão gênica da
α2M nas 48h pós-infecção precede o aumento da atividade biológica da
proteína, que ocorre nas 72h.
Outra conclusão relevante é que a depleção gênica da α2M
encontrada nos animais sob forte infecção viral nas 72h após o desafio
com WSSV, coincide com a depleção na expressão de outros genes
imunológicos analisados (caspase-3, ALF, proPO e TGase),
demonstrando claramente que esses animais apresentavam uma
condição de debilidade imunológica generalizada, resultando em morte
massiva dos mesmos, 48h após. Já, aqueles animais que foram
previamente injetados com dsRNA vp28 tiveram um perfil de expressão
da α2M muito diferente e semelhante ao do grupo controle, ou seja,
próximo da normalidade. Esses achados indicam, portanto, que esse
inibidor de proteases é superexpresso em função da progressão da
infecção do camarão por WSSV.
71
De modo contrário ao encontrado para as expressões da α2M e da
LGBP, o número de transcritos da proPO foi em geral muito diminuído
em L. vannamei desafiados por WSSV, indicando que esse vírus module
negativamente a expressão desse zimógeno, por consequência,
depletando a ativação do sistema proPO, que é um importante
mecanismo de reconhecimento do não-próprio em crustáceos. Diversos
trabalhos também relataram uma diminuição tanto na expressão gênica
da proPO [16,18,19], quanto na atividade da forma enzimática ativa, PO
[16,18,72,74,104,105] em camarões submetidos a infecções virais. Em
relação à atividade enzimática diminuída, alguns autores sugerem que a
infecção ativaria a expressão de inibidores de proteases, que por sua vez,
impediriam a ativação da proPO, levando a inibição de todo o sistema
proPO. De certa maneira, nossos resultados corroboram essa hipótese,
uma vez que um dos reguladores negativos do sistema proPO, a α2M
[24,92], foi superexpresso após desafio dos camarões com WSSV. No
entanto, cabe salientar que existem outros inibidores de protease que
atuam na regulação do sistema propor, tais como a pacifastina [20], os
inibidores da família Kazal [21,22,106] e a serpina [23].
De maneira interessante, os animais que foram tratados com
dsRNA vp28 apresentaram valores de expressão da proPO sempre
superiores aos animais apenas desafiados com vírus, sendo que o pico
de expressão em ambos grupos ocorreu nas 24h após desafio viral. O
motivo pelo qual o pico de superexpressão da proPO ocorreu no período
de 24h pós-desafio é, ainda, um fato desconhecido. Outros picos de
expressão também foram detectados em nosso estudo, em diferentes
momentos e dependendo do gene em questão (argonauta, caspase-3,
α2M, ALF e TGase), e análises futuras com qPCR poderiam auxiliar o
esclarecimento destes pontos. Contudo, podemos inferir que o maior
grau de expressão da proPO nos animais cujo sistema RNAi tenha sido
ativado, demonstra novamente que o tratamento com dsRNA vp28
propiciou aos animais melhores condições gerais de saúde, uma vez que
a infecção viral foi limitada, possibilitando a superexpressão de um
zimógeno do sistema proPO, um mecanismo de reconhecimento do não
próprio dos mais importantes dos crustáceos.
E por fim, a depleção na expressão da enzima de coagulacao
transglutaminase (TGase) nos animais não tratados com dsRNA vp28 e,
por consequência, com infecção severa por WSSV, também modulou
negativamente a expressão desta enzima, a exemplo da maioria dos
genes analisados nesse estudo. Estudos anteriores relatam uma
diminuição na expressão da TGase em F. chinensis a partir de 24h pósinfecção pelo WSSV [37]. Por outro lado, um aumento no tempo de
coagulação da hemolinfa é normalmente observado em camarões
72
severamente infectados por WSSV [38-40], o que é compatível com
uma expressão diminuída de TGase. Enquanto isso, nos animais tratados
com dsRNA vp28 os níveis de expressão deste gene permaneceram, em
geral, elevados durante o período experimental, indicando a recuperação
imunológica dos mesmos. Sabe-se que a coagulação da hemolinfa é um
mecanismo imunológico essencial para a sobrevivência dos crustáceos,
pois previne a perda de hemolinfa e a disseminação dos patógenos pela
cavidade corpórea do animal [12]. Recentemente, Maningas et al. [107]
demonstraram que o silenciamento pós-transcricional tanto da TGase
quanto da proteína de coagulação (CP) leva a um aumento na taxa de
mortalidade de M. japonicus infectados pelo WSSV, indicando um forte
envolvimento destas moléculas na resposta imune desses animais contra
vírus. Em vista disso, uma possível participação da transglutaminase nos
mecanismos de resistência dos crustáceos frente às infecções virais pode
ocorrer e necessita ser mais bem investigada no futuro.
Sendo assim, diante dos resultados obtidos neste estudo, podemos
concluir que em L. vannamei infectados severamente com WSSV, mas
não tratados com dsRNA vp28, ocorre uma superexpressão dos genes
LGBP e α2M e uma depleção na expressão gênica da argonauta,
caspase-3, ALF, proPO e TGase, além de promover uma queda
acentuada na quantidade de hemócitos circulantes. Já, o tratamento dos
animais com dsRNA vp28 antes do desafio com WSSV induziu um
aumento na expressão gênica do ALF, da proPO, da TGase e da
argonauta na maioria dos períodos analisados.
A superexpressão da LGBP e da α2M em animais infectados por
WSSV demonstra o envolvimento dessas proteínas na fase aguda de
uma infecção por WSSV, não sabendo, contudo se essas proteínas
participam de mecanismos antivirais, uma vez que os animais não
sobreviveram à infecção. Por outro lado, a modulação positiva dos
genes argonauta, ALF, proPO e TGase em L. vannamei cuja infecção
viral foi limitada via RNAi, sugere a participação destas proteínas na
defesa do organismo contra o WSSV, através da manutenção da sua
imunidade. Com exceção da argonauta, cuja participação na defesa
antiviral via RNAi já está bem estabelecida, a participação efetiva das
outras proteínas acima em mecanismos antivirais permanece
desconhecida, necessitando de maiores estudos para que se comprove
essa hipótese.
Além disso, o tratamento prévio de L. vannamei com dsRNA
vp28, na concentração utilizada nesse estudo mostrou-se eficaz para
induzir uma resposta antiviral contra o WSSV, via RNAi. A alta taxa de
sobrevivência e a restituição das boas condições imunológicas dos
animais desafiados, indicam que o tratamento com dsRNA vp28 levou
73
ao controle da infecção viral em sua fase inicial, limitando-a e
promovendo a ausência do vírus em 80% dos sobreviventes. Dessa
maneira, o dsRNA vp28 poderia ser utilizado, mediante estudos futuros
de viabilidade econômica, como um agente terapêutico para
prevenir/limitar infecções por WSSV em camarões.
Apesar da aplicação prática da tecnologia de RNAi ser ainda
inviável na carcinicultura, devido aos elevados custos e à dificuldade de
aplicação nos animais via sistêmica, recentes estudos apontam para uma
adaptação menos onerosa da técnica, o que viabilizaria a sua utilização
futura em larga escala [108]. Quanto à forma de administração do
dsRNA nos animais, um dos métodos alternativos a serem testados seria
a administração oral de bactérias produzindo o dsRNA (bactérias
transformadas, deficientes em RNAse III), método este já estabelecido
para o nematódo C. elegans [109]. Recentemente, Sarathi et al. [69]
investigaram a possibilidade de proteger camarões contra WSSV pela
administração oral de ração coberta com bactérias produzindo dsRNA
vp28 ou com nanopartículas de quitosana contendo dsRNA da vp28.
Cabe, contudo salientar que, a ativação do sistema RNAi depende da
entrada do dsRNA/siRNA na célula do hospedeiro e, portanto, a
administração via oral deve garantir que os constructos atravessem de
fato o trato digestório do camarão sem serem degradados e atinjam os
tecidos do animal. Embora estudos nessa área sejam ainda escassos,
existe uma forte demanda de pesquisas com o intuito de validar técnicas
e diminuir os custos de aplicação, já que é consensual que o mecanismo
antiviral baseado na técnica de RNAi representa atualmente a
abordagem terapêutica melhor fundamentada, efetiva e promissora no
controle das viroses de crustáceos cultivados.
AGRADECIMENTOS
Os autores gostariam de agradecer à CAPES pela bolsa concedida
à primeira autora, ao professor Maurício Lehmann do Instituto Federal
Catarinense-Campus Araquari pelo auxílio durante a fase do desafio
experimental, e ao Laboratório de Camarões Marinhos (UFSC), em
especial ao Dr. Felipe Vieira, pelo fornecimento dos animais utilizados
neste estudo.
74
6. REFERÊNCIAS
[1] Lightner, DV. The Penaeid Shrimp Viral Pandemics due to IHHNV,
WSSV, TSV and YHV. In: Sakai Y, Mcvey JP, Jang D, Mcvey E,
Caesar (Eds.), History in the Americas and Current Status, Japan:
Proceedings of the US-Cooperative Program in Natural Resources
(UJNR) 2003; 6-24.
[2] Flegel TW. Detection of major penaeid shrimp viruses in Asia, a
historical perspective with emphasis on Thailand. Aquaculture 2006;
258:1-33.
[3] Escobedo-Bonilla CM, Alday-Sanz V, Wille, M, Sorgeloos, P,
Pensaert, MB, Nauwynck, HJ. A review on the morphology, molecular
characterization, morphogenesis and pathogenesis of white spot
syndrome virus. J Fish Dis 2008; 31: 1–18.
[4] Chou HY, Huang CY, Wang H, Chiang HC, Lo CF. Pathogenicity of
a baculovirus infection causing White spot syndrome in cultured
penaeid shrimp in Taiwan. Dis Aquat Organ 1995; 23: 165-173.
[5] Chang CF, Su MS, Chen HY, Lo CF, Kou GH, Liao IC. Effect of
dietary beta-1,3-glucan on resistance to white spot syndrome virus
(WSSV) in postlarval and juvenile Penaeus monodon. Dis Aquat Organ
1999; 36:163–8.
[6] OIE World organisation for animal health. Manual of Diagnostic
Tests
for
Aquatic
Animals
2006.
Disponível
em
<http://www.oie.int/eng/en_index.htm> Acesso em 15/02/2010.
[7] Seiffert WQ, Beltrame E, Andreatta ER, Magionni DS.
Enfermidades: uma oportunidade para repensar o cultivo de camarões
marinhos. Panorama da Aqüicultura 2006; 16: 32-38.
[8] Cerenius, L., Söderhäll, K. The prophenoloxidase-activating system
in invertebrates. Immun Rev 2004; 198: 116-126.
[9] Johnson KN, van Hulten MC, Barnes AC. "Vaccination" of shrimp
against viral pathogens: phenomenology and underlying mechanisms.
Vaccine 2008; 8:4885-4892.
75
[10] Iwanaga S, Lee BL. Recent advances in the innate immunity of
invertebrate animals. Bioch Mol Biol 2005; 38: 128-150.
[11] Lee SY, Söderhäll K. Early events in crustacean innate immunity.
Fish Shellfish Immunol 2002; 12:(5) 421-437.
[12] Barracco MA, Perazzolo LM, Rosa RD. Inmunologia del camarón.
In: Vielka MQ, Cuéllar-Anjel J. (Eds.), Guía Práctica de Patología y
inmunología de Camarones Penaeidos, Ed. CYTED 2008; 169-224.
[13] Lee SY, Wang R, Söderhäll K. A lipopolysaccharide-and β-1,3glucan binding protein from hemocytes of the freshwater crayfish,
Pacifastacus leniusculus. Purification, characterization and cDNA
cloning. J Biol Chemistry 2000; 275:1337-1343.
[14] Cheng W, Liu CH, Tsai CH, Chen JC. Molecular cloning and
characterization of a pattern recognition molecule, lipopolysaccharideand b-1,3-glucan binding protein (LGBP) from the white shrimp
Litopenaeus vannamei. Fish Shellfish Immunol 2005;18:297–310.
[15] Du XJ, Zhao XF, Wang JX. Molecular cloning and characterization
of a lipopolysaccharide and β-1,3-glucan binding protein from fleshy
prawn (Fenneropenaeus chinensis). Mol Immunol 2007; 44:1085-1094.
[16] Roux MM, Pain A, Klimper KR, Dhar AY. The lipopolysaccharide
and β-1,3-glucan binding protein gene is upregulated in white spot
virus-infected shrimp (Penaeus stylirostris). J Virol 2002; 76:71407149.
[17] Dhar AK, Dettori A, Roux MM, Klimpel KR, Read B.
Identification of differentially expressed genes in shrimp (Penaeus
stylirostris) infected with white spot syndrome virus by cDNA
microarrays. Arch Virol 2003;148:2381–96.
[18] Yeh S, Chen Y, Hsieh S, Cheng W, Liu C. Immune response of
white shrimp, Litopenaeus vannamei, after a concurrent infection with
white spot syndrome virus and infectious hypodermal and hematopoietic
necrosis virus. Fish Shellfish Immunol 2009; 26: 582-588.
[19] Ai HS, Huang YC, Li SD, Weng SP, Yu XQ, He JG.
Characterization of a prophenoloxidase from hemocytes of the shrimp
Litopenaeus vannamei that is down-regulated by WSSV. Fish Shellfish
Immunol 2008; 25:28–39.
76
[20] Liang Z, Sottrup-Jensen L, Söderhäll K. Pacifastin, a novel 155kDa heterodimeric proteinase inhibitor containing a unique transferrin
chain. Proceedings of the National Academy of Sciences USA 1997;
94:6682-6687.
[21] Johansson MW, Keyser P, Söderhäll K. Purification and cDNA
cloning of a four-domains Kazal proteinase inhibitor from crayfish
blood cells. European Journal of Biochemistry 1994; 223:389-394.
[22] Jiménez-Vega F, Vargas-Albores F. A four-Kazal domain protein
in Litopenaeus vannamei hemocytes. Dev Comp Immunol 2005;
29:385- 391.
[23] Liang Z, Söderhäll K. Isolation of cDNA encoding a novel serpin
of crayfish hemocytes. Comparative Biochemistry and Physiology 1995;
2:385-391.
[24] Hergenhahn HG, Hall M, Söderhäll K. Purification and
characterization of an alpha2-macroglobulin-like proteinase inhibitor
from plasma of the crayfish Pacifastacus leniusculus. The Biochem J
1988; 255:801-806.
[25] Gross PS, Bartlett TC, Browdy CL, Chapman RW, Warr GW.
Immune gene discovery by expressed sequence tag analysis of
hemocytes and hepatopancreas in the Pacific White Shrimp,
Litopenaeus vannamei, and the Atlantic White Shrimp, L. setiferus. Dev
Comp Immunol 2001; 25:565-577.
[26] Rattanachai A, Hirono I, Ohira T, Takahashi Y, Aoki T. Molecular
cloning and expression analysis of alpha2-macroglobulin in the kuruma
shrimp, Marsupenaeus japonicus. Fish Shellfish Immunol 2004; 16:
599-611.
[27] Armstrong PB. Proteases and protease inhibitors: a balance of
activities in hostpathogen interaction. Immunobiology 2006; 211:263281.
[28] Destoumieux D, Bulet P, Loew D, Van Dorsselaer A, Rodriguez J,
Bachère E. Penaeidins: A new family of antimicrobial peptides in the
shrimp Penaeus vannamei (Decapoda). J Biol Chemistry 1997;
272:28398-28406.
77
[29] Bartlett TC, Cuthbertson BJ, Shepard EF, Chapman RW, Gross PS,
Warr GW. Crustins, homologues of an 11.5-kDa antibacterial peptide,
from two species of penaeid shrimp, Litopenaeus vannamei and
Litopenaeus setiferus. Marine Biotechnology NY 2002; 4:278-293.
[30] Supungul P, Klibunga S, Pichyangkura R, Jitrapakdee S, Hirono I,
Aoki T, Tassanakajon A. Identification of immune-related genes in
hemocytes of black tiger shrimp (Penaeus monodon). Marine
Biotechnology 2002; 4:487-494.
[31] Somboobwiwat K, Marcos M, Tassanakajon A, Klinbunga S,
Aumelas A, Romestand B, Guenguen Y, Boze H, Moulin G, Bachère E.
Recombinant expression and anti-microbial activity of antilipopolysaccharide factor (ALF) from the black tiger shrimp Penaeus
monodon. Dev Comp Immun 2005; 29:841-851.
[32] Nagoshi H, Inagawa H, Morii K, Harada H, Kohchi C, Nishizawa
T, Taniguchi Y, Uenobe M Honda T, Kondoh M, Takahashi Y, Soma G.
Cloning and characterization of a LPS-regulatory gene having an LPS
binding domain in kuruma prawn Marsupenaeus japonicus. Mol
Immunol 2006; 43: 2061-2069.
[33] Liu H, Jiravanichpaisal P, Soderhall I, Cerenius L, Soderhall K.
Antilipopolysaccharide factor interferes with white spot syndrome virus
replication in vitro and in vivo in the crayfish Pacifastacus leniusculus. J
Virol 2006; 80:10365–10371.
[34] Tharntada S, Ponprateep S, Somboonwiwat K, Liu H, Soderhall I,
Soderhall K, Tassanakajon A. Role of anti-lipopolysaccharide factor
from the black tiger shrimp, Penaeus monodon, in protection from white
spot syndrome virus infection Journal of General Virology 2009;
90:1491-1498.
[35] Lorand L, Conrad SM. Transglutaminases. Mol Cel Bioch 1984;
58:9-35.
[36] Perazzolo LM, Lorenzini DM, Daffre S, Barracco MA. Purification
and partial characterization of the plasma clotting protein from the pink
shrimp Farfantepenaeus paulensis. Comp Bioch Phys 2005; 142:302307.
78
[37] Liu Y-C, Li F-H, Wang B, Dong B, Zhang Q-L, Luan W, Zhang XJ , Xiang J-H. A transglutaminase from Chinese shrimp
(Fenneropenaeus chinensis), full-length cDNA cloning, tissue
localization and expression profile after challenge. Fish Shellfish
Immunol 2007; 22: 576-588.
[38] Yoganandhan K, Thirupathi S, Sahul Hameed AS. Biochemical and
hematological changes in white spot syndrome virus (WSSV)-infected
shrimp, Penaeus indicus. Aquaculture 2003; 221: 1–11.
[39] Sahul Hameed AS, Sarathi M, Sudhakaran R, Balasubramanian G,
Syed Musthaq S. Quantitative assessment of apoptotic hemocytes in
white spot syndrome virus (WSSV)-infected penaeid shrimp, Penaeus
monodon and Penaeus indicus, by flow cytometric analysis.
Aquaculture 2006; 256:111–120.
[40] Sarathi M, Ishaq Ahmed VP, Venkatesan C, Balasubramanian G,
Prabavathy J, Sahul Hameed AS. Comparative study on immune
response of Fenneropenaeus indicus to Vibrio alginolyticus and white
spot syndrome virus. Aquaculture 2007; 271:8–20.
[41] Robalino J, Bartlett TC, Chapman RW, Gross PS, Browdy CL,
Warr GW. Double-stranded RNA and antiviral immunity in marine
shrimp: inducible host mechanisms and evidence for the evolution of
viral counter-responses. Dev Comp Immunol 2007; 31:539-547.
[42] Liu H, Söderhäll K, Jiravanichpaisal P. Antiviral immunity in
crustaceans. Fish Shellfish Immunol 2009; 27:79-88.
[43] Roulston A, Marcellus RC, Branton PE. Viruses and apoptosis.
Annual Review of Microbiology 1999; 53:577-628.
[44] Menze MA, Fortner G, Nag S, Hand SC. Mechanisms of apoptosis
in Crustacea: what conditions induce versus suppress cell death?
Apoptosis 2010; 15:293–312.
[45] Chowdhury I, Tharakan B, Bhat GK. Caspases – an update. Comp
Bioch Physiology- Biochem Mol Biol 2008; 151:10-27.
[46] Phongdara A, Wanna W, Chotigeat W. Molecular cloning and
expression of caspase from white shrimp Penaeus merguiensis.
Aquaculture 2006; 252:114-120.
79
[47] Wongprasert K, Sangsurya P, Phongdar A. Cloning and
characterization of a caspase gene from black tiger shrimp (Penaeus
monodon) infected with white spot syndrome virus (WSSV). J
Biotechnol 2007; 131: 9-19.
[48] Rijiravanich, A.; Browdy, C. L ; Withyachumnarnkul, B.
Knocking down caspase-3 by RNAi reduces mortality in Pacific white
shrimp Penaeus (Litopenaeus) vannamei challenged with a low dose of
white-spot syndrome virus Fish Shellfish Immunol 2008; 24: 308-313.
[49] Wang L, Zhi B, Wu W, Zhang X. Requirement for shrimp caspase
in apoptosis against virus infection. Dev Comp Immunol 2008; 32:706715.
[50] Sahtout AH, Hassan MD, Shariff M. DNA fragmentation, an
indicator of apoptosis, in cultured black tiger shrimp Penaeus monodon
infected with white spot syndrome virus (WSSV). Dis Aquat Org 2001;
44:155-159.
[51] Wu JL, Muroga K. Apoptosis does not play an important role in the
resistance of ‘immune’ Penaeus japonicus against white spot syndrome
virus. J Fish Dis 2004; 27:15-21.
[52] Hannon GJ. RNA interference. Nature 2002; 418:244-251.
[53] Hamilton AJ, Baulcombe DC. A species of small antisense RNA in
posttranscriptional gene silencing in plants. Science1999; 286: 950-952.
[54] Hammond SM, Boettcher AA, Caudy R, Kobayashi GL, Hannon
GJ. Argonaute2, a link between genetic and biochemical analyses of
RNAi. Science 2001; 293:1146–1150.
[55] Carmell MA, Xuan Z, Zhang MQ, Hannon GJ. The Argonaute
family: tentacles that reach into RNAi, developmental control, stem cell
maintenance, and tumorigenesis. Genes Dev 2002; 16: 2733–2742.
[56] Unajak S, Boonsaeng V, Jitrapakdee S. Isolation and
characterization of cDNA encoding Argonaute, a component of RNA
silencing in shrimp (Penaeus monodon). Comp Bioch Physiol 2006;
145:179–187.
80
[57] Dechklar M, Udomkit A, Panyim S. Characterization of Argonaute
cDNA from Penaeus monodon and implication of its role in RNA
interference. Bioch Bioph Res Commun 2008; 367: 768–774.
[58] Robalino J, Bartlett T, Shepard E, Prior S, Jaramillo G, Scura E,
Chapman RW, Gross PS, Browdy CL, Warr GW. Double-stranded RNA
induces sequence-specific antiviral silencing in addition to nonspecific
immunity in a marine shrimp: convergence of RNA interference and
innate immunity in the invertebrate antiviral response? J Virol 2005;
79:13561-13571.
[59] Shekhar M, Lu Y. Application of nucleic-acid-based therapeutics
for viral infections in shrimp aquaculture. Mar Biotechnol 2009; 11:1-9.
[60] Kim CS, Kosuke Z, Nam YK, Kim SK, Kim KH. Protection of
shrimp (Penaeus chinensis) against white spot syndrome virus (WSSV)
challenge by doublestranded RNA. Fish Shellfish Immunol 2007; 23:
242-246.
[61] Xu J, Han F, Zhang X. Silencing shrimp white spot syndrome virus
(WSSV) genes by siRNA. Antiviral Res 2007; 73:126-131.
[62] Mejía-Ruíz CH, Vega-Peña S, Escobedo-Bonilla CM. Sequencespecific double-stranded (ds) RNA against white spot syndrome virus
(WSSV) protects Litopenaeus vannamei from consecutive reinfections.J Invertebrate Pathol 2010; in press.
[63] Yi G, Wang Z, Qi Y,Yao L, Qian J, Hu L. VP28 of shrimp White
Spot Syndrome Virus is involved in the attachment and penetration into
shrimp cells. J Biochem Mol Biol 2004; 37:726-734.
[64] Prior S, Browd CL, Shepard EF, Lamore R, Parnell PG. Controlled
bioassay systems for determination of lethal infective doses of tissue
homogenates containing Taura syndrome or white spot syndrome virus.
Dis Aquat Org 2003; 54:89-96.
[65] Beçak W, Paulete J. Técnicas de citologia e histologia. Rio de
Janeiro: Livros técnicos e científicos. 1976; v1, 327p.
[66] Tirasophon W, Roshorm Y, Panyim S. Silencing of yellow head
virus replication in penaeid shrimp cells by dsRNA. Biochem Biophys
Res Commun 2005; 334:102-107.
81
[67] Yodmuang S, Tirasophon W, Roshorm Y, Chinnirunvong W,
Panyim, S. YHV protease dsRNA inhibits YHV replication in Penaeus
monodon and prevents mortality. Biochem Biophys Res Commun 2006;
341:351-356.
[68] Assavalapsakul W, Chinnirunvong W, Panyim, S. Application of
YHV-protease dsRNA for protection and therapeutic treatment against
yellow head virus infection in Litopenaeus vannamei. Dis Aquat Org
2009; 84:167-171.
[69] Sarathi M, Simon MC, Venkatesan C, Hameed AS. Oral
administration of bacterially expressed VP28dsRNA to protect Penaeus
monodon from white spot syndrome virus. Mar Biotechno 2008;
10:242-249.
[70] Wang YT, Liu W, Seah JN, Lam CS, Xiang JH, Korzh V. White
spot syndrome virus (WSSV) infects specific hemocytes of the shrimp
Penaeus merguiensis. Dis Aquat Org 2002; 52:249–59.
[71] Hennig O, Itami T, Maeda M, Kondo M, Natsukari Y, Takahashi
Y. Analyses of hemolymph immunoparameters in Kuruma shrimp
infected with penaeid rodshaped DNA virus. Fish Pathology 1998;
33:389–93.
[72] Song YL, Yu CI, Lien TW, Huan CC, Lin MN. Haemolymph
parameters of Pacific white shrimp (Litopenaeus vannamei) infected
with Taura syndrome virus. Fish Shellfish Immunol 2003; 14:317-331.
[73] Wongprasert K, Khanobdee K, Glunukarn SS, Meeratana P,
Withyachumnarnkul B. Time-course and levels of apoptosis in various
tissues of black tiger shrimp Penaeus monodon infected with white-spot
syndrome virus. Dis Aquat Org 2003; 55:3-10.
[74] Cantelli L. Avaliação do efeito de polissacarídeos sulfatados da
macroalga Gracillaria birdiae sobre as condições de imunocompetência
de camarões Litopenaeus vannamei infectados com o vírus da síndrome
da mancha branca (WSSV). Dissertação de Mestrado, Universidade
Federal de Santa Catarina 2009; 68p.
[75] Chang C-C, Yeh M-S, Lin H-K, Cheng W. The effect of Vibrio
alginolyticus infection on caspase-3 expression and activity in white
82
shrimp Litopenaeus vannamei. Fish Shellfish Immunol 2008; 25:672–
678.
[76] Korfali N, Ruchaud S, Loegering D, Bernard D, Dingwall C,
Kaufmann SH, et al. Caspase-7 gene disruption reveals an involvement
of the enzyme during the early stages of apoptosis. J Biol Chem 2004;
279:1030–9.
[77] Jiranvanichpaisal P, Lee BL, Söderhäll K. Cell-mediated immunity
in arthropods: hematopoiesis, coagulation, melanization and
opsonization. Immunobiology 2006; 211:213-236.
[78] Okumura T. Effects of lipopolysaccharide on gene expression of
antimicrobial peptides (penaeidins and crustin), serine proteinase and
prophenoloxidase in haemocytes of the Pacific white shrimp,
Litopenaeus vannamei. Fish Shellfish Immunol 2007; 22:68-76.
[79] Dong C, Zhao J, Song L, Wang L, Qiu L, Zheng P, Li L, Gai Y,
Yang G. The immune responses in Chinese mitten crab Eriocheir
sinensis challenged with double-stranded RNA. Fish Shellfish Immunol
2009; 26:438–442.
[80] Martinez J, Patkaniowska A, Urlaub H, Hrman RL, Tuschl T.
Single-stranded antisense siRNAs guide target RNA cleavage in RNAi.
Cell 2002; 110:563-74.
[81] Carmell MA, Hannon GJ. RNase III enzymes and the initiation of
gene silencing. Nature Struct Mol Biol 2004;11:214-8.
[82] Khanobdee K, Soowannayan C, Flegel TW, Ubol S,
Withyachumnarnkul B. Evidence for apoptosis correlated with mortality
in the giant black tiger shrimp Penaeus monodon infected with yellow
head virus. Dis Aquat Organ 2002; 48:79-90.
[83] Clem RJ. Regulation of programmed cell death by baculoviruses.
In: Miller, L.K. (Ed.). The baculoviruses. New York: Plenum Press
1997; p 237-266.
[84] Maguire T, Harrison P, Hyink O, Kalmakoff J, Ward VK. The
inhibitors of apoptosis of Epiphyas postvittana nucleopolyhedrovirus. J
Gen Virol 2000; 81:2803-2811.
83
[85] Tschopp J, Thome M, Hofmann K, Meinl E. The fiht of viruses
against apoptosis. Current Opin Genet Dev 1998; 18:82-87.
[86] Wang Z, Hu L, Yi G, Xu H, Qi Y, Yao L. ORF390 of white spot
syndrome virus genome is identified as a novel anti-apoptosis gene.
Bioch Bioph Res Commun 2004; 325:899-907.
[87] Leu JH, Wang HC, Kou GH, Lo CF. Penaeus monodon caspase is
targeted by a white spot syndrome virus anti-apoptosis protein. Dev
Comp Immunol 2008; 32:476-86.
[88] de-la-Vega E, O’Leary NA, Shockey JE, Robalino J, Payne C,
Browdy CL, Warr GW, Gross PS. Antilipopolysaccharide factor in
Litopenaeus vannamei (LvALF): A broad spectrum antimicrobial
peptide essential for shrimp immunity against bacterial and fungal
infection. Mol Immunol 2008; 45:1916–1925.
[89] Carriel-Gomes MC, Kratz JM, Barracco MA, Bachére E, Barardi
CRM, Simões CMO. In vitro antiviral activity of antimicrobial peptides
against herpes simplex virus 1, adenovirus, and rotavirus. Mem Inst
Oswaldo Cruz 2007; 102(4):469-472.
[90] Yeh M-S, Chang C-C, Cheng W. Molecular cloning and
characterization of lipopolysaccharide- and β-1,3-glucan-binding protein
from the giant freshwater prawn Macrobrachium rosenbergii and its
transcription in relation to foreign material injection and the molt stage.
Fish Shellfish Immunol 2009; 27:701–706.
[91] Alexopoulou L, Holt AC, Medzhitov R, Flavell RA. Recognition of
double-stranded RNA and activation of NF-kB by Toll-like receptor 3.
Nature 2001; 413(6857):732-738.
[92] Aspán A, Hall M, Söderhäll K. The effect of endogenous
proteinase inhibitors on the prophenoloxidase activating enzyme, a
serine proteinase from crayfish haemocytes. Insect Biochem1990;
20:485-492.
[93] Armstrong PB. Proteases and protease inhibitors: a balance of
activities in host pathogen interaction. Immunobiology 2006; 211:263281.
84
[94] Tonganunt M, Phongdara A, Chotigeat W, Fuji K. Identification
and characterization of syntenin binding protein in the black tiger
shrimp Penaeus monodon. J Biotech 2005; 120:135-145.
[95] Rojtinnakorn J, Hirono I, Itami T, Takahashi Y, Aoki T. Gene
expression in haemocytes of kuruma prawn, Penaeus japonicus, in
response to infection with WSSV by EST approach. Fish Shellfish
Immunol 2002; 13:69–83.
[96] Tsai JM, Wang HC, Leu JH, Hsiao HH, Wang AHJ, Kou GH, Lo
CF. Genomic and proteomic analysis of thirty-nine structural proteins of
shrimp white spot syndrome virus. J Virol 2004; 78:11360–11370.
[97] Vaseeharan B, Lin YC, Ko CF, Chiou TT, Chen JC. Molecular
cloning and characterisation of a thioester-containing alpha2macroglobulin (alpha2-M) from the haemocytes of mud crab Scylla
serrata. Fish Shellfish Immunol 2007; 22:115-130.
[98] Lin YC, Vaseeharan B, Ko CF, Chiou TT, Chen JC. Molecular
cloning and characterisation of a proteinase inhibitor, alpha 2macroglobulin (alpha2-M) from the haemocytes of tiger shrimp Penaeus
monodon. Mol Immunol 2007; 44:1065-1074.
[99] Lin YC, Vaseeharan B, Ko CF, Chen JC. Molecular cloning and
phylogenetic analysis on α2-macroglobulin (α2-M) of white shrimp
Litopenaeus vannamei. Dev Comp Immunol 2008; 32:317–329.
[100] Ho P-Y, Cheng C-H, Cheng W. Identification and cloning of the
α2-macroglobulin of giant freshwater prawn Macrobrachium rosenbergii
and its expression in relation with the molt stage and bacteria injection.
Fish Shellfish Immunol 2009; 26:459–466.
[101] Lu K-Y, Sung H-J, Liu C-L, Sung H-H. Differentially enhanced
gene expression in hemocytes from Macrobrachium rosenbergii
challenged in vivo with lipopolysaccharide. J Invertebrate Pathol 2009;
100:9–15.
[102] Rattanachai A, Hirono I, Ohira T, Takahashi Y, Aoki T.
Molecular cloning and expression analysis of alpha2-macroglobulin in
the kuruma shrimp, Marsupenaeus japonicus. Fish Shellfish Immunol
2004; 16:599-611.
85
[103] Dieguez-Uribeondo J, Cerenius J. The inhibition of extracellular
proteinases from Aphanomyces spp by three different proteinase
inhibitors from crayfish blood. Mycological Res 1998; 102:821-824.
[104] Mathew S, Kumar KA, Anandan R, Nair PGV, Devadasan K.
Changes in tissue defence system in white spot syndrome virus (WSSV)
infected Penaeus monodon. Comp Biochem Physiol 2007; 145:315–320.
[105] Costa AM, Buglione CC, Bezerra FL, Martins PCC, Barracco
MA. Immune assessment of farm-reared Penaeus vannamei shrimp
naturally infected by IMNV in NE Brazil. Aquaculture 2009; 291:141146.
[106] Somprasong N, Rimphanitchayakit V, Tassanakajon A. A
fivedomain Kazal-type serine proteinase inhibitor from black tiger
shrimp Penaeus monodon and its inhibitory activities. Dev Comp
Immunol 2006; 30:998-1008.
[107] Maningas MBB, Kondo H, Hirono I, Saito-Taki T, Aoki T.
Essential function of transglutaminase and clotting protein in shrimp
immunity. Mol Immunol 2008; 45:1269–1275.
[108] Saksmerprome V, Charoonnart P, Gangnonngiw W,
Withyachumnarnkul B. A novel and inexpensive application of RNAi
technology to protect shrimp from viral disease. J Virol Methods 2009;
162:213-217.
[109] Timmons L, Court DL, Fire A. Ingestion of bacterially expressed
dsRNAs can produce specific and potent genetic interference in
Caenorhabditis elegans. Gene 2001; 263:103-112.
86
REFERÊNCIAS BIBLIOGRÁFICAS DA INTRODUÇÃO
AI, H-S. et al. Characterization of a prophenoloxidase from hemocytes
of the shrimp Litopenaeus vannamei that is down-regulated by white
spot syndrome virus. Fish and Shellfish Immunology, v. 25, p. 28-39,
2008.
ALEXOPOULOU L. et al. Recognition of double-stranded RNA and
activation of NF-kB by Toll-like receptor 3. Nature, v. 413(6857), p.
732-8, 2001.
ALIYARI, R.; DING, S-W. RNA-based viral immunity initiated by the
Dicer family of host immune receptors. Immunology Review, v.
227(1), p. 176–188, 2009.
ARMSTRONG, P.B. Proteases and protease inhibitors: a balance of
activities in hostpathogen interaction. Immunobiology, v. 211, p. 263281, 2006.
ARTS, J.A. et al. Molecular cloning and expression of a Toll receptor in
the giant tiger shrimp, Penaeus monodon. Fish and Shellfish
Immunology v. 23(3), p. 504–13, 2007.
ASPÁN, A.; HALL, M.; SÖDERHÄLL, K. The effect of endogenous
proteinase inhibitors on the prophenoloxidase activating enzyme, a
serine proteinase from crayfish haemocytes. Insect Biochemistry, v. 20,
p. 485-492, 1990.
ASSAVALAPSAKUL, W.; CHINNIRUNVONG, W.; PANYIM, S.
Application of YHV-protease dsRNA for protection and therapeutic
treatment against yellow head virus infection in Litopenaeus vannamei.
Diseases of Aquatic Organisms, v. 84, p. 167-171, 2009.
BACHÈRE, E. Shrimp immunity and disease control. Aquaculture
v.191, p. 3-11, 2000.
BACHÈRE, E. et al. Insights into the anti-microbial defense of marine
invertebrates: the penaeid shrimps and the oyster Crassostrea gigas.
Immunological Reviews, v. 198, p. 149-168, 2004.
BARBIERI, R.C.J; OSTRENSKY, A.N. Camarões marinhos:
Engorda. 1. ed. Viçosa: Aprenda Fácil, 2002, 352p.
87
BARRACCO, M.A; PERAZZOLO, L.M; ROSA, R.D. Inmunologia del
camarón. In: VIELKA, M.Q; CUÉLLAR-ANJEL, J. (Eds.), Guía
Práctica de Patología y Inmunología de Camarones Penaeidos,
CYTED, 2008, pp. 169-224.
BARTLETT, T.C. et al. Crustins, homologues of an 11.5-kDa
antibacterial peptide, from two species of penaeid shrimp, Litopenaeus
vannamei and Litopenaeus setiferus. Marine Biotechnology NY, v. 4,
p. 278-293, 2002.
BONAMI J-R. Shrimp Viruses. In: MAHY, B.W.J; VAN
REGENMORTEL, M.H.V. (Eds.), Encyclopedia of Virology, Oxford:
Elsevier, 2008, pp. 567-576.
BORGHETTI, N.R.B.; OSTRENSKY, A. BORGHETTI, J.R.
AQUICULTURA: uma visão geral sobre a produção de organismos
aquáticos no Brasil e no mundo. Curitiba: Grupo Integrado de
Aqüicultura e Estudos Ambientais, 2003. 12p.
BOYD, C.E. Water quality in Warnwater fish ponds. Opelika:
Craftmaster Printers Inc., 1979, 359p.
BUCHELI, P.; GARCIA, F. O vírus da síndrome da mancha branca.
Revista panorama da aqüicultura, v. 87, p. 43-49, jan/fev. 2005.
BUGLIONE NETO, C. C. Coeficiente de digestibilidade aparente da
proteina, energia e matéria seca em ração comercial suplementada
com probiótico para Litopenaeus vannamei. 2009. 41f. Dissertação
(Mestrado em Aquicultura) – Universidade Federal de Santa Catarina,
Florianópolis.
BULET, P.; STÖCKLIN, R.; MENIN, L. Anti-microbial peptides: from
invertebrates to vertebrates. Immunological Reviews, v. 198, p. 169184, 2004.
BURGE, E. J. et al. Lysozyme gene expression by hemocytes of Pacific
white shrimp, Litopenaeus vannamei, after injection with Vibrio. Fish
and Shellfish Immunology, v. 22, p. 327-339, 2007.
CAI, S.L. et al. Epidemiological studies on the explosive epidemic
disease of prawn in 1993-1994. Journal of Fish China, v. 19, p. 112–
117, 1995.
88
CAMARGO, S.G.O; POUEY, J.L.O. Aquicultura- um mercado em
expansão. Revista Brasileira de Agrociência. Pelotas, v. 11 (4), p. 393396, out/dez. 2005.
CHANG, P.S. et al. Identification of white spot syndrome virus
associated baculovirus (WSBV) target organs in the shrimp Penaeus
monodon by in situ hybridization. Diseases of Aquatic Organisms, v.
27, p. 131–139, 1996.
CHANG, P.S. et al. Detection of white spot syndrome associated
baculovirus in experimentally infected wild shrimp, crabs and lobsters
by in situ hybridization. Aquaculture, v. 164, n. 1-4, p. 233–242, 1998.
CHANG, C. F. et al. Effect of dietary beta-1,3-glucan on resistance to
white spot syndrome virus (WSSV) in postlarval and juvenile Penaeus
monodon. Diseases of Aquatic Organisms, v. 36, p.163–8, 1999.
CHANG, C-C. et al. Trichlorfon, an organophosphorus insecticide,
depresses the immune responses and resistence to Lactococcus garvieae
of the giant freshwater prawn Macrobrachium rosembergii. Fish and
Shellfish Immunology v. 20, p. 574-585, 2006.
CHEN, L.L. et al. Transcriptional analysis of the DNA polymerase gene
of shrimp white spot syndrome virus. Virology, v. 301, p. 136–147,
2002.
CHENG W. et al. Molecular cloning and characterization of a pattern
recognition molecule, lipopolysaccharide- and b-1,3-glucan binding
protein (LGBP) from the white shrimp Litopenaeus vannamei. Fish and
Shellfish Immunology, v. 18, p. 297–310, 2005.
CHOU, H.Y. et al. Pathogenicity of a baculovirus infection causing
White spot syndrome in cultured penaeid shrimp in Taiwan. Diseases of
Aquatic Organisms, v. 23, p. 165-173, 1995.
CHOWDHURY, I.; THARAKAN, B.; BHAT, G.K. Caspases–an
update. Comparative Biochemistry and Physiology (B) Biochem.
Mol. Biol., v. 151, p. 10-27, 2008.
de-la-VEGA, E. et al. Anti-lipopolysaccharide factor in Litopenaeus
vannamei (LvALF): A broad spectrum antimicrobial peptide essential
89
for shrimp immunity against bacterial and fungal infection. Molecular
Immunology, v. 45, p. 1916–1925, 2008.
de-LORGERIL, J. et al. A relationship between antimicrobial peptide
gene expression and capacity of a selected shrimp line to survive a
Vibrio infection. Molecular Immunology, v. 45, p. 3438–3445, 2008.
DECHKLAR, M.; UDOMKIT, A.; PANYIM, S. Characterization of
Argonaute cDNA from Penaeus monodon and implication of its role in
RNA interference. Biochemical and Biophysical Research
Communications, v. 367, p. 768–774, 2008.
DESTOUMIEUX, D. et al. Penaeidins: A new family of antimicrobial
peptides in the shrimp Penaeus vannamei (Decapoda). The Journal of
Biological Chemistry, v. 272, p. 28398-28406, 1997.
DESTOUMIEUX, D. et al. Penaeidins, antimicrobial peptides with
chiting-binding activity, are produced and stored in shrimp granulocytes
and released after microbial challenge. Journal of Cell Science, v. 113,
p. 461-469, 2000.
DHAR, A.K. et al. Use of cDNA microarray to isolate the differentially
expressed genes in white spot syndrome virus infected shrimp (Penaeus
stylirostris). Arch Virol, v. 148, p. 2381–96, 2003.
DIEGUEZ-URIBEONDO, J.; CERENIUS, J. The inhibition of
extracellular proteinases from Aphanomyces spp by three different
proteinase inhibitors from crayfish blood. Mycological Research, v.
102, p. 821-824, 1998.
DU, X.J.; ZHAO, X.F.; WANG, J.X. Molecular cloning and
characterization of a lipopolysaccharide and β-1,3-glucan binding
protein from fleshy prawn (Fenneropenaeus chinensis). Molecular
Immunology, v. 44, p. 1085-1094, 2007.
DURAND, S. et al. Application of gene probes as diagnostic tools for
white spot baculovirus (WSBV) of penaeid shrimp. Diseases of Aquatic
Organisms, v. 27, p. 59-66, 1996.
DURAND, S. et al. Ultrastructure and morphogenesis of white spot
syndrome baculovirus. Diseases of Aquatic Organisms, v. 29, p. 205–
211, 1997.
90
DURAND, S. V.; LIGHTNER, D. V. Quantitative real time PCR for the
measurement of white spot syndrome virus in shrimp. Journal of Fish
Diseases, v. 25, p. 381–389, 2002.
EPAGRI/CEPA. Síntese Anual da Agricultura de Santa Catarina, 20082009. Disponível em: <http://www.cepa.epagri.sc.org.br>. Acesso em
15/03/2010.
ESCOBEDO-BONILLA, C.M. et al. A review on the morphology,
molecular characterization, morphogenesis and pathogenesis of white
spot syndrome virus. Journal of Fish Diseases, v. 31, p. 1–18, 2008.
FAO-Fisheries and Aquaculture Department, 2007. Aquaculture
Newsletter, 36p. Disponível em <http://www.fao.org.br>. Acesso em
05/03/2010.
FIGUEIREDO, M.C.B. et al. Avaliação da demanda hídrica da
carcinicultura em águas interiores. In: Simpósio Brasileiro de
Aqüicultura, v. 13, p. 64, 2004.
FIRE, A. et al. Potent and specific genetic interference by doublestranded RNA in Caenorhabditis elegans. Nature, v. 391, p. 806-811,
1998.
FLEGEL, T.W. Special topic review: major viral diseases of the black
tiger prawn (Penaeus monodon) in Thailand. World Journal of
Microbiology & Biotechnology, v. 13, p. 433–442, 1997.
FLEGEL, T.W. Detection of major penaeid shrimp viruses in Asia, a
historical perspective with emphasis on Thailand. Aquaculture, v. 258,
p. 1-33, 2006.
GANTIER, M.P.; WILLIAMS, B.R. The response of mammalian cells
to double stranded RNA. Cytokine Growth Factor Rev., v.18, p. 363371, 2007.
GARGIONI, R; BARRACCO, M.A. Hemocytes of the palaemonids
Macrobrachium rosenbergii and M. acanthurus, and of the penaeid
Penaeus paulensis. Journal of Morphology, v. 236, p. 209-221, 1998.
GELEY, S.; MULLER, C. RNAi: ancient mechanism with a promising
future. Exp. Gerontol., v. 39, p. 985–998, 2004.
91
GILADI, H. et al. Small interfering RNA inhibits hepatitis B virus
replication in mice. Mol. Ther., v. 8, p. 769-776, 2003.
GITLIN, L.; KARELSKY, S.; ANDINO, R. Short interfering RNA
confers intracellular antiviral immunity in human cells. Nature, v. 418,
p. 430-434, 2002.
GREENBERG, C.C.; BRICKBICHLER, P.J.; RICE, Y.H.
Transglutaminases: multifunctional cross-linking enzymes that
stabilized tissues. The FASEB Journal, v. 5, p. 3071- 3077, 1991.
GRIVICICH, I.; REGNER, A.; ROCHA, A. B. Apoptosis: Programmed
Cell Death. Revista Brasileira de Cancerologia, v. 53(3), p. 335-343,
2007.
GROSS, P.S. et al. Immune gene discovery by expressed sequence tag
analysis of hemocytes and hepatopancreas in the Pacific White Shrimp,
Litopenaeus vannamei, and the Atlantic White Shrimp, L. setiferus.
Developmental and Comparative Immunology, v. 25, p. 565-577,
2001.
HAMMOND, S.M. et al. Argonaute 2, a link between genetic and
biochemical analyses of RNAi. Science, v. 293, p. 1146–1150, 2001.
HANNON, G.J. RNA interference. Nature, v. 418, p. 244-251, 2002.
HERGENHAHN, H.G.; HALL, M.; SÖDERHÄLL, K. Purification and
characterization of an alpha 2-macroglobulin-like proteinase inhibitor
from plasma of the crayfish Pacifastacus leniusculus. The Biochemical
Journal, v. 255, p. 801-806, 1988.
HOLMBLAD, T. SODERHALL, K. Cell adhesion molecules and
antioxidative enzymes in a crustacean, possible role in immunity.
Aquaculture, v. 172, p. 111-123, 1999.
HOSE, J.E; MARTIN, G.G; GERARD, A.S. A decapod hemocyte
classification scheme integrating morphology, cytochemistry, and
function. Biological Bulletin, v. 178, p. 33-45, 1990.
HUNTER, A. M.; LACASSE, E. C.; KORNELUK, R. G. The inhibitors
of apoptosis (IAPs) as cancer targets. Apoptosis, v. 12, p.1543–1568,
2007.
92
HUTVAGNER, G.; ZAMORE, P.D. A microRNA in a multipleturnover
RNAi enzyme complex. Science, v. 297, p. 2056-60, 2002.
IDEMA-Instituto de Desenvolvimento Econômico e Meio Ambiente do
Rio Grande do Norte. Desenvolvimento Sustentável – RN Melhor
para Carcinicultura. Ano I (1), 1-6, fev. 2006.
JACOBS, B.L.; LANGLAND, J.O. When two strands are better than
one: the mediators and modulators of the cellular responses to doublestranded RNA. Virology v. 219, p. 339-349, 1996.
JIMÉNEZ-VEGA, F.; VARGAS-ALBORES, F. A four-Kazal domain
protein in Litopenaeus vannamei hemocytes. Developmental and
Comparative Immunology, v. 29, p. 385- 391, 2005.
JIRAVANICHPAISAL, P; LEE, B.L; SÖDERHÄLL, K. Cell-mediated
immunity in arthropods: hematopoiesis, coagulation, melanization and
opsonization. Immunobiology, v. 211, p. 213-236, 2006.
JIRAVANICHPAISAL, P. et al. Antibacterial peptides in hemocytes
and hematopoietic tissue from freshwater crayfish Pacifastacus
leniusculus: characterization and expression pattern. Developmental
and Comparative Immunology, v. 31, p. 441-455, 2007.
JOHANSSON, M.W.; KEYSER, P.; SÖDERHÄLL, K. Purification and
cDNA cloning of a four-domains Kazal proteinase inhibitor from
crayfish blood cells. European Journal of Biochemistry, v. 223, p.
389-394, 1994.
JOHANSSON, M.W. et al. Crustacean haemocytes and haematopoiesis.
Aquaculture, v. 191, n.1-3, p. 45-52, 2000.
JOHNSON, K.N.; van HULTEN, M.C.; BARNES, A.C. "Vaccination"
of shrimp against viral pathogens: phenomenology and underlying
mechanisms. Vaccine, v. 8, p. 4885-4892, 2008.
KIM, C.S. et al. Protection of shrimp (Penaeus chinensis) against white
spot syndrome virus (WSSV) challenge by double stranded RNA. Fish
and Shellfish Immunology, v. 23, p. 242-246, 2007.
KIM, V. N.; HAN, J.; SIOMI, M. C. Biogenesis of small RNAs in
animals. Molecular cell Biology, v.10, p. 126-140, 2009.
93
KOU, G.T. et al, Tissue distribution of white spot syndrome virus
(WSSV) in shrimp and crabs. In: FLEGEL, T.W.l (Ed). Advances in
Shrimp Biotechnology National Center for Genetic Engineering and
Biotechnology, Bangkok: Publicação, 1998, p. 267–271.
KUMAR, S.R. et al. Immunological responses of Penaeus monodon to
DNA vaccine and its efficacy to protect shrimp against white spot
syndrome virus (WSSV). Fish and Shellfish Immunology, v. 24, p.
467-478, 2008.
KURTZ, J.; FRANZ, K. Innate defense: evidence for memory in
invertebrate immunity. Nature, v. 425, p. 37-38, 2003.
KURTZ, J.; ARMITAGE, S.A. Alternative adaptative immunity in
invertebrates. Trends Immunology, v. 27, p. 493-496, 2006.
LABREUCHE, Y.et al. Lack of evidence for Litopenaeus vannamei
Toll receptor (lToll) involvement in activation of sequence independent
antiviral immunity in shrimp. Developmental and Comparative
Immunology, v. 33, p. 806-810, 2009.
LE MOULLAC, G.; HAFFNER, P. Environmental factors affecting
immune responses in Crustacea. Aquaculture, v.191, p. 121-131, 2000.
LEE, S.Y; SÖDERHÄLL, K. Early events in crustacean innate
immunity. Fish and Shellfish Immunology, v. 12, n. 5, p. 421-437,
2002.
LEU, J. H. et al. Molecular cloning and characterization of an inhibitor
of apoptosis protein (IAP) from the tiger shrimp, Penaeus monodon.
Developmental and Comparative Immunology, v. 32, p. 121–133,
2008.
LIANG, Z.; SÖDERHÄLL, K. Isolation of cDNA encoding a novel
serpin of crayfish hemocytes. Comparative Biochemistry and
Physiology, v. 2, p. 385-391, 1995.
LIANG, Z.; SOTTRUP-JENSEN, L.; SÖDERHÄLL, K. Pacifastin, a
novel 155-kDa heterodimeric proteinase inhibitor containing a unique
transferrin chain. Proceedings of the National Academy of Sciences
USA, v. 94, p. 6682-6687, 1997.
94
LIGHTNER, D.V. The Penaeid Shrimp Viral Pandemics due to
IHHNV, WSSV, TSV and YHV. History in the Americas and
Current Status. In: Y. Sakai, J.P. McVey, D. Jang, E. McVey and M.
Caesar (ed), Proceedings of the US-Japan Cooperative Program in
Natural Resources (UJNR) Aquaculture Panel Meeting, 2003. p. 6-24.
LIN, Y.C. et al. Molecular cloning and characterisation of a proteinase
inhibitor, alpha 2-macroglobulin (alpha2-M) from the haemocytes of
tiger shrimp Penaeus monodon. Molecular Immunology, v. 44, p.
1065-1074, 2007a.
LIN, Y.C. et al. Molecular cloning and phylogenetic analysis on α2macroglobulin (α2-M) of white shrimp Litopenaeus vannamei.
Developmental and Comparative Immunology, v. 44, p. 1065-1074,
2007b.
LISTON, P.; FONG, W. G.; KORNELUK, R. G. The inhibitors of
apoptosis: there is more to life than Bcl2. Oncogen, v. 22, p. 8568–
8580, 2003.
LIU, F.; LIU, Y.; LI, F.; DONG, B.; XIANG, J. Molecular cloning and
expression profile of putative antilipopolysaccharide factor in Chinese
shrimp (Fenneropenaeus chinensis). Marine Biotechnology NY, v. 7,
p. 600-608, 2005.
LIU, H. et al. Antilipopolysaccharide factor interferes with white spot
syndrome virus replication in vitro and in vivo in the crayfish
Pacifastacus leniusculus. Journal of Virology, v. 80, p. 10365–10371,
2006.
LIU, W.J. et al. White spot syndrome virus annexes a shrimp STAT to
enhance expression of the immediateearly gene ie1. Journal of
Virology, v. 81, p. 1461-1471, 2007.
LIU, H.; SÖDERHÄLL, K.; JIRAVANICHPAISAL, P. Antiviral
immunity in crustaceans. Fish and Shellfish Immunology, v. 27, p. 7988, 2009.
LO, C.F. et al. Detection and tissue tropism of White spot Baculovirus
(WSBV) in captured brooders of Penaeus monodon with special
emphasis on reproductive organs. Diseases of Aquatic Organisms, v.
30, p. 53-72, 1997.
95
LORAND, L. et al. Dansylcadaverine specific staining for
transamidating enzymes. Analytical Biochemistry, v. 93, p. 453- 458,
1979.
LORAND, L.; CONRAD, S.M. Transglutaminases. Molecular and
Cellular Biochemistry, v. 58, p. 9-35, 1984.
MARIN, L.J. et al. Desenvolvimento de uma metodologia de PCR
semiquantitativa utilizando plasmídeo clonado com parte do gene Gb de
citomegalovírus. Medicina, v. 35, p. 85-94, 2002.
MARTINEZ, J. et al. Single-stranded antisense siRNAs guide target
RNA cleavage in RNAi. Cell, v. 110, p. 563-74, 2002.
MARTINS, F.A. Genotipagem de endosperma como estratégia
auxiliar no mapeamento e detecção de QTLs em populações
exogâmicas. 2006. 73p. Dissertação (Mestrado em Genética e
Melhoramento) Universidade Federal de Viçosa.
MARTORELLI, S.R; OVERSTREET, R.M; JOVONOVICH, J. A. First
report of viral pathogens WSSV and IHHNV in Argentine crustaceans.
Bulletin of Marine Science, v. 86, p. 117-131, 2010.
MEKATA, T. et al. Identification of cDNA encoding Toll receptor,
MjToll gene from kuruma shrimp, Marsupenaeus japonicus. Fish and
Shellfish Immunology, v. 24(1), p. 122–33, 2008.
MILLAR, D.A.; RATCLIFFE, N.A. Invertebrates. In: TURNER R. J.
(Ed.) Immunology: a comparative approach. Chichester, 1994, p. 4651.
MOURELATOS, Z. et al. miRNPs: a novel class of ribonucleoproteins
containing numerous microRNAs. Genes and Development, v. 16, p.
720-8, 2002.
NAGOSHI, H. et al. Cloning and characterization of a LPS-regulatory
gene having an LPS binding domain in kuruma prawn Marsupenaeus
japonicus. Molecular Immunology, v. 43, p. 2061-2069, 2006.
NAPOLI, C.; LEMIEUX, C.; JORGENSEN, R. Introduction of a
chimeric chalcone synthase gene into petunia results in reversible
96
cosuppression of homologous genes in trans. Plant Cell, v. 2, p. 279-89,
1990.
NOVINA, C.D. et al. siRNA-directed inhibition of HIV-1 infection.
Nat. Med., v. 8, p. 681-686, 2002.
NUNES, A. J. P. O cultivo do camarão Litopenaeus vannamei em águas
oligohalinas. Panorama da Aqüicultura, Rio de Janeiro, v. 11, p. 3751, 2001.
NUNES, A. J. P.; MARTINS, C. C.; GESTEIRA, T. C. V.
Carcinicultura Ameaçada: produtores sofrem com as mortalidades
decorrentes do Vírus da Mionecrose Infecciosa (IMNV). Panorama da
Aqüicultura, Rio de Janeiro, v. 14, p. 37-51, 2004.
OIE World organisation for animal health. Manual of Diagnostic Tests
for
Aquatic
Animals
2006.
Disponível
em
<http://www.oie.int/eng/en_index.htm>. Acesso em 15/02/2010.
OKUMURA, T. Effects of lipopolysaccharide on gene expression of
antimicrobial peptides (penaeidins and crustin), serine proteinase and
prophenoloxidase in haemocytes of the Pacific white shrimp,
Litopenaeus vannamei. Fish and Shellfish Immunology, v. 22, p. 6876, 2007.
OSTRENSKY, A. Estudos para viabilização dos cultivos comerciais
de camarões marinhos no litoral do Estado do Paraná, Brasil. 1997.
235p. Tese (Doutorado em Ciências Biológicas), Universidade Federal
do Paraná, Curitiba.
PARKER, J.S.; BARFORD, D. Argonaute: a scaffold for the function of
short regulatory RNAs. Trends in Biochemical Sciences, v.31, p.11,
2006.
PERAZZOLO, L.M; ROSA, R.D.; BARRACCO, M.A. Defesas
antivirais em crustáceos: Estado da Arte. In: SILVA-SOUZA, A.T;
LIZAMA, M.A.P; TAKEMOTO, R.M. Patologia e Sanidade de
Organismos Aquáticos, Maringá-PR: ABRAPOA 2010 (no prelo).
PEREIRA, A.M.L; LEGAT, A.P.; LEGAT, J.F.A. As enfermidades
ainda ameaçam: o sucesso da carcinicultura pressupõe boas práticas de
manejo. Panorama da Aquicultura, v. 95, p. 46-47. Maio/junho 2006
97
PÉREZ-FARFANTE, J.; KENSLE, B. Penaeoid and Sergestoid shrimps
and prawns on the world. In: Key of diagnoses for the families and
genera. Éditions du Museum National d’Histoire Naturelle, Paris, 1997
p 233.
PHONGDARA, A.; WANNA, W.; CHOTIGEAT, W. Molecular
cloning and expression of caspase from white shrimp Penaeus
merguiensis. Aquaculture, v. 252, p. 114-120, 2006.
RANDALL, G.; GRAKOUI, A.; RICE, C.M. Clearance of replicating
hepatitis C virus replicon RNAs in cell culture by small interfering
RNAs. Proc. Natl. Acad. Sci. USA, v. 100, p. 235-240, 2003.
RAJEDRAN, K.V. et al. Experimental host range and histopathology of
white spot syndrome virus (WSSV) infection in shrimp, prawns,
crayfish and lobsters from India. Journal of Fish Diseases, v. 22, p.
183–191, 1999.
RATTANACHAI, A. et al. Molecular cloning and expression analysis
of alpha 2-macroglobulin in the kuruma shrimp, Marsupenaeus
japonicus. Fish and Shellfish Immunology, v. 16, p. 599-611, 2004.
REDDY, K.V.; YEDERY, R.D.; ARANHA, C. Antimicrobial peptides:
premises and promises. International Journal of Antimicrobial
Agents, v. 24, p. 536-547, 2004.
RIJIRAVANICH, A.; BROWDY, C. L ; WITHYACHUMNARNKUL,
B. Knocking down caspase-3 by RNAi reduces mortality in Pacific
white shrimp Penaeus (Litopenaeus) vannamei challenged with a low
dose of white-spot syndrome virus. Fish and Shellfish Immunology, v.
24, p. 308-313, 2008.
ROBALINO, J. et al. Induction of antiviral immunity by doublestranded RNA in a marine invertebrate. Journal of Virology, v. 78, p.
10442-10448, 2004.
ROBALINO, J. et al. Double-stranded RNA induces sequence-specific
antiviral silencing in addition to nonspecific immunity in a marine
shrimp: convergence of RNA interference and innate immunity in the
invertebrate antiviral response? Journal of Virology, v. 79, p. 1356113571, 2005.
98
ROBALINO, J. et al. Double-stranded RNA and antiviral immunity in
marine shrimp: inducible host mechanisms and evidence for the
evolution of viral counter-responses. Developmental and Comparative
Immunology, v. 31, p. 539-547, 2007.
ROCHA, I. P. Carcinicultura Brasileira: Desenvolvimento Tecnológico,
Sustentabilidade Ambiental e Compromisso Social; Associação
Brasileira dos Criadores de Camarão; setembro de 2007; disponível
em:
<http://www.abccam.com.br/download/CarciniculturaBrasileira.pdf>;
acesso em 9 de fevereiro de 2009.
ROSA, R.D.; BARRACCO, M.A. Shrimp interferon is rather a portion
of the mitochondrial F0-ATP synthase than a true α-interferon.
Molecular Immunology, v. 45, p. 3490-3493, 2008.
ROSA, R.D.; STOCO, P.H.; BARRACCO, M.A. Cloning and
characterisation of cDNA sequences encoding for antilipopolysaccharide factors (ALFs) in Brazilian palaemonid and penaeid
shrimps. Fish and Shellfish Immunology, v. 25, p. 693-696, 2008.
ROSEMBERRY, B. Aquaculture accounts for 28% of world shrimp
production. Aquaculture Magazine, v. 18 (1), p. 90-91, 1992.
ROSEMBERRY, B. World shrimp farming,
Aquaculture Digest, San Diego, California. 2002.
Anual
Report.
ROULSTON, A.; MARCELLUS, R.C.; BRANTON, P.E. Viruses and
apoptosis. Annu. Rev. Microbiol., v. 53, p. 577-628, 1999.
ROUX, M.M. et al. The lipopolysaccharide and β-1,3-glucan binding
protein gene is upregulated in white spot virus-infected shrimp (Penaeus
stylirostris). Journal of Virology, v. 76, p. 7140-7149, 2002.
RUPNARAIN, C. et al. Colon cancer: genetics and apoptotic events.
Biol Chem, v. 385, p. 449-64, 2004.
SAITO, T.; GALE Jr, M. Differential recognition of double-stranded
RNA by RIG-I – like receptors in antiviral immunity. J. Exp. Med., v.
205(7), p. 1523-1527, 2008.
99
SEIFFERT, W.Q.; WINCLER, S.; MAGGIONI, D. A mancha branca
em Santa Catarina. Panorama da Aquicultura, Rio de Janeiro, v. 87, p.
51-53 jan/fev, 2005.
SEIFFERT, W.Q. et al. Enfermidades: uma oportunidade para repensar
o cultivo de camarões marinhos. Panorama da Aquicultura, Rio de
Janeiro, v. 16, p. 32-38 set/out, 2006.
SHEKHAR, M.; LU, Y. Application of nucleic-acid-based therapeutics
for viral infections in shrimp aquaculture. Marine Biotechnology, v. 11,
p. 1-9, 2009.
SÖDERHÄLL, K., SMITH, V.J. Separation of the hemocyte population
of Carcinus maenas and the other marine decapods and
prophenoloxidase distribution. Developmental and Comparative
Immunology, v. 7, p. 229-239, 1983.
SOMBOONWIWAT, K. et al. Recombinant expression and antimicrobial activity of anti-lipopolysaccharide factor (ALF) from the
black tiger shrimp Penaeus monodon. Developmental and
Comparative Immunology, v. 29, p. 841-851, 2005.
SRITUNYALUCKSANA, K. et al. PmRab7 is a VP28-binding protein
involved in white spot syndrome virus infection in shrimp. Journal of
Virology, v.80, p. 10734-10742, 2006.
SU, J. et al. A key gene of the RNA interference pathway in the black
tiger shrimp, Penaeus monodon: identification and functional
characterization of Dicer-1. Fish and Shellfish Immunology, v. 24 (2),
p. 223–233, 2008.
SUPUNGUL, P. et al. Identification of immune-related genes in
hemocytes of black tiger shrimp (Penaeus monodon). Marine
Biotechnology, v. 4, p. 487-494, 2002.
SYED-MUSTHAQ, S.S. et al. Localization of VP28 on the baculovirus
envelope and its immunogenicity against white spot syndrome virus in
Penaeus monodon. Virology, v. 391, p. 315-324, 2009.
TAVARES, E.C.B; SANTOS, M.A.S. Estudo exploratório da cadeia
produtiva da carcinicultura no estado do Pará: o caso do Litopenaeus
vannamei. Amazônia Ciência e Desenvolvimento, v. 1 (2), p. 85-96,
2006.
100
THARNTADA, S. et al. Role of anti-lipopolysaccharide factor from the
black tiger shrimp, Penaeus monodon, in protection from white spot
syndrome virus infection. Journal of General Virology, v. 90, p. 14911498, 2009.
TIRASOPHON, W.; ROSHORM, Y.; PANYIM, S. Silencing of yellow
head virus replication in penaeid shrimp cells by dsRNA. Biochem.
Biophys. Res. Commun., v. 334, p. 102-107, 2005.
TIRASOPHON, W. et al. Therapeutic inhibition of yellow head virus
multiplication in infected shrimps by YHV-protease dsRNA. Antiviral
Research, v. 74, p. 150-155, 2007.
TONGANUNT, M. et al. Identification and characterization of syntenin
binding protein in the black tiger shrimp Penaeus monodon. Journal of
Biotechnology, v. 120, p. 135-145, 2005.
UNAJAK, S.; V. BOONSAENG, S. JITRAPAKDE, E. Isolation and
characterization of cDNA encoding Argonaute, a component of RNA
silencing in shrimp (Penaeus monodon). Comparative Biochemistry
and Physiology, Part B v. 145, p. 179–187, 2006.
Van de BRAAK, C.B. et al. The roles of haemocytes and the lymphoid
organ in the clearance of injected Vibrio bacteria in Penaeus monodon
shrimp. Fish and Shellfish Immunology, v. 13 (4), p. 293-309, 2002.
VAN DER KROL, A.R. et al. Flavonoid genes in petunia: Addition of a
limited number of gene copies may lead to a suppression of gene
expression. Plant Cell, v. 2, p. 291-9, 1990.
Van HULTEN, M.C.W.; GOLDBACH, R.W.; VLAK, J.M. Three
functionally diverged major structural proteins of white spot syndrome
virus evolved by gene duplication. J. Gen. Virol., v. 8, p. 2525-2529,
2000.
Van HULTEN, M.C.W. et al. The white spot syndrome virus DNA
genome sequence. Virology, v. 286, p. 7–22, 2001.
VASEEHARAN, B. et al. Molecular cloning and characterisation of a
thioester-containing alpha2-macroglobulin (alpha2-M) from the
haemocytes of mud crab Scylla serrata. Fish and Shellfish
Immunology, v. 22, p. 115-130, 2007.
101
VERDEL, A. et al. RNAi-mediated targeting of heterochromatin by the
RITS complex. Science, v. 303, p.672-676, 2004.
WADHWA, R. et al. Know-how of RNA interference and its
applications in research and therapy. Mutation Research, v. 567, p. 71–
84, 2004.
WANG, Y.G. et al. Histopathology and cytopathology of white spot
syndrome virus (WSSV) in cultured Penaeus monodon from peninsular
Malaysia with emphasis on pathogenesis and the mechanism of white
spot formation. Diseases of Aquatic Organisms, v. 39, p. 1–11, 1999.
WANG, Y.T. et al. White spot syndrome virus (WSSV) infects specific
hemocytes of the shrimp Penaeus merguiensis. Diseases of Aquatic
Organisms, v. 52, p. 249–59, 2002.
WANG, Z. et al. ORF390 of white spot syndrome virus genome is
identified as a novel anti-apoptosis gene. Biochem. Biophys. Res.
Commun., v. 325, p. 899-907, 2004.
WANG, Y.-C.; CHANG, P.-S.; CHEN, H.-Y. Tissue expressions of
nine genes important to immune defence of the Pacific white shrimp
Litopenaeus vannamei. Fish and Shellfish Immunology, v. 23, p.
1161-1177, 2007.
WANG, L. et al. Requirement for shrimp caspase in apoptosis against
virus infection. Developmental and Comparative Immunology, v.32,
p. 706-71, 2008.
WANG, K.C H-C. et al. RNAi knock-down of the Litopenaeus
vannamei Toll gene (LvToll) significantly increases mortality and
reduces bacterial clearance after challenge with Vibrio harveyi.
Developmental and Comparative Immunology, v. 34, p. 49–58, 2010.
WASIELESKY, W. J. Cultivo de juvenis do camarão-rosa
Farfantepenaeus paulensis (Decapoda Penaeidae) no estuário da
Lagoa dos Patos: Efeitos de parâmetros ambientais e manejo de
cultivo. 2000, 199 f. Tese (Doutorado em Aquicultura) - Fundação
Universidade Federal do Rio Grande, Rio Grande.
102
WESTENBERG, M. et al. siRNA injection induces sequenceindependent protection in Penaeus monodon against white spot
syndrome virus. Virus Research, v. 114, p. 133-139, 2005.
WITTEVELDT, J.; VLAK, J.M.; van HULTEN, M.C. Protection of
Penaeus monodon against white spot syndrome virus using a WSSV
subunit vaccine. Fish and Shellfish Immunology, v. 16, p. 571-579,
2004.
WONGPRASERT, K.; SANGSURIYA, P.; PHONGDARA, A. Cloning
and characterization of a caspase gene from black tiger shrimp (Penaeus
monodon) infected with white spot syndrome virus (WSSV). J.
Biotechnol., v. 131, p. 9-19, 2007.
WU, W.; WANG, L.; ZHANG, X. Identification of white spot
syndrome virus (WSSV) envelope proteins involved in shrimp infection.
Virology, v. 332, p. 578-583, 2005.
WU, Y. et al. Inhibition of white spot syndrome virus in Litopenaeus
vannamei shrimp by sequence-specific siRNA. Aquaculture, v. 271, p.
21-30, 2007.
XU, J.; HAN, F.; ZHANG, X. Silencing shrimp white spot syndrome
virus (WSSV) genes by siRNA. Antiviral Research, v. 73, p. 126-131,
2007.
XU, H. et al. The interaction of white spot syndrome virus envelope
protein VP28 with shrimp Hsc70 is specific and ATPdependent. Fish
and Shellfish Immunology, v. 26, p. 414-421, 2009.
YANG, F. et al. Complete genome sequence of the shrimp white spot
bacilliform virus. Journal of Virology, v. 75, p. 11811–11820, 2001.
YANG, L.S. et al. A Toll receptor in shrimp. Molecular Immunology,
v. 44, p. 1999-2008, 2007.
YANG, C. et al. A Toll receptor from Chinese shrimp Fenneropenaeus
chinensis is responsive to Vibrio anguillarum infection. Fish and
Shellfish Immunology v. 24, p. 564–74, 2008.
YEH, S-P. et al. Immune response of white shrimp, Litopenaeus
vannamei, after a concurrent infection with white spot syndrome virus
103
and infectious hypodermal and hematopoietic necrosis virus. Fish and
Shellfish Immunology, v. 26, p. 582–588, 2009.
YI, G. et al. VP28 of shrimp White Spot Syndrome Virus is involved in
the attachment and penetration into shrimp cells. J. Biochem. Mol.
Biol., v. 37, p. 726-734, 2004.
YODMUANG, S. et al. YHV protease dsRNA inhibits YHV replication
in Penaeus monodon and prevents mortality. Biochem. Biophys. Res.
Commun., v. 341, p. 351-356, 2006.
ZHAO, Z.Y. et al. Profiling of differentially expressed genes in
hepatopancreas of white spot syndrome virus-resistant shrimp
(Litopenaeus vannamei) by suppression subtractive hybridization. Fish
and Shellfish Immunology, v. 22, p. 520-534, 2007.
ZERBINI, F. M.; ALFENAS, P. F.; ANDRADE, E. C. O silenciamento
de RNA como um mecanismo de defesa de plantas a vírus. RAPP, v.
13, p. 191-244, 2005.