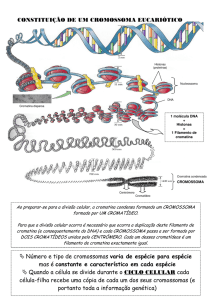
Programa de Pós-Graduação em Genética e
Melhoramento de Plantas
LGN 5799 - SEMINÁRIOS EM
GENÉTICA E MELHORAMENTO DE PLANTAS
Mecanismos de Formação de
Heterocromatina
Mestranda Larissa M. de Andrade
Orientador Prof. Dr. Mateus Mondin
Departamento de Genética
Avenida Pádua Dias, 11 - Caixa Postal 83, CEP: 13400-970 - Piracicaba - São Paulo - Brasil
Telefone: (0xx19) 3429-4250 / 4125 / 4126 - Fax: (0xx19) 3433-6706 - http://www.genetica.esalq.usp.br/semina.php
O que é Heterocromatina
Emil Heitz (1928) descreveu a heterocromatina
como o material nuclear que se mantém
condensado em todo o ciclo celular, ao contrário
do resto do cromossomo, que apresenta um ciclo
de condensação-descondensação
(Grewal e Jia, 2007)
Introdução
• Classificação da heterocromatina:
Heterocromatina facultativa: ocorre em apenas um dos
cromossomos do par, e é ativo em apenas um curto período
de tempo do ciclo de vida; mantendo-se epigeneticamente
reprimidos e heterocromáticos no restante da vida do
indivíduo
Heterocromatina constitutiva: ocorre no par de cromossomos
homólogos, e não é transcrito devido à sua composição do
DNA
(Sumner, 2003)
Introdução
Humanos
• Localização:
Centromérica:
Praticamente
todos os cromossomos têm
blocos de heterocromatina na
região centromérica e estes
blocos podem variar em
tamanho entre grande e muito
pequeno
Exemplo: Arabidopsis
(Sumner, 2003)
Introdução
Intersticial e Terminal: são menos comuns; comuns em plantas
com cromossomos grandes e insetos
Milho
Aguiar-Perecin, M. L. R. et al., 2000
Introdução
Características da Heterocromatina:
•
•
•
•
•
Aparência na intérfase
Densidade de gene
Replicação
Recombinação meiótica
Acessibilidade a nuclease
Altamente condensada
Baixa
Final da fase S
Baixa frequência
Baixa
(Sumner, 2003)
Introdução
Características da Heterocromatina:
é composta por seqüências de DNA repetitivo que tem pouca ou
nenhuma atividade
silenciadas por modificações covalentes no DNA ou nas histonas;
Auxilia na recombinação entre elementos repetitivos;
Auxilia no controle da segregação dos centrômeros;
Promove interações entre regiões de cromatina;
Introdução
• Formação e manutenção integram 2 tipos de informações:
localização cromossômica e nuclear
presença e densidade de elementos de DNA repetitivo
seqüências de DNA satélite e elementos transponíveis
encontrados em centrômeros, telômeros e ‘knobs'
são os principais alvos de formação da heterocromatina
(Grewal e Jia, 2007)
Mecanismos de Formação da Heterocromatina
Metilação do
DNA
Modificações
nas Histonas
Interferência
por RNA
Metilação do DNA
Metilação do DNA
• A metilação é uma modificação comum no DNA dos
eucariotos
Carbono 5 da citosina (5- methylcytosine,
5mC)
http://www.scq.ubc.ca/human-cloning-science-fiction-or-reality/
Metilação do DNA
• Em Plantas ocorre predominantemente nos sítios GC, mas
também em sítios CHG e CHH (onde H é A, T, or C)
(Teixeira et al., 2009)
• O nível de 5-MC é variável em Plantas:
6% das citosinas em Arabidopsis (Kakutani et al. 1999)
25% das citosinas em Milho
(Papa et al. 2001)
Metilação do DNA
• Está ausente ou quase ausente na levedura, moscas e
nematóides
• A interação entre a metilação do DNA e a metilação de
histonas é bem estabelecida em animais e plantas
(Lippman & Martienssen, 2004)
• Em Plantas, os transposons são metilados, os quais resultam
na regulação dos genes vizinhos
(Lippman e Martienssen, 2004;
Vaughn et.al, 2007)
• Os blocos de heterocromatina estão metilados e hipometilados
O repeat de 178 pb associado a CEN na cromatina são
hipometilados em
comparação com o mesmo repeat
localizado na heterocromatina pericentromérica em milho e
Arabidopsis
Epigenetic Modification of Centromeric Chromatin: Hypomethylation of DNA Sequences in the
CENH3-Associated Chromatin in Arabidopsis thaliana and Maize
5-MC em Arabidopsis
•
•
•
•
Marca cromocentros
Sinais com centros ocos em alguns núcleos
Os centros ocos são ocupados por cromatina associada a CENH3
As seqüências de DNA na cromatina CENH3 são hipometiladas
comparada as seqüências de DNA da cromatina pericentromérica
Epigenetic Modification of Centromeric Chromatin: Hypomethylation of DNA Sequences in the
CENH3-Associated Chromatin in Arabidopsis thaliana and Maize
5-MC em Milho
• Redução significativa do sinal de metilação foi observada nos
domínios ocupado por CENH3
• As seqüências de DNA na cromatina CENH3 são hipometiladas
comparada as seqüências de DNA da cromatina pericentromérica
Epigenetic Modification of Centromeric Chromatin: Hypomethylation of DNA Sequences in the
CENH3-Associated Chromatin in Arabidopsis thaliana and Maize
• Conclusões:
• Em Arabidopsis e no Milho os repeats associados
com a cromatina CENH3 são hipometiladas em
comparação com o mesmo repeat localizado nas
regiões pericentromérica
• Os resultados sugerem que a metilação do DNA
desempenham
um
papel
importante
na
demarcação epigenética da cromatina CENH3
Modificações nas Histonas
Modificações nas Histonas
• As modificações nas histonas ocorrem, normalmente, nas
caudas N-terminais
• Muitas destas alterações são específicas para:
o Heterocromatina: metilação de histonas H3 na lisina 9
o Eucromatina: metilação de histonas H3 na lisina 4
(Lippman e Martienssen, 2004)
Modificações nas Histonas
•
As modificações estão implicadas na regulação dos genes,
formação e manutenção das heterocromatinas
http://chemistry.gsu.edu/faculty/Zheng/pictures/nucleosome.jpg
Modificações nas Histonas
• Histonas são modificadas em muitos sítios: Há mais de 60
resíduos diferentes de modificações em histonas que foram
detectados por meio de anticorpos específicos
(Kouzarides, 2007)
(Fuchs et al., 2008)
Modificações nas Histonas
• Há 3 tipos de metilação:
(Sumner, 2003)
Modificações nas Histonas
• A heterocromatina é, normalmente, transcricionalmente inativa
• hipoacetilada nas histonas H3 e H4;
• hipometilada na lisina 4 da H3;
• altamente metilada na lisina 9 da
histona H3
em levedura, Drosophila, mamíferos e Arabidopsis
(Jasencakova et. al, 2003)
Modificações nas Histonas
• Proteínas que auxiliam na formação da heterocromatina
Essas proteínas não
foram descritas em
Plantas, mas estudos
mostram que
existem proteínas
análogas
(Sumner, 2003)
Modificações nas Histonas
• Essas proteínas (Swi6p, HP1) quando ligada a H3K9 me2, serve
como uma plataforma de montagem de fatores modificadores da
cromatina que são envolvidos na estabilização (manutenção) e
disseminação de heterocromatina
(Grewal e Jia, 2007)
Modificações nas Histonas
Bandeiras vermelhas indicam H3 metilada na lisina 9
HMT - Histona metiltransferase
HDAC – Histona deacetilase
• Esta estratégia geral da metilação da histona, funcionando
como uma âncora para recrutar efetores, não se restringe a
heterocromatina, sendo utilizados em loci eucromático, onde
H3K4me e H3K36me recrutam HDAC para a remodelação da
cromatina
(Grewal e Jia, 2007;
Jasencakova et. al, 2003)
Modificações nas Histonas
“Código de Histonas”
A alta diversidade de modificações de histonas, bem como
o elevado número de resíduos que podem ser modificados
dentro de caudas das histonas, e a correlação das
alterações individuais com vários processos nucleares,
levam à hipótese de que combinações específicas de
modificações de histonas fornecem um código de histonas,
que determina funções específicas
(Jasencakova et. al, 2003)
Comparar a distribuição das modificações de histonas ao
longo dos cromossomos durante o ciclo celular em espécies
de plantas que diferem no tamanho do genoma e na filogenia
Chromosomal histone modification patterns – from conservation to diversity
Padrões de distribuição das modificações nas histonas não são
conservados entre as plantas
Chromosomal histone modification patterns – from conservation to diversity
• Arabidopsis - 160 Mbp/1C
Eucromatina: marca específica da eucromatina, enquanto que o
nucléolo e o cromocentros permanecem não marcados
Heterocromatina: anticorpos contra as marcas de metilação
mostra sinais específicos preferencialmente no cromocentros
Chromosomal histone modification patterns – from conservation to diversity
• H. vulgare – 5100 Mbp/1C (Orientação de Rabl):
Eucromatina: marcas de metilação específicas mostram uma
redução gradual da marcação dos telômeros em direção ao
pólo centromérico
Heterocromatina: marcas de metilação mostram uma redução
de intensidades de sinal detectável nas regiões eucromáticas
FISH com sondas para
centrômeros (BAC7 cyan) e os telômeros
(HvT01 - verde)
(Fuchs et al., 2006)
Chromosomal histone modification patterns – from conservation to diversity
• Vicia faba - 740 Mb/1C
Eucromatina: marcas específicas de eucromatina são ausentes
nos conjuntos de heterocromatina
Heterocromatina: marcas específicas apresentam uma
distribuição dispersa
(Fuchs et al., 2006)
Chromosomal histone modification patterns – from conservation to diversity
• Dinâmica do ciclo celular e a intensidade de acetilação em vários
resíduos de lisina da H3 e H4 em domínios distintos da
cromatina:
A intensidade de acetilação em eucromatina e heterocromatina
aumenta durante a replicação
Chromosomal histone modification patterns – from conservation to diversity
• A distribuição cromossômica das modificações (acetilação e
metilação) podem variar ao longo do ciclo celular, bem como
dentro e entre grupos de eucariontes
possibilidade de divergência evolutiva na leitura do "código das
histonas”
• Conclusões:
a metilação da H3K4 me2 são restritas a eucromatina
H3K9me1, 2, e H3K27me1 H4K20me1 são marcas conservadas da
heterocromatina e exibem padrões de distribuição diferentes
dependendo do tamanho do genoma
Silenciamento de RNA
RNA de interferência
• Conceito:
É um mecanismo encontrado em uma grande
variedade de eucariontes, e sua principal
característica é a utilização de pequenas moléculas de
RNA (21-28 nucleotídeos) que conferem alta
especificidade a seqüência homóloga
(Lippman e Martienssen, 2004)
Silenciamento
RNA de interferência
de RNA
• Histórico:
Plantas: descrito em Petunia
Animais: Fire e colaboradores (1998) descreveram o
mecanismo em Caenorhabditis elegans como "interferência
por RNA ” (RNAi)
(Almeida e Allshire, 2005; Novina e Sharp, 2004)
RNAi foi originalmente definido como um mecanismo de
silenciamento pós-transcricional
• A maquinaria de RNAi é composta por: RNA-dependente de
RNA polimerase (RdRP), Dicer (Dcr) e Argonauta (Ago)
(Grewal e Jia, 2007)
RNA de interferência
•
Atuação do RNAi está envolvida
heterocromatina em sítios de:
na
manutenção
da
o elementos transponíveis (‘jumping genes’)
o DNAs repetitivos (incluindo transgenes)
altera a estrutura da cromatina
Garantem a estabilidade do genoma
(Lippman e Martienssen, 2004; Novina e Sharp, 2004)
Mecanismo
Recrutamento de:
•Histona Metiltransferase
•De novo DNA metiltransferase
•Proteínas que modificam cromatina
RNA de interferência
Heterochromatin siRNA: hc-siRNA
(Vaucheret, 2006)
Heterocromatina em Plantas
• A heterocromatina em plantas envolve RNAi, mas a metilação
no DNA e modificações são fatores importante na sua formação;
• 90%-95% dos siRNAs (pequenos RNAi) em Arabidopsis
correspondem a transposons e sequências repetitivas, nas
quais as histonas e o DNA são facilmente metilados;
RNAi e metiltransferases
das histonas
DNA metiltransferases e
histonas deacetilases
participam da formação e
manutenção da heterocromatina
(Lippman e Martienssen, 2004)
Conclusão
• As modificações nas histonas e a metilação do DNA são
mecanismos interdependentes na formação e manutenção da
heterocromatina
(Fuchs et al., 2008)
• O papel da metilação do DNA na formação da
heterocromatina foi reconhecido antes das modificações das
histonas
(Lippman & Martienssen, 2004)
• Diferenças na indexação heterocromatina correspondem a
diferenças na composição de seqüência ao longo dos
cromossomos e, conseqüentemente, diferenças na
organização do cromossomo
(Fuchs et al., 2008)
Conclusão
• Nas plantas, transposons são inativos e altamente metilados,
enquanto genes raramente têm qualquer metilação do DNA
(Rabinowicz et al., 2005)
• A heterocromatização dos elementos móveis serve como um
mecanismo de defesa, silenciando sua atividade
potencialmente deletéria
• Tem papel na regulação da expressão gênica durante o
desenvolvimento e diferenciação celular
Conclusão
• Estes 3 mecanismos grande importância no processo evolutivo
das plantas
http://www.nature.com/scitable/nated/content/19804/10.1038_nature02625-f1_mid_1.jpg
Referencial Bibliográfico
• ALMEIDA, R. E ALLSHIRE, R.C. 2005 RNA silencing and genome regulation.
TRENDS in Cell Biology 15: 251- 258.
• FUCHS, J et al. 2006 Chromosomal histone modification patterns – from
conservation to diversity. TRENDS in Plants Science 11:199-208.
• GREWAL, S. I. S. e JIA, S. 2007 Heterochromatin revisited. Nature Reviews
8: 35-46.
• GRAFI G, ZEMACH, A e PITTO, L 2007 Methyl-CpG-binding domain (MBD)
proteins in plants. Biochimica and Biophysica Acta 1769:287-294.
• JASENCAKOVA, Z. et al. 2000 Histone H4 Acetylation of Euchromatin and
Heterochromatin Is Cell Cycle Dependent and Correlated with Replication
Rather Than with Transcription. The Plant Cell 12: 2087-2100.
• JASENCAKOVA, Z.;
MEISTER, A. E SCHUBERT, I. 2001 Chromatin
organization and its relation to replication and histone acetylation during
the cell cycle in barley. Chromosoma 110: 83-92.
Referencial Bibliográfico
• LIPPMAN, Z. and MARTIENSSEN, R. 2004 The role of RNA interference in
heterochromatic silencing. Nature 431: 364-370.
• NOVINA,C.D. e SHARP, P.A. 2004 The RNAi revolution. Nature 430: 161164.
• RABINOWICZ, P.D. et al. 2005 Differential methylation of genes and
repeats in land plants. Genome Research 15:1431–1440.
• RICHARDS, E.J. 2006. Inherited epigenetic variation -revisiting soft
inheritance Nature Reviews 7: 395-401.
• SUMNER, A. 2003 Chromosomes Organization and Functions. Blackwell
Science Ltd. Cap 7- pag.:84-96.
• VAUCHERET, H. 2006 Post-transcriptional small RNA pathways in plants:
mechanisms and regulations. Genes Dev. 20: 759-771.
OBRIGADA!!!
[email protected]