PROGRAMA DE APRIMORAMENTO
PROFISSIONAL
SECRETARIA DE ESTADO DA SAÚDE
COORDENADORIA DE RECURSOS HUMANOS
FUNDAÇÃO DO DESENVOLVIMENTO ADMINISTRATIVO
HOSPITAL DAS CLÍNICAS FACULDADE DE MEDICINA DE
RIBEIRÃO PRETO - USP
ARIANE JOANA D’ARC RUSSO DE OLIVEIRA
Frequência de associação de haplótipos de HLA-B/HLA-C E HLA-DRB1/HLADQB1 de doadores voluntários de medula óssea da região de Ribeirão Preto
Ribeirão Preto
2014
PROGRAMA DE APRIMORAMENTO
PROFISSIONAL
SECRETARIA DE ESTADO DA SAÚDE
COORDENADORIA DE RECURSOS HUMANOS
FUNDAÇÃO DO DESENVOLVIMENTO ADMINISTRATIVO
HOSPITAL DAS CLÍNICAS FACULDADE DE MEDICINA DE
RIBEIRÃO PRETO - USP
ARIANE JOANA D’ARC RUSSO DE OLIVEIRA
Frequência de associação de haplótipos de HLA-B/HLA-C E HLA-DRB1/HLADQB1 de doadores voluntários de medula óssea da região de Ribeirão Preto
Monografia apresentada ao Programa de
Aprimoramento Profissional/CRH/SESSP e FUNDAP, elaborada no Hospital das
Clínicas da Faculdade de Medicina de
Ribeirão Preto da Universidade de São
Paulo – USP/ para conclusão do programa
de aprimoramento profissional em
Imunogenética.
Orientadora: Neifi Hassan Saloum Deghaide
Ribeirão Preto
2014
OLIVEIRA, ARIANE JOANA D’ARC RUSSO DE OLIVEIRA
BIBLIOTECA CENTRAL DA USP DE RIBEIRÃO PRETO
FACULDADE DE MEDICINA
DE RIBEIRÃO PRETO _ USP
TOMBO: ___________ SYSNO.: _______________
MONOGRAFIA 2014
Frequência de associação de haplótipos de HLA-B/HLA-C E HLA-DRB1/HLA-DQB1
de doadores voluntários de medula óssea da região de Ribeirão Preto
ALUNO: ARIANE JOANA D’ARC RUSSO DE OLIVEIRA DE OLIVEIRA
ORIENTADOR: NEIFI HASSAN SALOUM DEGHAIDE
RESUMO
OLIVEIRA, A. J.D.R. Frequência de associação de haplótipos de HLA-B/HLA-C
E HLA-DRB1/HLA-DQB1 de doadores voluntários de medula óssea da região
de Ribeirão Preto. Monografia apresentada ao Programa de Aprimoramento
Profissional/CRH/SES-SP e FUNDAP, Ribeirão Preto, 2014.
O número de transplantes realizados tem crescido, devido à compreensão
dos mecanismos envolvidos na rejeição do enxerto, moléculas de MHC (HLA) e o
advento de terapias imunossupressoras eficientes, e de acordo com a Associação
Brasileira de Transplantes de Órgãos (ABTO), o número de transplantes aumentou
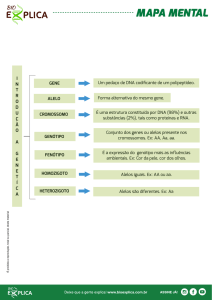
450% em 10 anos. Os genes que compõe a molécula de HLA apresentam duas
propriedades importantes: uma é a expressão dos genes de forma codominante, ou
seja, todos os genes de origem materna como os paternos são expressos. O
conjunto de alelos do MHC presentes em cada cromossomo é herdado em conjunto
e é denominado de haplótipo, assim o genótipo de um indivíduo é constituído por
dois alótipos HLA. O presente trabalho tem por objetivo Investigar a frequência de
associação entre os haplótipos de HLA-B e HLA-C e entre os haplótipos HLA-DRB1
e HLA-DQB1 de doadores voluntários da região de Ribeirão Preto bem como
estabelecer quais são os alelos mais comuns da mesma população. Para isso foi
realizada uma busca de doadores voluntários de medula óssea registrados no banco
de dados interno (TMO Voluntário) do Centro Regional de Hemoterapia do Hospital
das Clínicas da Faculdade de Medicina de Ribeirão Preto da Universidade de São
Paulo, que possuíam a tipificação HLA de alta resolução, totalizando 603 doadores.
Esses dados encontravam-se dispostos em planilhas do aplicativo Microsoft Office
Excel e foram organizados em tabelas no mesmo aplicativo, e através de contagem
direta, utilizando-se da ferramenta “Filtro”, foram calculadas as frequências. Através
delas foi possível observar a predominância de alguns Alelos, como: HLA B7, B15,
B35, B44 e B51, e HLA DRB1 03:01, DRB107:01, DRB1 11:01 e DRB1 13:01. Além
de caracterizar populações, o sistema HLA, tem sido muito utilizado para
caracterizar doenças em relação à susceptibilidade/resistência. Desde o final dos
anos 60 os antígenos HLA tem sido estudados em uma grande variedade de
doenças de distintas etiologias, incluindo as auto-imunes, as infecciosas, as
neoplásicas e as idiopáticas. Atualmente muitos estudos tem sido desenvolvidos em
relação a associação de haplótipos de HLA a doenças específicas, a fim de
determinar marcadores moleculares que proporcionem um melhor prognóstico.
Palavras Chave: Tipificação HLA, Haplótipos HLA, Transplantes.
SUMÁRIO
1. INTRODUÇÃO ................................................................................................. 6
1.1 Transplantes de órgãos sólidos, tecidos e medula óssea ........................... 6
1.2 Complexo principal de histocompatibilidade ............................................. 13
1.3 Tipificação HLA ......................................................................................... 16
2. OBJETIVO ...................................................................................................... 17
3. METODOLOGIA ............................................................................................. 17
4. RESULTADOS ............................................................................................... 18
4.1 Gráficos de frequência de associação entre HLA-B COM HLA-C ............ 18
4.2 Gráficos de frequência de associação entre HLA-DRB1 COM HLA-DQB1
.................................................................................................................. 24
5. DISCUSSÃO/CONCLUSÃO ........................................................................... 32
6. REFERÊNCIAS BIBLIOGRÁFICAS ............................................................... 33
6
1. INTRODUÇÃO
1.1 TRANSPLANTES DE ÓRGÃOS SÓLIDOS, TECIDOS E MEDULA ÓSSEA
Transplante é o processo de retirada de células, tecidos ou órgãos de um
indivíduo e sua inserção em outro indivíduo ou nele próprio. Essas células, tecidos
ou órgãos retirados de um indivíduo são chamados de enxertos e este indivíduo de
doador. Já o indivíduo que recebe o enxerto é denominado receptor (ABBAS et
al.,2008).
A terapia baseada em transplantes tem sido adotada há muito tempo. A
descrição do primeiro transplante de medula óssea ocorreu em 1959, por Edward
Donnall Thomas, nos Estados Unidos (GELLER, SCHEINBERG, 2005). Em 1912,
Alexis Carrel – com o seu trabalho “Transplantation of Veins and organs”
desenvolveu métodos de anastomose de vasos e fluidos para a preservação dos
órgãos a serem transplantados, lançando as bases para os futuros transplantes. Em
1939, Peter Gorer descreveu o Complexo Principal de Histocompatibilidade (MHC)
em camundongos, posteriormente estudados por Snell e Benacerraf. A partir disso,
Peter Brian Medawar e MacFarlane Burnet estabeleceram os mecanismos de
rejeição e tolerância aos transplantes depele entre camundongos (ABBAS et al.,
2008).
Em
1952,
Jean
Dausset
descreveu
o
primeiro
Antígeno
de
Histocompatibilidade em humanos, e posteriormente, obteve-se a identificação do
Complexo Principal de Histocompatibilidade Humano, conhecido como Sistema HLA
– “Human Leukocyte Antigen” (GOLDMAN, AUSIELLO, 2009). Após isso, Edward D.
Thomas, em 1968, realizou com sucesso o primeiro transplante de medula óssea
entre irmãos não gêmeos (GELLER, SCHEINBERG, 2005).
Atualmente, os transplantes com finalidade terapêutica tornaram-se comuns
devido à compreensão dos mecanismos envolvidos na rejeição do enxerto e o
advento de terapias imunossupressoras eficientes (BENJAMIN et al., 2002).
Consequentemente o número de transplantes realizados tem crescido (Figura 1) e
de acordo com a Associação Brasileira de Transplantes de Órgãos (ABTO), no ano
de 2002 foram realizados 10.578 transplantes (Tabela 1), tendo aumentado, em 10
anos, para 47.701. Dos transplantes realizados em 2012, 15,57% foram transplantes
de órgãos sólidos (rim, coração, fígado, pâncreas isolado, pulmão, intestino e
pâncreas/rim), 80,76% de tecidos (córnea, ossos e pele) e 3,67% de células
7
progenitoras hematopoiéticas (medula óssea). Esses dados podem ser observados
na Tabela 2.
Figura 1. Medula Óssea e Tecidos: número de transplantes de 2002 até 2012.
Fonte: RBT . Acesso em: 10 Jul. 2013
8
Tabela 1. Número de Transplantes de Órgãos Sólidos e Tecidos durante 2002.
ÓRGÃOS
Órgãos
Vivo
Falecido
Coração
0
146
Fígado
143
515
Intestino
0
0
Pâncreas/Rim
6
137
Pâncreas Isolado
0
42
Pulmão
3
30
Rim
1.806
1.184
Total
1.953
2.049
TECIDOS
Córneas
0
4.976
Ossos
80
544
Total
80
5.520
MEDULA ÓSSEA
Alogênico
562
Autólogo
404
Total
966
-
Total
146
658
0
143
42
3
2.990
4.012
4.976
624
5.600
562
404
966
Fonte: RBT. Acesso em: 10 Jul. 2013
Tabela 2. Número de Transplantes de Órgãos Sólidos e Tecidos durante 2012.
ÓRGÃOS
Vivo
0
117
0
0
0
1
1.488
Órgãos
Coração
Fígado
Intestino
Pâncreas/Rim
Pâncreas Isolado
Pulmão
Rim
Total
TECIDOS
Córnea
15.281
Ossos
23.211
Pele
30
Total
38.522
Falecido
227
1.478
0
31
119
68
3.897
Total
227
1.595
0
31
119
69
5.820
MEDULA ÓSSEA
Alogênico
630
Autólogo
1.123
Total
Fonte: RBT. Acesso em: 10 Jul. 2013
1.753
9
O TMO é o transplante de células-tronco hematopoiéticas pluripotentes, com
o objetivo de corrigir um defeito quantitativo ou qualitativo da medula óssea, pois é
conhecido que a medula óssea mantém-se em atividade intensa e ininterrupta para
produzir células sanguíneas e quando existe qualquer anormalidade ela precisa ser
substituída para que seja promovida reconstituição hematopoiética de forma
quantitativa ou qualitativa. É utilizado no tratamento de doenças hematológicas
neoplásicas e não neoplásicas, imunodeficiências, erros inatos do metabolismo e
tumores sólidos (ABBAS et al.,2008; ZAGO et al.,2004).
O fundamento lógico para o transplante de células-tronco está baseado no
fato de que todas as células maduras que circulam no sangue, tais como glóbulos
vermelhos, glóbulos brancos e plaquetas, provêm de uma única célula contida na
medula óssea denominada célula progenitora ou “stem cell”. Assim, após o
transplante, as células progenitoras infundidas na corrente sanguínea se implantam
na medula óssea iniciando a reconstituição hematopoiética do paciente, após regime
de condicionamento. O condicionamento é o uso de altas doses de quimioterapia
associado ou não à radioterapia corporal para que o paciente seja tratado de sua
doença hematológica. Com uma infusão suficiente de células progenitoras do
paciente ou de um doador próximo e compatível, a função da medula e a produção
das células do sangue são restauradas de maneira suficiente a permitir a
recuperação de um tratamento intensivo (ABBAS et al.,2008; ABRALE, 2011).
Atualmente, o termo “Transplante de Medula Óssea” denominado TMO, vem
sendo modificado por um termo mais específico: Transplante de Células
Progenitoras Hematopoiéticas (TCPH), pois tal denominação reflete melhor o tipo de
procedimento realizado e o tipo de célula que o paciente irá receber para reconstituir
sua medula óssea (ABRALE, 2011).
O transplante é classificado de acordo com o tipo de doador, sendo:
transplante autogênico, quando utiliza as células do próprio paciente, coletadas
previamente; transplante singênico, no qual o doador é um irmão gêmeo idêntico,
sendo a modalidade mais rara de transplante devido à baixa frequência de gêmeos
idênticos na população, porém é considerado o doador ideal; transplante
alogênico, no qual o paciente recebe a medula de outra pessoa, que pode ser
algum familiar, ou seja, um doador aparentado, ou sem vínculo familiar, um doador
não aparentado (CASTRO JR et al.,2011).
10
As células progenitoras hematopoiéticas (CPH) são encontradas em
diferentes concentrações na medula óssea, sangue periférico e sangue de cordão
umbilical (SCU) (ZAGO et al.,2004).
A coleta da medula óssea é realizada em centro cirúrgico, sob anestesia, e
tem duração de aproximadamente duas horas. São realizadas múltiplas punções,
com agulhas, nos ossos posteriores da bacia (cristas ilíacas). Geralmente, a
quantidade de medula óssea necessária para o transplante é estimada em 10 mL/kg
de peso do receptor, que geralmente corresponde a um número adequado de
células progenitoras suficiente para permitir a pega do enxerto. Esta retirada não
causa qualquer comprometimento à saúde do doador. O índice de complicações
graves deste procedimento é baixo (cerca de 0,4%). Estas complicações ocorrem,
em sua maioria, em doadores com história de doença prévia, e metade delas pode
ser atribuída à anestesia. A maioria dos doadores recebe alta 24 horas após a coleta
e a grande maioria dos transplantes alogênicos ainda são realizados utilizando-se
desta forma de coleta (CASTRO JR et al.,2011; INCA, 2011).
As células progenitoras hematopoiéticas periféricas são coletadas com o
auxílio de equipamentos de aférese, após a mobilização das mesmas a partir da
medula óssea para o sangue periférico, através da utilização de fator de
crescimento, sendo mais usado o fator de crescimento de granulócitos, o G-CSF, ou
através de quimioterapia; no caso de pacientes submetidos ao transplante
autogênico, pode ser combinada uma quimioterapia prévia (ABRALE, 2011). Foi
durante a década de 80 que a coleta de células do sangue periférico se consagrou,
sendo utilizada em mais de 90% dos transplantes autogênicos e em cerca de 20%
dos transplantes alogênicos (CASTRO JR, 2011). As complicações mais frequentes
da coleta de células progenitoras hematopoiéticas periféricas são relacionadas à
passagem do cateter (pneumotórax), preferindo-se desta maneira que cirurgiões
experientes façam o procedimento. O G-CSF pode provocar efeitos colaterais como
dor óssea, cefaléia e febre; entretanto, é pouco frequente a não realização da coleta
por este motivo (ABRALE, 2011; CASTRO JR et al.,2011).
A primeira experiência bem sucedida no uso do sangue de cordão umbilical
(SCU) como fonte de células para reconstituição de medula óssea ocorreu em 1988
(ABRALE, 2011). É coletado logo após o nascimento da criança, sendo
posteriormente processado e mantido congelado até a infusão. O número de
células-tronco provenientes do cordão e da placenta é geralmente insuficiente para
11
transplantar pessoas adultas. Portanto, normalmente quem recebe as células
progenitoras do SCU são crianças e adultos de tamanho pequeno ou médio (até 50
Kg) (ABRALE, 2011; CASTRO JR et al., 2011).
A Tabela 3 resume os tipos de transplante de medula óssea e as fontes de
células utilizadas.
Tabela 3. Tipos de transplantes, células e doadores.
TIPO DE
TRANSPLANTE
FONTE DE CÉLULAS
PROGENITORAS
DOADOR
Autogênico
Medula Óseea
Sangue Periférico
Próprio Paciente
Singênico
Medula Óseea
Sangue Periférico
Irmão Gêmeo Idêntico
Alogênico
Medula Óseea
Sangue Periférico
Sangue de Cordão
Umbilical
Relacionado: Irmão ou
outro familiar
Não relacionado:
qualquer pessoa sem
laços familiares com o
paciente
Fonte: CASTRO JR et al., 2011.
O TMO consiste de várias etapas e inicia-se pela decisão da necessidade e
viabilidade do transplante. A indicação depende da superioridade desta terapêutica
às outras disponíveis, levando em conta a idade do paciente e do doador, doença de
base e estadio clínico, sendo que complicações prévias e presentes na fase prétransplante estão estabelecidos em protocolos aprovados pelos Comitês de Ética
(ABRALE, 2011; ZAGO et al., 2004).
O TMO pode ser usado para tratar deficiências apresentadas pelo sistema
hematopoiético ou pelo sistema imunológico, uma vez que células sanguíneas,
incluindo os linfócitos, desenvolvem-se a partir de uma célula-tronco em comum. É
indicado para doenças neoplásicas e não neoplásicas. Dentre as doenças não
neoplásicas destaca-se a Anemia Aplástica Severa (AAS), compreendendo mais de
75% dos transplantes realizados neste grupo de doenças. O transplante na AAS
está indicado para os pacientes com idade inferior a 40 anos e com citopenia de
risco. Dentre as doenças neoplásicas destaca-se a Leucemia Mielóide Crônica
(LMC), a qual o TMO alogênico é o único procedimento capaz de determinar a cura
12
definitiva (ABBAS et al., 2008; ZAGO et al., 2004). A Tabela 4 apresenta as doenças
neoplásicas e não neoplásicas, nas quais o TMO é indicado.
Tabela 4. Tipos de doenças neoplásicas e não neoplásicas.
DOENÇAS NEOPLÁSICAS
Leucemias
- Leucemia Mielóide Aguda
- Leucemia Linfóide Aguda
- Leucemia Mielóide Crônica
- Leucemia Linfóide Crônica
- LMC juvenil
- Síndromes Mielodisplásicas
- Leucemias e SMD secundárias
Doenças linfoproliferativas
- Linfoma de Hodgkin
- Linfoma não-Hodgkin
- Mieloma Múltiplo
Tumores sólidos
- Neuroblastoma
- Câncer de mama
- Câncer testicular
- Câncer de ovário
- Câncer pulmonar de pequenas
células
DOENÇAS NÃO NEOPLÁSICAS
Falências Medulares Adquiridas
- Anemia Aplástica Severa (AAS)
- Hemoglobinúria paroxística noturna
(HPN)
Falências Medulares Hereditárias
- Anemia de Fanconi
- Síndrome de Diamond-Blackfan
- Agranulocitose de Kostmann
- Histiocitose eritrofagocítica familiar
- Disceratose congênita
- Síndrome de Shwachman-Diamond
Hemoglobinopatias
- Talassemia maior
- Anemia Falciforme
Deficiências Imunológicas
- Imunodeficiência combinada grave
(SCID)
- Síndrome de Wiskott-Aldrich
- Doença granulomatosa crônica
infantil
Erros Inatos do Metabolismo
- Doença de Gaucher
- Síndrome de Hunter
Fonte: ZAGO et al., 2004.
Definida a necessidade e o doador, a etapa seguinte constitui-se no regime
preparativo ou de condicionamento. Os pacientes são preparados para o transplante
de medula óssea com uma combinação de altas doses de agentes quimioterápicos,
associados ou não com radioterapia corporal (ABRALE, 2011; BOUZAS, 2000;
ZAGO et al., 2004).
Logo após o transplante de medula óssea, ocorre a deficiência imunológica
humoral e celular que pode perdurar por meses e até anos. A regeneração do
sistema imunológico exige a recuperação de componentes adaptativos e inatos. O
sistema imune inato é restaurado rapidamente, de dois a seis meses após o
transplante de medula óssea, enquanto a recuperação dos linfócitos T e B é muito
mais difícil de ser alcançada, sendo por volta de dois anos (MOTA et al., 2011).
13
Além da imunodeficiência, outro problema frequente associado ao transplante de
medula óssea é a Doença do Enxerto Contra o Hospedeiro (DECH), que está
envolvida em torno de 40% a 50% dos pacientes submetidos a um TMO alogênico.
Também aumenta as taxas de mortalidade e morbidade do procedimento e tem
como órgãos-alvo o fígado e as células epiteliais do intestino e pele (MOTA et al.,
2011; SOARES et al., 2007).
A DECH consiste no ataque das células T presentes no enxerto contra os
tecidos do receptor, podendo ser crônica ou aguda. Isso ocorre quando linfócitos T
maduros, histoincompatíveis e viáveis, que são transplantados em um organismo
incapaz de reconhecê-los como estranhos, são diretamente estimulados pelas
proteínas HLA do hospedeiro. As condições para ativação, replicação e ação desses
linfócitos são promovidas pelo próprio hospedeiro e destinadas contra ele (ABBAS,
LICHTMAN, 2007; LANDI, OLIVEIRA, 1999). Atualmente tem sido feito um grande
esforço para prevenção da DECH, e o conhecimento sobre os genes e as moléculas
de histocompatibilidade faz-se extremamente necessário.
1.2 COMPLEXO PRINCIPAL DE HISTOCOMPATIBILIDADE
O Complexo Principal de Histocompatibilidade ou “Major Histocompatibility
Complex” (MHC) representa o conjunto de genes responsável por codificar as
moléculas de histocompatibilidade em uma determinada espécie, sendo chamado no
ser humano de sistema HLA (“Human Leukocyte Antigen” – Antígeno Leucocitário
Humano) (FERNANDES et al., 2003).
Esses genes apresentam duas propriedades importantes: uma é a expressão
dos genes de forma codominante, ou seja, todos os genes de origem materna como
os paternos são expressos. O conjunto de alelos do MHC presentes em cada
cromossomo é herdado em conjunto e é denominado de haplótipo, assim o genótipo
de um indivíduo é constituído por dois alótipos HLA que o outro é de 25%, e neste
caso, diz-se HLA idênticos. Há a probabilidade de 50% serem haploidênticos
(apenas um haplótipo comum) e de 25% serem HLA distintos (nenhum haplótipo
comum). E a outra característica, é o polimorfismo, sendo estes os genes mais
polimórficos do genoma. O polimorfismo genético é a variabilidade em um lócus
gênico, isto é, existem muitas versões alternativas de cada gene que codificam
proteínas ligeiramente diferentes. Essa propriedade leva a consequências positivas
14
e negativas. Como positivo, tem-se a formação de diferentes moléculas de MHC o
que faz com que o sistema imune possa reconhecer o maior número de peptídeos,
ampliando assim a capacidade de resposta imunológica, em nível individual e
populacional, aos inúmeros agentes patogênicos do meio ambiente. E como
negativa, tem-se a dificuldade de transplante de órgãos e tecidos, pois é muito difícil
encontrar dois indivíduos escolhidos aleatoriamente que expressem grupos de
moléculas MHC idênticas. Assim, o polimorfismo é a base para a rejeição rápida de
enxerto entre indivíduos geneticamente diferentes. Dentre os genes mais
polimórficos do complexo gênico HLA estão os HLA-A, B e DR. Conforme a última
estatística realizada pela “European Bioinformatics Institute”, já foram descritos
1.698 alelos para HLA-A, 2.271 para HLA-B e 975 para DRB1 (ABBAS et al., 2008;
BOUZAS, 2011; ZAGO et al., 2004).
O MHC está localizado no braço curto do cromossomo 6 e apresenta um
número excepcionalmente grande de genes, os quais são agrupados em três
regiões. A região mais distal, ou seja, em direção do telômero corresponde à classe
I, a região mais centromérica corresponde à classe II, e a região intermediária
corresponde à classe III (GELLER, SCHEINBERG, 2005).
A região de classe I engloba os loci HLA-A, B, C, E, F, G, H, J, K e L. Os
genes HLA-A, B e C codificam as moléculas clássicas de histocompatibilidade de
classe I que estão presentes na superfície de todas as células nucleadas do
organismo e apresentam peptídeos gerados intracelularmente aos linfócitos TCD8+
citotóxicos. Os genes HLA-E e F codificam moléculas encontradas apenas em
tecidos fetais e em alguns tecidos da fase adulta. Os genes HLA-G codificam
moléculas presentes apenas em tecidos placentários. E os loci HLA-H, J, K e L não
codificam proteínas, sendo denominados pseudogenes (ABBAS et al., 2008;
DONADI, 2000).
A região de classe II engloba os loci HLA-DRA; DRB1-9; DQA1, 2; DQB1, 3;
DOA; DOB; DMA; DMB; DPA1, 2; DPB1, 2; TAP1, 2; LMP2, 7 e MICA-E. Os genes
HLA-DR, DQ e DP codificam moléculas clássicas de histocompatibilidade de classe
II que apresentam distribuição celular mais restrita, estando presentes na superfície
de células que participam diretamente da resposta imune, tais como macrófagos,
monócitos, células dendríticas, células de Langerhans, linfócitos B e linfócitos T
ativados, e apresentam peptídeos extracelulares, que sofreram processo de
endocitose, aos linfócitos TCD4+ auxiliares. Os genes DOA e DOB codificam as
15
cadeias α e β, respectivamente, produzindo moléculas estruturalmente semelhantes
àquelas codificadas pelos genes clássicos de classe II. Os loci DRB2, 6, 7, 8 e 9;
DQA2; DQB2; DQB3; DPA2 e DPB2 são pseudogenes. Os genes LMP2 e 7
codificam proteossomas que funcionam como endopeptidases. Os produtos dos
genes TAP1 e 2 transportam os peptídeos degradados no citosol para o retículo
endoplasmático. As moléculas HLA-DMA e DMB auxiliam no processamento e
inserção do peptídeo às moléculas HLA de classe II. Os genes MIC estão
relacionados com a codificação de moléculas relacionadas com as de classe II
(ABBAS et al., 2008; DONADI, 2000).
Os genes de classe III, embora situados dentro do MHC, não codificam
moléculas de histocompatibilidade. Codificam outros tipos de moléculas que atuam
no sistema imune, como alguns componentes do sistema complemento (C4a, C4b e
o fator B), as enzimas 21-hidroxilase (21B, 21A), a proteína do choque térmico
(Hsp70) e os fatores de necrose tumoral TNF-α e β (DONADI, 2000).
A nomenclatura dos genes HLA segue as normas determinadas por um
comitê internacional que se reúne periodicamente para nomear alelos recentemente
descobertos e rever a nomenclatura vigente, essas normas são: os genes de classe
I são denominados pela sigla HLA seguida da letra que indica a região (locus), por
exemplo: HLA-A. Os genes de classe II são designados pela sigla HLA seguida das
letras que identificam a sub-região (locus), e estas pelas letras A ou B conforme o
gene codifique uma cadeia α ou β, respectivamente. Estas últimas são
acompanhadas de um número arábico, caso haja, mais de um gene de cadeia α ou
β na mesma sub-região, por exemplo: HLA-DRB1. Os alelos, tanto de classe I como
de classe II, segue-se após um asterisco por um código de quatro dígitos separados
por (:) em que os dois primeiros dígitos correspondem ao grupo alélico e os dois
últimos representam o alelo propriamente dito, caracterizado por uma sequência
específica de nucleotídeos. HLA-A*02:01 e HLA-DRB1*01:01, são exemplos de
alelos de classe I e classe II, respectivamente. A letra W é utilizada apenas para os
epítopos públicos, ou seja, epítopos comuns a mais de um antígeno como o Bw4 e o
Bw6 e para as especificidades do locus C a fim de diferenciá-las das proteínas do
complemento (BOUZAS, 2011; VOLTARELLI et al., 2008; ZAGO et al., 2004).
As moléculas clássicas de histocompatibilidade, além de estarem envolvidas
na resposta imunológica como apresentadoras de peptídeos aos linfócitos T e serem
importantes para a compreensão de mecanismos associados à suscetibilidade ou
16
resistência a determinadas doenças, elas estão fortemente associadas ao
alorreconhecimento de antígenos em transplantes de órgãos que desencadeiam a
ativação de linfócitos e o processo de rejeição do enxerto. Portanto, a tipificação do
HLA é importante, principalmente para o TMO, pois diferente do que ocorre dentre
alguns transplantes de órgãos sólidos, o grau de compatibilidade imunológica entre
o doador e o paciente é crucial para o sucesso dos transplantes de medula óssea
(BORTOLOTTO et al., 2009; CASTRO JR et al., 2011).
1.3 TIPIFICAÇÃO HLA
Inicialmente, a tipificação das moléculas de HLA era realizada por métodos
sorológicos,
utilizando
um
ensaio
de
microcitotoxicidade
dependente
de
complemento e painéis de antissoros alogênicos contendo anticorpos anti-HLA
(BOUZAS, 2011).
Atualmente, com advento das técnicas que utilizam biologia molecular,
tornou-se possível definir cada classe de molécula HLA pela identificação de sua
sequência específica. Nesse caso, não são os antígenos expressos nas superfícies
celulares que são tipificados, e sim, os grupos de alelos ou os alelos individualmente
do DNA genômico. Para tal, o DNA é extraído das células nucleadas do sangue
periférico, utilizando um kit comercial que se baseia em cinco etapas: a hemólise; a
degradação e a retirada das proteínas; a lise de leucócitos; a precipitação do DNA; e
a purificação do DNA (BORTOLOTTO et al., 2009).
O DNA é amplificado pela Reação em Cadeia da Polimerase (“Polymerase
Chain Reaction” - PCR) utilizando oligonucleotídeos específicos para amplificar a
região do genoma a ser estudada. Dois métodos têm sido bastante utilizados para
tipificação dos alelos HLA de classe I ou II: através do emprego de iniciadores
(“primers”) ou sondas (“probes”) de oligonucleotídeos com sequências conhecidas,
sendo denominados PCR-SSP ou PCR-SSO, respectivamente (FERNANDES et al.,
2003).
No método PCR-SSP são realizadas várias reações de amplificação, cada
uma contendo um par de iniciadores capaz de detectar um grupo de alelos ou um
alelo. Os produtos de amplificação são submetidos a uma eletroforese em gel de
agarose, contendo brometo de etídio que é uma substância fluorescente capaz de
se intercalar no DNA, tornando fluorescentes os produtos de amplificação quando o
17
gel é submetido à luz ultravioleta, identificando assim, o grupo alélico ou o alelo
propriamente dito (FERNANDES et al., 2003).
No método PCR-SSO, utiliza-se um par de iniciadores construídos para
amplificar uma região genérica de um gene (por exemplo, HLA-DR). Parte do
produto da PCR também é submetida à corrida eletroforética em gel de agarose
para verificar o sucesso da amplificação. Em seguida, o DNA amplificado é
hibridizado com sondas de oligonucleotídeos (microesferas) marcadas por
fluorescência capazes de reconhecer os diversos grupos de alelos do gene. Para
identificar a intensidade da fluorescência em cada microesfera utiliza-se um
analisador de fluxo (LABScan™100). Os dados gerados pelo analisador de fluxo são
analisados pelo aplicativo HLA Fusion 2.0 para a tipagem HLA (BOUZAS, 2011).
Outro método que poderá ser muito utilizado futuramente é o sequenciamento
automático das bases nitrogenadas. A identificação dos alelos é realizada através da
comparação com um banco de dados referente às sequências já conhecidas
(FERNANDES et al., 2003).
A tipificação das moléculas de HLA tem grande importância no transplante de
células progenitoras hematopoiéticas, já que o grau de compatibilidade HLA entre o
paciente e o doador é um dos pontos cruciais para o sucesso do transplante. Quanto
maior o grau de compatibilidade HLA, maior a probabilidade de pega do enxerto
contra o hospedeiro (VOLTARELLI et al., 2008).
2. OBJETIVO
Investigar a frequência de associação entre os haplótipos de HLA-B e HLA-C
e entre os haplótipos HLA-DRB1 e HLA-DQB1 de doadores voluntários da região de
Ribeirão Preto bem como estabelecer quais são os alelos mais comuns da mesma
população.
3. METODOLOGIA
Foi realizada uma busca de doadores voluntários de medula óssea
registrados no banco de dados interno (TMO Voluntário) do Centro Regional de
Hemoterapia do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto
da Universidade de São Paulo, que possuíam a tipificação HLA de alta resolução,
totalizando 603 doadores.
18
Esses dados encontravam-se dispostos em planilhas do aplicativo Microsoft
Office Excel e foram organizados em tabelas no mesmo aplicativo, e através de
contagem direta, utilizando-se da ferramenta “Filtro”, foram calculadas as
frequências mencionadas e demonstradas em gráficos neste estudo.
4. RESULTADOS
Utilizando o cadastro de doadores voluntários que tiveram a tipificação HLA
feita em alta resolução foi possível organizar as frequências de associação entre os
grupos alélicos HLA-B e HLA-C bem como as frequências entre os grupos HLADRB1 e HLA-DQB1 que seguem expostos nos gráficos abaixo.
4.1 GRÁFICOS DE FREQUÊNCIA DE ASSOCIAÇÃO ENTRE HLA-B COM HLA-C
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
C*01 C*02 C*03 C*04 C*05 C*06 C*07 C*15
**Figura 2 – Frequência de associação do Alelo B7
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
C*02 C*03 C*04 C*05 C*06 C*07 C*12 C*16
**Figura 3 – Frequência de associação do Alelo B8
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
19
100,0%
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*01
C*03
C*04
C*05
C*06
C*12
**Figura 4 – Frequência de associação do Alelo B13
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*02 C*03 C*04 C*05 C*06 C*07 C*08 C*12
**Figura 5 – Frequência de associação do Alelo B14
50,0%
45,0%
40,0%
35,0%
30,0%
25,0%
20,0%
15,0%
10,0%
5,0%
0,0%
**Figura 6 – Frequência de associação do Alelo B15
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
20
40,0%
35,0%
30,0%
25,0%
20,0%
15,0%
10,0%
5,0%
0,0%
C*02 C*03 C*04 C*05 C*06 C*07 C*08 C*12 C*16
**Figura 7 – Frequência de associação do Alelo B18
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*01
C*02
C*03
C*04
C*05
C*06
**Figura 8 – Frequência de associação do Alelo B27
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*01 C*02 C*03 C*04 C*05 C*06 C*07 C*08 C*12
**Figura 9 – Frequência de associação do Alelo B35
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
21
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*03
C*04
C*06
C*07
C*08
C*12
**Figura 10 – Frequência de associação do Alelo B38
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*03
C*04
C*07
C*08
C*12
C*15
**Figura 11 – Frequência de associação do Alelo B39
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*02 C*03 C*04 C*06 C*07 C*08 C*12 C*14 C*15
**Figura 12 – Frequência de associação do Alelo B40
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
22
40,0%
35,0%
30,0%
25,0%
20,0%
15,0%
10,0%
5,0%
0,0%
C*02 C*03 C*04 C*05 C*06 C*07 C*08 C*16
**Figura 13 – Frequência de associação do Alelo B44
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*03
C*04
C*05
C*06
C*07
C*16
C*17
**Figura 14 – Frequência de associação do Alelo B45
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
C*02
C*03
C*04
C*06
C*07
C*12
**Figura 15 – Frequência de associação do Alelo B49
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
23
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*02
C*03
C*04
C*05
C*06
C*07
**Figura 16 – Frequência de associação do Alelo B50
35,0%
30,0%
25,0%
20,0%
15,0%
10,0%
5,0%
0,0%
C*02 C*03 C*04 C*05 C*07 C*12 C*14 C*15 C*16
**Figura 17 – Frequência de associação do Alelo B51
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*02 C*03 C*04 C*05 C*06 C*07 C*16 C*18
**Figura 18 – Frequência de associação do Alelo B57
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
24
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
C*02 C*03 C*04 C*06 C*07 C*08 C*12 C*15
**Figura 19 – Frequência de associação do Alelo B58
4.2 GRÁFICOS DE FREQUÊNCIA DE ASSOCIAÇÃO ENTRE HLA-DRB1 COM
HLA-DQB1
120,0%
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
DQB1 03:01
DQB1 03:02
DQB1 05:01
**Figura 20 – Frequência de associação do Alelo DRB1 01:01
120,0%
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
DQB1 02:02
DQB1 03:01
DQB1 05:01
**Figura 21 – Frequência de associação do Alelo DRB1 01:02
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
25
120,0%
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
DQB1 02:01 DQB1 03:01 DQB1 05:01 DQB1 06:03
**Figura 22 – Frequência de associação do Alelo DRB1 03:01
60,0%
58,0%
56,0%
54,0%
52,0%
50,0%
48,0%
DQB1 03:01
DQB1 03:02
**Figura 23 – Frequência de associação do Alelo DRB1 04:01
120,0%
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
DQB1 03:01
DQB1 03:02
DQB1 04:02
**Figura 24 – Frequência de associação do Alelo DRB1 04:02
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
26
100,0%
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1
02:02
DQB1
03:01
DQB1
03:02
DQB1
04:02
DQB1
05:01
DQB1
06:03
**Figura 25 – Frequência de associação do Alelo DRB1 04:04
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1
02:02
DQB1
03:01
DQB1
03:02
DQB1
04:01
DQB1
05:01
**Figura 26– Frequência de associação do Alelo DRB1 04:05
100,0%
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 03:01
DQB1 03:02
DQB1 04:02
**Figura 27 – Frequência de associação do Alelo DRB1 04:11
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
27
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 02:01
DQB1 02:02
DQB1 03:01
DQB1 03:03
**Figura 28 – Frequência de associação do Alelo DRB1 07:01
120,0%
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
DQB1 03:01
DQB1 04:02
DQB1 06:02
DQB1 06:04
**Figura 29 – Frequência de associação do Alelo DRB1 08:01
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 03:01
DQB1 03:02
DQB1 04:02
DQB1 06:03
**Figura 30 – Frequência de associação do Alelo DRB1 08:04
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
28
100,0%
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 02:02
DQB1 03:01
DQB1 04:02
DQB1 05:01
**Figura 31 – Frequência de associação do Alelo DRB1 08:07
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 02:02DQB1 03:01DQB1 03:02DQB1 03:03DQB1 04:02
**Figura 32 – Frequência de associação do Alelo DRB1 09:01
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1
02:02
DQB1
03:01
DQB1
03:02
DQB1
05:01
DQB1
06:02
DQB1
06:03
**Figura 33 – Frequência de associação do Alelo DRB1 11:01
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
29
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 03:01
DQB1 03:19
DQB1 04:02
DQB1 06:02
**Figura 34 – Frequência de associação do Alelo DRB1 11:02
100,0%
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 03:01 DQB1 03:02 DQB1 05:02 DQB1 06:03
**Figura 35 – Frequência de associação do Alelo DRB1 11:04
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 02:02
DQB1 03:01
DQB1 05:01
**Figura 36 – Frequência de associação do Alelo DRB1 12:01
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
30
100,0%
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1
03:01
DQB1
03:02
DQB1
03:03
DQB1
05:01
DQB1
06:03
DQB1
06:04
**Figura 37 – Frequência de associação do Alelo DRB1 13:01
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1
03:01
DQB1
05:01
DQB1
06:02
DQB1
06:03
DQB1
06:04
DQB1
06:09
**Figura 38 – Frequência de associação do Alelo DRB1 13:02
120,0%
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
DQB1
05:01
DQB1
05:02
DQB1
05:03
DQB1
06:02
DQB1
06:03
**Figura 39 – Frequência de associação do Alelo DRB1 14:01
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
31
100,0%
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1 02:01 DQB1 03:01 DQB1 05:01 DQB1 06:03
**Figura 40 – Frequência de associação do Alelo DRB1 14:02
90,0%
80,0%
70,0%
60,0%
50,0%
40,0%
30,0%
20,0%
10,0%
0,0%
DQB1
02:01
DQB1
02:02
DQB1
03:01
DQB1
03:02
DQB1
05:01
DQB1
06:02
**Figura 41 – Frequência de associação do Alelo DRB1 15:02
120,0%
100,0%
80,0%
60,0%
40,0%
20,0%
0,0%
DQB1 DQB1 DQB1 DQB1 DQB1 DQB1 DQB1
02:02 03:01 03:02 03:03 04:02 05:02 06:02
**Figura 41 – Frequência de associação do Alelo DRB1 15:02
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
32
5. DISCUSSÃO/CONCLUSÃO
Neste estudo foi analisada a frequência de associação entre os Haplótiplos de
HLA Classe I e Classe II de doadores voluntários da população de Ribeirão Preto e
região, onde foi possível observar a predominância de alguns Alelos, como: HLA B7,
-15, -35, -44 e -51, e HLA DRB1- 03:01, - 07:01, -11:01 e -13:01.
O sistema HLA é altamente informativo em estudos de genética de
populações, devido ao seu elevado polimorfismo e ao forte desequilíbrio de ligação
entre alelos de locos próximos. Essas propriedades permitem que a tipificação HLA
seja utilizada como um instrumento de investigação para caracterizar a composição
genética de diferentes povos, uma vez que a freqüência dos alelos HLA e o padrão
de haplótipos são característicos de cada etnia e população (MONTE et al., 2004).
Além de caracterizar populações, o sistema HLA, tem sido muito utilizado
para caracterizar doenças em relação à susceptibilidade/resistência.
No final dos anos 60, os primeiros estudos referentes à associação entre os
antígenos de histocompatibilidade e doenças mostraram resultados inconsistentes
na avaliação de doenças como linfoma de Hodgkin e a leucemia linfóide aguda
(WALFORD, 1970 in DONADI, 2000). No início dos anos 70, foi relatada a
importante associação do antígeno HLA-B27 com a espondilite anquilosante
(SCHLOSSTEIN, 1973 in DONADI, 2000). Desde então, os antígenos HLA tem sido
estudados em uma grande variedade de doenças de distintas etiologias, incluindo as
auto-imunes, as infecciosas, as neoplásicas e as idiopáticas.
Atualmente muitos estudos tem sido desenvolvidos em relação a associação
de haplótipos de HLA a doenças específicas, a fim de determinar marcadores
moleculares que proporcionem um melhor prognóstico.
**As frequências expostas nos gráficos não somam 100%, pois os alelos vem em pares, sendo assim
as 2 combinações foram levadas em consideração.
33
6. REFERÊNCIAS BIBLIOGRÁFICAS
ABBAS, A.K.; LICHTMAN, A.H. Imunologia básica: funções e distúrbios do sistema
imunológico. 2. ed. Rio de Janeiro: Elsevier, 2007. 345 p.
ABBAS, A.K.; LICHTMAN, A.H.; PILLAI, S. Imunologia celular e molecular. 6. ed. Rio
de Janeiro: Elsevier, 2008. 564 p.
ASSOCIAÇÃO BRASILEIRA DE LINFOMA E LEUCEMIA (ABRALLE). Transplantes
de medula óssea. São Paulo. Disponível em: http://www.abrale.org.br/doencas/
transplante/index.php. Acesso em: 11 NOV. 2013.
ASSOCIAÇÃO BRASILEIRA DE TRANSPLANTES DE ÓRGÃOS (ABTO). Registro
Brasileiro de Transplante, Ano VIII Num. 3 Jan/Dez – 2002 – São Paulo. Disponível
em http://www.abto.org.br/abtov03/default.aspx?mn=457&c=900&s=0
_________Registro
Jan/Dez
-
Brasileiro
2012
de
–
Transplante,
São
Paulo.
Ano
XVIII
Num.
Disponível
4
em
http://www.abto.org.br/abtov03/Upload/file/RBT/2012/rbt2012-parciall.pdf
BENJAMINI, E.; COICO, R.; SUNSHINE, G. Imunologia. 4. ed. Rio de Janeiro:
Guanabara Koogan, 2002. 288 p.
BORTOLOTTO, A.S.; PETRY, M.F.; KRIEGER, E.A.G; BONDAR, F.; FERNANDES,
S.R.; NEUMANN, J.; BONORINO, C. Frequência de alelos HLA A, B e DRB1 em
uma amostra de doadores voluntários de medula óssea do estado do Rio Grande do
Sul. In: MOSTRA DE PESQUISA DA PÓS-GRADUAÇÃO, 4., 2009, Rio Grande do
Sul. Anais... Rio Grande do Sul: PUCRS, 2009. p. 77- 79.
BOUZAS, L.F.S. Análise da capacidade do REDOME/RENACORD em suprir as
necessidades dos pacientes registrados no REREME: programa de pós-graduação
em oncologia. Rio de Janeiro: Instituto Nacional do Câncer, 2011.
34
BOUZAS, L.F.S. Transplante de medula óssea em pediatria e transplante de cordão
umbilical. In: SIMPÓSIO DE TRANSPLANTE DE MEDULA ÓSSEA, 3., 2000,
Ribeirão Preto. Anais... Ribeirão Preto: USP, 2000. p.241-263.
CASTRO JR., C.G.; GREGIANIN, I.J.; BRUNETTO, A.L. Transplante de medula
óssea e transplante de sangue de cordão umbilical em pediatria. J. Pediatr., v. 77, n.
5, p. 345-360, 2011.
DONADI, E.A. Como entender a nomenclatura e os mecanismos de associação
entre antígenos e os alelos de Histocompatibilidade com as doenças. In: SIMPÓSIO
DE ASSOCIAÇÃO DO SISTEMA HLA COM DOENÇAS NO BRASIL. 1., 2000,
Ribeirão Preto. Anais... Ribeirão Preto: USP, 2000. p 7-18.
FERNANDES, A.P.M.; MACIEL, L.M.Z.; FOSS, M.C.; DONADI, E.A. Como entender
a associação entre o sistema HLA e as doenças autoimunes endócrinas. Arq. Bras.
Endocrinol. Metabol. São Paulo, v. 47, n. 5, p. 601-611, Oct. 2003.
GELLER,
M.;
SCHEINBERG,
M.
Diagnóstico
e
tratamento
das
doenças
imunológicas: para clínicos, pediatras e residentes. Rio de Janeiro: Elsevier, 2005.
497 p.
INSTITUTO NACIONAL DE CÂNCER (INCA). Caracterização dos tipos de
transplantes. Rio de Janeiro. Disponível em: http://www.inca.gov.br/conteudo_
view.asp?ID=125. Acesso em: 11 NOV. 2013.
LANDI, E.P.; OLIVEIRA, J.S.R. Doença do enxerto contra hospedeiro póstransfusional-guia para irradiação gama de hemocomponentes. Rev. Assoc. Méd.
Bras., São Paulo, v. 45, n. 3, p. 261-272, jul./set. 1999.
MONTE, et al. Polimorfismo do sistema HLA em uma amostra de mestiços da
população de teresina, piauí. Rev Assoc Med Bras 2004; 50(4): 422-6.
35
MOTA, F.B.; RODOLFO, P.; ABRAHAO, S.T.; LARA, G.M. Aspectos imunológicos
dos transplantes alogênicos de células-tronco hematopoiéticas. NewsLab, Rio
Grande do Sul,ed.105, p. 90-98, 2011.
SOARES, D.S.; FREITAS, K.; BARBOSA, G.M.; ARAUJO, M.C.S. Doenças enxerto
contra hospedeiro: relato de caso. Disc. Sci. Série: Ciências da Saúde, Santa Maria,
v. 8, n.1, p. 91-113, 2007.
VOLTARELLI, J.C.; DONADI, E.A.; CARVALHO, I.F.; ARRUDA, L.K.; LOUZADA JR.,
P.; SARTI, W. Imunologia clínica na prática médica. Rio de Janeiro: Atheneu, 2008.
1070 p.
ZAGO, M. A.; FALCÃO, R.P.; PASQUINI, R. Hematologia: Fundamentos e prática.
São Paulo: Atheneu, 2004. 1081 p.