54º Congresso Brasileiro de Genética
Resumos do 54º Congresso Brasileiro de Genética • 16 a 19 de setembro de 2008
Bahia Othon Palace Hotel • Salvador • BA • Brasil
www.sbg.org.br - ISBN 978-85-89109-06-2
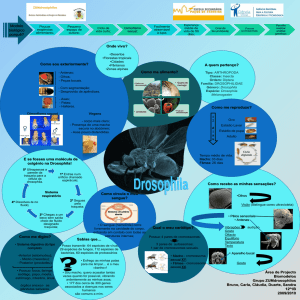
Estudo do elemento Mar, um MITE,
no gênero Drosophila
Ludwig, A1; Deprá, M1; Loreto, ELS1,2; Valente, VLS1,3
PPGBM – UFRGS
Departamento de Biologia – UFSM
3
Departamento de Genética – UFRGS
[email protected]
2
Palavras-chave: MITEs, Mar, Drosophila, superfamília hAT, transposon
MITEs (Miniature inverted repeat transposable elements) são elementos de transposição curtos, não-autônomos e
freqüentemente presentes em alto número de cópias nos genomas. Vários trabalhos têm sugerido que esses elementos
originam-se a partir de transposons autônomos que sofrem deleções internas, mas mantém as repetições terminais
invertidas (TIRs) conservadas, possibilitando mobilização in trans. O elemento Mar de Drosophila willistoni possui
610 pb, sítio alvo de duplicação (TSDs) de 8 pb, TIRs perfeitas de 11 pb e não apresenta capacidade codificadora.
Esse elemento foi classificado como um MITE da superfamília hAT com base na similaridade das TIRs e TSDs, e
foi encontrado em apenas uma linhagem de D. willistoni, analisada em um trabalho anterior de outros autores. Na
tentativa de caracterizar a representatividade deste elemento e suas implicações evolutivas nos genomas de Drosophila,
iniciamos um trabalho de análise da distribuição do elemento Mar em um grande número de espécies. Um total de
58 espécies de Drosophila de diferentes grupos dos dois principais subgêneros, Sophophora e Drosophila, juntamente
com Zaprionus indianus, Z. sepsoides, Scaptodrosophila latifasciaeormis e S. lebanonensis foram testadas por PCR para
a presença do elemento Mar. Fragmentos de tamanho esperado (400 pb) foram encontrados em todas as linhagens
do subgrupo willistoni investigadas (exceto em D. tropicalis onde encontramos um fragmento maior): 3 linhagens
de D. willistoni, 3 semiespécies de D. paulistorum, D. equinoxialis e D. insularis. Além disso, a amplificação de um
fragmento maior também foi detectada em D. saltans e D. prosaltans. Os amplicons de cada espécie foram clonados e
serão seqüenciados para a realização de análise evolutiva do elemento. Adicionalmente às análises por PCR, realizamos
buscas in silico por seqüências homólogas ao Mar, nos 12 genomas de Drosophila atualmente disponíveis. Através
da ferramenta BLAST, inúmeras cópias deste elemento (mais de 30) foram encontradas no genoma de D. willistoni.
Nenhuma seqüência foi encontrada nas outras espécies. A maioria das cópias encontradas em D. willistoni possui TIRs
e TSDs conservadas indicando recente amplificação dessas seqüências neste genoma. Dessa forma, podemos sugerir a
existência de seqüências ativas de elementos da superfamília hAT no genoma de D. willistoni capazes de mobilizar esses
MITEs. A análise das seqüências dos clones das diferentes espécies deverá contribuir para o entendimento da evolução
do elemento Mar no gênero Drosophila.
Apoio financeiro: CNPq.
282