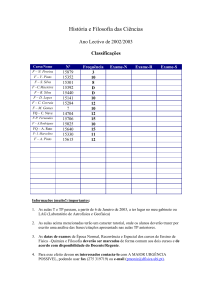
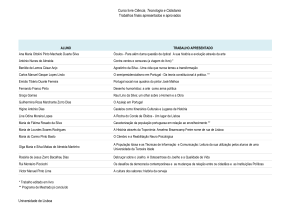
Fabiana G. S. Pinto
Anotação de Genomas
Fabiana G. S. Pinto
Obtenção de Seqüências geradas pelo MegaBace 1000
• Dados brutos (medidas analógicas) de saída do seqüênciamento→ Base calling
BIOINFORMÁTICA
* PHRED:
- Transforma os dados brutos em seqüências de bases, atribuí valores
de qualidade a cada base na seqüência e gera arquivos de saída FASTA
e PHD
* PHRAP:
- Leitura Montagem dos pequenos fragmentos de DNA seqüenciados em
seqüências maiores: CONTIG
* CONSED:
- Visualização e edição das montagens das seqüências de alta qualidade
Fabiana G. S. Pinto
Valores de qualidade gerados pelo PHRED
Quando arquivos de seqüências de DNA são analisados pelo phred
→ a cada base é assinada um valor de qualidade, o qual é uma
estimativa da probabilidade de erro para essa base.
Bases com um valor de qualidade de 20 são consideradas com um
alto valor de qualidade.
q = -10 log10(pe) onde pe= erro estimado
q20 = 1/100 probabilidade de erro
q30= 1/1000 probabilidade de erro
q40= 1/10000 probabilidade de erro
Fabiana G. S. Pinto
Regiões genômicas que podem ser melhoradas re-seqüênciamento.
Fabiana G. S. Pinto
Análise e Montagem das Seqüências
Seqüências shotgun analisadas Phred, Phrap e Consed
Resultado
Seqüências ordenadas com consenso formam um “CONTIG”
Fabiana G. S. Pinto
Anotação de Genomas
ANOTAÇÃO DE
NUCLEOTÍDEOS
Onde estão os genes?
ANOTAÇÃO DE
PROTEÍNAS
Quais são os genes?
ANOTAÇÃO DE
PROCESSOS
Cómo os genes interagem?
GLIMMER - GENEMARK
Predições de ORF – Open Reading Frame
“6 frames”
Fabiana G. S. Pinto
Gerar todas as possíveis ORFs (open reading frames) nos seis sentidos e sobrepostas
CONTIG GERADO APÓS ANOTAÇÃO PELO GLIMMER OU GENEMARK..........
Fabiana G. S. Pinto
CONTIG APÓS A ANOTAÇÃO MANUAL .........................
O anotador decide qual ORF tem que eliminar ou validar. Requere-se de conhecimento
e de muito critério.
Fabiana G. S. Pinto
Análise de Seqüências: Bioinformática
Fabiana G. S. Pinto
CONTIG APÓS A ANOTAÇÃO MANUAL .........................
O anotador decide qual ORF tem que eliminar ou validar. Requere-se de conhecimento
e de muito critério.
Fabiana G. S. Pinto
Banco de Dados Biológicos
Banco de Dados (DataBase)
Banco de Dados Públicos
Bancos Primários
INSDC – International
Nucleotide Sequence
Database Colaboration
Bancos Secundários
Bancos Funcionais
Fabiana G. S. Pinto
Sistema de
Gerenciamento de Banco
de Dados - SGBD
Bancos primários
Bancos secundários
GenBank
EBI European Bioinformatics Institute
DDBJ – DNA DataBase of Japan
PDB Protein Data Bank
PIR – Protein Information Resource
SIWSS-PROT. InterPro.
COG – Orthologous
KEGG – Kyoto Encyclopedia of Genes and
Genomes
Atualizações em Nucleic Acids Research
http:// www3.oup.co.uk/nar/database/
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Procurar pelo gene polC (dnaE, DNA Polimerase III)
Fabiana G. S. Pinto
Procurar pelo gene polC (dnaE,DNA Polimerase III)
Fabiana G. S. Pinto
Entrez: procurar pelo gene polC (dnaE) AND E coli
Fabiana G. S. Pinto
Entrez: procurar pelo gene polC (dnaE) AND E coli
Fabiana G. S. Pinto
Resgatar seqüências de genes em formato FASTA
Fabiana G. S. Pinto
Resgatar seqüências de genes em formato FASTA
Seqüência
FASTA em
nucleotídeos
Seqüência
FASTA do
produto gênico
em aminoácidos
Coordenadas do
gene no contexto do
genoma total
Fabiana G. S. Pinto
seqüência FASTA do gene dnaE
Começa com codon de
iniciação, no caso ATG
(metionina) e continua sem
espaços até um codon de
finalização
Começa com símbolo
maior e descrição do
gene e do organismo
sem espaços
Fabiana G. S. Pinto
Informação adicional do gene dnaE, Polimerase III
subunidade alfa
Link para referencias
bibliográficas no
PubMed
Processo celular
envolvido e
localização do
produto gênico
Fabiana G. S. Pinto
Informação adicional do gene dnaE, Polimerase III
subunidade alfa
EcoCyc. Informação do site de
enciclopédia e genes de E coli
http://ecocyc.org/
Classificação de
COG. Genes
Ortólogos
Classificação EC
number. Número de
Enzima
Fabiana G. S. Pinto
CONCEITOS PARA
LEMBRAR
HOMÓLOGOS
PARÁLOGOS
ORTÓLOGOS
E. coli
S.meliloti
R. tropici
ORTÓLOGOS
R. tropici
S.meliloti
E coli
Gene dnaE
Delta subunit
Gene dnaE
Alpha subunit
Duplicação gênica
Gene DNA Polimerase III inicial
Fabiana G. S. Pinto
Classificação de Gene Ortólogo para dnaE COG0587
Relação Filogenética
para COG0587 entre
50 espécies
Fabiana G. S. Pinto
Link para Procurar números de enzimas
http://www.genome.jp/dbget-bin/get_htext?ECtable
EC para Polimerase III : 2.7.7.7
Fabiana G. S. Pinto
EC para Polimerase III : 2.7.7.7
Fabiana G. S. Pinto
EC para Polimerase III: 2.7.7.7
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Alinhamento das Seqüências
•Finalidade: procurar determinar o grau de similaridade entre duas ou mais
seqüências. Alinhamento entre mais de uma seqüência é Múltiplo.
•Programas on-line: ClustalW, Multialin, FASTA, BLAST.......
•Qualidade: soma dos pontos obtidos por cada unidade (match) menos a
penalidades pela introdução dos gaps (mismatch)
Alinhamento global ou Local
Global: ClustalW-X
Multialin
•Local: BLAST (Basic Local
Alignment Search Tool)
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Blast no NCBI: http://www.ncbi.nlm.nih.gov/BLAST/
Fabiana G. S. Pinto
Vamos procurar similaridades através do Blastx do gene
dnaE de E coli (resgatar seq FASTA do entrez gene)
Fabiana G. S. Pinto
Colar a seqüência
FASTA com os
dados iniciais. Este
é nosso query
Fabiana G. S. Pinto
“Blastar”
Fabiana G. S. Pinto
Clicar Format para
abrir nova janela
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Qualidade do
Alinhamento
Score elevado
Ver Subject: Salmonella
E value máximo: 0.0. Fabiana G. S. Pinto
Ótimo: a partir de e-60 (e diminuindo
para mais negativo)
Aceitável: entre e-30 a e-60
E value maiores indicam alinhamento
ao acaso: maior a 0.0 até e-10 - e-30
typhymurium
Identidade difere de positivos.
Positivo procura analogia do
aminoácido na seq da proteína
Fabiana G. S. Pinto
http://www.ebi.ac.uk/clustalw/
Fabiana G. S. Pinto
http://www.ebi.ac.uk/clustalw/
Fabiana G. S. Pinto
Resgatar seq FASTA em Entrez Genes: RNA Polymerase
sigma subunit AND alpha proteobacteria
Fabiana G. S. Pinto
Copiar quatro seqüências FASTA (1, 6, 10 e 17) e colar em
bloco de notas
Fabiana G. S. Pinto
Selecionar tudo copiar e colar na janela do ClastalW. Run!
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
O asterisco mostra identidade. Este alinhamento auxilia
quando é preciso desenhar primers PCR para amplificar
um gene de interesse. Buscamos seqüências do gene em
espécies próxima da que estamos estudando
Fabiana G. S. Pinto
No final mostra uma
árvore filogenética
Fabiana G. S. Pinto
Banco de proteínas
http://us.expasy.org/sprot/sprot-top.html
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Banco Funcional: enciclopédia de genes e genomas
http://www.genome.jp/kegg/kegg2.html
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Procariotos
Polimerase III
subunidade alfa
Fabiana G. S. Pinto
Fabiana G. S. Pinto
Genes para 2.7.7.7 seqüenciados
em vários organismos
Fabiana G. S. Pinto
Gene dnaE em E. coli K-12 com
muita informação e links !
Fabiana G. S. Pinto
Seqüência FASTA em
aminoácidos
Seqüência FASTA em
nucleotídeos
Fabiana G. S. Pinto
Também podem navegar na lista de genomas seqüenciados
http://www.genome.jp/kegg/catalog/org_list.html