UBA III - BM
PROTOCOLOS
UBA III – Biologia Molecular
Aula Laboratorial 1
ENZIMAS DE RESTRIÇÃO
As endonucleases de restrição são enzimas que cortam a molécula de DNA em cadeia dupla através do
reconhecimento de sequências nucleotídicas específicas, atuando como “tesouras moleculares”. Foram
primeiramente identificadas e isoladas em bactérias que as usam como mecanismo de defesa natural
para degradar DNA de bacteriófagos (vírus que infectam bactérias). Podem ser usadas para a
degradação de moléculas de DNA com obtenção de fragmentos de menor tamanho, cujas extremidades
são conhecidas em termos de sequência nucleotídica e que podem ser separados por eletroforese em
gel de agarose. As sequências das extremidades e o tamanho dos fragmentos obtidos variam de acordo
com a enzima de restrição usada. Dada a sua especificidade, estas enzimas são rotineiramente
utilizadas como ferramentas moleculares na tecnologia do DNA recombinante, sendo aplicadas na
transformação genética em diversas áreas da biotecnologia, desde a indústria alimentar, agricultura
(produção de espécies transgénicas), à medicina (terapia genica), à indústria farmacêutica (ex.:
produção de insulina).
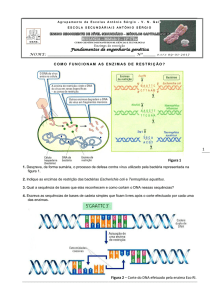
Outra propriedade das enzimas de restrição está relacionada com o tipo de terminais gerados após o
corte. Assim, há enzimas que produzem extremidades sem nenhuma das cadeias protuberante,
gerando terminais cegos (SmaI, DraI). Outras, que não cortam ao longo do eixo de simetria da
sequência de reconhecimento, deixam uma das extremidades com uma cadeia protuberante, que pode
ser a extremidade 3’ (PstI) ou a 5’ (EcoRI, HindIII), produzindo terminais coesivos.
Fig.1 - Exemplo de enzimas de restrição que produzem terminais coesivos
Endonucleases com a mesma sequência de reconhecimento que cortam a mesma ligação são
designadas isosquisómeros. Como exemplo, as enzimas MboI e NdeII reconhecem e cortam a
sequência GATC. Se diferentes enzimas tiverem atividade sobre outro local de restrição, da mesma
sequência de reconhecimento, denominam-se neosquisómeros. Por exemplo, as enzimas SacI e
EcoICRI reconhecem a sequencia 5’…GAGCTC…3’, mas cortam em GAGCTC e em GAGCTC,
respetivamente.
AS Duarte
1
UBA III - BM
PROTOCOLOS
PROTOCOLO EXPERIMENTAL
A. Preparação das amostras:
1. Coloque os tubos com as diferentes enzimas de restrição (PstI, EcoRI e HindIII), com o DNA λ e
com tampão de restrição (RB) num contentor com gelo.
Enzimas de restrição
2. Preparar um tubo de cada cor e identificá-los segundo a tabela:
amarelo
violeta
verde
laranja
L = DNA λ
P = PstI (Digestão do DNA lambda)
E = EcoRI (Digestão do DNA lambda)
H = HindIII (Digestão do DNA lambda)
3. Pipetar os reagentes de acordo com a tabela, usando pontas estéreis entre cada reagente.
Tubo DNA RB
PstI
EcoRI HindIII
4. Misturar bem batendo, gentilmente,
com o dedo, como mostra a figura:
5. Colocar as reações de digestão num banho a 37ºC,
durante 30 minutos.
37ºC
6. Reservar a 4ºC para, posteriormente, proceder à separação dos fragmentos gerados, por
eletroforese em gel de agarose.
AS Duarte
2