DEQB
4. Actividade e Estabilidade de Enzimas
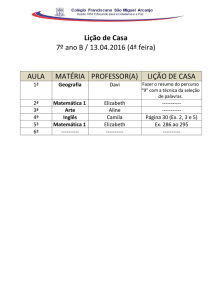
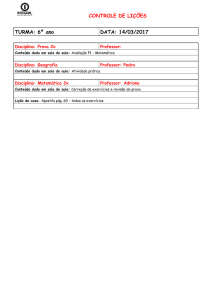
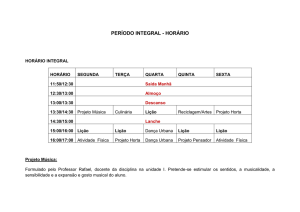
Lição de Biocatálise
ESTRUTURA DE ENZIMAS
DEQB
ENZIMA – Entidade de natureza proteica e de origem biológica que levam a
cabo in vivo as inúmeras transformações químicas e bioquímicas
necessárias à vida dos seres vivos.
Os enzimas, tal como as proteínas, são polímeros complexos constituídos
por sequências de aminoácidos, as unidades estruturais básicas, ligadas
entre si por uma ligação peptídica.
O
Cα
C
Cα
O-
N
Cα
C
H
Cα
N+
H
O homem procura utilizar os ENZIMAS para levarem a cabo in vitro
inúmeras transformações químicas e bioquímicas do qual resultam produtos
de alto valor acrescentado.
Lição de Biocatálise
ESTRUTURA DE ENZIMAS
DEQB
Os aminoácidos constituintes das proteínas são constituídos por 4 grupos
diferentes ligados a um carbono central (Cα):
H
H3N+
C
α
COO -
R
- um grupo amino (NH3+);
- um grupo carboxílico (COO-);
- um átomo de hidrogénio (H);
- um grupo variável (R);
- um átomo de carbono (carbono-α, Cα), a que se ligam os quatro grupos
anteriores.
Lição de Biocatálise
As propriedades físico-químicas dos aminoácidos estão associadas
ao grupo R variável do amínoácido
H
DEQB
H3N+
C
COO -
R
- Têm propriedades ácidas e básicas semelhantes, à excepção da prolina.
- As cadeias laterais têm uma enorme variedade de propriedades funcionais
(carga eléctrica, polaridade, hidrofobicidade, reactividade química) e estruturais,
conferindo aos aminoácidos a respectiva individualidade.
- O grupo α-amino de um aminoácido pode reagir com o grupo β-carboxilo de outro,
com formação de cadeias polipeptídicas.
- Podem sintetizar-se quimicamente polipéptidos com uma sequência pre-determinada.
- A sequência de aminoácidos numa cadeia pode ser determinada por hidrólise da cadeia
em fragmentos (péptidos) e análise sequencial da composição dos péptidos a partir de um
dos extremos da cadeia.
Lição de Biocatálise
DEQB
Tabela – Classificação, nomenclatura e constante de dissociação das cadeias
laterais R dos aminoácidos
Classificação
Abreviatura Abreviatura
de 3 letras
de 1 letra
Alanina
Ala
A
Valina
Val
V
Leucina
Leu
L
Grupo I
Isoleucina
Ile
I
Aminoácidos
Metionina
Met
M
Apolares
Prolina
Pro
P
Fenilalanina
Phe
F
Triptofano
Trp
W
Glicina
Gly
G
Serina
Ser
S
Grupo II
Treonina
Tre
T
Aminoácidos
Cisteína
Cis
C
Polares
Tirosina
Tyr
Y
Asparagina
Asn
N
Glutamina
Gln
Q
Lisina
Lys
K
Grupo III
Arginina
Arg
R
Aminoácidos
Histidina
His
H
Com Grupos R Ácido Aspártico
Asp
D
Carregados
Ácido Glutâmico
Glu
E
Lição de Biocatálise
Nome
pKR da cadeia
lateral
------------------------~ 13
~ 13
8.33
10.13
10.79
12.48
6.04
3.90
4.07
Estrutura química da cadeia lateral (R) de aminoácidos
Ácido aspártico
Glicina
O
C
H2
DEQB
H
+
H3N
C
COO
-
R
CH 3
CH
CH 3
Alanina
Leucina
O grupo R variável
CH
Hidrolixados
CH
C
H2
CH 3
pKa ~ 13.0
C
H2
C
H2
CH 3
CH 3
OH
pKa ~ 13.0
CH 3
CH
Lição de Biocatálise
C
NH 2
Ácido glutâmico
C
H2
O
C
H2
OH
pKa ~ 4.1
C
O
-
O
C
H2
C
NH 2
Lisina
C
H2
C
H2
C
H2
C
H2
Arginina
C
H2
pKa ~ 3.9
O
C
H2
CH 3
Isoleucina
Treonina
O-
Glutamina
C
H2
Serina
C
Asparagina
CH 3
Básicos
Aminoácidos
Alifáticos
Valina
Dicarboxilicos
H
pKa ~ 10.8
NH 3+
N+H2
C
H2
C
H2
N
H
C
NH 2
pKa ~ 12.5
Histidina
C
H2
C
H
C
N
NH
C
H
pKa ~ 6.0
Heterocíclicos
Histidina
C
H
C
H
C
N
NH
2
DEQB
H
H3N
C
Prolina
COO
-
NH 2
H
C
H2C
CH 2
+
R
O
C
C
H2
O-
N
H
Fenilalanina
Cisteina
pKa ~ 8.3
Sulfurados
C
HC
C
H2
O grupo R variável
Triptofano
Aromáticos
+
C
H
C
H2
SH
C
H2
Metionina
Tirosina
pKa ~ 10.1
Lição de Biocatálise
C
H2
C
H2
S
CH 3
C
H2
OH
Efeito do pH na Reacção Enzimática
v
DEQB
pH óptimo
pH
Tabela 2-1 - Exemplos de aminoácidos envolvidos no centro
activo de enzimas e respectivos valores de pKa.
Enzima
Lisozima
Acetoacetato descarboxilase
Quimotripsina
Papaína
Lição de Biocatálise
Resíduo
Glu-35
Lys
α-NH3 (Ile-16)
His-159
pKa
6,5
5,9
10,0
3,4
As modificações químicas mais comuns de alguns resíduos dos
aminoácidos durante ou após a sua biossíntese são:
DEQB
- Acetilação do grupo N-terminal.
- Carboxilação (Asp e Glu).
- Fosforilação (Ser, Thr, Tyr e His).
- Formação de pontes dissulfureto (Cys).
- Glicosilação (Asn: pontes N-glicosídicas; Ser e Thr: ligações O-glicosídicas).
- Metilação (Lys e Glu).
- Ligação covalente:
. De grupos prostéticos (grupos de natureza não proteica que se ligam
covalentemente (irreversível) ou não-covalentemente (reversível) à
proteína na sua forma tridimensional, (p.ex. grupos hemo do citocromo).
. Substituição/modificação de grupos funcionais das cadeias laterais
(p.ex. –NH2), alterando assim as propriedades de carga, polaridade ou
hidrofobia desse aminoácido. A modificação química específica de alguns
resíduos (p.ex. lisinas) é um dos métodos possíveis para melhorar a
actividade e estabilidade enzimáticas.
Lição de Biocatálise
Estrutura Primária e Secundária de proteínas
A)
DEQB
B)
C
H N
O
N
C
H
N
H
C
O
HC-R
Folha β Paralela
H N
HC-R
O
H N
C
14 Å
N -H
O =C
N
C
O
H
O
H N
C
HC-R
O
H N
HC-R
C
R-CH
Folha β Antiparalela
Figura 2-1 – A) Representação esquemática dos dois tipos de estrutura
secundária mais frequentemente encontrados em proteínas e enzimas:
hélice a e folha β. B) A folha β paralela e antiparalela, com indicação do
avanço por cada dois aminoácidos.
Lição de Biocatálise
O
R-CH
R-CH
R-CH
H N
Folha β
H N
HC-R
O =C
O =C
R-CH
Hélice α
H N
C
O
HC-R
HC-R
13 Å
C
HC-R
R-CH
R-CH
R-CH
O
Estrutura Terciária de proteínas
DEQB
H 188
S 120
Ligações covalentes
-S–S-
D 175
Interacções electrostáticas
Pontes de Hidrogénio
Interacções hidrófobas
Figura 2-1– Representação esquemática da estrutura terciária da cutinase de
Fusarium solani pisi, com indicação de alguns resíduos que limitam o centro
activo, e, em particular, da tríade catalítica (Ser120-His188-Asp175).
Lição de Biocatálise
Estrutura Quaternária de proteínas
DEQB
Pode-se observar em enzimas multiméricos, i.e., enzimas compostos por duas ou
mais cadeias polipeptídicas associadas entre si, por exemplo, penicillin V acylase
de B. sphaericus.
Domínio α
Domínio β
Lição de Biocatálise
Horseradish peroxidase - HRP
DEQB
Monomeric enzyme
Prosthetic group - heme
2 calcium ions
Glycoprotein – 8 specific sites
Molecular weight - 44 kDa
Isoelectric point - 7.5
Catalyses the oxidation of several
compounds by the H2O2, e.g. phenols,
aromatic amines such as phenolsulfonic
acid (PSA) and 4-aminoantipyrine (4-AAP)
FUNÇÃO DOS ENZIMAS
DEQB
Os enzimas (catalisadores biológicos ou biocatalisadores) catalisam
reacções bioquímicas, aumentando a sua velocidade, por exemplo:
2H O —> 2H O + O
2 2
2
2
______________________________________________________________________
CATALISADOR
Ea (kJ/mol)
Velocidade reacção
(Relativa)
SEM CATALISADOR
75,4
1,0
PLATINA (INORGÂNICO)
50,2
2x104
CATALASE (ENZIMA)
8,4
3x1011
______________________________________________________________________
Em termos de propriedades gerais, comuns aos catalisadores
químicos, os biocatalisadores caracterizam-se por:
i)
baixarem a energia de activação para que a reacção que catalisam
ocorra;
ii) não serem destruídos pelo efeito da reacção;
iii) não alterarem o equilíbrio químico das reacções em que participam.
Lição de Biocatálise
FUNÇÃO DOS ENZIMAS
DEQB
Por outro lado, os biocatalisadores em relação aos catalisadores
químicos são:
-
Muitíssimo específicos:
-
tipo de reacção que catalisam,
-
tipo(s) substrato(s) que reconhecem,
-
ligação química em que actuam,
-
bem como à estereo-especificidade dos átomos ou grupo de
átomos no substrato.
-
especificidade absoluta em relação a único substrato
Lição de Biocatálise
Elevada especificidade em relação ao tipo de reacção que catalisam,
DEQB
Lição de Biocatálise
Elevada especificidade em relação ao tipo de substrato que catalisam,
DEQB
Sacarose
Maltose
Lactose
Lição de Biocatálise
Elevada especificidade em relação ao tipo de substrato que catalisam,
DEQB
Lactose
Maltose
Sacarose
Trehalose
Lição de Biocatálise
Elevada especificidade em relação ao tipo de ligação química em que
actuam,
DEQB
Lição de Biocatálise
Elevada especificidade em relação ao tipo da estereoespecificidade
do substrato em que actuam,
DEQB
Lição de Biocatálise
Elevada especificidade em relação ao comportamento dos compostos
do tipo Cx1x2yz nas reacções enzimáticas
DEQB
Estes compostos Cx1x2yz são do ponto de vista da química orgânica substâncias simétricas
em solução (pois possuem um plano de simetria) mas comportam-se assimétricas enquanto
substratos de certas reacções enzimáticas (quer dizer que x1 pode ser preferencialmente
atacado em relação ao x2 apesar de serem grupos funcionais iguais)
Lição de Biocatálise
FUNÇÃO DOS ENZIMAS
DEQB
Por outro lado, os biocatalisadores em relação aos catalisadores
químicos são:
-
A actividade catalítica regulável,
-
Mas devido à sua natureza proteica são lábeis e inactivados por
efeito da temperatura, pH, ou na presença de alguns compostos
(designados por agentes desnaturantes).
Lição de Biocatálise
Características do centro activo
DEQB
O centro activo corresponde geralmente a uma fenda ou cavidade existente
na estrutura da molécula proteica onde se localiza o centro catalítico
(centro activo) e locais de reconhecimento do substrato (centro de ligação
do substrato) e caracteriza-se por:
- Ocupa uma parte relativamente pequena do volume total de um enzima;
- É uma entidade tridimensional formada por grupos que pertencem a
partes distintas da sequência linear de aminoácidos;
- Os substratos ligam-se às enzimas através de atracções fracas
múltiplas (electrostáticas, de van der Waals, hidrófobas, ligações de
hidrogénio);
- A especificidade da ligação do substrato depende do arranjo dos
átomos no centro activo (modelo da chave e fechadura - E. Fischer, 1890;
modelo do ajuste induzido - D. Koshland, 1958).
Lição de Biocatálise
Características do centro activo
DEQB
a)
+
substrato
enzima
complexo
enzima - substrato
enzima
complexo
enzima - substrato
b)
+
substrato
Figura 2-1– a) Modelo chave-fechadura; b) Modelo do ajuste induzido
Lição de Biocatálise
Características do centro activo
DEQB
Muitas vezes os enzimas exigem pequenas moléculas ou iões (ex: Zn, Fe,
Cu, Ca…) para exercerem uma actividade catalítica; a essas espécies
chama-se co-factores. Ao enzima sem cofactor chama-se apoenzima, e
ao enzima activo intacto (enzima + cofactor) chama-se holoenzima.
- Quando os co-factores são moléculas orgânicas (ex: vitaminas B) ou de
natureza nucleotídica (ex: nicotinamida-adenina-dinucleótido - NAD e NAD
fosfato - NADP) denominam-se co-enzimas.
NAD
Lição de Biocatálise
NADP
Características do centro activo
DEQB
- Quando são necessários co-factores os enzimas têm que conter centros
de ligação adicionais.
- A catálise enzimática pode portanto fazer intervir, não só um número
limitado de grupos funcionais que se encontram em cadeias laterais de
resíduos de aminoácidos (His, Ser, Tyr, Cys, Lys, Glu, Asp), mas também
N
N
co-enzimas e catiões metálicos.
III
Fe
N
N
N
HOOH
A + H2O
N
H 2O
HRP
AH
O
N
O
IV
Fe
N
N
N
N
Compound II
IV
Fe
H+
N
A
N
Lição de Biocatálise
N
AH
N
+
N
N
N
Compound I
PSA + 4-AAP + 2H2O2 → Quinoneimine + H2O + NaHSO4
λ = 490 nm
Classificação de Enzimas
NOMENCLATURA:
DEQB
- Nomes empíricos (ex: tripsina, pepsina, quimotripsina).
- 1ª designação normalizada: nome do substrato + sufixo ase (ex: urease, amilase).
- 2ª designação normalizada: nome do substrato + tipo de reacção que catalisam
(ex: malato desidrogenase).
- Enzyme Comission EC: nome de acordo com tipo de reacção + substrato + o tipo
de aceitador do hidrogénio (ex: malato-NAD-óxido-redutase).
1º dígito
Classe da enzima
Tipo de reacção catalisada
1
Oxido-redutases
Reacções de oxidação-redução
2
Transferases
Transferência de 1 átomo ou grupo entre
moléculas
3
Hidrolases
Reacções de hidrólise
4
Liases
Remoção de um grupo de uma molécula
(sem ser por hidrólise)
5
Isomerases
Reacções de isomerização
6
Ligases ou
sintetases
Reacções de síntese acoplada à hidrólise de 1
molécula de ATP
Lição de Biocatálise
• Nomenclatura e Classificação de Enzimas
Assim, cada enzima recebeu,
DEQB
NOME CURTO (NOME RECOMENDADO),
NOME SISTEMÁTICO (IDENTIFICA A REACÇÃO QUE O ENZIMA CATALISA)
NÚMERO CLASSIFICACÃO
EXEMPLO:
ATP + CREATINA < ------ > ADP + FOSFOCREATINA
NOME CURTO: CREATINA CINASE
NOME SISTEMÁTICO: ATP CREATINA FOSFOTRANSFERASE
NÚMERO CLASSIFICAÇÃO: E.C. 2.7.3.2
Lição de Biocatálise
2. GRUPO das Transferases
7. TRANSFERE GRUPOS FOSFATOS
3. GRUPO AZOTO COMO ACEITADOR
2. Nº DE ORDEM
Nomenclatura e Classificação de Enzimas
DEQB
k1
H2O
E + AX ↔ EAX → E + A + X
k−1
k2
Tabela 1-1- Subclassificação das hidrolases
Lição de Biocatálise
Classificação EC
Tipo de ligação
3.1
3.2
3.3
3.4
3.5
3.6
3.7
3.8
3.9
3.10
3.11
Éster
Glicosilo
Éter
Peptidica
C-N, não peptidica
Ácido anidro
C-C
Haleto, C-X
P-N
S-N
C-P
Quimosina (EC 3.4.23.4)
Nomenclatura e Classificação de Enzimas
GOx
DEQB
β-D-glucose + oxygen
Lição de Biocatálise
D-glucono-1,5-lactone + hydrogen peroxide
Óxido-redutases
DEQB
Lição de Biocatálise
• Nomenclatura e Classificação de Enzimas
DEQB
Aminotransferase
L-aspartate + 2-oxoglutarate
Lição de Biocatálise
oxaloacetate + L-glutamate
Transferases
DEQB
Lição de Biocatálise
• Nomenclatura e Classificação de Enzimas
DEQB
Hidrolase (quimosina ou renina)
κ-casein + water
Lição de Biocatálise
para-κ-casein + caseino macropeptide
Hidrolases
DEQB
Lição de Biocatálise
• Nomenclatura e Classificação de Enzimas
DEQB
Liase (L-histidina amónia-liase ou histidase)
L-histidine
Lição de Biocatálise
urocanate + ammonia
Liases
DEQB
Lição de Biocatálise
• Nomenclatura e Classificação de Enzimas
glucose isomerase
DEQB
α-D-glucopyranose
Lição de Biocatálise
α-D-fructofuranose
Isomerases
DEQB
Lição de Biocatálise
• Nomenclatura e Classificação de Enzimas
Ligase (glutathione sintetase)
DEQB
γ-L-glutamyl-L-cysteine + ATP + glycine
Lição de Biocatálise
glutathione + ADP + phosphate
Ligases ou sintetases
DEQB
Lição de Biocatálise
Medição da actividade enzimática
DEQB
Os enzimas como catalisadores são testados em condições
reaccionais padrão correspondentes, em geral, aos óptimos de pH
e concentração do substrato e co-factores (não limitante), de forma
que a velocidade da reacção medida seja máxima, isto é, de
ordem zero em relação ao substrato.
Por exemplo, o método mais simples de medir a velocidade da
reacção efectuada pelo enzima ou determinar a sua actividade é
medir ao longo do tempo o aparecimento de um produto da
reacção ou desaparecimento de um substrato.
Lição de Biocatálise
Hidrólise da ureia pela urease
DEQB
Lição de Biocatálise
Medição da actividade enzimática
DEQB
Unidade de actividade enzimática (U): é definida pela quantidade
de enzima que a transformação do substrato em produto e produz
1 µmole de produto por minuto nas condições óptimas da reacção.
U = 1 µmole/min
(T=25°C)
KATAL- Quantidade de actividade enzimática que transforma 1
mole de substrato por segundo.
KAT = l mole/s
Lição de Biocatálise
U = l µmole/min
1U=16,67x10-3 KAT
Pureza da preparação enzimática
DEQB
(ACTesp) Actividade específica (U/mg) exprime o número de
unidades de actividade enzimática expresso por miligrama de
proteína da preparação enzimática.
Esta grandeza permite medir a pureza da preparação enzimática
quando se compara o seu valor com o do enzima puro e assim
permite avaliar o grau de pureza obtido num passo de isolamento ou
purificação durante a produção do enzima.
Lição de Biocatálise
Medir concentrações por espectrofotometria
DEQB
Lição de Biocatálise
Medição da actividade de desidrogenases
β-D-galactose dehidrogenase
DEQB
α-D-galactose + NAD+
Lição de Biocatálise
D
-galactono-1,4-lactona + NADH + H+
Desidrogenases + Co-factor
DEQB
Analito
1st Enzima
Alcoóis
-
Formaldeído
-
D-Fructose
D-Glucose / ATP
2
nd
Enzima Co-factor e detecção
Alcool dehidrogenase
+
NAD - λ340nm
+
Formaldeído dehidrogenase NAD - λ340nm
Fructose dehidrogenase Azul Prússia λ- 660nm
+
Glucose quinase
Glucose fosfat
NAD - λ340nm
dehidrogenase
+
D-Glucose
Glucose dehidrogenase
NAD - λ 340nm
+
Glicerol / ATP
Glicerol quinase
Glicerol fosfato
NAD - λ340nm
dehidrogenase
+
Glicerol
Glycerol dehydrogenase
NAD - λ340nm
Glutamate
Glutamato
NADPH λ
- 340nm
dehidrogenase
+
D-hidróxiibutirato
D-hidroxibutirato
NAD - λ340nm
dehidrogenase
+
Malato
Malato dehidrogenase
NAD - λ 340nm
+
Lactato / Piruvato
Lactato dehidrogenase
NAD - λ340nm
+
Sarcosina/ creatinina / creatina
Sarcosina dehidrogenase
NAD - λ340nm
Lição de Biocatálise
Reacções com desidrogenases
DEQB
Lição de Biocatálise
Eléctrodos enzimáticos
DEQB
Clark electrode
Reacções electroquímicas
- +
Cátodo Pt (-600mV) O2 + 4H+ + 4eÁnodo 4Ag + 4Cl-
4AgCl + 4e-
Reacções enzimáticas
e-
Glucose + FAD + H2O
P
t
e-
FADH2 + O2
A
g-
Cl
GOx
Ácido Glucónico + H2O2 Glucose + O
2
Lição de Biocatálise
2H2O
Membrana de teflon,
permeável à glucose e ao O2,
Ácido glucónico + FADH2
FAD + H2O2
DEQB
Actividade da Glucose Oxidase (GOx)
pelo consumo de O2
Act = - d[% O2]dis / dt
Lição de Biocatálise
Actividade da Glucose Oxidase (GOx)
pela formação de H2O2
DEQB
Os ensaios de determinação da actividade da GOx pela formação de
H2O2.
Glucose + FAD + H O
Ácido glucónico + FADH
2
FADH2 + O2
2
FAD + H2O2
Pela combinação com a HRP que oxida por sua vez compostos
fenólicos e percursores de um corante (quinoneimine λmax = 490-520nm)
com ε490nm = 5,56 mM-1 cm-1.
Lição de Biocatálise
Actividade da Glucose Oxidase (GOx)
pela formação de H2O2
DEQB
Neste caso, a actividade de glucose oxidase é definida como número
de micromole de H2O2 formados por minuto determinado
indirectamente pela medição do aparecimento da cor a 490nm
devido á formação do corante (quinoneimine).
Neste caso, a mistura reaccional contêm 0,4mM de 4-AAP, 25mM de
PSA e 2 U/ml de HRP em tampão fosfato 0,1M pH 7,0. Caso típico é
medir a actividade da GOx pela adição de 25µl da amostra da
preparação enzimática a 950 µl da mistura reaccional ao qual foi
adicionado previamente 25µl de uma solução de glucose a 10% em
água.
Lição de Biocatálise
Actividade de esterases pela hidrólise de
substratos sintéticos
DEQB
O p-nitrofenol absorve a 400 nm (cor intensa amarela), o que
permite quantificar a quantidade de produto presente na mistura
reaccional.
Lição de Biocatálise
Actividade da cutinase pela hidrólise de
substratos sintéticos
DEQB
Os ensaios de determinação da actividade da cutinase são
efectuados a partir da hidrólise do p-nitrofenilbutirato (pNPB) em pnitrofenol (pNP).
pNPB + H2O Cutinase
pNP + ácido butírico
Quando efectuado em descontínuo, numa cuvette de 1 ml, o ensaio
caracteriza-se pela adição de 20µl de amostra da preparação de
enzima a 980µl de mistura reaccional e a reacção enzimática deve
ocorrer à temperatura ~ 22ºC.
Lição de Biocatálise
Actividade da cutinase pela hidrólise de
substratos sintéticos
DEQB
A actividade enzimática na mistura reaccional é calculada a partir da
derivação da lei de Beer-Lambert em ordem ao tempo, de acordo com a
seguinte equação:
= d [ pNP ] × V = dAbs(400nm ) × 1 × V ×103
Act (U ) = U µmol
ml
ml
ml. min
dt
Ve
dt
ε × l Ve
Em que:
- U é a unidade de actividade enzimática definida como a
concentração de enzima necessário para produzir um µmole de pNP por
minuto;
- [pNP] é a concentração milimolar de p-nitrofenol;
- V é o volume reaccional em ml;
- Ve é o volume do extracto enzimático em ml;
- Abs(400nm) é a absorvância a 400 nm;
- ε é o coeficiente de extinção molar (ε=1,84x104 M.cm-1);
- l é a espessura da célula (l=1cm).
Este método é válido para variações de absorvância (dAbs/dt) entre
0.1 e 0.9 min-1.
Lição de Biocatálise