Carlos Capela
ÁCIDOS NUCLEICOS
ÁCIDOS NUCLEICOS ................................................ 1
A.
INTRODUÇÃO ......................................................... 4
1.
OS NUCLEÓTIDOS ......................................................... 4
1.a.
Estrutura dos Nucleótidos....................................................... 4
1.A.I.
1.A.II.
1.A.III.
1.b.
2.
AS BASES NITROGENADAS .............................................................. 4
AS PENTOSES ................................................................................... 4
O FOSFATO ...................................................................................... 4
Principais funções dos Nucleótidos........................................ 4
O DNA........................................................................... 4
2.a.
A Ligação Fosfodiéster ........................................................... 4
2.b.
A Estrutura do DNA................................................................ 4
1.B.I.
1.B.II.
1.B.III.
2.c.
Organização do Material Genético Eucariótico .................... 4
2.C.I.
2.C.II.
2.C.III.
3.
ESTRUTURA PRIMÁRIA .................................................................... 4
ESTRUTURA SECUNDÁRIA – A DUPLA HÉLICE ............................... 4
ESTRUTURA TERCIÁRIA................................................................... 4
AS HISTONAS ................................................................................... 4
OS NUCLEOSSOMAS ......................................................................... 4
NÍVEIS DE ORGANIZAÇÃO DO DNA................................................. 4
O RNA........................................................................... 4
3.a.
A Estrutura do RNA................................................................ 4
3.A.I.
3.A.II.
3.A.III.
3.b.
ESTRUTURA PRIMÁRIA.................................................................... 4
ESTRUTURA SECUNDÁRIA ............................................................... 4
ESTRUTURA TERCIÁRIA................................................................... 4
Tipos de RNA........................................................................... 4
3.B.I.
3.B.II.
3.B.III.
RNA RIBOSSÓMICO – RRNA........................................................... 4
RNA DE TRANSFERÊNCIA – TRNA ................................................. 4
RNA MENSAGEIRO – MRNA ........................................................... 4
Ácidos Nucleicos página 1 de 60
Carlos Capela
B.
METABOLISMO DOS NUCLEÓTIDOS ..................... 4
1.
BIOSSÍNTESE DOS NUCLEÓTIDOS................................. 4
1.a.
Síntese dos nucleótidos com purinas...................................... 4
1.A.I.
1.A.II.
1.A.III.
1.A.IV.
1.b.
Síntese dos nucleótidos com pirimidina ................................. 4
1.B.I.
1.B.II.
1.B.III.
1.c.
FORMAÇÃO DA DTMP..................................................................... 4
Síntese das Coenzimas Nucleotídicas..................................... 4
1.D.I.
1.D.II.
1.D.III.
1.e.
SÍNTESE DO UMP ............................................................................ 4
SÍNTESE DE CMP............................................................................. 4
REGULAÇÃO DA SÍNTESE ................................................................ 4
Formação dos desoxirribonucleótidos ................................... 4
1.C.I.
1.d.
SÍNTESE DE IMP.............................................................................. 4
SÍNTESE DE AMP E GMP................................................................ 4
SÍNTESE DOS NUCLEÓSIDOS DI- E TRIFOSFATO PÚRICOS................ 4
REGULAÇÃO DA SÍNTESE ................................................................. 4
NAD+ E NADP+ ............................................................................... 4
FAD ................................................................................................. 4
COA ................................................................................................. 4
Inibidores ................................................................................. 4
1.E.I.
1.E.II.
SULFAS ............................................................................................. 4
METOTREXATO ............................................................................... 4
2. CATABOLISMO DOS ÁCIDOS NUCLEICOS E
NUCLEÓTIDOS ........................................................................ 4
2.a. Catabolismo dos Ácidos Nucleicos, Nucleótidos e
Nucleósidos ........................................................................................ 4
2.b.
Catabolismo das bases púricas ............................................... 4
2.c.
Catabolismo das bases pirimídicas ......................................... 4
3.
DOENÇAS ....................................................................... 4
3.a.
Alterações do metabolismo das purinas ................................. 4
3.A.I.
3.A.II.
3.A.III.
A GOTA ............................................................................................ 4
A SÍNDROME DE LESCH-NYHAN ..................................................... 4
IMUNODEFICIÊNCIA ASSOCIADA A DEFEITOS NA DEGRADAÇÃO DE
NUCLEÓTIDOS DE PURINA ................................................................................ 4
3.b.
Alterações do metabolismo das pirimidinas........................... 4
3.B.I.
ACIDÚRIA ORÓTICA HEREDITÁRIA ................................................ 4
Ácidos Nucleicos página 2 de 60
Carlos Capela
C.
BIOSSÍNTESE DOS ÁCIDOS NUCLEICOS E
TRANSFERÊNCIA DA INFORMAÇÃO GENÉTICA. .......... 4
1.
REPLICAÇÃO DO DNA.................................................. 4
1.a.
1.b.
1.c.
2.
Iniciação .................................................................................. 4
Elongação ................................................................................ 4
Terminação.............................................................................. 4
TRANSCRIÇÃO DO DNA................................................ 4
2.a.
2.b.
2.c.
2.d.
3.
Iniciação .................................................................................. 4
Elongação ................................................................................ 4
Terminação.............................................................................. 4
Maturação do mRNA ............................................................... 4
TRADUÇÃO – A SÍNTESE DE PROTEÍNAS ..................... 4
3.a.
Introdução ............................................................................... 4
3.b.
O código genético .................................................................... 4
3.B.I.
3.c.
Componentes necessários à tradução..................................... 4
3.C.I.
3.C.II.
3.C.III.
3.C.IV.
3.C.V.
3.C.VI.
3.d.
CARACTERÍSTICAS DO CÓDIGO GENÉTICO ..................................... 4
OS AMINOÁCIDOS: .......................................................................... 4
O RNA DE TRANSFERÊNCIA:.......................................................... 4
O RNA MENSAGEIRO: .................................................................... 4
RIBOSSOMAS: .................................................................................. 4
FACTORES PROTEICOS: .................................................................. 4
FONTES DE ENERGIA ....................................................................... 4
Etapas da Biossíntese Proteica:.............................................. 4
3.D.I.
3.D.II.
3.D.III.
3.D.IV.
3.D.V.
ACTIVAÇÃO DOS AMINOÁCIDOS:.................................................... 4
INICIAÇÃO DA CADEIA: ................................................................... 4
ELONGAÇÃO DA CADEIA:................................................................ 4
TERMINAÇÃO DA CADEIA POLIPEPTÍDICA:.................................... 4
MODIFICAÇÕES PÓS-TRANSLACIONAIS ......................................... 4
Ácidos Nucleicos página 3 de 60
Carlos Capela
A. INTRODUÇÃO
São as moléculas com a função de armazenamento e expressão da informação
genética.
Cromossoma
Genes
Dupla Hélice de DNA
Sequência
Exões
reguladora
Intrões
Transcrição do DNA
Splicing do RNA
RNA
Tradução
Proteína
Existem basicamente 2 tipos de ácidos nucleicos:
O Ácido Desoxirribonucleico – DNA
O Ácido Ribonucleico – RNA
Os ácidos nucleicos são macromoléculas formadas pela ligação tipo fosfodiéster
entre 5 nucleótidos diferentes. Podem-se encontrar tanto na forma livre como
associados a proteínas (histonas e protaminas).
Ácidos Nucleicos página 4 de 60
Carlos Capela
1.OS NUCLEÓTIDOS
São as unidades fundamentais dos ácidos nucleicos. Ligam-se uns aos outros
através de ligações fosfodiéster, formando cadeias muito longas com milhões de
resíduos de comprimento.
Além de participarem da estrutura dos ácidos nucleicos, os nucleótidos actuam
também como componentes na estrutura de coenzimas importantes no metabolismo
oxidativo da célula, e como forma de energia química – ATP, por exemplo.
Actuam ainda como activadores e inibidores importantes em várias vias do
metabolismo intermediário da célula.
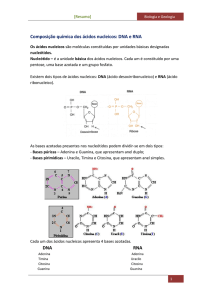
1.a. Estrutura dos Nucleótidos
Os nucleótidos são moléculas formadas por:
•
Uma pentose;
•
Uma base nitrogenada;
•
Um ou mais grupos fosfato.
Ao conjunto formado pela pentose e pela base azotada dá-se
o nome de Nucleósido.
1.a.i. AS BASES NITROGENADAS
Pertencem a 2 famílias de compostos, e são 5 no total:
•
Bases Púricas, ou Purinas (anel duplo): Adenina e Guanina;
•
Bases Pirimídicas, ou Pirimidinas (anel simples): Citosina, Timina e Uracilo.
Tanto o DNA como o RNA possuem as mesmas bases púricas, e a citosina como
base pirimídica.
A timina existe apenas no DNA, e no RNA, é substituída pela uracilo – que possui
um grupo metil a menos.
Em alguns tipos de DNA virais e no RNA de transferência podem aparecer bases
incomuns.
As bases azotadas unem-se ao carbono 1 das pentoses por ligações covalentes.
Ácidos Nucleicos página 5 de 60
Carlos Capela
DNA
RNA
1.a.ii. AS PENTOSES
A adição de uma pentose a uma base nitrogenada produz um nucleósido. Os
nucleósidos de A, C, G, T e U são denominados, respectivamente, Adenosina, Citosina,
Guanosina, Timidina e Uridina.
Se o açúcar em questão é a Ribose, temos um ribonucleósido, característico do
RNA.
Se o açúcar é a desoxirribose – 1 hidroxilo (-OH) a menos em C2 – temos um
desoxirribonucleósido, característico do DNA.
A ligação com a base nitrogenada ocorre sempre através do hidroxilo do carbono
anomérico da pentose.
Ácidos Nucleicos página 6 de 60
Carlos Capela
1.a.iii. O FOSFATO
A adição de um ou mais radicais fosfato à pentose, através de uma
ligação tipo éster com o hidroxilo do carbono 5 da mesma, dá origem aos
Nucleótidos.
Os grupos fosfato são responsáveis pelas cargas negativas dos
nucleótidos e dos ácidos nucleicos.
A adição do segundo ou terceiro grupo fosfato ocorre em sequência, dando origem
aos nucleótidos di e trifosfatados. A função principal destes nucleótidos di e
trifosfatados é armazenar energia.
1.b. Principais funções dos Nucleótidos
•
Metabolismo energético – os nucleótidos entram na constituição do ATP, que
é a principal forma de energia química para as células. Este está envolvido na
contracção muscular, no transporte activo e na manutenção de gradientes
iónicos;
•
Unidades monoméricas dos Ácidos Nucleicos;
•
Mediadores fisiológicos – nucleósidos e nucleótidos servem como mediadores
fisiológicos de processos metabólicos chave, como por exemplo, a adenosina é
importante no controlo do fluxo sanguíneo;
•
Função de Percursor – alguns nucleótidos são percursores para a formação de
cofactores;
•
Componente de Coenzimas – o AMP é um componente das coenzimas NAD+,
NADP+, FAD, suas formas reduzidas, e coenzima A.
•
Intermediários
activados
–
os
nucleótidos
também
servem
como
transportadores de intermediários activados necessários para várias reacções.
Por ex: a UDP-glicose é um intermediário chave para a síntese de glicogénio e
de glicoproteínas;
•
Efectores Alostéricos – muitas das etapas reguladoras das vias metabólicas são
controladas pelas concentrações intracelulares de nucleótidos.
Ácidos Nucleicos página 7 de 60
Carlos Capela
2.O DNA
Está presente no núcleo das células eucarióticas, nas mitocôndrias, nos cloroplastos,
e no citosol das células procarióticas.
Nas células germinativas e no óvulo fertilizado, dirige todo o desenvolvimento do
organismo, a partir da informação contida na sua estrutura. É duplicado cada vez que a
célula somática se divide.
O DNA é um polidesoxirribonucleótido formado por milhares de nucleótidos
ligados entre si através de ligações fosfodiéster 3’-5’.
2.a. A Ligação Fosfodiéster
Ocorre entre o fosfato do carbono 5 da pentose de um nucleótido e o hidroxilo do
carbono 3 da pentose do nucleótido seguinte. A cadeia resultante é bastante polar, e
possui:
•
Uma extremidade 5’ Fosfato, do carbono 5 da pentose, livre;
•
Uma extremidade 3’ Hidroxilo, do carbono 3 da pentose, livre
Por convenção, as bases de uma sequência são sempre descritas da extremidade 5’
para a extremidade 3’.
As ligações fosfodiéster podem ser quebradas enzimaticamente por enzimas
chamadas Nucleases, que se dividem em:
•
Endonucleases – quebram ligações no meio da molécula;
•
Exonucleases – quebram ligações nas extremidades da molécula.
2.b. A Estrutura do DNA
1.b.i. ESTRUTURA PRIMÁRIA
A estrutura primária é a sequência de nucleótidos que o formam. A sequência das
bases é característica de um ácido nucleico, pois é nela que reside a informação
genética.
Ácidos Nucleicos página 8 de 60
Carlos Capela
1.b.ii. ESTRUTURA SECUNDÁRIA – A DUPLA HÉLICE
Na dupla hélice do DNA, descrita pela primeira vez por Watson e Crick, as cadeias
da molécula dobram-se em torno de um eixo comum e de modo antiparalelo – a
extremidade 5’ de uma cadeia está emparelhada com a extremidade 3’ da outra cadeia.
No tipo mais comum de hélice – “B” – o esqueleto hidrofílico dos fosfatos e
pentoses fica na parte externa, enquanto que as bases hidrofóbicas, fixadas a este
esqueleto, ficam no lado interno da estrutura.
Existe uma complementaridade de Bases entre as cadeias da molécula do DNA.
Assim, temos sempre emparelhadas:
•
Adenina com Timina → A-T
•
Citosina com Guanina → C-G
As bases mantém-se emparelhadas devido à formação de pontes de hidrogénio, 2
entre “A” e “T” e 3 entre “C” e “G”.
Ácidos Nucleicos página 9 de 60
Carlos Capela
Existem 3 formas estruturais de DNA:
A forma “B” – descrita por Watson e Crick em 1953 e já citada acima, é a forma
mais comum; a hélice tem rotação para a direita, com 10 resíduos por volta e com
planos de bases perpendiculares ao eixo helicoidal.
A forma “A” – Obtida pela desidratação moderada da forma “B”, também possui
rotação para a direita, mas possui 11 resíduos por volta e as bases estão num ângulo de
20 graus em relação ao eixo helicoidal.
A forma “Z” – A hélice nesta forma possui rotação para a esquerda e contém cerca
de 12 resíduos por volta.
A transição entre as formas de DNA pode desempenhar um papel importante na
regulação da expressão genética.
1.b.iii. ESTRUTURA TERCIÁRIA
A molécula de DNA pode sofrer enrolamento no sentido da sua progressão e
também dobras sobre si própria – embora de uma forma menos nítida do que as
proteínas quando adquirem a estrutura terciária.
Essas conformações designam-se por “supercoiling” que se poderá traduzir por
sobre-enrolamento ou superenrolamento.
A estrutura terciária do DNA encontra-se nos eucariotas, nos seus nucleossomas,
situação em que o enrolamento se faz em torno das histonas.
Ácidos Nucleicos página 10 de 60
Carlos Capela
O DNA pode formar superenrolamento para a direita (negativo) ou para a esquerda
(positivo). O superenrolamento negativo comunica um “stress” torcional ao DNA que
favorece a separação das cadeias.
Nas células existem enzimas denominadas Topoisomerases que são capazes de
conferir superenrolamento ao DNA (DNA-girase) e também de o desenrolar
(Topoisomerase I).
2.c. Organização do Material Genético Eucariótico
O DNA total de uma célula mede em média 1 metro de comprimento. Para que um
volume tão grande de material genético esteja compreendido dentro do núcleo da
célula, o DNA interage com um grande número de proteínas com o objectivo da sua
compactação. Estas proteínas exercem funções importantes na organização e
mobilização deste material genético
2.c.i. AS HISTONAS
As histonas são pequenas proteínas básicas, ricas em lisina e arginina, e carregadas
positivamente em pH fisiológico, às quais se associa a molécula do DNA.
As suas cargas positivas, em associação com o catião Mg2+, facilitam esta ligação
com o esqueleto negativo do DNA e estabilizam o conjunto.
Existem 5 classes de histonas: H1, H2, H2B, H3 e H4.
2.c.ii. OS NUCLEOSSOMAS
São considerados as unidades estruturais dos
cromossomas.
São formados por 8 moléculas de histonas: 2
H2, 2 H2B, 2 H3 e 2 H4, formando um octâmero
regular sobre o qual se enrola a dupla cadeia do
DNA, cerca de 2 voltas (146 nucleótidos por
nucleossoma).
Nucleossoma
Os nucleossomas são ligados entre si por
Ácidos Nucleicos página 11 de 60
Carlos Capela
segmentos de DNA “ligante” de aproximadamente 50 nucleótidos de comprimento,
formando os polinucleossomas, ou nucleofilamentos.
Após vários níveis de organização espacial,
ancorados por vários tipos de proteínas, chegamos à
estrutura final dos cromossomas.
A histona H1 não participa da estrutura dos
nucleossomas, mas sim liga-se ao DNA “ligante” e
participa do processo de compactação das estruturas,
formando os solenóides.
Ácidos Nucleicos página 12 de 60
Carlos Capela
2.c.iii. NÍVEIS DE ORGANIZAÇÃO DO DNA
Ácidos Nucleicos página 13 de 60
Carlos Capela
3.O RNA
O RNA é criado a partir de um molde de DNA e é utilizado na expressão da
informação genética. A síntese de uma molécula de RNA a partir de um molde de DNA
chama-se transcrição. Nesta transcrição, modificações podem ocorrer sobre a molécula
de RNA transcrita, convertendo-a de uma cópia fiel numa cópia funcional do DNA.
Em relação ao DNA, o RNA possui uracilo no lugar da timina na sequência de
bases. A pentose do RNA é a Ribose.
O RNA é formado por uma cadeia única, com eventual emparelhamento de bases
intracadeia. A molécula do RNA é muito menor que a do DNA.
3.a. A Estrutura do RNA
3.a.i. ESTRUTURA PRIMÁRIA
Tal como no DNA, a estrutura primária é a sequência de nucleótidos que o formam.
3.a.ii. ESTRUTURA SECUNDÁRIA
Contrariamente ao que acontece com a molécula de DNA, o RNA é formado por
uma única cadeia polinucleotídica. No entanto, esta cadeia simples pode, em
determinadas zonas, dobrar-se sobre si própria, devido à formação de pontes de
hidrogénio entre as bases complementares.
3.a.iii. ESTRUTURA TERCIÁRIA
Além da estrutura secundária por pares de bases, as moléculas de RNA também
formam outras pontes de Hidrogénio, gerando a estrutura terciária.
3.b. Tipos de RNA
Existem 3 tipos fundamentais de RNA, cada um com características estruturais e
funcionais próprias:
Ácidos Nucleicos página 14 de 60
Carlos Capela
3.b.i. RNA RIBOSSÓMICO – RRNA
O rRNA é sintetizado no nucléolo, uma vez que é nessa zona que se encontram os
seus genes codificadores. Após a sua síntese vai agregar-se a proteínas específicas
sintetizadas previamente no citoplasma, dando origem às ribonucleoproteinas que
entram na constituição dos ribossomas, os organelos responsáveis pela síntese proteica.
Corresponde a cerca de 80% do total de RNA da célula.
3.b.ii. RNA DE TRANSFERÊNCIA – TRNA
É a menor molécula dos 3 tipos de RNA. Inclui na sua composição uma sequência
de 3 bases designadas anticodão e tem como função estabelecer a correspondência
entre os aminoácidos e os codões do mRNA, de modo a permitir a tradução da
informação codificada no mRNA em proteínas.
Possui um extenso emparelhamento de bases intracadeia, actuando no
posicionamento dos aminoácidos na sequência prevista pelo código genético, no
momento da síntese proteica. Está ligado de forma específica a cada um dos 20
aminoácidos encontrados nas proteínas (proteicos comuns).
Corresponde a cerca de 15% do RNA total da célula.
Estrutura terciária
Estrutura
secundária
Ácidos Nucleicos página 15 de 60
Carlos Capela
3.b.iii. RNA MENSAGEIRO – MRNA
É a molécula resultante da transcrição do DNA. Actua como transportador da
informação genética do núcleo da célula eucariótica para o citosol, onde ocorrerá a
biossíntese proteica, através da leitura de 3 bases – o codão.
Corresponde a apenas 5% do total de RNA da célula
Ácidos Nucleicos página 16 de 60
Carlos Capela
B. METABOLISMO DOS
NUCLEÓTIDOS
1.BIOSSÍNTESE DOS NUCLEÓTIDOS
Embora os animais ingiram ácidos nucleicos e consequentemente nucleótidos, nos
seus alimentos, a sua sobrevivência não depende da absorção e utilização desses
compostos, uma vez que o Homem e a maioria dos vertebrados podem sintetizar de
novo nucleótidos de purina e pirimidina. Também conseguimos reutilizar aquelas de
que já dispomos no nosso organismo, e que assim sofrem um “turnover” elevado.
Existe um grande turnover do RNA e dos nucleótidos, mas não de DNA. O
reaproveitamento de purinas, pirimidinas e nucleósidos envolve a sua conversão a
nucleótidos, evitando assim à célula um gasto desnecessário de energia.
Por outro lado, existe uma interconversão de nucleótidos de forma a manter uma
concentração apropriada de percursores para a síntese de RNA, DNA e outros
intermediários nucleotídicos.
A síntese de novo tem como fontes: átomos de carbono, azoto e oxigénio, os
aminoácidos glutamina, glicina e ácido aspártico, o CO2, a ribose-5-fosfato e o
amoníaco (NH4).
A síntese de novo de nucleótidos implica a síntese das pentoses (pela vias das
pentoses fosfato) e das bases azotadas.
As purinas e pirimidinas que não são reutilizadas são catabolisadas e os produtos
resultantes excretados. Nos primatas, o produto de excreção final é o ácido úrico,
enquanto que noutros mamíferos este é catalisado em alantoína.
Ácidos Nucleicos página 17 de 60
Carlos Capela
1.a. Síntese dos nucleótidos com purinas
As purinas são formadas a partir dos seguintes percursores:
1. Glicina – C4, C5 e N7.
2. Formilo (combinado ao THF) – C2 e C8.
3. Glutamina – fornece o N para os N3 e N9.
4. Ácido Aspártico – N1.
5. CO2 – C6.
Ácidos Nucleicos página 18 de 60
Carlos Capela
A síntese faz-se por um grande número de etapas. O primeiro nucleótido a formarse é o IMP, formando-se os outros a partir dele.
1.a.i. SÍNTESE DE IMP
As principais etapas são as seguintes:
1. Formação da forma activada da ribose – o 5-fosforribosil-1-pirofosfato
(PRPP) – por combinação da ribose-5-fosfato com o ATP. Esta reacção
catalisada pela Ribose-5-fosfato Pirofosfocinase, é inibida pelo ADP, GDP,
AMP e ácido 2,3-Bisfosfoglicérico. O ião Mg2+ e o grupo fosfato actuam
como activadores.
2. Formação da 5-fosforribosilamina, sendo o NH2 cedido pela glutamina.
Ácidos Nucleicos página 19 de 60
Carlos Capela
3. Formação do ribótido de glicinamida, por combinação com a glicina.
4. Transferência de um carbono, formando-se o ribótido da formilglicinamida.
O formilo é transportado pelo ácido tetrahidrofólico (THF).
5. Amidinação pela glutamina, formando-se o ribótido da formilglicinamidina.
A reacção faz-se na presença de ATP.
6. Ciclização em ribótido do 5-amino-amidazol, com gasto de ATP.
7. Carboxilação em ribótido do ácido 5-amino-imidazol carboxílico.
8. Formação de uma amida por combinação com o ácido aspártico, formandose o ribótido do 5-amino-4-imidazol-succino carboximida.
9. Transformação em ribótido da 5-aminoimidazol-carboxamida por perda de
ácido fumárico.
10. Formilação
em
ribótido
da
formil
aminoimidazol-carboxamida
e
desidratação deste em IMP.
Ácidos Nucleicos página 20 de 60
Carlos Capela
1.a.ii. SÍNTESE DE AMP E GMP
O IMP combinando-se com ácido aspártico formando ácido adenilo-succínico. A
energia para a síntese é fornecida pela hidrólise do GTP. O ácido adenilo-succínico
hidrolisa-se, em seguida, em ácido fumárico e AMP.
Para a síntese do GMP terá que ser introduzido um átomo de azoto no núcleo de
purina do IMP. O IMP é, inicialmente, oxidado em XMP e, depois, este é aminado pela
glutamina em GMP.
1.a.iii. SÍNTESE DOS NUCLEÓSIDOS DI- E TRIFOSFATO PÚRICOS
Os mononucleótidos AMP e GMP são convertidos em nucleósidos difosfato (ADP
e GDP) pela transferência de um grupo fosfato de ATP.
GMP/AMP + ATP → GDP/ADP + ADP
Nucleósido-5-monofosfato cinase
Ácidos Nucleicos página 21 de 60
Carlos Capela
O GDP é então convertido em GTP com gasto de outro ATP.
GDP + ATP → GTP + ADP
Nucleósido-5-difosfato cinase
A conversão do ADP em ATP é feita maioritariamente por fosforilação oxidativa,
mas também por reacções de glicólise e ciclo de Krebs.
O ATP e o GTP são os percursores púricos necessários para a síntese dos ácidos
nucleicos.
1.a.iv. REGULAÇÃO DA SÍNTESE
A síntese de nucleótidos de purina é controlada a vários níveis:
•
Os níveis de PRPP são regulados pelo AMP, GMP e IMP, do mesmo modo que
a conversão do PRPP em 5-fosforribosilamina.
•
O AMP e GMP regulam a sua própria síntese a partir do IMP, ou seja, uma
elevada concentração de AMP inibe a sua síntese, e uma concentração elevada
de GMP inibe a do GMP.
´
Ácidos Nucleicos página 22 de 60
Carlos Capela
1.b. Síntese dos nucleótidos com pirimidina
Os percursores do núcleo pirimídico são C4, C5, C6 e N1 do ácido aspártico, N3 da
glutamina e C2 do CO2.
Ácidos Nucleicos página 23 de 60
Carlos Capela
1.b.i. SÍNTESE DO UMP
Contrariamente à síntese de novo de nucleótidos de purina, este processo é mais
simples devido à estrutura anelar simples das pirimidinas.
1. Formação do Carbamil-fosfato (uma forma activada de azoto) através da
condensação de CO2, amoníaco e ATP, numa reacção catalisada pela Carbamil
fosfato sintetase II, tendo como coenzima a Biotina.
2. O Carbamil-fosfato, reage com o ácido aspártico formando o ácido carbamilaspártico.
3. Este ácido desidrata-se dando ácido di-hidro-orótico que é desidrogenado por
um enzima com NAD+ dando ácido orótico.
4. O ácido orótico recebe a ribose do PRPP, dando ácido orotidílico.
5. Seguidamente, o ácido orotidílico descarboxila-se em UMP.
Ácidos Nucleicos página 24 de 60
Carlos Capela
1.b.ii. SÍNTESE DE CMP
O UMP para se converter em CMP necessita de se transformar, primeiro, em UTP
(pela acção de cinases). Este, aminando-se pela glutamina na presença de ATP, origina
o CTP.
A formação e a presença de TMP é bastante rara.
1.b.iii. REGULAÇÃO DA SÍNTESE
Devido, ao seu papel de regulador
alostérico da Carbamil fosfato sintetase II, o
CTP intervêm decisivamente no controlo do
metabolismo dos nucleótidos pirimídicos.
Ácidos Nucleicos página 25 de 60
Carlos Capela
1.c. Formação dos desoxirribonucleótidos
Os desoxirribonucleótidos são formados a partir da redução do anel de ribose pela
Ribonucleósido difosfato redutase que catalisa a redução dos ribonucleótidos
difosfatados.
Esta enzima utiliza como dador de hidrogénio na redução da ribose uma proteína, a
Tioredoxina, com SH quando reduzida e S-S quando oxidada. A tioredoxina oxidada
passa a reduzida pela acção do NADPH, reacção catalisada pela tioredoxina redutase.
Desta forma, há uma manutenção de desoxirribonucleótidos necessários para a
síntese de DNA.
ADP, GDP, CDP e UDP são catabolisados respectivamente em dADP, dGDP,
dCDP e dUDP.
1.c.i. FORMAÇÃO DA DTMP
O uracilo não é componente do DNA porque este contêm timina (uracilo metilado).
O dUMP é metilado pelo N5-N10-metileno-THF que actua como dador de electrões e de
carbono, reacção catalisada pela Timidilato sintetase.
Ácidos Nucleicos página 26 de 60
Carlos Capela
Ácidos Nucleicos página 27 de 60
Carlos Capela
1.d. Síntese das Coenzimas Nucleotídicas
Os coenzimas FAD, NAD+, NADP+ e Coenzima A têm uma base nucleotídica.
Cada um destes coenzimas tem um resíduo de AMP como parte da molécula que, no
entanto, não está directamente envolvido na parte funcional da molécula.
1.d.i. NAD+ E NADP+
A síntese de NAD+ e NADP+ inicia-se a partir da condensação de Nicotinamida
com PRPP. A nicotinamida ou é ingerida ou deriva do Triptofano. A carência de
Nicotinamida provoca o Pelagra.
O PRPP reage com a nicotinamida para formar o nicotinamida-mononucleótido
que, recebendo um AMP do ATP, se transforma em NAD+. Este pode ser fosforilado
em NADP+, pela NAD+ quinase.
Ácidos Nucleicos página 28 de 60
Carlos Capela
O NAD+ e o NADP+ são transportadores de equivalentes redutores. O NAD+ é uma
coenzima nas reacções (geralmente catabólicas) em que os equivalentes redutores são
utilizados na síntese de ATP e actua como aceitador de 2 e- e um protão H+,
provenientes de um dador ou substrato que é oxidado. O NADP+ é utilizado em
reacções (geralmente anabólicas) em que os equivalentes redutores serão aproveitados
em reacções de síntese e actua de forma análoga ao NAD+.
1.d.ii. FAD
A síntese do FAD faz-se a partir da vitamina B2 (Riboflavina) que sofre 2
fosforilações pelo ATP.
Ácidos Nucleicos página 29 de 60
Carlos Capela
1.d.iii. COA
A síntese de CoA faz-se a partir de uma outra vitamina do grupo B, o ácido
pantoténico.
Ácidos Nucleicos página 30 de 60
Carlos Capela
1.e. Inibidores
Actualmente, muitos fármacos são fabricados para inibirem uma enzima específica,
numa via metabólica. Esta aplicação é mais facilmente observada com fármacos
antivirais, antibacterianas e antitumorais, que são administradas ao paciente, sob
condições de toxicidade limitada. Tal toxicidade é, frequentemente, inevitável porque,
existem poucas vias metabólicas críticas que são inerentes/particulares ao tumor, ao
vírus ou à bactéria. Assim, os fármacos que destroem estes organismos, frequentemente
matarão as células hospedeiras.
Uma característica de que se pode tirar vantagem é o tempo comparativamente mais
curto de geração, dos organismos indesejáveis. Eles são muito mais sensíveis aos
antimetabólitos e, em particular, àqueles que inibem as enzimas envolvidos na
replicação. Antimetabólitos são compostos com alguma diferença estrutural do
substrato natural. Como exemplo temos:
1.e.i. SULFAS
A quimioterapia moderna teve o seu início com compostos de fórmula geral R-SO2NHR’. A Sulfanilamida, o membro mais simples da classe, é um agente anti-bacteriano
porque compete com o ácido p-aminobenzóico (PABA), que é necessário para o
crescimento bacteriano.
A bactéria não pode absorver ácido fólico (uma vitamina essencial para o
hospedeiro), por isso deve sintetizá-la. Como a sulfanilamida é um análogo estrutural
do ácido p-aminobenzóico, a dihidropteroato sintetase bacteriana é levada a fazer um
intermediário contendo sulfanilamida, que não pode ser convertida a ácido fólico.
Assim, a bactéria é privada de ácido fólico necessário para o crescimento e divisão.
Como o homem requer ácido fólico da dieta diária, a sulfanilamida não é perigosa
nas doses que destroem a bactéria.
Ácidos Nucleicos página 31 de 60
Carlos Capela
1.e.ii. METOTREXATO
A biossíntese de purinas, bases heterocíclicas necessárias para a síntese de RNA e
DNA, requer ácido fólico, que serve como uma coenzima na transferência de unidades
de carbono de vários aminoácidos dadores.
Metotrexato (ácido 4-amino-N10-metil fólico) é um análogo estrutural do ácido
fólico. O seu mecanismo de acção é baseado na competição com o dihidrofolato (DHF)
pela dihidrofolato redutase. Este liga-se 1000 vezes mais fortemente que o substrato
natural e é um potente inibidor competitivo da enzima. A síntese de TMP para na
presença de metotrexato devido à falha na reacção de transferência de um carbono (a
Timidilato sintetase é particularmente sensível à presença de N5-N10-metileno-THF).
Como a divisão celular depende de TMP e de outros nucleótidos, a célula não se pode
multiplicar.
Um problema é que as células humanas que se dividem rapidamente, como as da
medula óssea e da mucosa intestinal, são sensíveis à droga pelas mesmas razões.
Também, o uso prolongado leva a uma amplificação do gene para a dihidrofolato
redutase, com níveis aumentados da enzima e crescimento preferencial de células
resistentes ao metotrexato.
Aminopterina (ácido 4-amino fólico) funciona de maneira similar ao metotrexato,
uma vez que é estruturalmente análoga ao ácido fólico, inibindo, assim, a dihidrofolato
redutase.
Ácidos Nucleicos página 32 de 60
Carlos Capela
2.CATABOLISMO DOS ÁCIDOS NUCLEICOS
E NUCLEÓTIDOS
2.a. Catabolismo
dos
Ácidos
Nucleótidos e Nucleósidos
Nucleicos,
As Nucleoproteínas são, primeiramente, cindidas por proteases em proteínas e
ácidos nucleicos. A digestão de ácidos nucleicos e o seu catabolismo tecidular faz-se
por vias relativamente semelhantes.
O processo inicial é a despolimerização por nucleases em nucleótidos. Estas são
ribonucleases ou desoxirribonucleases.
Os nucleótidos resultantes são desfosforilados por nucleotidases. As nucleotidases
cindem os nucleótidos formados por fosforólise, em pentose-1-fosfato e respectiva
base.
Ácidos Nucleicos página 33 de 60
Carlos Capela
2.b. Catabolismo das bases púricas
O catabolismo das purinas leva no homem à formação de ácido úrico.
O catabolismo faz-se por desaminação das bases púricas livres ou das bases
integradas em nucleótidos ou nucleósidos, seguindo-se uma oxidação dos produtos
formados.
Por desaminação, os nucleótidos de adenina origina a hipoxantina, enquanto os
nucleótidos de guanina a xantina. A hipoxantina é oxidada a xantina, e esta, pela
xantina oxidase, oxida-se a ácido úrico, um produto final exclusivo da degradação da
purina em seres humanos.
A xantina oxidase é uma enzima que tem, como, coenzima, o FAD e contém ferro e
molibdénio, e requer oxigénio como substrato.
Ácidos Nucleicos página 34 de 60
Carlos Capela
Como o ácido úrico não é muito solúvel em meio aquoso, existem condições
clínicas nas quais níveis elevados de ácido úrico resultam em depósito de cristais de
urato de sódio, principalmente nas articulações.
Na maior parte das espécies, o ácido úrico não é o produto final da excreção do
metabolismo púrico sendo descarboxilado oxidativamente em alantoína pela acção da
uricase. Esta enzima encontra-se no fígado e rins dos mamíferos mas não se encontra
no Homem e nos primatas.
Ácidos Nucleicos página 35 de 60
Carlos Capela
2.c. Catabolismo das bases pirimídicas
O catabolismo das pirimidinas, que ocorre principalmente no fígado, leva a
produtos finais altamente solúveis, o que contrasta com a produção a partir das purinas
de ácido úrico e de urato de sódio que são pouco solúveis.
No que se refere à citosina e uracilo, estes são catabolisados em β-alanina, a qual
pode ser eliminada pela urina, incorporada no ácido pantoténico ou catabolisada a ácido
acético.
No que se refere à timina esta é catabolisada em Ácido β-aminoisobutírico, que na
maior parte vai-se transformar em ureia, além disso origina algum ácido propiónico.
Ácidos Nucleicos página 36 de 60
Carlos Capela
3.DOENÇAS
3.a. Alterações do metabolismo das purinas
Existem várias doenças associadas a alterações no metabolismo dos nucleótidos de
purina entre as quais se destacam: a Gota, a síndrome de Lesh-Nyan e a
Imunodeficiência.
3.a.i. A GOTA
A Gota é caracterizada pelos níveis elevados de ácido úrico no sangue e urina
devido a várias anormalidades metabólicas que levam à produção aumentada de
nucleótidos púricos pela via de novo. A maioria dos sintomas clínicos resulta da baixa
solubilidade do ácido úrico em ambientes aquosos. Cristais de urato de sódio
depositam-se nas articulações (cartilagem e membranas sinoviais) e no tecido renal
intersticial.
Estudos de pacientes com gota demonstraram que uma heterogeneidade e
multiplicidade de defeitos são a causa da produção aumentada de ácido úrico. Em
alguns casos, os defeitos bioquímicos ainda não foram determinados.
Exemplos de defeitos bioquímicos que resultam numa síntese aumentada de
nucleótidos incluem os seguintes:
1. Actividade aumentada da PRPP sintetase, que resulta num aumento dos
níveis intracelulares de PRPP. O PRPP, age como um efector alostérico positivo
da glutamina-PRPP amidotransferase, levando ao fluxo aumentado da via de
novo, uma vez que a actividade da etapa limitante é acentuadamente aumentada;
2. Diminuição parcial da actividade da HGPRTase, tem duas vertentes com
respeito à via de novo para a síntese de nucleótidos de purina. Primeiro, como
há diminuição na recuperação de hipoxantina e guanina, o PRPP não é
consumido na reacção catalisada pela HGPRTase. O PRPP pode activar a
glutamina-PRPP amidotransferase. Segundo, com a diminuição da recuperação
de hipoxantina e guanina, IMP e GMP não são formados por essa via, assim, a
regulação da etapa da glutamina-PRPP amidotransferase por IMP e GMP como
efectores alostéricos negativos fica comprometida.
Ácidos Nucleicos página 37 de 60
Carlos Capela
3. Deficiências da glicose-6-fosfatase. Em pacientes que tem deficiência de
glicose-6-fosfatase (doença de von Gierke e doença de armazenamento de
glicogénio tipo I) também ocorre, frequentemente, hiperuricémia e gota. A
perda da actividade da glicose-6-fosfatase resulta na acumulação de glicose-6fosfato (principalmente nos hepatócitos), sendo posteriormente desviada para a
via das pentoses fosfato, no que resulta um aumento de ribose-5-fosfato e
consequentemente o nível de PRPP intracelular é aumentado. O PRPP é um
efector alostérico positivo da glutamina-PRPP amidotransferase.
Estes exemplos demonstram que factores que aumentam a actividade da etapa
limitante na síntese de novo de nucleótidos de purina levam a um aumento da síntese e
degradação a ácido úrico.
Existem diferentes abordagens para o tratamento da gota que incluem colchicina,
drogas anti-hiperuricémicas e alopurinol (um análogo da hipoxantina). O alopurinol é
um inibidor eficiente da xantina oxidase que o converte em oxipurinol, que, por sua
vez, se liga fortemente à enzima inactivando-a, resultando uma diminuição dos níveis
de ácido úrico.
Em pacientes que não possuem deficiência na HGRTPase, o tratamento com
alopurinol inibe a xantina oxidase, aumentando, assim, as concentrações de hipoxantina
e xantina. Essas bases púricas são, então, recuperadas para formar IMP e XMP. Estas
reacções consomem PRPP e geram inibidores da glutamina-PRPP amidotransferase. O
efeito global do alopurinol é a diminuição quer da formação de ácido úrico quer da
síntese de novo de nucleótidos de purina.
Ácidos Nucleicos página 38 de 60
Carlos Capela
3.a.ii. A SÍNDROME DE LESCH-NYHAN
A síndrome de Lesch-Nyhan é caracterizada clinicamente por hiperuricémia,
produção excessiva de ácido úrico e problemas neurológicos, que podem incluir
espasticidade, retardo mental e auto-mutilação.
Esta doença está associada a uma deficiência severa ou total da actividade da
HGPRTase. O gene para a HGPRTase está no cromossoma X, assim, a deficiência é
virtualmente limitada a homens, sendo as mulheres portadoras.
O papel da HGPRTase é catalisar reacções nas quais hipoxantina e guanina são
convertidas a nucleótidos. Assim, a hipoxantina e guanina não são recuperadas, levando
ao aumento intracelular de PRPP e diminuição dos níveis de IMP ou GMP. Ambos os
factores promovem a síntese de novo de nucleótidos de purina, mas sem a regulação
apropriada da via.
Ainda não está compreendido porque é que um defeito severo na via de recuperação
leva a problemas neurológicos? Argumenta-se que os produtos da degradação das
purinas (hipoxantina, xantina e ácido úrico) podem não ser tóxicos para o sistema
nervoso central (SNC). No entanto, é possível que esses metabólitos sejam tóxicos para
o SNC em desenvolvimento ou que a falta dessa enzima leve a um desequilíbrio nas
concentrações de nucleótidos de purina em épocas críticas de desenvolvimento. Se a
actividade da IMP desidrogenase no cérebro fosse extremamente baixa, a falta de
HGPRTase poderia levar ao decréscimo dos níveis intracelulares de GTP devido ao
decréscimo da recuperação de guanina. Como o GTP é percursor da tetrabiopterina, um
cofactor necessário para a biossíntese de neurotransmissores; e é necessário em outras
funções como a transdução de sinal através de proteínas-G e na síntese proteica; baixos
níveis de GTP durante o desenvolvimento poderiam ser o factor desencadeador das
manifestações neurológicas observadas.
Tratamento de pacientes com alopurinol diminui a quantidade de ácido úrico
formado, aliviando alguns dos problemas causados pelos depósitos de urato de sódio.
Entretanto, como os pacientes com Lesch-Nyhan tem uma redução acentuada na
actividade da HGPRTase, a hipoxantina e a guanina não são recuperadas, o PRPP não é
consumido e, consequentemente a síntese de novo de nucleótidos de purina não é
interrompida. Não existe tratamento para os problemas neurológicos. Estes pacientes,
geralmente, morrem de insuficiência renal, resultante dos elevados depósitos de urato
de sódio.
Ácidos Nucleicos página 39 de 60
Carlos Capela
3.a.iii. IMUNODEFICIÊNCIA
ASSOCIADA
A
DEFEITOS
NA
DEGRADAÇÃO DE NUCLEÓTIDOS DE PURINA
Duas doenças distintas com imunodeficiência são associadas a defeitos na
adenosina desaminase (ADA) e purina nucleósido fosforilase (PNP), respectivamente.
Estas enzimas estão envolvidas nas vias de degradação que levam à formação de ácido
úrico. Adenosina e desoxiadenosina são os substratos para a ADA, enquanto os
substratos para a PNP são a inosina, guanosina, desoxiinosina e desoxiguanosina.
Uma deficiência da ADA é associada a uma imunodeficiência severa combinada,
envolvendo as funções dos linfócitos T e B. Deficiência da PNP é associada a uma
imunodeficiência envolvendo as funções dos linfócitos T, com poucos efeitos sobre as
funções dos linfócitos B. Em ambos os casos desconhece-se o mecanismo através do
qual a falta destas enzimas leva à disfunção imunológica. No entanto, em pacientes com
a falta de ADA, as concentrações intracelulares de dATP e S-Adenosil-Homocisteína
estão grandemente aumentadas. Várias hipóteses foram levantadas para explicar as
consequências bioquímicas da falta de ADA:
1. Altos níveis de dATP inibem a actividade da ribonucleótido redutase e, como
consequência inibem a síntese de DNA;
2. A desoxiadenosina inactiva a S-adenosil-homocisteína hidrolase, levando à
diminuição da quantidade de S-adenosilmetionina necessária para a metilação
de bases do RNA e do DNA;
3. Níveis aumentados de adenosina resultam num aumento de cAMP.
É possível que cada um destes mecanismos contribua para o efeito total sobre a
disfunção imunológica. No entanto, não existe uma explicação adequada para a
especificidade dos efeitos somente sobre os linfócitos B e T.
Tratamento de crianças com deficiência de ADA, inclui transfusão de sangue,
transplantes de medula óssea, terapia de reposição da enzima com ADA-polietileno
glicol (ADA-PEG) e, mais recentemente, terapia genica.
Cada um dos tratamentos tem vantagens e desvantagens. Transfusões sanguíneas
produzem problemas de “excesso de ferro” e segurança da fonte. Transplante da
medula óssea, embora seja curativo, requer um doador compatível. A terapia com
reposição da enzima com ADA-PEG tem sido até ao momento o tratamento mais bem
sucedido, mas requer monitoriamento constante e injecções frequentes de ADA-PEG e
Ácidos Nucleicos página 40 de 60
Carlos Capela
há um custo considerável envolvido. A terapia genica é a esperança para o futuro,
consiste na transferência do gene ADA para células com deficiência de ADA.
Ácidos Nucleicos página 41 de 60
Carlos Capela
3.b. Alterações do metabolismo das pirimidinas
Existem várias doenças devidas a alterações no metabolismo dos nucleótidos de
pirimidina, entre os quais se destaca a Acidúria Orótica Hereditária. Contudo, as
anormalidades clinicamente detectáveis não são muito frequentes, ao contrário do que
acontece com as purinas, pelo facto dos produtos de catabolismo serem muito
hidrossolúveis.
3.b.i. ACIDÚRIA ORÓTICA HEREDITÁRIA
Resulta de um defeito na síntese de novo de nucleótidos de pirimidina. Esta doença
genética é caracterizada por anemia severa, retardamento no crescimento e elevados
níveis de excreção de ácido orótico. A base bioquímica para a acidúria orótica é um
defeito numa ou ambas actividades (orotato fosforribosiltransferase ou OMP
descarboxilase) associadas à UMP sintetase, uma proteína bifuncional. É uma doença
muito rara (somente 15 pacientes são conhecidos), mas a compreensão das bases
metabólicas dessa doença levaram a um tratamento bem sucedido. Os pacientes
recebem uridina, que leva não somente à reversão do problema hematolágico, mas
também à diminuição da formação de ácido orótico. A uridina é captada pelas células e
convertida, pela uridina fosfotransferase, a UMP, que é convertido a UDP e, então, a
UTP. O UTP formado a partir da uridina exógena, por sua vez, inibe a carbamil-fosfato
sintetase II, a principal etapa reguladora da via de novo. Como resultado, o ácido
orótico produzido através da via de novo é acentuadamente diminuído para níveis
essencialmente normais. UTP também é substrato para a síntese de CTP. De facto, a
uridina exógena desvia o metabolismo da UMP sintetase deficiente e fornece, para as
células, UTP e CTP necessários para a síntese de ácidos nucleicos e outras funções
celulares.
O tratamento bem sucedido da acidúria orótica hereditária com uridina fornece
dados, in vivo, sobre a importância da etapa da Carbamil-fosfato sintetase II, como um
ponto de regulação da síntese de nucleótidos de pirimidina no homem.
Ácidos Nucleicos página 42 de 60
Carlos Capela
C. BIOSSÍNTESE DOS ÁCIDOS
NUCLEICOS E TRANSFERÊNCIA
DA INFORMAÇÃO GENÉTICA.
1.REPLICAÇÃO DO DNA
O DNA genómico de cada célula contêm em si a capacidade de se auto-replicar,
resultando a formação de 2 moléculas filhas rigorosamente idênticas à molécula
original.
De facto, a replicação do DNA corresponde à copia integral de cada uma das
cadeias constituintes do genoma da célula original através da polimerização ordenada
dos nucleótidos em obediência à regra de complementaridade de bases que desta forma
conduz à produção das 2 moléculas filhas. A polimerização dá-se a partir dos quatro
percursores de DNA que são os nucleósidos trifosfato de desoxirribose.
A reacção de polimerização dos percursores consiste no ataque nucleofílico do
fosfato ligado ao carbono 5’ da respectiva pentose ao grupo hidroxilo em posição 3’ do
nucleótido imediatamente precedente, dando origem à formação de ligações
fosfodiéster que se vão estabelecendo sucessivamente entre os resíduos que constituem
os elos da cadeia polinucleotídica. Em cada uma destas reacções é libertado um grupo
pirofosfato formado por 2 fosfatos.
Esta reacção é catalisada pelas DNA polimerases (enzimas de Kornberg) que,
embora variando entre si, apresentam diversas propriedades comuns, nomeadamente:
•
Utilizam como percursores desoxirribonucleósidos trifosfatados;
•
Dependem do ião Mg2+ para a sua actividade;
•
Necessitam de um molde de DNA;
•
Catalisam a polimerização ordenada dos percursores, de forma sequencial e
unidireccional, já que a reacção se processa exclusivamente na direcção 5’→3’.
Ácidos Nucleicos página 43 de 60
Carlos Capela
A replicação é um processo semi-conservativo, já que cada uma das novas
moléculas resultantes deste processo é constituído por uma cadeia proveniente da
molécula original de DNA molde e por uma cadeia complementar e anti-paralela
neosintetizada.
A replicação inicia-se num único sítio bem definido da molécula, o sítio de
iniciação da replicação, designado por origem. As 2 cadeias do DNA são copiadas em
paralelo e de forma simultânea, até ao ponto de terminação da replicação, também este
correspondendo a uma região bem definida do genoma.
Na replicação ou duplicação distinguem-se 3 etapas: iniciação, elongação e
terminação.
1.a. Iniciação
Nos organismos superiores existem, em geral, múltiplos sítios de iniciação da
replicação, ou origens, de modo que o DNA nuclear seja, completamente duplicado no
período correspondente à fase de síntese do ciclo celular (6-8 horas). Cada unidade de
replicação é designada por replicão.
Ácidos Nucleicos página 44 de 60
Carlos Capela
Existem proteínas dotadas de grande afinidade para o DNA e especificidade para as
sequências características de cada origem, que determinam a iniciação da replicação
sendo por isso designada por primers.
O primer é um fragmento de RNA catalisado por uma RNA polimerase específica,
designada por Primase. Este facto é bastante lógico, já que o RNA pode ser sintetizado
sem um iniciador, enquanto a síntese do DNA o requer.
O primer de RNA e as moléculas de proteína da forquilha de replicação constituem
o primossoma.
A Girase e a Helicase são duas enzimas que abrem caminho. Estas 2 enzimas
preparam a molécula de DNA para a replicação:
•
A DNA-girase vai introduzir um ponto de torção logo adiante da progressão da
bifurcação. Trata-se de uma Topoisomerase – estas enzimas, essenciais no
metabolismo do DNA, não necessitam de um factor para fornecer energia, o que
significa que a reacção intermediária conserva a energia da ligação fosfodiéster;
•
A Helicase, uma enzima que destabiliza a hélice por ruptura das pontes de
hidrogénio, liga-se à cadeia de DNA na forquilha de replicação, e promove o
desenrolamento à custa de energia fornecida pelo ATP.
As regiões monocatenárias expostas do DNA molde são estabilizadas por uma
proteína específica – proteína de ligação ao DNA ou “binding protein”. É este
complexo (DNA monocatenário e proteína de ligação) que constitui o substrato para a
DNA polimerase.
Replicão
Cadeia leading
Topoisomerase
Helicase
Binding proteins
Primossoma
Primase
Ligase
Cadeia lagging
DNA Polimerase
Ácidos Nucleicos página 45 de 60
Carlos Capela
1.b. Elongação
A copia de DNA molde é então iniciada com a síntese de um fragmento de RNA
catalisada pela primase. O fragmento de RNA sintetizado vai ser o iniciador, sendo a
cadeia polinucleotídica sintetizada a partir do grupo 3’ OH do ultimo ribonucleótido do
primer, ao qual se vêm ligar o resíduo 5’ fosfato do primeiro desoxirribonucleótido da
cadeia de DNA que será elongada por cópia do molde. Esta reacção catalisada pela
DNA polimerase que, nesta fase, desloca a primase do sistema. Após o inicio da síntese
da cadeia de DNA, estas sequências iniciadoras de RNA são removidas pela DNA
polimerase e substituídas por desoxirribonucleótidos.
O processo dá-se simultaneamente sobre as duas cadeias molde do DNA, afastadas
uma da outra. Uma dessas cadeias oferece um molde, cuja orientação 3’→5’ é propícia
ao crescimento da neocadeia na direcção 5’→3’ por polimerização sequencial dos
nucleósidos trifosfato, denominando-se de cadeia “leading” ou condutora, por dirigir o
processo de replicação. Aqui a replicação é contínua.
Na cadeia orientada 5’→3’, denominada cadeia “lagging” ou atrasada, o processo
de replicação é mais complexo, uma vez que é iniciado apenas à mediada que essa
cadeia vai sendo libertada pelo próprio processo de cópia da cadeia condutora. Isto
deve-se ao facto da DNA polimerase actuar apenas em sentido 5’→3’. Assim a
replicação da cadeia atrasada resulta da adição de pequenos fragmentos sintetizados no
sentido 5’→3’, denominados fragmentos de Okazaki, em homenagem ao seu
descobridor. A cada fragmento de Okazaki corresponde um primer diferente que será
depois removido e substituído por DNA, por acção da DNA polimerase. Esta enzima
funciona como uma enzima de reparação das cadeias de DNA.
O crescimento simultâneo das duas cadeias neosintetizadas vai, assim, progredindo,
obrigando o afastamento das duas cadeias de DNA molde, designando-se por forquilha
de replicação o ponto de progressão do fenómeno.
Ácidos Nucleicos página 46 de 60
Carlos Capela
1.c. Terminação
A replicação da cadeia de DNA termina com a ligação do último
desoxirribonucleósido trifosfato à cadeia neosintetizada. A terminação, distinta da
reacção de condensação catalisada pelas DNA polimerases, é catalisada por enzimas
designadas por DNA ligases.
É importante ainda referir que esta terminação é condicionada pelo factor ρ, que
corresponde a um tripleto de bases, que dá a informação de que a replicação está
concluída.
Ácidos Nucleicos página 47 de 60
Carlos Capela
2.TRANSCRIÇÃO DO DNA
A concentração de RNA celular está relacionada com a maior actividade metabólica
do tecido em causa.
Os RNAs existem nas células exclusivamente como subprodutos directos dos genes.
Eles são sempre sintetizados por cópia de regiões específicas e bem delimitadas do
DNA, que constitui o genoma de cada espécie, obedecendo a sua síntese ao princípio da
complementaridade de bases.
Este fenómeno, em que a informação contida em segmentos do DNA genómico é
transferida para moléculas da mesma natureza, que reflectem a própria sequência
nucleotídica dos genes designa-se por Transcrição. Este consiste essencialmente na
polimerização orientada e sequencial dos percursores de RNA que são os nucleósidos
trifosfato de ribose.
A sequência de ribonucleótidos numa molécula de RNA é complementar à
sequência de desoxirribonucleótidos de uma das cadeias da molécula de DNA. A cadeia
que é transcrita para a molécula de RNA é denominada cadeia molde de DNA ou
cadeia -. A outra cadeia é frequentemente referida como cadeia codificadora ou cadeia
+. Ela é assim chamada porque apenas difere do RNA na medida em que neste a timina
é substituída pelo uracilo.
A transcrição dos genes processa-se sob a acção de enzimas designadas por RNA
polimerases, dependentes do DNA, que catalisam a condensação orientada e sequencial
dos ribonucleótidos por cópia da cadeia molde de DNA, baseada na complementaridade
estrutural das bases. A reacção ocorre entre o grupo 3’ OH do resíduo de ribose do 1º
nucleósido trifosfato percursor e o grupo 5’ fosfato do nucleósido trifosfato seguinte,
com a formação de uma ligação fosfodiéster, resultando no crescimento da cadeia de
RNA no sentido 5’→3’.
Existe uma grande diversidade de RNA polimerases, como as RNA polimerases I,
II e III, que são responsáveis pela síntese dos rRNA, mRNA e tRNA, respectivamente.
Existem alguns vírus que podem fazer uma síntese em direcção contrária à
transcrição, ou seja, formar DNA a partir de RNA do hospedeiro. Este processo,
catalisado pela enzima transcriptase inversa é designado por transcrição inversa.
Na transcrição existem 3 etapas distintas: iniciação, elongação e terminação.
Ácidos Nucleicos página 48 de 60
Carlos Capela
2.a. Iniciação
A copia de cada gene é iniciada pela RNA polimerase ao nível do 1º nucleótido que
irá ser copiado em RNA. É designado por Nucleótido +1 (Nt+1).
A transcrição inicia-se em regiões do DNA designadas promotores que são
reconhecidas pela RNA polimerase tendo esta enzima um papel fundamental no
desenvolvimento e abertura da cadeia de DNA. Estas regiões promotoras têm
particularidades estruturais, verificando-se que, na maior parte dos casos, a mesma
sequência de nucleótidos está presente nos diversos genes – sequências de consenso.
Existem ainda factores de transcrição, nomeadamente o factor σ, que se liga às
regiões promotoras e estimulam a ligação da RNA polimerase ao local promotor.
A iniciação da transcrição dá-se efectivamente quando os dois primeiros
ribonucleósidos trifosfatados são posicionados e condensados entre si, sob a acção da
RNA polimerase.
2.b. Elongação
Esta etapa consiste na polimerização sequencial de resíduos ribonucleótidos
ordenados segundo a sequência de DNA molde, por complementaridade de bases. O
superenrolamento do DNA induzido pela abertura localizada resultante da RNA
polimerase é resolvido, durante a elongação, através da acção de Topoisomerases.
2.c. Terminação
A terminação da síntese da molécula de RNA é determinado por uma sequência da
molécula de DNA, um sinal que é reconhecido por uma proteína de terminação, o
factor ρ. Após a terminação da síntese da molécula de RNA, a enzima separa-se da
cadeia molde.
Ácidos Nucleicos página 49 de 60
Carlos Capela
Hélice de DNA
Promotor
RNA Polimerase
Iniciação
Stop Sequência
Elongação
Terminação
2.d. Maturação do mRNA
A cadeia de RNA mensageiro neosintetizada não pode ser considerada como
definitiva
para
a
síntese
de
proteínas
designando-se nesta fase de RNA prémensageiro.
Neste
existem
2
tipos
Adição de um cap
de
sequências nucleotídicas:
PoliAdenilação
•
Exões – codificam aminoácidos;
•
Intrões – intercalados entre os exões,
intrões
sem significado proteico.
Por
um
mecanismo
designado
por
exões
splicing, realizado por uma grande complexo,
spliceossomas
cauda poliA
RNA Splicing
o spliceossoma, os intrões são removidos por
RNA Mensageiro
acção das maturases, seguida da reparação
maturado
dos cortes pelas ligases.
As etapas de processamento do mRNA envolvem adição de um cap à extremidade
5’ e a poliadenilação da extremidade 3’. Após estas transformações o mRNA é
considerado maturo e pronto para a tradução.
Ácidos Nucleicos página 50 de 60
Carlos Capela
3.TRADUÇÃO – A SÍNTESE DE PROTEÍNAS
3.a. Introdução
A biossíntese de proteínas também é chamada tradução, uma vez que envolve a
tradução bioquímica da informação de uma linguagem de 4 letras e estrutura dos ácidos
nucleicos numa linguagem de 20 letras e estrutura das proteínas.
Em suma, a biossíntese proteica é resultado do processo de tradução da informação
contida no material genético da célula; as proteínas são, portanto, a expressão da
informação genética.
O processo de tradução requer um Código Genético, através do qual a informação
contida numa sequência de nucleótidos de um ácido nucleico é expressa na forma de
uma sequência específica de aminoácidos de um peptído.
Uma alteração na sequência original de nucleótidos leva a uma alteração na
sequência de aminoácidos, potencialmente causando uma doença – as vezes letal – no
organismo.
Ácidos Nucleicos página 51 de 60
Carlos Capela
3.b. O código genético
O código genético dos seres vivos, hoje conhecido, funciona como uma linguagem
baseada em 4 “letras” – ou bases de nucleótidos –, que forma “palavras” de três letras
que, dependendo da maneira como são agrupadas, formam as “sentenças” – ou
peptídos.
Estas “palavras” de 3 letras são denominadas Codões, e são possíveis 64 codões
diferentes.
Os codões são expressos na linguagem do mRNA, sempre no sentido 5’→3’, e cada
codão pode ser traduzido num aminoácido específico.
3 Codões sinalizam o término da sequência polipeptídica, e são designados por
codões de terminação: UGA, AUG e UAA
O codão AUG codifica a Metionina e, a iniciação de uma cadeia polipeptídica,
sendo designado por codão de iniciação.
Podem ocorrer mutações pontuais, em consequência da alteração de uma das bases
de um codão, que podem levar a:
•
Mutação silenciosa – Quando o codão alterado codifica o mesmo aminoácido;
•
Mutação em “missense” – Quando o codão alterado codifica um aminoácido
diferente;
•
Mutação em “nonsense” – Quando o codão alterado sinaliza o termino da
cadeia
3.b.i. CARACTERÍSTICAS DO CÓDIGO GENÉTICO
•
Especificidade – Um codão específico codifica sempre o mesmo aminoácido
•
Universalidade – O código genético é válido para qualquer ser vivo, ou seja um
dado mRNA de uma espécie ao ser traduzido em células de outra espécie, iria
originar sempre a mesma proteína. Contudo encontram-se na mitocôndria
alguns codões que codificam aminoácidos diferentes, quando comparados com
o código genético mitocondrial, devendo por esta razão dizer-se que o código
genético citoplasmático é universal.
•
Redundância – Um mesmo aminoácido pode ter mais de um codão que o
codifica. Contudo um codão codifica sempre o mesmo aminoácido, pelo que se
diz que o código genético não é ambíguo.
Ácidos Nucleicos página 52 de 60
Carlos Capela
•
Linearidade – A leitura do código genético é feita sem sobreposições ou
pausas; se 1 ou 2 nucleótidos são delectados, a leitura torna-se completamente
diferente a partir do ponto da deleção. Esta mutação denomina-se “frameshift”,
ou mutação localizada.
Ácidos Nucleicos página 53 de 60
Carlos Capela
3.c. Componentes necessários à tradução
São vários, entre eles:
3.c.i. OS AMINOÁCIDOS:
Devem estar presentes todos os 20 aminoácidos proteicos comuns no momento da
síntese, inclusive os essenciais.
3.c.ii. O RNA DE TRANSFERÊNCIA:
São as moléculas “bilingues” do processo, e estão ligadas de forma específica aos
aminoácidos nas suas extremidades 3’.
Possuem o anticodão ou a sequência de bases complementar e anti-paralela ao
codão do mRNA.
Cada aminoácido pode ser transportado por mais de uma molécula de tRNA.
São em número de cerca de 50 tRNA no ser humano, para 64 codões diferentes.
A hipótese “wobble”, ou de oscilação, tenta explicar como um tRNA pode
reconhecer mais que um codão; nesta hipótese, o emparelhamento da primeira base do
anticodão (5’) não é necessariamente feita do modo tradicional. Assim, nesta posição, a
G pode emparelhar com a C ou U, e o U com a A ou G, por exemplo.
3.c.iii. O RNA MENSAGEIRO:
Produzido a partir de um molde de DNA no núcleo da célula, vai ao citosol para ser
traduzido numa sequência polipeptídica
Traz a sequência de codões que define o número e o posicionamento dos
aminoácidos da cadeia polipeptídica
Cada molécula de mRNA corresponde – quase sempre – a uma cadeia polipeptídica
(monocistrônicos).
3.c.iv. RIBOSSOMAS:
São organitos formados por uma complexa associação entre proteínas e RNA
Ribossómico, localizados no citosol na forma livre ou associados ao retículo
endoplasmático, quando sintetizam proteínas que serão “exportadas” da célula.
Ácidos Nucleicos página 54 de 60
Carlos Capela
São formados por 2 subunidades, uma maior e outra menor, cujas características são
expressas em termos de coeficientes de sedimentação, ou “S” (de Svedberg).
O ribossoma eucariota é formado por uma subunidade 60S e outra 40S; o
procariota, por uma subunidade 50S e outra 30S.
Compõem um ribossoma:
•
rRNA – 3 moléculas no procariota, 4 moléculas no eucariota.
•
Proteínas ribossómicas – Desempenham várias funções na tradução.
•
Sítio “A” e “P” – São locais de ligação para as moléculas de tRNA, e estendemse por ambas as subunidades:
o Os sítios A e P cobrem juntos dois codões vizinhos;
o Na tradução, o sítio A – de aminoacil – é ocupado pelo Aminoacil-tRNA
correspondente ao codão posicionado, e especifica o próximo
aminoácido a ser adicionado à cadeia peptídica em formação;
o O sítio P – de peptidil – é ocupado pelo Peptidil-tRNA, ligado à cadeia
peptídica que está sendo sintetizada.
Ácidos Nucleicos página 55 de 60
Carlos Capela
3.c.v. FACTORES PROTEICOS:
São factores de iniciação, elongação e libertação da cadeia polipeptídica que está
sendo sintetizada.
3.c.vi. FONTES DE ENERGIA
A adição de cada aminoácido à cadeia polipeptídica que está sendo sintetizada
utiliza 2 moléculas de ATP e 2 de GTP como fonte de energia.
Outras moléculas de ATP e GTP são necessárias nos processos de iniciação e
libertação da cadeia sintetizada.
Ácidos Nucleicos página 56 de 60
Carlos Capela
3.d. Etapas da Biossíntese Proteica:
3.d.i. ACTIVAÇÃO DOS AMINOÁCIDOS:
Refere-se à ligação dos aminoácidos ao(s) seu(s) tRNA específico(s).
Esta ligação é catalisada pelas Aminoacil-tRNA Sintetases, enzimas que
reconhecem os aminoácidos e os seus tRNA específicos com grande especificidade.
A reacção ocorre em 2 etapas, e é dependente de energia. Na primeira etapa, o
aminoácido liga-se ao AMP, obtido a partir da hidrólise do ATP, com libertação de PPi.
Na segunda etapa, o AA-AMP liga-se à extremidade 3’ do tRNA, libertando o AMP e
energia.
Ácidos Nucleicos página 57 de 60
Carlos Capela
3.d.ii. INICIAÇÃO DA CADEIA:
Depende da presença da subunidade ribossomal 40S, do mRNA, do tRNA com o
anticodão de iniciação AUG (metionina), e de uma série de proteínas denominadas
“factores de iniciação”, ou eIF, no mínimo, em número de 9, nos eucariotas.
Etapas:
a) O eIF-2 liga-se especificamente com uma molécula de GTP e com o MettRNAi;
b) A subunidade ribossomal 40S liga-se ao eIF-3, que a separa e a mantém
separada da subunidade 60S, que por sua vez se liga ao eIF-6, com o mesmo
propósito;
c) O complexo formado em (a) liga-se ao complexo 40S formado em (b), com a
ajuda de vários outros factores de iniciação, e este novo complexo associa-se ao
mRNA – complexo de pré-iniciação;
d) A associação do complexo de pré-iniciação com o factor de iniciação eIF-5 e
com a subunidade 60S do ribossoma dá origem ao complexo de iniciação
propriamente dito. O GTP é hidrolisado e os outros factores de iniciação são
libertados;
e) O complexo de iniciação é, portanto, formado por um ribossoma 80S associado
aos eIF-5 e eIF-4, ao mRNA correctamente posicionado, e ao tRNA de
iniciação também posicionado no sítio P, com o sítio A vazio.
3.d.iii. ELONGAÇÃO DA CADEIA:
Depende dos “factores de elongação” e do complexo de iniciação
Etapas:
a) O Factor de Elongação eEF-1 dirige a selecção do tRNA correcto para o
posicionamento no local A do complexo de iniciação. Este posicionamento
depende ainda da hidrólise de 1 GTP, e ocorre com posterior libertação do eEF1;
b) Há a formação da ligação peptídica entre os 2 aminoácidos adjacentes;
c) A enzima peptidiltransferase, componente da subunidade ribossómica 60S, faz
a transferência da metionina inicial do sítio P para o aminoácido do sítio A,
formando um dipeptidil-tRNA;
Ácidos Nucleicos página 58 de 60
Carlos Capela
d) O ribossoma move-se – às custas do GTP – na direcção 5’→3’ uma distância de
3 nucleótidos, com a ajuda do eEF-2, ou translocase, posicionando o péptido no
sítio P e libertando o sítio A para o posicionamento de um novo aminoácido;
e) O tRNA livre é libertado, e o processo repete-se até a tradução completa do
mRNA.
3.d.iv. TERMINAÇÃO DA CADEIA POLIPEPTÍDICA:
O aparecimento de um codão de terminação – UAG, UGA ou UAA – determina a
ligação de um “Factor de Libertação”, ou eRF, associado ao GTP.
Este factor de libertação determina a quebra da ligação entre o último AA e seu
tRNA.
A dissociação do eRF do ribossoma depende da hidrólise do GTP.
Ribossoma
unidade pequena (40S)
Ribossoma
Aminoácido
unidade grande (60S)
tRNA iniciador
Iniciação
Elongação
Polipéptido
Terminação
Ácidos Nucleicos página 59 de 60
Carlos Capela
3.d.v. MODIFICAÇÕES PÓS-TRANSLACIONAIS
Várias modificações podem ser impostas à cadeia polipeptídica após a sua
biossíntese. As principais modificações são:
•
Redução do Tamanho: Conversão de proteínas inactivas e zimogênios nas suas
formas activas, por remoção de sequências de aminoácidos catalisadas por
endoproteases específicas.
•
Alterações Covalentes: Fosforilação, glicosilação, hidroxilação, etc.
•
Algumas proteínas são ainda endereçadas a organelos.
Ribossomas
Polissoma
Polipéptidos
Ácidos Nucleicos página 60 de 60