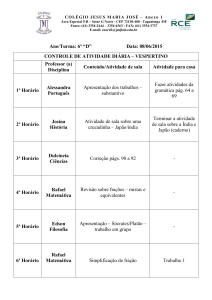
Proteínas
Enovelamento, Desnaturação e Função
Rafael Mesquita
Ligação peptídica – limitações estruturais
Limitações estruturais impostas pelas ligações peptídicas
• Impedimento estérico
Rafael Mesquita
Radical – limitações estruturais
Limitações estruturais impostas pelo radical
• Volume (impedimento estérico)
• Repulsões de carga
Rafael Mesquita
Enovelamento
Rafael Mesquita
Enovelamento
• A estrutura tridimensional das proteínas é determinada
diretamente pela estrutura primária
• Existem forças que direcionam o enovelamento
• Predição puramente computacional ainda não é viável
http://www.wiley.com/legacy/college/boyer/0470003790/animations/protein_folding/
protein_folding.htm
Rafael Mesquita
Enovelamento
O Paradoxo de Levinthal, 1968:
Uma proteína não pode experimentar todas as possíveis conformações entre o
estado desenovelado e o estado nativo.
Imagine uma proteína com 150 aa que experimentam apenas as 3 conformações
previstas no plote de Ramachandran. Cada conformação se interconverteria na
outra em picoseg (10-12s). Essa proteína, então, possuiria 3150 possíveis
conformações (= 1068). Para experimentar todas essas conformações seriam
necessários
1068 x 10-12 seg = 1056 seg = 1048 anos !!!
O enovelamento demora de 0.1 a 1000 seg in vivo e in vitro.
O enovelamento é, portanto, dirigido passando por rotas cinéticas e
intermediários bem definidos escapando de conformações irrelevantes.
Rafael Mesquita
Rafael Mesquita
Rafael Mesquita
Os três mecanismos clássicos de enovelamento
Rafael Mesquita
A. Fersht and V. Daggett, 2002, Cell
Enovelamento: Estado nativo e Estabilidade
Rafael Mesquita
Problemas que dificultam a passagem I → N
• agregação devido a superfícies hidrofóbicas expostas
• formação de pontes dissulfeto incorretas
• isomerização dos resíduos de prolina
ê
Proteínas Chaperones
Proteína-dissulfeto-isomerase
Proteína-peptidil-prolil-cis-trans-isomerase
Rafael Mesquita
Enovelamento Assistido : Chaperonas
• São caixas de enovelamento
• Antes de enovelar, algumas proteínas expõem segmentos
hidrofóbicos que tendem a agregar
• as chaperones podem ser induzidas pelo choque térmico
• chaperones são chamadas de heat shock proteins
• DNA K (Hsp 70)
• GroEL e GroES (Hsp 60 e 10) chamada de chaperoninas
-se ligam a polipeptídeos desenovelados ou
parcialmente enovelados
- são promíscuos
Rafael Mesquita
Enovelamento Assistido : Chaperonas - GroEL
• 14 subunidades que formam 2 anéis com uma cavidade central
• cada subunidade tem 3 domínios: equatorial, intermediário e apical
• contem 7 sítios de ligação a ATP
• Gro ES se liga formando um tampa
Rafael Mesquita
Paul Sigler and Arthur Horwich,1994
Enovelamento Assistido : GroEL + GroES
Rafael Mesquita
Enovelamento Assistido : Dissulfeto isomerases
• Nas bactérias existem as Dsb (disulfide-bridge forming
enzimes) que ficam no periplasma
• Em eucariotos existem as PDI (protein-disulfide isomerases)
no retículo endoplasmático que corrigem proteínas com pontes
incorretas
Exemplo de enzima que precisa deste “serviço”:
BPTI (bovine pancreatic tripsin inhibitor): possui 6 resíduos
de cisteína que formam 3 pontes de S (58 aa).
30-51
Rafael Mesquita
5-55
14-38
Enovelamento: Peptidil-prolil-cis-trans-isomerase
• Na forma trans, os peptídeos são mais estáveis (C=O e N-H apontando
para direções opostas)
• para a maioria dos petídeos, a forma cis é 1000 vezes mais instável
que a forma trans
Rafael Mesquita
Enovelamento: Peptidil-prolil-cis-trans-isomerase
• Entretanto, quando existem prolinas, a forma cis é apenas 4 vezes mais
instável que a forma trans
• em algumas proteínas as prolinas aparecem na forma cis
• a isomerização cis-trans é muito lenta in vitro.
• in vivo, a proteína peptidil prolil isomerase (procariotes e eucariotes)
aumenta a isomerização de prolinas em 1.000.000 de vezes
• essa proteína está envolvida na imunosupressão inibindo a proliferação
das células T (chamada de ciclofilina, pois liga ciclosporina A)
Rafael Mesquita
Mudança de estrutura do príon
Rafael Mesquita
Desnaturação
• “As proteínas são marginalmente estáveis” (10-20 kcal/
mol)
• Isso é importante pois garante turnover das proteínas e
flexibilidade
• Interações que mantém a estrutura nativa
• pontes de hidrogênio
• interações iônicas - entre resíduos carregados
• interações hidrofóbicas - entre resíduos apolares
• pontes dissulfeto
Rafael Mesquita
Desnaturação
• Perda da estrutura X solubilidade X atividade
• Reversibilidade
• Agentes desnaturantes:
– pH
– Força iônica
– Temperatura
– Polaridade
– Meio redutor (pontes dissulfetos)
– Agentes caotrópicos (uréia, guanidina, etc)
Rafael Mesquita
Experimento de Christhian
Anfinsen (década de 50)
Ribonuclease: 124 aa com 4
pontes de S
Rafael Mesquita
Desnaturação térmica e química
Rafael Mesquita
Desnaturação: pH e força iônica
Rafael Mesquita
Proteínas: Estrutura e Função
As proteínas são macromoléculas e têm funções muito variadas
Rafael Mesquita
Exemplo de funções de proteínas
Rafael Mesquita
Colágeno
Aproximadamente 25% do total das
proteínas do nosso corpo é colágeno
Rafael Mesquita
Mioglobina
Rafael Mesquita
Hemoglobina
Heme
Rafael Mesquita
Transporte e Transferência de O2
Cooperatividade da hemoglobina
Rafael Mesquita
Mutação e anemia falciforme
Rafael Mesquita
Mutação e anemia falciforme
Rafael Mesquita
Queratina
Rafael Mesquita
Mudança de estrutura do príon
Perda da função fisiológica è dano celular
Rafael Mesquita
Endereçamento de proteínas:
Rafael Mesquita
Endereçamento de proteínas: Peptídeo sinal
Rafael Mesquita
http://www.cbs.dtu.dk/services/
>SALSA_197631813 CATHEPSIN D [SALMO SALAR]
MKVLYLCLFAALALASDALVRIPLRKFRSIRRTLTDSGRAAEELLAGKEHTKYNNLGFPSSSNGPTPETLKNFMDAQYYG
EIGLGTPAQTFTVVFDTGSSNLWVPSVHCSFTDIACLLHHKYNGAKSSTYVKNGTAFAIQYGSGSLSGYLSQDTCTIG
GLSIEEQVFGEAIKQPGVAFIAAKFDGILGMAYPRISVDGVAPPFDNIMSQKKVEQNVFSFYLNRNPESEPGGELLLGGT
DPKYYSGDFQYLNVSRQAYWQVHMDGMGVGSQLSLCKGGCEAIVDTGTSLITGPTAEVKALQKAIGATPLIQGEYMV
NCDKIPTMPDITFNLGGQSYSLTAEQYVLKESQAGKTICLSGFMGLDIPAPAGPLWILGDVFIGQYYTVFDRDNNRVGF
AKSK
Rafael Mesquita
Rafael Mesquita
Modificações pós-traducionais: Glicosilação
Rafael Mesquita
http://www.cbs.dtu.dk/services/
Modificações pós-traducionais: Fosforilação
• Quinases são enzimas responsáveis pela adição de um
grupamento fosfato em resíduos de Ser, Thr e Tyr de
proteínas.
• Fosfatases são enzimas responsáveis pela remoção do
fosfato.
ATP
PO3 4-
Kinase
Proteína
Fosfatase
Proteína
PO3 4Rafael Mesquita
http://www.cbs.dtu.dk/services/
Agradecimentos:
Glória Braz (slides de estrutura-função de proteínas)
Ana Paula Salerno (slides de enovelamento e desnaturação)
Rafael Mesquita




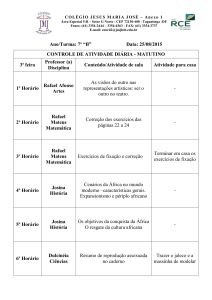
![[1] 22 Data Turma PROFESSOR / DISCIPLINA CONTEÚDO](http://s1.studylibpt.com/store/data/003499684_1-b8efb2362ad2902c95fc6c42f4f67a4c-300x300.png)