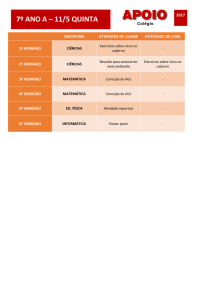
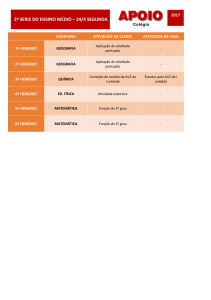
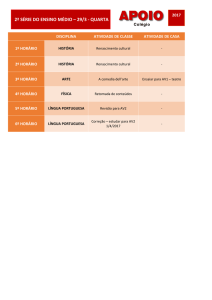
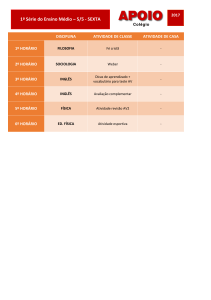
50º Congresso Brasileiro de Genética
Resumos do 50º Congresso Brasileiro de Genética • 7 a 10 de setembro de 2004
Costão do Santinho • Florianópolis • Santa Catarina • Brasil
www.sbg.org.br - ISBN 85-89109-04-6
[email protected]
Palavras-chave: Paracoccidioides brasiliensis,
genes completos, ATP sintase, filogenia
Pereira, TM; Moral, FAF; Nóbrega, FG
Laboratório de Genética Molecular e Genomas, Instituto de Pesquisa e Desenvolvimento (IP&D), Universidade do Vale do
Paraíba (UNIVAP)
Reconstrução filogenética conjunta
dos genes ATP4, ATP5 E ATP7
de Paracoccidioides brasiliensis
baseada no método de distância
O Paracoccidioides brasiliensis (Pb), agente etiológico da paracoccidioidomicose (PCM), é um organismo
pleomórfico dependente da temperatura para manifestar sua morfologia. Essa transição da fase micelial
para a leveduriforme, afeta a fisiologia mitocondrial de maneira marcante. Estamos estudando os cDNAs
que codificam para genes envolvidos na biogênese e atividade do complexo da ATP sintase, a enzima que
conserva a energia do gradiente eletroquímico mitocondrial sob forma de ATP. A ATP sintase é formada por
dois componentes, cada um constituído por várias cadeias polipeptídicas. Um porção esférica, chamada de
fator de acoplamento 1, contém os sítios de síntese de ATP. A segunda porção, que confere sensibilidade a
oligomicina, fica embebida na membrana interna, constituindo um canal através do qual os prótons retornam
à matriz mitocondrial. Neste trabalho apresentamos a seqüência completa de vários genes nucleares que
codificam proteínas da ATP sintase e um estudo filogenético conjunto comparando os polipeptídeos Atp4p,
Atp5p e Atp7p de Pb com as proteínas correspondentes de outros organismos através da construção de uma
topologia para o grupo pelo método de distância. O alinhamento múltiplo das seqüências foi gerado através
do programa CLUSTAL X com valores de penalidades alterados, tomando-se como base o trabalho de Hall
em 2001. Alterações manuais para busca de possíveis erros, tanto para inserções como para deleções, foram
feitos no programa BIOEDIT v.5.0.9. A análise filogenética foi gerada pelo método de distância, usando
o modelo “Neighbor-Joining”, com “bootstrap” para 1000 replicações dentro do programa computacional
MEGA 2 v.3.0 (Molecular Evolutionary Genetics Analisys.). A filogenia obtida coloca P. brasiliensis em
um clado consistente que inclui Aspergillus nidulans, ficando estes com uma ramificação independente de
Saccharomyces cerevisiae e também distante de Neurospora crassa.
Apoio financeiro: FAPESP e CNPq
919