Transcrição
Objetivos
RNA polimerase
Uma vez que o processo de transcrição é extremante conservado dentre todas as espécies
estudadas, os padrões destas proteínas também são conservados. Isto quer dizer que existem
muitas semelhanças em seqüência e estrutura nestas proteínas quando comparamos proteínas
de diversas espécies. Na figura a seguir portanto é mostrada a semelhança em estrutura de
enzimas procariota e eucariota.
Características da RNA polimerase
O sítio ativo da enzima é capaz de reconhecer especificamente os rNTPs (ribonucleotideos
trifosfatados), não reconhecendo portanto os dNTPs (desoxinucleotideos trifosfatados presentes
no DNA.
Contudo similarmente a DNA polimerase a RNA polimerase também possui dois domínios de
ligação a íons metálicos divalentes. Estes íons possuem a mesma função encontrada na DNA
polimerase, ou seja, atacar o hidroxila 3’ livre do nucleotideo no final da cadeia, facilitando o
ataque nucleofílico do radical hidroxila ao fosfato alfa do nulceotídeo entrante e assim formar a
liagação fosfodiester.
Nucleotídeos trifosfato (rNTPs)
Radical hidroxila
presente no carbono 2
do açúcar, no caso
ribose
A transcrição pode ser dividida didaticamente em três fases
1 – Inicio
2 - Elongação
3 – Término
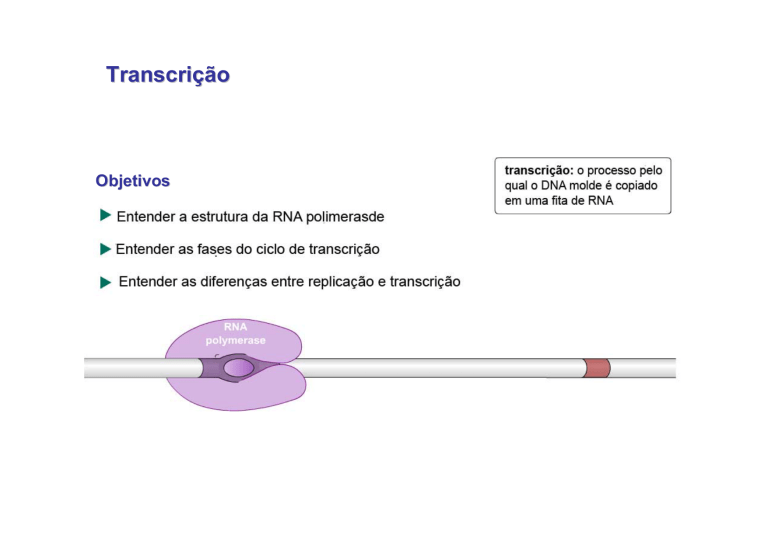
No inicio de transcrição o fator sigma (σ) se liga a RNA polimerase. É o fator
sigma que se liga à região promotora posicionando a enzima de forma correta
para inicio de transcrição. Esta precisão é necessária porque não pode haver
dúvida sobre qual fita deve ser transcrita. Na transcrição somente uma fita do
DNA é transcrita. No esquema mostrado a fita a ser transcrita seria aquela
esquematizada de vermelho.
Etapas da elongação
Atividade exonucleasica da RNA polimerase (correção de bases
polimerizadas erradamente).
Assim como a DNA polimerase a RNA polimerase também possui a capacidade de
perceber mudanças conformacionais decorrentes da adição de nucleotídeos errados.
A RNA polimerase possui um mecanismo de correção menos eficiente do que a DNA
polimerase, em média a DNA polimerase incorpora um nucleotídeo errado a cada 10
milhões de bases (1x107) e a RNA polimerase incorpora um nucleotídeo errado a cada
dez mil bases (1x104).
Similarmente a DNA polimerase, a RNA polimerase abaixa bastante sua velocidade após
perceber o nucleotídeo errado. A retirada do nucleotídeo errado pode ser feita de duas
maneiras. A) pelo mecanismo de edição fosforolítica que implica na retirada do
nucleotídeo errado pela atividade exonucleasica. Para isto a enzima volta uma base e
cliva o nucleotídeo. B) Edição hidrolítica, neste mecanismo a RNA polimerase faz uma
varredura de 1 ou mais nucleotídeos estimulada (em bactérias) pela proteína Fator GE
que promove também a elongação.
OBS: note que as regiões repetidas invertidas irão se ligar por
complementariedade quando forem transcritas
Região repetida
invertida
Região repetida
invertida










