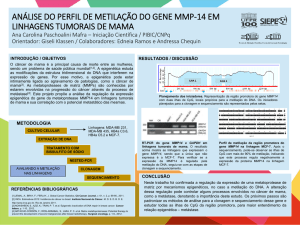
Clinical Chemistry
Q&A
Eleftherios P. Diamandis
Epigenomics-Based Diagnostics
Diagnóstico Baseado em Epigenômica
Eleftherios P. Diamandis, Chefe da Bioquímica Clínica, Departamento de Bioquímica Clínica, Mount Sinai Hospital and University Health Network, e Professor e Chefe, Divisão de Bioquímica Clínica, Departamento de Medicina Laboratorial e Patobiologia, University of Toronto, Toronto, Ontario, Canada.
O termo "epigenética," primeiro criado em
1942, se refere às características hereditárias
das células (após muitas rodadas de divisão celular) que não envolvem mudanças na sequência subjacente do DNA. Os 2 mecanismos epigenéticos predominantes são metilação do DNA
e modificação da histona.
Mudanças epigenéticas, especialmente metilação do DNA na posição 5 da citosina, que ocorre
nas regiões ricas em guanina/citosina (ilhas CpG
), podem ter grandes efeitos na transcrição dos
genes. Por exemplo, é sabido que extensiva metilação nas regiões ricas em GC dos promotores
dos genes ou outras áreas podem afetar dramaticamente a transcrição dos genes e consequentemente toda a biologia de uma célula específica. Mudanças epigenéticas são preservadas
quando as células se dividem. Embora seja bem
conhecido que o câncer pode ser causado pela
perda dos genes supressores de tumores (por
exemplo, através da mutação ou deleção), outro importante mecanismo de silenciar genes
críticos é através da metilação. Existem numerosos exemplos de genes que podem ser silenciados por metilação. Desse modo é razoável
sugerir que a metilação do DNA possa ser um
marcador do silenciamento do gene e que tais
modificações possam se correlacionar com o
início e progressão do câncer. Por essa razão, é
provável que mudanças epigenéticas no DNA
possam carregar informação diagnóstica, prognóstica ou preditiva. Desse modo não é surpreendente que mudanças epigenômicas/epigenéticas tenham atraído grande atenção recentemente para sua possível aplicação no câncer e
em outros diagnósticos de doenças. Nessa Q&A,
4 peritos no campo discutem o presente e o
futuro da epigenômica e como são usadas nas
aplicações diagnósticas.
Você poderia descrever
rapidamente o campo
da epigenômica?
David Sidransky2: O
campo da epigenômica
está focado em achar
alterações genômicas além das mudanças na
sequência do DNA.
Peter W. Laird3: Epigenômica é o estudo da
escala do genoma da distribuição das marcas epigenéticas, que são modificações estáveis do DNA
ou da conformação do
DNA que transmitem informação hereditária não
geneticamente codificada. Embora haja algum
debate quanto a que marcas são fielmente preservadas durante a divisão celular, é geralmente
aceito que as marcas epigenômicas incluem
metilação do DNA na citosina 5, modificações
das extremidades das histonas, e posicionamento do nucleossoma.
Paul Cairns4: O estudo
da metilação do DNA,
das modificações da histona, do estado da cromatina, a da interferência do RNA, e o efeito
relacionado sobre a expressão gênica em diferentes tipos de células normais que compartilham um genoma idêntico, entre a célula pro-
Clinical Chemistry
genitora normal e a célula tumoral, e em outras
doenças humanas.
Bharati Bapat5: Epigenômica se refere à
regulação do genoma
por mudanças hereditárias na expressão
gênica mediadas por
fatores de sequência
de não DNA. Essas
incluem metilação do DNA, regulação da estrutura e função da cromatina, e pequena regulação mediada por RNA não codificante. Uma característica única é que diferente das mutações
nas sequências do DNA, muitas dessas alterações são potencialmente reversíveis por natureza e desse modo fornecem alvos atraentes para
abordagens terapêuticas.
Quais técnicas são usadas para diagnóstico
epigenômico? Essas técnicas são facilmente
adaptáveis aos laboratórios diagnósticos?
David Sidransky: Existem muitas técnicas usadas para encontrar mudanças epigenômicas em
células cancerosas. Para metilação do promotor, essas incluem sequenciamento do DNA
modificado, abordagens baseadas nos arranjos,
e testes baseados no PCR quantitativo. Sim, todas essas abordagens podem ser adaptadas aos
laboratórios diagnósticos. Algumas, como o PCR
quantitativo, já estão integradas em muitos laboratórios.
Peter W. Laird: Técnicas da escala do genoma
para análise epigenética são tecnicamente desafiadoras e improváveis de ser adaptadas por
laboratórios diagnósticos no futuro próximo.
Testes específicos dos genes baseados no PCR
em tempo real são mais facilmente adaptados
às plataformas comerciais presentes nos laboratórios diagnósticos e oferecem a vantagem de
elevada sensibilidade e especificidade técnica.
Paul Cairns: PCR específico da metilação é a
tecnologia predominante usada para diagnóstico baseado na epigenômica. A disponibilidade
de kits comerciais de modificação de bissulfito e
da tecnologia quantitativa do PCR em tempo
real significa que a metilação é mais pronta-
Q&A
Eleftherios P. Diamandis
mente adaptável aos laboratórios diagnósticos.
Mais recentemente, PCR quantitativo de transcrição reversa tem sido usado para detecção da
expressão do microRNA como uma ferramenta
diagnóstica.
Bharati Bapat: Várias técnicas estão disponíveis
para detectar mudanças epigenômicas. Metilação do DNA é detectada por análise de sequência do DNA modificado de bissulfito. Para diagnóstico epigenético, metilação do DNA e microRNAs são detectados por testes quantitativos baseados no PCR e técnicas de alta produtividade baseadas nos microarranjos. Testes baseados no PCR são facilmente adaptáveis aos
laboratórios diagnósticos e já estão implementados no diagnóstico do câncer.
Quais são as distintas vantagens da epigenômica sobre outras técnicas para o diagnóstico?
Quais são as desvantagens mais sérias do diagnóstico baseado na epigenômica ?
David Sidransky: No câncer, metilação do promotor produz um sinal positivo, portanto achar
alelos metilados torna o trabalho de encontrar
células ou moléculas raras mais fácil do que
procurar por eventos que estejam ausentes nas
células. Ao contrário das mutações genéticas,
onde um grupo de tumores pode ter centenas
de diferentes mutações, definir metilação densa
em um local é mais fácil e mais sensível às técnicas de alta produtividade.
As desvantagens dessa abordagem são que mudanças epigenéticas são diversas e requerem
vários tipos de materiais e testes. Também, ainda temos que testar vários locais ou alterações
para cobrir um tipo de tumor.
Peter W. Laird: Perfis epigenômicos são bastante estáveis e relativamente impermeáveis às
flutuações no estado fisiológico e às condições
de coleta das amostras. Testes de metilação do
DNA possuem uma vantagem de que o analito
do DNA é resistente à degradação, tanto in vivo
quanto após se obter a amostra. As marcas epigenéticas em uma região do genoma determinam o potencial para expressão gênica, em vez
do estado real da expressão. Portanto, o epigenoma transmite informação não plenamente
Clinical Chemistry
capturada pelos perfis de expressão gênica. A
desvantagem mais séria do diagnóstico baseado
na epigenômica é a dificuldade técnica da análise epigenômica.
Paul Cairns: Para diagnóstico de câncer as vantagens distintas são que hipermetilação aberrante é frequente e precoce na carcinogênese,
que é uma mudança positiva, e que tecnologia
de detecção sensível está disponível. As desvantagens da metilação são que uma quantidade
relativamente grande de bioamostra é necessária (como com qualquer alvo de detecção baseado no ácido nucléico) e que a robustez do teste depende de cuidadoso design e operação.
Bharati Bapat: A detecção das mudanças epigenéticas, particularmente mudanças da metilação do DNA, possui várias vantagens sobre os
marcadores genéticos convencionais. Para um
dado gene, mutações pontuais frequentemente
ocorrem em vários locais nos tumores individuais. Em contraste, metilação do DNA geralmente ocorre sobre a mesma região do gene (por
exemplo, promotor), e isso grandemente simplifica o design e interpretação dos testes de seleção. Metilação do DNA constitui um sinal positivamente detectável, ao contrário de uma perda
de sinal, tais como deleções cromossômicas.
Metilação anormal do DNA geralmente não
ocorre em células normais. Portanto, DNA derivado do tumor pode ser detectado com um alto
grau de especificidade, particularmente em fluidos corporais tais como saliva, soro, ou amostras de urina.
A principal desvantagem do diagnóstico baseado na epigenômica é que vários diferentes testes podem ser necessários para detectar diferentes tipos de mudanças epigenéticas num
câncer.
Quais são os cânceres mais preferidos para
diagnóstico por epigenômica?
David Sidransky: Todos os cânceres possuem
alterações epigenômicas. Câncer de pulmão,
mama, próstata, cabeça e pescoço, e cólon
também pode ser sensível a abordagens precoces de detecção.
Q&A
Eleftherios P. Diamandis
Peter W. Laird: Anormalidades epigenômicas
têm sido descritas para uma ampla variedade
de tipos de câncer. Perfis epigenômicos podem
ser usados para identificar subtipos moleculares
de um câncer específico, com resultados clínicos associados. Um exemplo disso seria os fenótipos metiladores da ilha CpG, descritos para
vários tipos de câncer. Padrões anormais da
metilação do DNA podem ser usados para detectar a presença de células malignas ou préneoplásicas em amostras luminais ou biópsias
de tecidos para cânceres em locais acessíveis.
Metilação do DNA anormal também pode ser
detectada no DNA flutuante derivado do tumor
na corrente sanguínea de alguns pacientes com
câncer. Tais testes são melhores aplicados aos
cânceres com procedimentos de detecção de
acompanhamento bem estabelecidos, tais como colonoscopia para câncer colorretal.
Paul Cairns: Existem estudos de viabilidade do
diagnóstico de todos os tipos câncer na literatura. Cânceres onde as biópsias dos tecidos são
obtidas, provavelmente possuem mais atração
porque eles geralmente contêm uma maior
proporção de células neoplásicas comparada a
uma amostra do fluido corporal.
Bharati Bapat: Todos os cânceres carregam alterações epigenéticas. Atualmente, testes de
detecção da metilação do DNA são oferecidos
para a detecção de cânceres de próstata, pulmão, cérebro, e também certos subtipos (mismatch-repair deficiente) de câncer do cólon.
Testes de detecção do microRNA também estão
atualmente sendo desenvolvidos para a detecção de câncer pancreático e outros cânceres.
Existem aplicações diagnósticas epigenômicas
além do câncer que tenham sido consideradas?
David Sidransky: Eu não sou perito nessa área.
Peter W. Laird: Caracterizações epigenômicas
estão em andamento para um grande número
de condições crônicas, incluindo doenças psiquiátricas e neurodegenerativas, doença cardiovascular, e diabetes. Dois principais problemas
confrontam o uso do diagnóstico epigenômico
para tais doenças. Primeiro, essas doenças ge-
Clinical Chemistry
ralmente não têm expansão clonal das células
com uma definida anormalidade epigenética, e,
segundo, o tecido alvo da doença pode não estar prontamente acessível para testes diagnósticos. Até o ponto em que a doença é reflexiva
de alterações epigenômicas sistêmicas, tecidos
substitutos tais como leucócitos podem ser fontes diagnósticas apropriadas de DNA ou cromatina.
Paul Cairns: Sim, para duas doenças, síndromes
de Prader–Willi e Angelman, onde uma minoria dos casos surge de um defeito na impressão.
Bharati Bapat: Outras aplicações diagnósticas
epigenéticas não são tão comuns quanto aquelas vistas para o diagnóstico do câncer. Entretanto, análise da metilação do DNA é um dos
testes diagnósticos oferecidos para doenças
genéticas hereditárias associadas com "impressão genômica," tais como síndrome de Angelman, síndrome de Prader–Willi, e síndrome de
Beckwith–Wiedemann. As duas últimas doenças
estão associadas com desenvolvimento do tumor.
Existem testes comerciais para diagnóstico
epigenômico no momento? Eles são usados na
clínica?
David Sidransky: A metilação da MGMT6 (O-6metilguanina-DNA metiltransferase) no glioblastoma e a metilação da GSTP1 (glutationa Stransferase pi 1) nos cânceres de próstata possuem estudos bem discutidos e são oferecidas
agora nos laboratórios da CLIA. Ambas provavelmente serão aprovadas relativamente em
breve pela Food and Drug Administration dos
Estados Unidos.
Peter W. Laird: Eu tenho um conflito de interesse em responder essa pergunta.
Paul Cairns: Hipermetilação do promotor gênico da MGMT para previsão de resposta para
temozolomida no tecido do glioma, GSTP1 e
APC (polipose coli adenomatosa) hipermetilação no tecido da biópsia da próstata para detecção de câncer, metilação da vimentina em
teste baseado nas fezes para câncer colorretal,
e SEPT9 (septina 9) metilação no DNA sérico
Q&A
Eleftherios P. Diamandis
para câncer colorretal. Também existem testes
para as síndromes de Prader–Willi e Angelman.
Bharati Bapat: Sim, detecção da metilação da
GSTP1 no câncer de próstata, MLH1 [mutL homólogo 1, câncer de cólon, metilação da não
polipose do tipo 2 (E. coli)] com deficiente
mismatch-repair para câncer de cólon, e metilação da MGMT no glioblastoma são alguns
exemplos de tais testes usados na clínica.
Qual é o seu ponto de vista sobre o futuro do
diagnóstico baseado na epigenômica para os
próximos 10 anos?
David Sidransky: Testes epigenômicos serão
incorporados à maioria dos tipos de câncer para
detecção precoce, prognóstico, e previsão para
resposta terapêutica.
Peter W. Laird: A aplicação clínica do diagnóstico epigenético provavelmente ficará restrita aos
marcadores de metilação do DNA no futuro
próximo. O custo rapidamente declinante do
sequenciamento genômico pode possibilitar o
perfil epigenômico de todo o genoma na próxima década. Entretanto, é mais provável que
abordagens de todo o genoma serão usadas
para descoberta do marcador em cenários de
pesquisa e que testes clínicos epigenéticos serão baseados nos genes individuais, ou em painéis de genes.
Paul Cairns: Para o câncer, dependerá do nosso
entendimento da carcinogênese precoce e em
particular a interface entre envelhecimento e
câncer. É possível que metilação aberrante se
torne um marcador de risco do câncer. Para
outras doenças comuns, como Andy Feinberg
ressaltou, alterações no epigenoma acumuladas
durante a vida são pelo menos uma causa subjacente para diferentes susceptibilidades à doença entre indivíduos quanto, digamos, polimorfismos de nucleotídeo único, e epigenômica
na doença com exceção do câncer está se tornando uma área de intenso estudo.
Bharati Bapat: Diagnóstico baseado na epigenômica é uma área emergente com grande potencial para aplicação clínica. No futuro, metila-
Clinical Chemistry
ção do DNA e testes baseados no microRNA
progressivamente serão incorporados como
biomarcadores do câncer de detecção e prognóstico precoces e como preditores de resposta
à terapia.
No contexto da medicina personalizada, como
a epigenômica irá interagir com outros desenvolvimentos diagnósticos moleculares?
David Sidransky: Epigenômica será uma das
várias alterações comumente testadas para definir a melhor maneira para lidar com ou tratar
um paciente com câncer.
Peter W. Laird: Perfis epigenômicos transmitem
informação que complementa a expressão gênica e perfis de mutação. Alguns subtipos moleculares, tais como fenótipos dos metiladores da
ilha CpG são mais facilmente reconhecidos pelo
perfil epigenômico do que pela expressão, número de cópias, ou perfil de mutação. Portanto,
eu antecipo que análise epigenômica será uma
ferramenta indispensável em nosso arsenal diagnóstico para selecionar as estratégias terapêuticas mais apropriadas para cada paciente.
Paul Cairns: Para o câncer, por exemplo, é muito provável que quimioresistência intrínsica e
adquirida terá um componente epigenético.
Inativação do BRCA1 (câncer de mama 1, início
precoce) por hipermetilação no câncer esporádico de mama ou de ovário deve conferir sensibilidade semelhante aos inibidores polimerase
poli(ADP-ribose) como as mutações pontuais de
inativação fazem nos cânceres hereditários. Para outras doenças humanas comuns é possível,
até mesmo provável, que conhecimento epigenômico seja tão importante quanto o genoma
na susceptibilidade.
Bharati Bapat: Detecção de um ou de vários
biomarcadores epigenéticos estará integrada
com outros testes de diagnósticos moleculares
para determinar as estratégias ideais para tratamentos de pacientes com câncer.
Notas de Rodpé
2 David Sidransky, Diretor, Pesquisa do Câncer
de Cabeça e Pescoço, Johns Hopkins Sidney
Kimmel Cancer Center, e Professor, Otolaringo-
Q&A
Eleftherios P. Diamandis
logia e Oncologia, Johns Hopkins University,
Baltimore, MD; e Presidente, Rede de Pesquisa
de Detecção Precoce, National Cancer Institute,
Rockville, MD.
3 Peter W. Laird, Diretor, USC Epigenome Center, Norris Comprehensive Cancer Center, Keck
School of Medicine, University of Southern California, e Professor Adjunto, Departamentos de
Cirurgia e de Bioquímica e Biologia Molecular,
University of Southern California, Keck School of
Medicine, Los Angeles, CA.
4 Paul Cairns, Professor Adjunto, Departamentos de Oncologia Cirúrgica e Patologia, Fox
Chase Cancer Center, Philadelphia, PA.
5 Bharati Bapat, Cientista, Samuel Lunenfeld
Research Institute, Mount Sinai Hospital, e Professor, Departamento de Medicina Laboratorial
e Patobiologia, University of Toronto, Toronto,
Ontario, Canada.
6 Genes humanos: MGMT, O-6-metilguaninaDNA metiltransferase; GSTP1, glutationa Stransferase pi 1; APC, coli polipose adenomatosa; SEPT9, septina 9; MLH1, mutL homólogo 1,
câncer de cólon, não polipose do tipo 2 (E. coli);
BRCA1, câncer de mama 1, início precoce.
Contribuições dos Autores: Todos os autores
confirmaram que eles contribuíram para o conteúdo intelectual desse paper e satisfizeram os
3 seguintes requisitos: (a) contribuições significantes para a concepção e design, aquisição de
dados, ou análise e interpretação dos dados; (b)
rascunhando ou revisando o artigo para conteúdo intelectual ; e (c) aprovação final do artigo
publicado.
Revelações dos Autores de Potenciais Conflitos
de Interesse: Na submissão do manuscrito, todos os autores completaram o formulário de
Revelações de Potenciais Conflitos de Interesse.
Potenciais conflitos de interesse:
Emprego ou Liderança: Nada a declarar.
Consultor ou Papel Consultivo: D. Sidransky,
Oncomethylome Sciences; P.W. Laird, Epigenomics; P. Cairns, Oncomethylome Sciences.
Clinical Chemistry
Q&A
Eleftherios P. Diamandis
Posse dos Valores: D. Sidransky, Oncomethylome Sciences; P. Cairns, Oncomethylome Sciences.
design do estudo, escolha dos pacientes inscritos, revisão e interpretação dos dados, ou preparação ou aprovação do manuscrito.
Honorários: P.W. Laird, Epigenomics.
1 Eleftherios P. Diamandis, Chefe da Bioquímica
Clínica, Departamento de Bioquímica Clínica,
Mount Sinai Hospital and University Health
Network, e Professor e Chefe, Divisão de Bioquímica Clínica, Departamento de Medicina Laboratorial & Patobiologia, University of Toronto,
Toronto, Ontario, Canada.
Fundo de Pesquisas: D. Sidransky, Oncomethylome Sciences.
Testemunho Hábil: Nada a declarar.
Papel do Patrocinador: As organizações patrocinadoras não desempenharam papel algum no
“This article has been translated with the permission of AACC. AACC is not responsible for the accuracy of the translation. The views
presented are those of the authors and not necessarily those of the AACC or the Journal. Reprinted from Clin Chem, 20 10; 56 no. 8
1216-1219, by permission of AACC. Original copyright © 2010 American Association for Clinical Chemistry, Inc. When citing this article,
please refer to the original English publication source in the journal, Clinical Chemistry.”
“Este artigo foi traduzido com a permissão da AACC. AACC não é responsável pela acurácia da tradução. Os pontos de vista apresentados são aqueles dos autores e não necessariamente os da AACC ou do Jornal. Reimpresso da ClinChem, 2010; 56 no. 8 1216-1219, por
permissão da AACC. Cópia original © 2010 American Association for Clinical Chemistry, Inc. Quando citar este artigo, por favor refira-se
à fonte de publicação original em inglês na revista,Clinical Chemistry.”