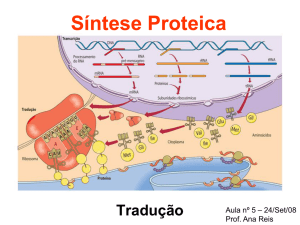
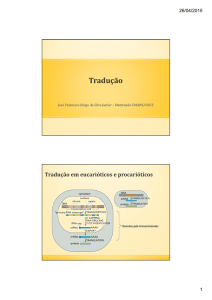
Tradução: do mRNA à Proteína
Faculdades de Medicina e Medicina Dentária da Universidade do Porto
Disciplina de Biologia Celular e Molecular
Penas, Ana; Silva, Ana A.; Silva, Ana N.; Fontes, Andreia; Ferreira, Luís
Introdução
As proteínas são efectores de um grande número de processos celulares, executando uma enorme variedade de tarefas. Uma célula eucariótica típica requer centenas de proteínas diferentes num dado momento. Assim, a questão impõe-se: como é feita a
síntese de proteínas? Para responder a esta questão, é oportuno debruçarmo-nos sobre a tradução, processo subjacente à síntese proteica. A tradução do mRNA é o primeiro passo na formação de uma proteína funcional. Seguidamente, a cadeia polipeptídica
deve dobrar-se na conformação tridimensional apropriada e passar por diversas etapas de processamento antes de atingir a sua forma activa. Da tradução, devemos analisar as suas 3 fases - iniciação, alongamento e terminação - e, de seguida, atender ao
modo como estas são reguladas. Embora a expressão da maioria dos genes seja regulada primariamente ao nível da transcrição, a expressão genética também pode ser controlada ao nível da tradução, e esse controle é um elemento fundamental da regulação
genética. Finalmente, afigura-se indispensável a ligação de todo o processo à prática da Medicina. Comecemos então esta viagem do mRNA à Proteína...
Alongamento
Iniciação
• Ligação dos Eucariotic Initiation Factors
eIF-1, eIF-1A, eIF-3 e eIF-5 à subunidade
40s do ribossoma.
• Ligação do eIF-2 (num complexo com
GTP) ao tRNA metionil iniciador.
• Reconhecimento do mRNA e transporte
deste ao ribossoma pelo grupo eIF-4,
sendo o 5’ cap do mRNA reconhecido
pelo eIF-4E.
• Ligação do eIF-4G ao eIF-4E e a uma
proteína (designada PABP- poly-A binding
protein) associada à cauda poli-A da
extremidade 3’.
Terminação
• Ribossoma com três locais para a
ligação do tRNA: locais P (peptidil), A
(aminoacil) e E (exit).
• Ligação do tRNA metionil iniciador ao
local P.
• Ligação do próximo tRNA aminoacil ao
local A, por emparelhamento com o
segundo codão do mRNA; este tRNA é
acompanhado até ao ribossoma por um
Eucariotic Elongation Factor – eEF-1α –
que forma um complexo com o GTP.
• Hidrólise do GTP a GDP (quando o
tRNA aminoacil correcto é inserido no
local A) e libertação do factor de
alongamento ligado ao GDP.
• Estabelecimento de ligação peptídica
entre a metionina iniciadora e o
segundo aminoácido, por acção da
subunidade 60s, após quebra da
ligação do eEF-1α ao ribossoma.
• Transferência da metionina para o
tRNA aminoacil do local A, seguida de
formação de um tRNA peptidil;
permanência do tRNA iniciador, sem a
metionina, no local P.
• Reconhecimento de ambas
as extremidades dos mRNAs
pelos eIFs, o que contribui
para o estímulo da tradução.
• Transporte do mRNA pelos
eIF-4E, eIF-4G, eIF-4A e eIF4B
à
subunidade
40s
ribossomal, ocorrendo uma
interacção entre o eIF-4G e o
eIF-3.
• Associação do tRNA metionil
e eIFs à subunidade 40s, que
percorre o mRNA até identificar
o codão de iniciação AUG.
• Ausência de tRNAs com anticodões complementares aos
codões STOP implica o reconhecimento destes pelo factor de
libertação eRF-1 (Eucatiotic Release Factor). Conjuntamente
com o eRF-1, actua o factor eRF-3, não envolvido no
reconhecimento dos codões STOP.
• Translocação, com intervenção do eEF2, acoplada à hidrólise do GTP.
• Durante a translocação, deslocamento,
em três nucleótidos, do ribossoma; este
posiciona o próximo codão num local A
vazio.
• Transferência do tRNA peptidil do local A
para o P e do tRNA iniciador desprovido
de metionina do local P para o E.
• Após identificação do codão
AUG, há o desencadear, pelo
eIF-5, da hidrólise do GTP ligado
ao eIF-2.
• Libertação dos factores de
iniciação (incluindo eIF-2 ligado
ao GDP) e junção da subunidade
60s à subunidade 40s para
formar o complexo de iniciação.
• Ligação de novo tRNA aminoacil ao local
A, induzindo à libertação do tRNA iniciador
do local E: ribossoma pronto para a
inserção de um próximo aminoácido na
cadeia polipeptídica em formação.
• Reconversão, durante o alongamento,
do eEF-1α libertado do ribossoma ligado
ao GDP à sua forma GTP, havendo
intervenção de um terceiro factor de
alongamento - eEF-1βγ; este une-se ao
complexo eEF-1 α/GDP e promove a troca
do GDP por GTP: o eEF-1α/GTP pode
acompanhar um novo tRNA aminoacil ao
local A, começando um novo ciclo de
alongamento.
• Translocação, no local A, de codão
STOP (UAA, UAG e UGA) determina fim
do alongamento.
Regulação da Síntese Proteica
Quer nos Eucariontes quer nos Procariontes, há uma regulação do processo de tradução. Assim, enumeramos alguns mecanismos de Regulação da
Tradução:
1. Ligação de proteínas repressoras: As proteínas repressoras vão ligar-se à extremidade 5’ do mRNA, inibindo deste modo a Iniciação.
2. Regulação da Actividade de Factores de Iniciação eIF-2 e eIF - 4E: No final da Iniciação, para que o eIF-2 se dissocie do GDP e fique disponível
para nova ligação ao GTP, tem que intervir o factor eIF-2B. Na inibição, que se sucede é que o eIF-2 sofre fosforilação e, de seguida, liga-se ao eIF-2B,
conduzindo à inactivação deste. Estando o eIF-2B inactivado, o eIF-2 não se dissocia do GDP, logo, não pode ser reutilizado. Por conseguinte, a síntese
de proteínas é inibida. No que se refere ao factor eIF – 4E, existem eIF - 4E binding proteins, que quando desfosforiladas se ligam a este factor impedindo
a sua ligação ao eIF – 4G. Por conseguinte, a regulação da síntese proteica resulta da fosforilação ou desfosforilação das eIF – 4E binding proteins.
3. Iniciação nos codões AUG downstream do local de início da tradução: A Iniciação começa não no primeiro codão AUG, mas no segundo ou
terceiro; é um mecanismo que conduz à produção de proteínas que diferem entre si apenas no terminal amino, sendo, por conseguinte, uma “estratégia
utilizada pelo gene” para produzir proteínas semelhantes, mas que vão ter, por exemplo, diferentes localizações celulares.
4. Locais de entrada internos no ribossoma: A tradução pode também iniciar-se em sequências especializadas de RNA - IRES (internal ribosome entry
site). Quando células eucariotas entram na fase M do ciclo celular regista-se uma redução de 25% na taxa total de tradução, uma vez que existe uma
desfosforilação do eIF-4E (diminui a afinidade para o 5’ cap). No entanto os mRNAs com IRES não sofrem o efeito do eIF-4E de modo que a sua tradução
aumenta quando a célula entra na fase M.
5. Mudança de estabilidade de mRNA: A síntese de proteínas codificadas por mRNA estável persiste muito tempo para além da repressão da transcrição
do gene. Nos Eucariontes, existem proteínas com um tempo de semi-vida muito longo. Outras, porém, são apenas necessárias por curtos períodos de
tempo e têm que ser expressas breve e intensamente, como acontece, por exemplo, com os factores de tradução. Nestas proteínas, o mRNA
correspondente tem um tempo de semi-vida curto, sendo que a sua expressão é controlada por activação e desactivação da transcrição dos seus mRNAs,
que se traduzirá, naturalmente, numa activação e desactivação da tradução. Muitos destes mRNAs têm múltiplas cópias da sequência AUUUA, geralmente
localizadas na UTR (untranslated region) da extremidade 3’.
6. Regulação do tamanho da cadeia poli-A dos mRNAs dos Ovócitos: Nos ovócitos, existem mRNAs cuja cadeia poli-A tem cerca de 20 nucleotídeos;
estes mRNAs armazenados são utilizados para a tradução num estádio de desenvolvimento apropriado através do alongamento da cadeia poli-A até
centenas de nucleotídeos. É, pois, através da regulação do tamanho da cadeia poli-A que se faz a regulação da tradução.
7. RNA interference que silencia a expressão de genes: Nas células eucarióticas existe um mecanismo de degradação de RNA, designado RNAi, que é
utilizado como mecanismo de defesa, destruindo moléculas de RNA estranho. Este actua com a formação de cadeias duplas de RNA que não são
traduzidas e, por isso, são facilmente degradadas.
• Ligação dos factores eRF-1 e eRF-3 a um codão de
terminação no seu local A que conduz à hidrólise da ligação
entre o tRNA e a cadeia polipeptídica no local P, resultando na
libertação do polipeptídeo completo do ribossoma.
• Libertação do tRNA e dissociação
ribossomais e do mRNA molde.
das
subunidades
Ligação à Medicina
Wang, C. e Pflugheber, J. (2003): Alpha Interferon Induces Distinct Translational Control Programs To Suppress
Hepatitis C Virus RNA Replication
• A infecção pelo vírus de hepatite C (HCV) é um grave problema de saúde pública. Dados recentes revelam que
mais de 170 milhões de pessoas em todo o mundo são infectadas, sendo que estudos epidemiológicos
identificaram o vírus da hepatite C como a principal causa de hepatite crónica e de doença hepática na população
humana.
• A infecção com o HCV é frequentemente tratada com uma terapia que recorre a um alfa interferão (α-IFN). Os
IFNs são produzidos pela célula em resposta à infecção vírica. A exposição da célula aos IFNs leva, por sua vez, à
expressão induzida de genes estimulados pelo IFNs (ISG), estando a maioria deles envolvidos em funções
antivíricas. Assim, acredita-se que a acção dos ISGs pode limitar a replicação do vírus.
• Há, pelo menos, três vias de controlo de tradução a nível celular desencadeados como resposta à presença de
vírus e, por conseguinte, de INFs: a via da sintétase do 2’-5’ oligoadenilato (OAS)/ RNase L; a via da cínase da
proteína R (PKR) – subunidade alfa do factor de iniciação eucariótico (eIF-2α); a via do P56-eIF-3.
• A activação da enzima RNase L pelo OAS pode levar a uma supressão na tradução através da clivagem e
consequente inactivação do rRNA 28s.
• A activação da PKR durante a infecção vírica conduz à reacção de fosforilação na serina-51 do factor de iniciação
eIF-2α. Esta fosforilação pode bloquear a iniciação na tradução, uma vez que reduz o pool de eIF-2α, bloqueando a
ligação do tRNA metionil à subunidade 40s.
• A P56 foi identificada como sendo uma proteína de ligação ao eIF-3, supressora da tradução.
• Em suma, este estudo caracterizou os mecanismos moleculares de actuação do IFN contra a replicação do vírus
HCV, caracterizando as vias da PKR e P56 enquanto reguladores dos programas de tradução do HCV através da
restrição no recrutamento de RNA viral nos ribossomas.
Fig.1: Mecanismos de Repressão da Tradução do RNA do HCV (Wang, C., Pflugheber, J. (2003))
Este estudo é, pois, um exemplo elucidativo de que, actuando ao nível da tradução, pode encontrar-se a chave para
a cura de muitas doenças.
Bibliografia
• Wang, C., Pflugheber, J..(2003) Alpha Interferon Induces Distintc Translational Control Programs to Supress Hepatitis C Virus RNA Replication. Journal of Virology.
pp. 3898-3912, Vol. 77, No. 7
• Nelson D.L., Cox M.M. Lehningher Principles of Biochemistry (4th Ed.). W.H. Freeman and Company. New York. 2005.(p.1034).
• G.M. Cooper. "The Cell. A Molecular Approach. (3rd Ed.). AMS Press. U.S.A. 2004. (pp. 281-317).
• Alberts et al. "Molecular Biology of the Cell." - (4th Ed.). Inc. New York. 2002.
• http://apps.bioneq.qc.ca/twiki/bin/view/knowledgebase/Translation