Um comparativo entre ontologias relacionadas ao Câncer: a
interoperabilidade destas ontologias com a Ontocancro
A comparison between ontologies related to cancer: the interoperability of
these ontologies with Ontocancro
Laís Falcade, Universidade Federal de Santa Maria, Brasil, [email protected]
Karine Rodrigues de Souza, Universidade Federal de Santa Maria, Brasil,
[email protected]
Karlise Soares Nascimento, Universidade Federal de Santa Maria, Brasil,
[email protected]
Giovani Rubert Librelotto, Universidade Federal de Santa Maria, Brasil, [email protected]
Resumo
A complexidade existente nas pesquisas sobre o câncer produz uma grande quantidade de
informação, que está distribuída sem padronização de alguns termos dificultando a busca por
dados genéticos. Com base nisso, a Ontocancro foi desenvolvida através da implementação de
uma ontologia para o estudo de vias metabólicas envolvidas no processo carcinogênico. Este
trabalho tem como objetivo reconhecer ontologias existentes sobre câncer visualizando os
métodos e ferramentas utilizadas, comparando-as com a ontologia Ontocancro 2.0, que estuda
dados genéticos de quatro tipos de câncer, a fim de buscar o aperfeiçoamento da mesma. Dentro
dos projetos encontrados, foi ressaltada a ontologia ACGT que traz a proposta de formar um
estudo semântico sobre os dados genéticos e clínicos de três tipos de câncer. Esta proposta será
comparada a Ontocancro, visando os benefícios de sua união com o projeto.
Palavras chave: Ontologias, Ontocancro, Câncer.
Abstract
The existing complexity in cancer research produces a lot of information, which is distributed
without standardization of some terms thus hindering the search for genetic data. Based on this,
the Ontocancro was developed through the implementation of an ontology for the study of
metabolic pathways involved in the carcinogenic process . This work aims to recognize existing
ontologies on cancer by analyzing the used methods and tools, comparing them with Ontocancro
2.0 ontology , which studies genetic data of four types of cancer, in order to seek the
improvement of it . Within the found projects, it was highlighted the ACGT ontology that
proposes the semantic study of genetic and clinical data from three types of cancer. This
proposal will be compared to Ontocancro , seeking the benefits of its union with the project.
Key Words: Ontologies , Ontocancro , Cancer
1. INTRODUÇÃO
No início dos anos 1950, começou uma revolução científica com a determinação da estrutura do
DNA. Com isso, foi iniciada uma corrida para o mapeamento e interpretação dessa estrutura a fim
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
03 e 04 de Outubro de 2014, Évora, Portugal
ISSN 2183-489X
DOI http://dx.doi.org/10.18803/capsi.v14.308-325
308
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
de entender melhor os processos fisiológicos dos seres vivos e buscar soluções para a cura de
determinadas doenças Junior et al. [2012]. A partir da necessidade de guardar tais informações de
maneira eficiente, os cientistas da computação foram inseridos nesse contexto, dando origem a
bioinformática.
As informações coletadas por biólogos nesses últimos anos estão disseminadas por diversos bancos
de dados, que trabalham com vários tipos de informações, nem sempre dispostas em formato padrão.
Cada banco pode possuir um formato de apresentação desses dados e por tal motivo, dificulta a
utilização dos mesmos para outras pesquisas.
Para filtrar esses dados com vocabulários controlados que restringem as palavras, alguns bancos
foram criados a partir de repositórios públicos utilizando ontologias, que são especificações
explicitas de uma conceitualização [Gruber 1993], ou seja, elas permitem uma “especificação formal
de termos e seus relacionamentos” [Simão et al. 2010]. Esses bancos buscam informações mais
especificas de determinado grupo de informações, como é o exemplo da Ontocancro [Librelotto et
al. 2009] [Nascimento et al. 2009], que traz dados de quatro tipos de Câncer, bem como outros
elementos que os acompanham, como genes, vias e amostras, além de outras informações relevantes
para seu estudo.
Na mesma linha da Ontocancro, pode ser encontrada a ACGT Master Ontology [Brochhausen et al.
2011] que busca um formato padrão de conceitos para o câncer de mama, além de dados técnicos e
clínicos para tratamentos. Também podemos encontrar na rede, ontologias para estudos biológicos
e ferramentas que auxiliam na comparação de ontologias, como é o exemplo do SBO (Systems
Biology Ontology) [Courtot & Juty 2011], TEDDY (TErminology for the Description of Dynamics)
[Courtot & Juty 2011] e AgreementMaker v0.23 [Cruz et. al. 2009].
Com base nestas ontologias, foi realizada uma pesquisa para averiguar as diferenças e semelhanças
destas com a Ontocancro. Além disso, foi analisado o objetivo de cada ontologia com o intuito de
reutilizar métodos comprovados como eficazes no estudo oncológico e fazer uma comparação da
Ontocancro com a ACGT, que está disposta na mesma linha de conhecimento.
O artigo está organizado da seguinte forma: Seção 2 será mostrado um estudo de Sanchez et al.
[2004] sobre os tipos de informações encontradas em bancos de dados e ontologias, e o que eles
podem ajudar na pesquisa sobre o câncer. Na Seção 3 serão encontradas especificações sobre as
ontologias, facilitando o entendimento sobre o que ela representa e no que pode ajudar no estudo de
doenças. Na Seção 4 poderá ser vista uma determinação do câncer, foco deste trabalho. A Seção 5
apresenta as ontologias já citadas anteriormente. Na Seção 6 é realizado um comparativo entre as
ontologias estudadas na Seção 5. Seguido da Seção 7 que traz a interoperabilidade das mesmas com
a Ontocancro, finalizando com a Seção 8 que traz algumas considerações sobre o trabalho.
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
309
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
2. BIOINFORMÁTICA E INFORMÁTICA MÉDICA
Sanchez et al. [2004], em sua pesquisa, revisou os resultados do BIOINFOMED, um estudo
financiado pela Comissão Europeia e, através deste estudo, foram identificados dois ramos que
buscam, através do tratamento de dados, a prevenção e diagnóstico de doenças de forma antecipada.
Um desses ramos é a Bioinformática que trabalha com dados laboratoriais, estudados por biólogos
pesquisadores e informatas. São, em sua maioria, dados genômicos encontrados em repositórios do
projeto Genoma Humano. Outro ramo é a Informática Médica, que trabalha com dados de tratamento
clínico hospitalar. Esta busca gerar informações através de estudos de casos reais.
No trabalho de Sanchez et. al. [2004] é sugestionado uma união dessas duas áreas, em favor da
complementariedade das informações obtidas dentro do estudo genômico da bioinformática com as
análises de ensaios clínicos reais da informática médica. Essa união geraria informações mais
completas possibilitando um estudo mais apurado de doenças, formas de cura, prevenção e
resultados negativos e positivos de tratamentos.
Para que os dados dessas duas áreas sejam unidos de forma a gerar um conhecimento mais completo
sobre possíveis doenças, a utilização de ontologias e axiomas vem sendo estudada pelos informatas.
Hoje, na rede de internet, já existem algumas ontologias que trazem o estudo biológico como fator
importantíssimo para o estudo e prevenção de diversas doenças que é o caso da ACGT e da
Ontocancro, que tem como foco principal o câncer.
Sanchez et al. [2004] ainda propôs uma série de requisitos para um sistema que abranja os dados
genômicos e clínicos, categorizando-se como um sistema mais completo para ser utilizado por
médicos clínicos e pesquisadores, bem como bioinformatas que estudam a evolução de doenças.
Esses requisitos estariam dispostos dentro de uma única ontologia, facilitando a busca por
informações de casos já ocorridos, agilizando os resultados de tratamentos.
3. ONTOLOGIA
Tem se tornado cada vez mais difundido na área de ciência da computação, a pesquisa sobre
ontologias, sendo utilizada em diversas áreas de investigação, como: engenharia do conhecimento,
representação do conhecimento, modelagem qualitativa, engenharia da linguagem, design de banco
de dados, modelagem da informação, integração de informações, objeto orientado a análise,
recuperação e extração de informação, gestão e organização do conhecimento e projeto de sistemas
baseados em agentes. A ontologia tem sido utilizada nas mais diversas áreas de aplicação, incluindo,
integração empresarial, tradução da linguagem natural, medicina, engenharia mecânica,
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
310
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
padronização do produto do conhecimento, sistemas de comércio eletrônico, sistemas de informação
geográfica, sistemas de informação jurídica e sistemas de informação biológica [Guarino 1998].
Segundo Guarino [1998], ontologia pode ser considerada como um conjunto de axiomas lógicos
projetados para explicar o significado de um vocabulário pretendido, podendo especificar uma
conceituação única de forma muito indireta, uma vez que só podem aproximar-se de um conjunto
de modelos destinados, e este conjunto pode ter apenas uma fraca caracterização de uma
conceituação. Assim, as ontologias podem ser classificadas de acordo com sua precisão na
caracterização de um conceito. Uma ontologia pode aproximar-se de um conceito desenvolvendo
um axioma mais rico ou adotando um domínio mais rico de relações conceituais relevantes.
Gruber [1993] fala sobre a importância de definir um vocabulário comum para a representação do
conhecimento compartilhado, para que sistemas possam compartilhar e reutilizar esta representação
formal. Esta definição, na qual é especificado um vocabulário para representação de um domínio
através de classes, relações e funções, é chamado de ontologia. Uma ontologia é uma especificação
explícita de uma conceitualização, e uma conceitualização é uma ‘visão simplificada do mundo que
desejamos representar para algum propósito’.
A integração de informações é uma grande área de aplicação para ontologias, porém ainda existem
problemas para realizar esta integração. Mesmo dois sistemas adotando o mesmo vocabulário, não
existem garantias que ambos irão concordar com determinada informação, a menos que se
comprometam com a mesma conceitualização [Guarino 1998].
Gruber [1993] ressalta que a ontologia de um sistema pode ser descrita através da definição de um
conjunto de termos de representação, redigida de acordo com algum formalismo comum, no entanto,
diferentes aplicações requerem diferentes tipos de raciocínio, línguas e tem propósitos especiais,
tornando um problema a portabilidade de ontologias entre múltiplos sistemas.
Como resolução deste problema, Guarino [1998] sugere que se entre em acordo sobre uma única
ontologia de mais alto nível, ao invés de depender de acordos com base no cruzamento de diferentes
ontologias, desta forma, podem ser desenvolvidos diferentes tipos de ontologias de acordo com seu
nível de generalidade.
A Figura 1 ilustra os diferentes tipos de ontologias propostas por Guarino [1998], que podem ser
consideradas como ontologia de nível superior ou genérica, ontologia de domínio, ontologia de
tarefa e ontologia de aplicação. As ontologias de nível superior descrevem conceitos gerais que são
independentes de um problema ou domínio particular. Já as ontologias de domínio e ontologias de
tarefa, descrevem, respectivamente, o vocabulário relacionado com o domínio genérico (como
medicina ou automóveis) ou uma tarefa ou atividade genérica (como diagnosticar ou vender),
especializando os termos introduzidos na ontologia de nível superior. Por último, as ontologias de
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
311
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
aplicação descrevem conceitos dependendo tanto de um domínio específico como tarefa, que muitas
vezes são especializações de ambas as ontologias relacionadas.
Figura 1: Tipos de ontologias
Estes conceitos frequentemente correspondem a papéis desempenhados por entidades de domínio
durante a execução de uma determinada atividade, como unidade substituível ou componente de
reposição. É importante ter em mente que uma ontologia pode ser utilizada não somente na
construção de um novo sistema, como também ser igualmente utilizada para a reengenharia, a fim
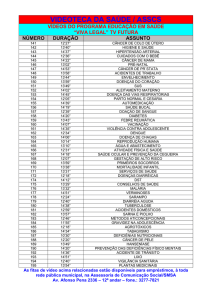
de aumentar o reuso e a facilidade de manutenção.
4. CÂNCER
O crescimento desordenado de células, que invadem órgãos e tecidos, é o que caracteriza o câncer,
podendo espalhar-se para outras regiões do corpo, conhecido como metástase. Estas células
dividem-se rapidamente, sendo muito agressivas e incontroláveis, o que determina a formação de
tumores ou neoplasias malignas.
Segundo Librelotto et al. [2009], a neoplasia ocorre devido a um desequilíbrio entre a proliferação
celular e o desgaste celular. O câncer se desenvolve através da mutação de um ou mais genes que
regulam o crescimento e morte celular programada. Esta morte celular programada é chamada de
apoptose, que é extremamente regulado e eficiente, na qual requer a interação de inúmeros fatores
[Grivicich et al. 2007].
De acordo com Grivicich et al. [2007], o câncer tem características muito heterogêneas, porém todos
os tumores malignos crescem além dos limites impostos às células normais. Além disso, evidências
demonstram que a característica mais marcante da maioria dos tumores malignos é a resistência à
apoptose.
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
312
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
Segundo a Agência Internacional para Pesquisa sobre Câncer, da ONU [Wild 2014], o câncer é a
doença que mais mata no mundo, sendo de total importância o seu diagnóstico antecipado e, por que
não, a sua prevenção, através de estudos de fenótipo de cada pessoa. Este estudo pode vir desde a
revisão genômica do indivíduo (dados bioinformáticos), até seus hábitos e vícios rotineiros
(avaliação clínica na informática médica), que por conseguinte levarão a uma possível mutação
gênica resultando em uma metástase, o câncer.
5. ESTUDOS BIOLÓGICOS E ONTOLOGIAS
As ontologias vem sendo incluídas em vários ramos para auxiliar no estudo de informações de
grande porte, que é o caso de dados biomédicos. Essas ontologias auxiliam na interpretação de
informações através da web semântica, possibilitando visualizar grupos de dados correlacionados,
no caso da bioinformática auxiliar na detecção e prevenção de doenças hereditárias e de mutação
gênica que já foram sequenciados pelo Projeto Genoma e estão disponíveis em bases de dados
públicas.
Esse tipo de pesquisa ainda não traz dados de tratamentos juntamente com os dados de detecção e
prevenção pois, para o primeiro, é necessária informação médica de pessoas doentes que fizeram o
tratamento hospitalar, já no segundo e terceiro caso, as informações coletadas são a nível genético
antecipado, sendo observadas a partir do DNA do indivíduo, coletando probabilidades.
Nas seções a seguir serão apresentadas as ontologias citadas anteriormente, destacando suas
peculiaridades, características e definições.
5.1. Ontocancro
A Ontocancro é um trabalho que vem sendo desenvolvido por pesquisadores da área da biologia
molecular, física e computação. Na primeira etapa, chamada de Ontocancro 1.0, foi realizada uma
pesquisa para centralizar dados a fim de permitir uma análise consistente de informações extraídas
de bancos de dados públicos referentes ao estudo de genes humanos potenciais ao câncer, buscando
a padronização dos dados encontrados através de uma ontologia [Simão et al. 2010].
Na Ontocancro 2.0, foram realizadas investigações do processo de ativação da barreira anticâncer
proposto pelo pesquisador Halazonetis et al. [2008], sendo acrescentado ao estudo, um cálculo
estatístico que verifica as redes de manutenção do genoma, identificando as vias metabólicas que se
encontram mais ativas na fase inicial do câncer, possibilitando resultados que comprovam a
expressão de genes no impedimento do processo de evolução tumoral [Nascimento et al. 2009]. Na
Figura 2 podemos verificar a estrutura da ontologia Ontocancro 2.0.
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
313
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
Figura 2 – Principais classes da ontologia Ontocancro 2.0
Dentre as informações encontradas no banco de dados, podemos visualizar 977 genes distribuídos
em 48 vias metabólicas ou pathways, acompanhadas das amostras de estudos em tecidos (ou
samples) de quatro tipos de câncer que ocorrem nos seres humanos.
5.1.1 Sistemas de reparo
Nas células cancerosas podemos encontrar grandes e pequenas alterações no seu material genético,
incluindo a falta ou adição de cromossomos, além de genes mutantes e alterações genéticas devido
a caminhos disfuncionais ocorridos durante o reparo do DNA e estabilidade cromossômica, essas
alterações são decorrentes da perda de Mecanismos de Manutenção Genômica (GMM).
Esses GMMs são mecanismos de reparo para proteger o DNA contra danos, como as quebras de
cadeias de DNA ocasionado pela radiação ultravioleta. Os sistemas de reparo se constituem em redes
genéticas especializadas nesta proteção, uma vez que impedem que diferentes tipos de danos sejam
fixados no material genético. Em células cancerosas, essas redes de proteção não funcionam
corretamente, ocasionando uma série de mutações.
A disfunção destas vias pode provocar inúmeros tipos de anomalias celulares, onde uma delas é o
câncer. As vias se dividem de acordo com a sua função. Por exemplo, na via de Reparação por
Excisão de Base (BER) encontra-se um conjunto de genes envolvidos diretamente no reparo das
bases do DNA, assim como as outras vias de reparação, tais como, Reparação por Excisão de
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
314
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
Nucleotídeos (NER), Reparação por Mau Pareamento (MMR) e Recombinação Homóloga (HR),
entre outras, também auxiliam na manutenção e no reparo do DNA.
Em outros casos, aparecem as vias de Ciclo Celular (CC), responsáveis pelo processo de duplicação
do DNA. Outra via importante é a via de Estabilidade Cromossômica (CS), que coordena todas as
proteínas responsáveis pela estabilidade dos cromossomos. Se algumas destas vias não são capazes
de corrigir ou atuar de forma adequada, entra em função a via de Apoptose, que tem como função
destruir a célula ou então o DNA.
5.2. ACGT Master Ontology
Nas pesquisas em respeito ao câncer, são utilizadas ferramentas de alto rendimento que resultam em
uma “explosão” de informações e conhecimento sobre os diversos tipos de câncer e seu tratamento
[Brochhausen et al. 2011]. Isto é devido a complexidade deste grupo de doenças que afetam uma
porção significativa da população. A falta de estrutura de informação aberta e compartilhada muitas
vezes impede que instituições de pesquisa possam visualizar e integrar dados de diferentes níveis de
investigação, por exemplo, para integrar dados de estudos voltados para os elementos moleculares
de câncer com aqueles focados no que ocorre no nível de órgãos.
A visão do projeto ACGT (Advancing Clinico-genomic Trials on Cancer – Open Grid Services for
Improving Medical Knowledge Discovery) é contribuir para a resolução destes problemas através
de uma ontologia orientada. Seu objetivo final é o desenvolvimento de uma rede de serviços de
infraestrutura semântica para facilitar o acesso de forma transparente e segura a bancos de dados
heterogêneos distribuídos em vários níveis, com o intuito de fornecer uma gama de semânticas
reutilizáveis, utilizando ferramentas abertas para análise dos mesmos integrando dados clínicos
genômicos de vários níveis.
A ACGT Master Ontology é um projeto financiado pela União Européia, teve início em 2006, foi
implementada em OWL e tem sido ampliada para atender as necessidades tanto técnicas quanto
clínicas. Representa de forma abrangente o domínio de pesquisa e tratamento de câncer, em especial
o câncer de mama, tumor de Wilms [Garvin et al. 1993] e tumor Rhabdoid [Biegel et al. 1999].
Atualmente possui 1667 classes, 288 propriedades de objetos, 15 propriedades de dados e 61
indivíduos.
5.3. SBO
A SBO (Systems Biology Ontology - Ontologia de Sistemas Biológicos) descreve as entidades
usadas na modelagem computacional, fornecendo um conjunto de conceitos inter-relacionados que
podem ser utilizados para especificar, por exemplo, o tipo de componente que está sendo
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
315
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
representado em um modelo, ou a função destes componentes em descrições de sistemas biológicos
[Courtot & Juty 2011].
É uma ontologia aberta e está disponível em diferentes formatos como OBO, OWL e SBO-XML,
podendo ser acessada através de web services. A documentação relacionada e recursos associados
estão disponíveis gratuitamente em http://biomodels.net/sbo/. É desenvolvida como uma ontologia
padrão, respeitando o conjunto de princípios comuns de desenvolvimento.
5.4. TEDDY
TEDDY é uma ontologia projetada para o alinhamento e comparação de sequências biológicas
[Courtot & Juty 2011]. É composta por quatro ramos: a classificação dos comportamentos concretos
temporais observados em uma simulação (trajetórias), as diversificações e as relações entre os
comportamentos, as características de comportamentos específicos e os motivos funcionais
geradores de determinados tipos de comportamentos. Tem como objetivo fornecer uma ontologia
para comportamentos dinâmicos, fenômenos dinâmicos observáveis e elementos de bio-modelos e
sistemas biológicos. Os termos TEDDY podem ser suficientes para qualificar, com níveis variáveis
de detalhe, as características críticas de resultados numéricos obtidos a partir de simulações, bem
como aqueles a partir de medições experimentais. Está codificada em OWL.
5.5. Agreement Maker
O Agreement Maker é um sistema composto de uma grande variedade de métodos de
correspondência entre ontologias, abordando diferentes níveis de granularidade dos componentes
que estão sendo correspondidos. Consiste em “definir mapeamentos entre esquema ou elementos da
ontologia que são semanticamente relacionados”, considerando alguns domínios e aplicações, como
geoespacial, ambiental e biomédica. As informações conceituais destas aplicações são armazenadas
em forma de ontologia [Cruz et al. 2009].
6. OBJETO DE ANÁLISE
Segundo Bodenreider [2008], a função da ontologia na área biomédica é padronizar o
armazenamento de dados para que os mesmos tornem-se interoperáveis entre sistemas eletrônicos
de saúde. Esta padronização é fundamental para facilitar a manutenção, compartilhamento e
interoperabilidade entre diversos sistemas, além da integração heterogênea de dados a partir de
diferentes fontes de dados. De acordo com Sanchez et al. [2004], o conhecimento gerado por grandes
bases de dados biomédicos permitirá que as organizações de cuidados de saúde possam identificar
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
316
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
cidadãos que, não apenas tenham risco genético, mas também doenças cujos riscos de desenvolver
sintomas possam ser reduzidos por uma ou mais intervenções.
O maior conhecimento do genoma humano apoia a teoria de que as doenças devem ser entendidas
considerando as complexas interações entre genes e fatores ambientais que desencadeiam processos
patológicos e definem o fenótipo. Novas terapias para doenças estabelecidas e novas intervenções
para a prevenção de doenças estão sendo desenvolvidas e discutidas, porém estas abordagens
inovadoras não podem ser sustentadas sem efetivamente trabalhar com os dados gerados em
laboratório nas áreas de genômica funcional, estrutural e proteômica. Desta forma, torna-se
importante a integração de dados clínicos, que são gerados por registros médicos, vinculando o
atendimento ao paciente e a saúde pública. A informática biomédica visa criar uma informação
conceitual comum para promover a descoberta de novos diagnósticos e métodos terapêuticos,
integrando informações médicas e biológicas [Sanchez et al. 2004].
Ontologias tem sido utilizadas em uma grande variedade de aplicações e tornaram-se um recurso
importante na investigação biomédica. Muitas aplicações utilizam ontologias como fonte de
conhecimento computável, incluindo aplicações de processamento de linguagem e sistemas de apoio
à decisão, sendo fundamentais também para a descoberta de conhecimento em uma abordagem
direcionada para a pesquisa biomédica [Bodenreider 2008].
A ACGT consiste na adoção de uma BFO (Ontologia Básica Formal), a taxonomia BFO faz uso de
uma base de nível superior que faz distinção entre dois tipos de entidades: continuants e ocurrents.
As entidades continuants podem ser subdividas em dependente, independente ou região espacial,
enquanto as ocurrents podem ser subdividas em região temporal, região espaço temporal ou entidade
processual. As entidades continuants perduram através do tempo, mantendo sua identidade,
enquanto as ocurrents são entidades que podem acontecer, se desdobram ou desenvolvem com o
tempo [Cocos 2008]. Neste primeiro momento serão nosso alvo de interesse as entidades continuants
dependentes, continuants independentes e ocurrents entidade processual.
A característica que define continuants dependentes é que são conceitos inerentes de outro conceito
(ou seja, continuants independentes), por exemplo, o tamanho da superfície de um fígado, o sexo de
um ser humano, a doença de um organismo, etc. As continuants independentes são conceitos
definidos pelas dependentes, por exemplo, ser humano, fígado, laboratório, etc. Entidades
processuais são entidades que podem ocorrer e sempre depende de uma entidade continuant, por
exemplo, a vida de um organismo, o processo de meiose, os processos de biópsia, exame clínico,
desenvolvimento do tumor, processo de divisão celular, etc [Cocos 2008].
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
317
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
7. COMPARATIVO ENTRE AS ONTOLOGIAS
Conforme a pesquisa realizada, foi possível observar que hoje em dia existem poucos estudos na
área de genética, principalmente relacionados ao câncer, não sendo encontradas muitas ontologias.
Entre as ontologias citadas anteriormente, somente a Ontocancro e a ACGT tratam especificamente
de genes relacionados à doença, as demais propostas apresentadas trazem expectativas diferentes
quanto a criação e a atualização de uma ontologia já existente referente ao estudo biológico, o que
favorece com o intuito desse trabalho que é aperfeiçoar a ontologia Ontocancro.
O Quadro 1 denota as propriedades de cada ontologia a ser comparada, podendo assim ter uma visão
geral do que as mesmas representam, qual seu intuito e o que realmente desejam alcançar.
Ontologia
Ontocancro 2.0
ACGT
TEDDY
SBO
Agrement Maker
Master
Tipo de Estudo
Carcinomas humanos
adrenocorticais ,
Carcinomas humanos
pancreáticos,
Câncer de
Mama,
Tumor de
Wilms,
Tumor de
Carcinomas humanos Rhabdoid
da tireoide
Descreve as
Realiza
Define
comparações de entidades usadas mapeamentos entre
na modelagem
sequências
esquema ou
computacional
biológicas com
elementos das
ontologias que são
comportamentos
dinâmicos
semanticamente
relacionados
Carcinomas humanos
colorretal
Sintaxe
OWL
OWL
OWL
OBO OWL
OWL
SBO-XML
Download
Inferência/
Sim
Sim
Sim
Sim
Sim
Não
Não
Não
Não
Não
68
1667
-
-
-
58
288
-
-
-
42
15
-
-
-
Reasoning
Classes
Propriedades
de Objetos
Propriedades
de Dados
Quadro 1 – Propriedades das Ontologias.
Atualmente, a Ontocancro armazena informações de vias genéticas humanas, que possuem várias
sub-vias do ciclo celular, mecanismos de reparo do DNA, apoptose e estabilidade cromossômica,
tendo como principal objetivo visualizar a interação dos genes com cada uma das situações citadas,
e a partir desse estudo, detectar com antecedência fatores de risco para o desenvolvimento dos quatro
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
318
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
tipos de câncer vistos no Quadro 1. Em seu estudo de dados biológicos e clínicos, Sanchez et al.
[2004] considera uma ontologia completa aquela que armazena tanto dados genéticos quanto
clínicos, portanto, podemos considerar a ACGT-MO como uma ontologia completa. O objetivo da
mesma é alcançar a existência de apenas uma ontologia relacionada a câncer na qual se torne possível
a integração semântica, distribuição e armazenamento de dados clínicos e genômicos, sendo possível
o estudo desde a doença detectada até seu tratamento.
Essas duas ontologias buscam especificamente o estudo de carcinomas, cada um com seus
resultados, mas com um mesmo objetivo: unir as informações de diversas fontes de dados em uma
mesma ontologia. Esses dados são de fundamental importância para cada um dos projetos, pois
disponibilizam os resultados de cada tratamento, para cada tipo de câncer, podendo, em trabalhos
futuros, chegar à prevenção e detecção de doenças letais
Segundo Sanchez et al. [2004], os resultados do projeto genoma humano tem atraído o interesse de
muitos profissionais e está introduzindo novos desafios que irão transformar a pesquisa biomédica
e assistência à saúde. As pesquisas nas áreas de bioinformática e informática médica possuem
interesses e objetivos diferentes, desta forma, uma característica da era pós-genômica, será
correlacionar informações genotípicas essenciais com informações fenotípicas expressas. Este
correlacionamento é buscado através da integração entre as ontologias ACGT e Ontocancro.
As ontologias SBO e TEDDY são relacionadas a área de modelagem computacional, onde a SBO
descreve as entidades usadas na modelagem computacional e TEDDY é projetada para a comparação
e alinhamento de sequências biológicas. As atividades desenvolvidas por essas duas ontologias, em
linhas gerais, visam o aperfeiçoamento de outras ontologias, possibilitando extrair informações mais
significativas, facilitando o mapeamento dos termos que estão sendo utilizados. Pode-se ver pelo
Quadro 1, que todos os projetos possibilitam o download dos estudos realizados, cada um em seu
formato, como por exemplo a estrutura e os dados das ontologias Ontocancro e ACGT, e ferramentas
de alinhamento que é o caso do SBO, do TEDDY e do AgreementMaker. Nenhum destes, entretanto,
possui propriedades de inferência e raciocínio, impossibilitando a troca de informações entre o banco
de dados e os pesquisadores.
8. INTEROPERABILIDADE DAS ONTOLOGIAS RELACIONADAS AO CÂNCER COM A
ONTOCANCRO
Após o comparativo realizado na seção anterior, percebe-se que a Ontocancro é a ontologia
relacionada ao câncer que mais possui informação relacionada a esta doença em linhas laboratoriais
e bioinformáticas. Entretanto, sabe-se que ela não contempla o domínio em questão completo. Desta
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
319
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
forma, esta seção traça a integração do conhecimento da Ontocancro com as demais ontologias
encontradas.
A ontologia TEDDY fornece classes e propriedades relacionadas ao alinhamento de sequências
genéticas que serão úteis no processo de comparação de sequências genéticas encontradas na
Ontocancro com resultados laboratoriais. Desta forma, criou-se propriedades de objetos para
relacionar as classes da TEDDY com a classe Gene, da Ontocancro.
Este processo de alinhamento pode ser realizado através da modelagem computacional descrita na
ontologia SBO. Portanto, faz-se necessário a agregação destas classes da SBO para relacionar com
as doenças que a Ontocancro descreve, como o câncer do pâncreas e da tireoide, entre outros.
De todas as ontologias relacionadas ao câncer encontradas, a que mais se aproxima da Ontocancro
é a ACGT. Esta é rica em classes e relações que descrevem as informações clínicas relacionadas ao
câncer. Contudo, a ACGT é composta apenas pela representação deste domínio, não contendo
instâncias relacionadas a nenhuma das suas classes. No caso da Ontocancro, são quatro tipos de
câncer que estão mapeados até agora, mostrando informações genéticas de uma série de pacientes a
partir de seus tecidos sadios, com pré-câncer e com câncer.
Desta forma, para que a proposta de dados clínicos da ACGT seja incluída no projeto Ontocancro,
faz-se necessária a inclusão de novos dados encontrados a partir de um estudo dentro de um hospital,
de forma a popular as classes que foram obtidas desta ontologia. Estes dados serão obtidos junto ao
Hospital Universitário de Santa Maria, em seu departamento de Oncologia.
Assim, a visão geral da ontologia Ontocancro, integrada com as demais ontologias relevantes ao
câncer, pode ser visualizada na Figura 3.
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
320
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
Figura 3 – Integração das ontologias relacionadas ao câncer com a Ontocancro 2.0
Durante essa coleta de dados, pretende-se desenvolver um algoritmo que busque padrões dentro dos
dados já existentes, podendo ser comparado aos dados reais, formando assim resultados mais
completos no estudo do pós-câncer.
9. CONSIDERAÇÕES FINAIS
Este artigo teve como objetivo uma revisão das ontologias relacionadas ao estudo biológico, mais
especificamente sobre câncer, existentes atualmente, para prover a melhoria e aperfeiçoamento da
ontologia Ontocancro. Com o presente estudo foi possível observar que, embora se tenha muita
atenção voltada para estudos relacionados ao câncer, não existem muitos estudos utilizando
ontologia para representação deste domínio de conhecimento.
O maior conhecimento do genoma humano apoia a teoria de que as doenças devem ser entendidas
considerando as complexas interações entre genes e fatores ambientais que desencadeiam processos
patológicos e definem o fenótipo. Novas terapias para doenças estabelecidas e novas intervenções
para a prevenção de doenças estão sendo desenvolvidas e discutidas, porém estas abordagens
inovadoras não podem ser sustentadas sem efetivamente trabalhar com os dados gerados em
laboratório nas áreas de genômica funcional, estrutural e proteômica. Desta forma, torna-se
importante a integração de dados clínicos, que são gerados por registros médicos, vinculando o
atendimento ao paciente e a saúde pública. A informática biomédica visa criar uma informação
conceitual comum para promover a descoberta de novos diagnósticos e métodos terapêuticos,
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
321
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
integrando informações médicas e biológicas. É necessária uma base de dados sólida para
diagnóstico, estratificação de tratamento e testes de previsão de doenças [Sanchez et al. 2004].
De forma que seja possível promover a descoberta de novos diagnósticos e tratamentos terapêuticos,
analisou-se as ontologias encontradas para obter uma nova visão na evolução da Ontocancro de
maneira que seja possível auxiliar também outros grupos de pesquisa e reutilizar representações
existentes sempre que possível. Através desta pesquisa, foi pensado na inserção da parte clínica na
Ontocancro, vista na ontologia ACGT, para que o estudo dos carcinomas da Ontocancro seja mais
completo, possibilitando o estudo desde a tentativa de prevenção até o tratamento clínico e
possivelmente o resultado pós-tratamento.
O resultado obtido até o momento é uma ontologia que está Inter operável com as demais ontologias
que descrevem este domínio (ou parte dele), através do uso da sintaxe OWL que descreve a
representação de todas estas ontologias. A Ontocancro, além de descrita em OWL, possui um banco
de dados contendo todas as informações relacionadas às suas classes e instâncias. Esse banco de
dados também já está adaptado aos conceitos obtidos a partir das demais ontologias e disponível em
seu sítio http://ontocancro.org/.
Em relação a trabalhos futuros, pretende-se obter e analisar os dados reais, vindo de estudos clínicos
do câncer, para fomentar as instâncias da ontologia. De posse destes dados, o próximo passo será a
criação de uma série de regras de consultas e inferências que possam facilitar o raciocínio sobre o
conhecimento desta ontologia.
Outro processo que será realizado será a análise dos dados armazenados na Ontocancro, após essa
integração, usando técnicas de datamining. Pretende-se, com isso, descobrir algum conhecimento
que não esteja claro aos médicos oncologistas e especialistas, a partir do mapeamento de todo o
domínio e a consequente população das instâncias da ontologia, seja com dados obtidos no Hospital
Universitário de Santa Maria, seja a partir dos bancos de dados biológicos disponíveis, como o GEO.
10. AGRACEDIMENTOS
Os autores agradecem à Fundação de Amparo à Pesquisa do Rio Grande do Sul (FAPERGS) pelo
apoio ao desenvolvimento deste projeto sob número FAPERGS PqG 2012 121256-7.
REFERÊNCIAS
Biegel, J. A.; Zhou, JY.; Rorke, L. B.; Stenstrom, C.; Wainwright, L. M.; Fogelgren, B. Germ-Line
and Acquired Mutations of INI1 in Atypical Teratoid and Rhabdoid Tumors. Cancer Research
59,
74–79,
January
1,
1999.
Disponível
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
em
322
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
<http://cancerres.aacrjournals.org/content/59/1/74.full.pdf+html> Acesso em 10 de dezembro
de 2013.
Bodenreider, O. Biomedical Ontologies in Action: Role in Knowledge Management, Data
Integration and Decision Support. IMIA Yearbook of Medical Informatics, 2008. Disponível
em <http://www.ncbi.nlm.nih.gov/pubmed/18660879> Acesso em 10 de junho de 2014.
Brochhausen, M., Spear, A. D., Cocos, C., Weiler G., Martín L., Anguita A., Stenzhorn H.,
Daskalaki E., Schera F., Schwarz U., Sfakianakis S., Kiefer S., Dörr M., Graf N., Tsiknakis
M., The ACGT Master Ontology and its applications – Towards an ontology-driven cancer
research and management system. Journal of Biomedical Informatics 8-25. 2011.
Cocos, C. Design Principles of the ACGT Master Ontology: Examples and Discussion. Information
Society
Tecnologies,
Dezembro
2008.
Disponível
em
<http://www.unisaarland.de/fileadmin/user_upload/Institute/ifomis/documentation/ACGT_
D7.7_IFO MIS_Final.pdf> Acesso em 16 de julho de 2014.
Courtot M., Juty N., Knüpfer C., Waltemath D., Zhukova A., Dräger A., Dumontier M., Finney A.,
Golebiewski M., Hastings J., Hoops S., Keating S., Kell D. B., Kerrien S., Lawson J., Lister
A., Lu J., Machne R., Mendes P., Pocock M., Rodriguez N., Villeger A., Wilkinson D. J.,
Wimalaratne S., Laibe C., Hucka M., Le Novère N. Controlled vocabularies and semantics in
systems biology. Molecular Systems Biology. 2011.
Cruz, I. F.; Antonelli, F. P.; Stroe, C., AgreementMaker: Ecient Matching for Large RealWorld
Schemas and Ontologies. VLDB ‘09, August 24-28, 2009, Lyon, France 2009
VLDB
Endowment,
ACM.
Disponível
em
<http://disi.unitn.it/~p2p/RelatedWork/Matching/vldb2009-revised.pdf> Acesso em 27 de
outubro de 2013.
Garvin, A.J., Re, G.G., Tarnowski, B.I., Hazen-Martin, D.J., Sens, D.A. The G401 Cell Line,
Utilized for Studies of Chromosomal Changes in Wilms' Tumor, Is Derived from a Rhabdoid
Tumor of the Kidney. American journal of Pathology, Vol. 142, No. 2, 1993. Disponível em
<http://www.ncbi.nlm.nih.gov/pmc/articles/PMC1886739/pdf/amjpathol
000740031.pdf>
Acesso em 10 de dezembro de 2013.
Grivicich, I., Regner, A., Rocha, A. B. Morte Celular por Apoptose. Revista Brasileira de
Cancerologia,
Vol.
53(3):
335-343,
2007.
Disponível
em
<http://www.inca.gov.br/rbc/n_53/v03/pdf/revisao4.pdf> Acesso em 10 de dezembro de
2013.
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
323
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
Gruber, T. R. A Translation Approach to Portable Ontology Specifications. Journal Knowledge
Acquisition - Special issue: Current issues in knowledge modeling, Volume 5, Issue 2, June
1993, Pages 199 - 220, ISSN: 1042-8143
Guarino, N. Formal Ontology and Information Systems. Book Formal Ontology in Information
Systems: Proceedings of the 1st International Conference, June 1998, Pages 3 - 15,
ISBN:9051993994
Halazonetis, T. D.; Gorgoulis, V. G.; Bartek, J. An oncogene-induced DNA damage model for
cancer development. Science (New York, N.Y.), v. 319, n. 5868, p. 1352–55, 7 mar. 2008.
Disponível em: <http://www.sciencemag.org/content/319/5868/1352.abstract>. Acesso em 7
de abril de 2014.
Junior, H.L.R.; Oliveira, R.T.G.; Ceccatto, V.M. Bioinformática como recurso pedagógico para o
curso de ciências biológicas na Universidade Estadual do Ceará – UECE – Fortaleza, Estado
do Ceará. Acta Scientiarum. Education, Maringá, v. 34, n. 1, p. 129-140, Jan-Jun, 2012.
Disponível em: <http://periodicos.uem.br/ojs/index.php/ActaSciEduc/article/ view/14584>.
Acesso em 7 de abril de 2014.
Librelotto, G. R.; Mombach, J. C.; Sinigaglia, M.; Simão, É. M.; Cabral, H. B.; Castro, M. A. A. An
Ontology to Integrate Transcriptomics and Interatomics Data Involved in Gene Pathways of
Genome Stability. Advances in Bioinformatics and Computational Biology, volume 5676,
2009, pag 164-167, ISBN 978-3-642-03222-6, http://dx.doi.org/10.1007/978-3-642-032233_18.
Nascimento, K. S.; Cabral, H. B.; Librelotto, G. R. Uma Metodologia para a Visualização das Redes
Metabólicas da Ontocancro. VIII Simpósio de Informática da Região Centro/RS – UNIFRA,
2009. Disponível em <http://www.sirc.unifra.br/artigos2009/artigo17.pdf> Acesso em 10 de
abril de 2014.
Sanchez, F. M.; Iakovidis, I.; Norager, S.; Maojo, V.; Groen, P. de; Lei, Van der; Jones, T.; Fuchs,
K. A.; Apweiler, R.; Babic, A.; Baud, R.; Breton, V.; Cinquin, P.; Doupi, P.; Dugas, M.; Eils,
R.; Engelbrecht, R.; Ghazal, AL, P.; Jehenson, P.; Kulikowski, C.; Lampe, K.; Moor, G. De;
Orphanoudakis, S.; Rossing, N.; Sarachan, B.; Sousa, A.; Spekowius, G.; Thireos, G.;
Zahlmann, G.; Zvarov, J.; Hermosilla, I.; Vicente, F.J. Synergy between medical informatics
and bioinformatics: facilitating genomic medicine for future health care. Journal of
Biomedical Informatics. Volume 37, Issue 1, Pages 30–42, February 2004. Disponível em
<http://www.j-biomedinform.com/article/S1532-0464%2803%2900085-6/pdf> Acesso em
15 de junho de 2014.
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
324
Falcade et al. / Um comparativo entre ontologias relacionadas ao Câncer
Simão, É. M.; Cabral, H. B.; Castro, M. A. A.; Sinigaglia, M.; Mombach, J. C. M.; Librelotto, G. R.
Modeling the Human Genome Maintenance network. Physica A: Statistical Mechanics and
its Applications, Volume 389, Issue 19, 1 October 2010, Pages 4188-4194, ISSN 0378-4371,
http://dx.doi.org/10.1016/j.physa.2010.05.051.
Wild, C. Câncer é hoje a doença que mais mata mundo, com o número de casos devendo explodir
nos próximos anos. Tempo e leitura: Bahia Negócios. 2014. Disponível em
<http://www.bahianegocios.com.br/saude/cancer-e-hoje-a-doenca-que-mais-matamundocom-o-numero-de-casos-devendo-explodir-nos-proximos-anos/> Acesso em 14 de junho de
2014.
14ª Conferência da Associação Portuguesa de Sistemas de Informação (CAPSI 2014)
325