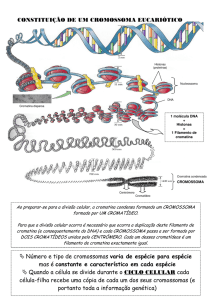
Epigenética
Como diferentes células tronco adultas “sabem” seu destino celular?
Diferentes tipos celulares mas
sequência de DNA idêntica
Como organismos geneticamente idênticos podem apresentar diferentes
fenótipos?
Como organismos geneticamente idênticos podem apresentar diferentes
fenótipos?
Como é possível que haja expressão alélica de apenas um dos genitores?
Como balancear a dose de cromossomos X entre os sexos?
Como as fêmeas de mamíferos podem expressar apenas um de seus
cromossomos X em suas células?
A Epigenética
O termo “Epigenética” foi utilizado pela primeira vez por Conrad H.
Waddington, em meados da década de 40 – “Epigenetic landscape”.
Emergência de um eventual fenótipo em um organismo através do
processo de desenvolvimento, este iniciado a partir de um perfil
genético específico.
A relação entre fenótipo e genótipo não é linear e probabilística, não
há um determinismo. Há influência de efeitos aleatórios ou ambientais.
Antigamente...
Epigenética: diferenciação das células a partir de um estado inicial
totipotente no desenvolvimento embrionário.
Epigenética e Epigenoma
EPIGENÉTICA
◦ Refere-se ao estudo dos fatores herdados que modificam a expressão dos genes sem alterar
a sequência de DNA.
EPIGENOMA (“acima” do genoma)
◦ É o conjunto de modificações bioquímicas da cromatina que determina uma
informação genética, refletindo o status epigenético da cromatina de uma célula.
Marcas epigenéticas
O que, onde e quando fazer...
A informação epigenética
Nem todos os genes são necessariamente expressos em todas as
células de um organismo. A maioria está programada para permanecer
reprimida.
Modificações epigenéticas são mecanismos moleculares que permitem
preservar o estado inativo por regenerar a estrutura repressiva da
cromatina a cada rodada de replicação do DNA.
A informação epigenética
Mecanismos epigenéticos
Metilação de citosinas
Mecanismos
epigenéticos
Modificação de histonas
Remodelamento de cromatina
RNA de interferência
Metilação de citosinas no
DNA
Modificação covalente do DNA: substituição do H5 de citosinas por um
grupo metil (doador: S-adenosilmetionina - SAM).
DNA metiltransferases em mamíferos: DNMT1, DNMT2, DNMT3A,
DNMT3B, DNMT3L.
Tipos:
- de manutenção
- de novo
5-metilcitosina
Metilação de citosinas no
DNA
Ocorre nas sequências CpG in tandem, em ambas as fitas do DNA.
Ilhas CpG (promotores e regiões intergênicas)
Em mamíferos, 60% - 90% de todas as CpGs estão metiladas
CpG não metilada:
ocorre transcrição
CpG metilada: inibe
transcrição
Metilação no gene
facilita transcrição
Gene não metilado: início de
transcrição espúrios
ON
Sequências repetitivas metiladas:
previnem instabilidade cromossômica
OFF
Metilação de citosinas no
DNA
Ocorre nas sequências CpG in tandem, em ambas as fitas do DNA.
Ilhas CpG (promotores e regiões intergênicas)
Em mamíferos, 60% - 90% de todas as CpGs estão metiladas
Não interfere no pareamento das bases nitrogenadas, mas adiciona
informações: grupo metil posicionado no sulco maior do DNA
Controle da ligação de proteínas ao DNA
CpG não
metilada
ON
Organização da cromatina e expressão gênica
(frequentemente silenciamento gênico)
OFF
CpG
metilada
Metilação de citosinas no
DNA
Funções:
◦ Expressão gênica
◦ Estabilidade cromossômica
◦ Diferenciação celular
◦ Imprinting
◦ Inativação do X
ON
OFF
Alterações no padrão de metilação
e a carcinogênese
A função normal da célula depende do equilíbrio entre dois eventos:
HIPOMETILAÇÃO
Ilhas CpG
X
HIPERMETILAÇÃO
Regiões intergênicas
Alterações neste equilíbrio podem comprometer a expressão de genes
críticos, resultando em diversos tipos de câncer.
- Hipometilação generalizada do genoma
frequente em sequências do DNA
repetidas (DNA satélite, LINE e SINE), retrotransposons e genes cópia única
- Hipermetilação que se apresenta em áreas localizadas dentro da região
promotora de genes supressores de tumor, fatores de transcrição; genes de
controle do ciclo celular, genes anti-apoptóticos e genes de reparo do DNA
Alterações no padrão de metilação
e a carcinogênese
METILAÇÃO NORMAL DO DNA
HIPOMETILAÇÃO GLOBAL
INSTABILIDADE CROMOSSÔMICA
ATIVAÇÃO DE ONCOGENES
HIPERMETILAÇÃO DE ILHAS CpG
INATIVAÇÃO DE GENES
SUPRESSORES TUMORAIS
INATIVAÇÃO DE
GENES DE REPARO
A metilação de C atenua a
sequencia patogênica em
diferentes níveis,
resultando em um
espectro caracterizado
por: cor amarela, tumores
e obesidade.
Com suplementação
dietética na gestação
Sem suplementação
dietética na gestação
“Memória epigenética”
A metilação em ambas as fitas permite reproduzir o padrão após a
replicação do DNA.
Metilases de manutenção reconhecem fitas hemimetiladas e
adicionam grupos metil às citosinas correspondentes.
Importância da hemimetilação para o reconhecimento de pareamentos
errados durante a replicação.
Modificação de histonas
Classes de histonas
H1, H2, H3 e H4
Modificações pós-traducionais :
acetilação, metilação,
fosforilação e ubiquitinação
Funções:
Empacotamento do DNA e
Regulação da expressão gênica
Código de histonas
Código de histonas:
Conformação da cromatina
Acetilação de histonas
A acetilação de histonas ocorre em resíduos de lisina
Histona acetiltransferase
(HAT)
HAT (acetilação de
histonas)
Cromatina inativa
Hipoacetilação de H3 e H4
–
regiões
de
baixa
atividade trascricional
Histonas desacetilases
(HDAC)
HDAC (desacetilação de
histonas)
Lisin
a
Cromatina ativa
Hiperacetilação de H3 e
H4 – regiões ativas
transcricionalmente
Fosforilação de histonas
A fosforilação de histonas pode ocorrer em resíduos de serina, treonina e
tirosina.
Funções:
Sinalização para reparo de dano ao DNA (H2A, H2B...)
Regulação transcricional
Compactação da cromatina associada com a mitose/ meiose
Condensação da cromatina – fosforilação de T3, S10, T11 e S28
na H3
Descondensação da cromatina – fosforilação da H1
Compactação da cromatina e apoptose
Resíduo fosforilado pode determinar o reparo do dano ou a
apoptose da célula
Os mesmos eventos de fosforilação podem estar relacionados com múltiplos processos de
modulação da cromatina;
Existe uma interação entre as modificações pós-traducionais (MTPs) que ocorrem nas
histonas – caudas das histonas representam uma “plataforma” para várias MTPs.
Metilação de histonas
A metilação de histonas ocorre em resíduos de lisina.
O grau de metilação e o resíduo metilado estão envolvidos na
regulação transcricional – ativação ou repressão dependente da lisina
em questão.
- H3K9me e H3K27me são marcadores de silenciamento de DNA
(regiões de heterocromatina, X inativado e promotores silenciados)
enquanto H3K4me sugere DNA transcricionalmente ativo (promotores
ativos).
H3K4me3 – promotores ativos
Fatores de remodelamento de
cromatina
Complexo SWI/SNF – 9 a 12 subunidades que são recrutadas para
promotores específicos e atuam no remodelamento da cromatina:
“Deslizamento” do octâmero de histonas ao
longo do DNA, reposicionando o nucleossomo;
Afrouxar a fita do DNA sobre o nucleossomo;
Alteração da conformação do nucleossomos,
reorganizando seus componentes;
“Construir” um local promotor
acessível para a maquinaria
transcricional
Perfil de modificação de
histonas
Inúmeras enzimas atuam em conjunto para adicionar e remover
modificações covalentes nas histonas, interagindo entre si e com outros
mecanismos para manter a conformação da cromatina e o controle da
transcrição.
Relação entre acetilação e metilação de histonas
Cromatina “fechada”
transcricional
–
“aberta”
repressãoCromatina
transcricional
–
ativação
Perfil de modificação de
histonas
Inúmeras enzimas atuam em conjunto para adicionar e remover
modificações covalentes nas histonas, interagindo entre si e com outros
mecanismos para manter a conformação da cromatina e o controle da
transcrição.
Marcas epigenéticas recrutam demais mecanismos para
manutenção do estado da cromatina
HP1 é repressor de
transcrição
“Memória Epigenética”
Durante a replicação do DNA, o octâmero de histonas não se dissocia
completamente, de forma que as fitas recém-sintetizadas têm seus
nucleossomos montados a partir de histonas novas e antigas.
Replicação do DNA e das características da cromatina
Replicação de cromatina
Diferentes tipos de cromatina são
replicadas em momentos distintos
(origens de replicação ativadas
distintamente) para manter o
padrão de cromatina de cada uma.
RNAs não codificadores
(siRNA e miRNA)
RNAs não codificadores atuam no silenciamento transcricional e póstranscricional.
Regulam a expressão de genes ao interagir com seus mRNAs
Há interferência na expressão gênica:
Regulação por RNA de interferência
Epigenética...
Replicação de cromatina
Conjunto das modificações nucleares herdadas e que modulam a
expressão gênica, mas não envolvem modificações na sequência de DNA.
Grau de compactação da cromatina
Acesso aos promotores gênicos
Adição/Remoção de grupos funcionais
Recrutamento de outros fatores e mecanismos que
promovem a manutenção do estado de cromatina
Remodelamento da cromatina
Fenômenos epigenéticos
Inativação do X
Imprinting genômico
Inativação do X
Organismos com sistema de determinação sexual XX/XY e XX/X0
precisam equalizar a dosagem dos genes ligados ao X em ambos os sexos.
Compensação de dose
Inativação do X nas fêmeas (mamíferos)
Hipótese de Lyon:
Nas células somáticas femininas, apenas um cromossomo
X é transcricionalmente ativo;
A inativação do X ocorre entre o 13º-16º dia da vida
embrionária;
É aleatório e fixo para todas as células descendentes;
Determina um mosaicismo somático
Exemplo: Cor de pelagem ligada ao X – gatos Tortoiseshell
Processo de inativação do
X
XIC (Xq13)
Centro de Inativação do X – contém o gene XIST
XIST
Transcrito específico de inativação do X
Inativa o X
Essencial para iniciar o processo de inativação, posteriormente
mantido por outros mecanismos
No início do desenvolvimento embrionário ambas as cópias de XIST
estão ativas – Metilação inativa um deles
XIST silenciado = cromossomo NÃO pode ser inativado
X ativo tem XIST hipermetilado
Processo de inativação do
X
XIST é expresso somente pelo cromossomo inativo (para inativá-lo).
ncRNA XIST se associa a toda a extensão do o cromossomo X a ser
inativado = INATIVAÇÃO DO X – corpúsculo de Barr.
A inativação é mantida pela metilação ao longo desse cromossomo.
Vários fatores estão relacionados a este processo, controlando e auxiliando a
inativação. Ex.: Tsix regula a expressão de XIST: Tsix ativo = XIST reprimido = X ativo
Inativação do X
A inativação não é completa:
Região pseudoautossômica (homologia com cromossomo Y)
Região pericentromérica
30% dos genes do braço curto
Inativação variável entre os indivíduos
Síndrome de Turner (X0)
Falhas da inativação do X:
Em células embrionárias, após o 13º16º dia, a não inativação de um dos
cromossomos X é um evento letal.
Em células somáticas é um evento
raro, podendo ocorrer em células
normais ou neoplásicas.
Imprinting genômico
Expressão gênica diferencial: o alelo é expresso na dependência de sua
origem (materna ou paterna).
Exceção ao padrão de herança Mendeliana.
Mediado por metilação das ICRs (Região de Controle de Imprinting).
O padrão de genes metilados da mãe e do pai são transmitidos para o filho
Espermatogênese - Padrão de metilação paterno
Oogênese - Padrão de metilação materno
Gene imprintado: expressão condicionada por sua origem parental.
Hemizigose:
apenas uma
cópia metilada
Influências ambientais
Epigenética e nutrição
Determinados componentes bioativos de alimentos podem alterar a expressão
gênica via alterações na metilação do DNA e modificações de histonas.
Disponibilidade do doador de grupos metil (SAM):
É dependente de vias metabólicas relacionadas às vitaminas B6 e B12 – deficiência
de vitamina B compromete a metilação do DNA;
Envolve folato, betaina e colina e aminoácidos – consumo crônico de álcool pode
alterar a disponibilidade de SAM através do desperdício de metionina e colina ou por
diminuir a disponibilidade de vitamina B;
EGCG (polifenol - chá verde): pode diminuir a metilação global do DNA, em
linhagens celulares de câncer, pela inibição competitiva de DNMT; também
aumenta o nível de miR-210 – diminuição da proliferação celular.
Redução de selênio (grãos e vegetais): diminuição da metilação global do DNA;
expressão diminuída de DNMT1 em linhagens celulares de câncer de cólon e
próstata.
Epigenética e a dieta materna
A transmissão materna de marcas epigenéticas pode ser alterada por
nutrientes:
Consumo materno de vitamina B está associado com mudanças no
risco de carcinogênese mamária e de cólon na prole;
↑ consumo materno de colina, em ratas, aumenta os níveis de
metilação de histonas (H3K9me2 e H3K27me3) – possivelmente
devido ao aumento na expressão de HMT;
Dieta rica ou pobre em proteínas, em porcas, pode alterar a
metilação global do DNA na prole, através de mudanças na
expressão das DNMTs;
Epigenética e experiências na
infância
Influência do ambiente de infância no sistema nervoso central e
resposta ao estresse na vida adulta:
Comparação de padrões epigenéticos do hipocampo entre vítimas
de suicídio com e sem histórico de abuso na infância – ↑metilação
do promotor de NR3C1 = ausência do receptor
Estresse social (frustração social) em camundongos jovens está
relacionado a mudanças epigenéticas no hipocampo – prole exibe
comportamento depressivo e ansiedade.
Experiências na infância resultam em alterações moleculares na
resposta cerebral ao estresse via mudanças nos padrões epigenéticos.
Epigenética e envelhecimento
As “assinaturas” epigenéticas tendem a mudar naturalmente com a idade:
Hipermetilação de
determinados promotores
ao longo do genoma
Diminuição da metilação
global de DNA
Alterações no padrão de
modificação de histonas
indicando região
heterocromática –
repressão da transcrição
No entanto, não está claro se os padrões epigenéticos são programados ou
estocásticos.
O mesmo padrão de metilação do DNA observado no envelhecimento
aparece no desenvolvimento do câncer – hipermetilação de genes supressores
de tumor.
Terapêutica: compreensão das marcas epigenéticas relacionadas
envelhecimento e proposta de intervenção farmacêutica e de dietas.
ao
Epigenética e doenças
Epigenética e doenças
Epigenética como fonte de
variabilidade
Epigenética como fonte de
variabilidade