IDENTIFICAÇÃO E CARACTERIZAÇÃO DO AGENTE CAUSAL DA DOENÇA AZUL DO
ALGODOEIRO
Régis Lopes Corrêa (UFRJ / [email protected]), Tatiane da Silva Franca (UFRJ), Marcos De
Bonis (UFRJ), Paulo Augusto Vianna Barroso (Embrapa Algodão), Márcia Soares Vidal (Embrapa
Algodão), Maite Vaslin Freitas Silva (UFRJ).
RESUMO - Apesar da importância da Doença azul ou azulão do algodoeiro, seu agente causal ainda
não havia sido identificado. Neste trabalho, após sucessivas passagens da Doença azul em casa de
vegetação, conseguiu-se a identificação de seu agente causal utilizando a técnica de RT-PCR e
oligonucleotídeos degenerados. As análises comprovaram tratar-se de um vírus da família Luteoviridae,
gênero Polerovirus. Foi possível também seu isolamento e caracterização por microscopia eletrônica.
Além disto, parte do genoma viral correspondente a CP, a região intergênica, parte da replicase e a
proteína do movimento do vírus foi clonada e seqüenciada.
Palavras-chave: algodão, Gossypium spp., Doença azul do algodoeiro
IDENTIFICATION AND CARACTERIZATION OF COTTON BLUE DISEASE CAUSAL AGENTTÍTULO
EM INGLÊS
ABSTRACT - Despite Cotton blue disease importance its causal agent wasn’t identified until now. In
this work, after successive passges of Cotton blue disease in greenhouse conditions we were able to
identify it using RT-PCR and degenerated primers. Sequence analysis shown that it’s a virus from
Luteoviridae family. Sequence analysis of the entire capsid amino acid sequence and partial RdRp
sequence revealed a virus probably belonging to the Polerovirus genus. We also purify virus particule
and observed it by electronic microscope. Based on ours results we propose that cotton blue disease is
actually caused by a virus with the putative name Cotton leafroll darf virus (CLRDV).
Key words: cotton, Gossypium spp., Cotton blue disease
INTRODUÇÃO
O algodão (Gossypium spp.) é um dos produtos agrícolas mais antigos e importantes no
mundo. No Brasil, uma doença do algodoeiro chamada Mosaico das Nervuras foi descrita no estado de
São Paulo por Costa e Forster em 1938. Alguns anos mais tarde, em 1962, uma estirpe mais virulenta,
denominada Mosaico das Nervuras var. Ribeirão Bonito foi encontrada no mesmo estado. Estudos
sobre sintomatologia e o modo de transmissão levantaram a hipótese de que esta doença está
relacionada com a doença azul do algodoeiro, uma moléstia de algodão mundialmente disseminada. A
doença azul do algodoeiro foi inicialmente descrita na África em 1949 e em seguida foi encontrada
também na Ásia e nas Américas (CAUQUIL e VAISSAYRE, 1971). A doença é transmitida pelo pulgão
Aphis gossypii e os sintomas são caracterizados por nanismo devido ao encurtamento dos entrenós,
enrolamento e escurecimento foliar e amarelecimento das nervuras (CAUQUIL, 1977). Os sintomas e a
transmissão restrita ao afídeo levaram a especulação de que a doença azul poderia estar relacionada a
um vírus da família Luteoviridae. Entretanto, nenhuma evidência molecular suportava essa hipótese até
então. Os Luteoviridae são classificados em três gêneros baseados na organização genômica,
estratégia de replicação e mecanismos de expressão: Luteovirus, Polerovirus e Enamovirus. Todos os
gêneros possuem regiões 3` conservadas compreendendo a proteína do capsídeo e de transmissão.
Diferenças são observadas na região 5` dos genomas (MAYO e ZIEGLER-GRAFF, 1996).
Nesse trabalho mostrou-se a caracterização molecular de um vírus da família Luteoviridae
associado à doença azul do algodoeiro. Utilizando a metodologia de RT-PCR foi possível amplificar e
posteriormente obter a seqüência de 1405 nucleotídeos do genoma viral, correspondendo à parte da
polimerase e seqüências inteiras do capsídeo, do movimento e região intergênica. Análises das
seqüências de aminoácidos claramente posicionaram o vírus dentro da família Luteoviridae. Além
disso, a comparação de seqüências virais em diferentes plantas de algodão mostrou alta conservação
intra-populacional. Baseados em sintomas de campo nós propomos o nome Cotton leafroll dwarf virus
(CLRDV) para o vírus identificado.
MATERIAL E MÉTODOS
Para a caracterização inicial do vírus, plantas de algodão da variedade CNPA ITA 90
apresentando sintomas característicos da doença azul (aqui denominadas de MT1) foram coletadas em
uma plantação no estado de Mato Grosso. Nos ensaios de infecção, pulgões Aphis gossypii não
virulíferos foram transferidos e mantidos por dois dias em plantas infectadas para a aquisição viral. Em
seguida aproximadamente dez pulgões foram transferidos para plantas saudáveis e mantidos por dois
dias de inoculação viral.
RNA total de plantas infectadas e saudáveis foram obtidos por extração segundo Vervoed,
1989 ou com o Plant RNeasy Kit da Qiagen. Cerca de 2,5 µg de RNA foram utilizados para a síntese
de cDNA usando os oligos degenerados PLF (5`-ACDGAYTGYTCYGGTTTYGACTGG-3`) e PLR (5`TCTGAWARASWCGGCCCGAASGTGA-3`), resultando na amplificação de um fragmento de 1058
nucleotídeos. Oligos internos PL2F (5`-AACAATTAGGTTTTAAAGTCGAGG-3`) e PL2R (5`TTCTACCCACGACCGTATTCAT-3`), foram usados para o seqüenciamento completo do fragmento. O
fragmento inteiro da CP foi obtido com os oligos PL4F (5` TGCGACAAATAGTTAATGAATACGGT 3`) e
o 3R (5` GTCTACCTATTTBGGRTTNTGGAA 3`). Os oligos estão esquematicamente representados na
Figura 1. Todos os fragmentos amplificados foram clonados em pGem-T Easy (Promega) e três a nove
clones em cada planta foram seqüenciados com oligos universais T7 e M13 usando o seqüenciador
automático Mega Bace.
Seqüências das espécies de Luteoviridae foram obtidas no Genbank para comparação.
Seqüências de aminoácidos foram analisadas com o programa Multalin e análises filogenéticas foram
realizadas com o programa MEGA 2.0.
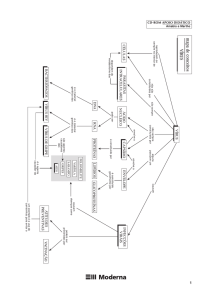
Figura 1: Representação esquemática da estrutura genômica dos Polerovirus. Números representam ORFs 0 a 5. ORF 0 –
proteína supressora de silenciamento; ORF 1 + ORF 2 – Replicase; ORF 3 – capsídeo; ORF 4 – proteína do movimento;
ORF 5 – proteína de transmissão. As posições dos oligos PLF, PLR, PL2F, PL2R, PL4F e o3R usados para amplificar 1405
nt do CLRDV estão indicados por setas.
RESULTADOS E DISCUSSÃO
Pulgões virulíferos coletados em campo foram transferidos para plantas de algodão saudáveis
CNPA ITA 90 e em seguida eliminados com o uso do inseticida Orthene. Durante a primeira passagem
foi possível observar sintomas típicos da doença como encurtamento dos entrenós, folhas rugosas e
com curvatura nos bordos, enrolamento das folhas em sua superfície inferior, associados a uma
coloração verde-escura com tonalidade azulada. Após algumas passagens, sintomas de mosaico nas
nervuras com amarelecimento das mesmas começaram a se apresentar de forma mais evidente,
principalmente nas folhas jovens das plantas (Fig. 2).
Folhas apicais de três plantas de algodão com aproximadamente um mês após o inoculo viral e
apresentando sintomas de mosaico nas nervuras foram coletadas para a extração de RNA total. Os
oligonucleotídeos desenhados foram utilizados em reações de RT-PCR com o objetivo de verificar a
existência de Polerovirus e/ou Luteovirus associados aos sintomas observados. A análise do produto
de amplificação em gel de agarose mostrou a existência de bandas com o tamanho esperado nas três
plantas infectadas quando amplificadas com os oligonucleotídeos PLF e PLR (Fig. 3). Nenhuma banda
foi observada nas RT-PCRs da planta não infectada e no tubo que continha apenas água. Resultados
negativos foram obtidos também nas amplificações realizadas com os oligonucleotídeos específicos
para o gênero Luteovirus (dados não mostrados). A partir da seqüência obtida, novos oligonucleotídeos
degenerados foram desenhados para amplificar todo o capsídeo viral.
Figura 2. Sintomas da doença azul do
algodoeiro em casa de vegetação. A Folha
expandida de uma planta saudável. B Sintoma
de enrolamento foliar após quinze dias de
inoculo viral. C Folha jovem de uma planta não
inoculada. D Sintoma de amarelecimento das
nervuras em uma folha jovem inoculada. E
Sintoma de nanismo em três plantas inoculadas
(direita) comparado com uma não inoculada
(esquerda) com a mesma idade. Barras
representam 1 cm.
.
Figura 3. Amplificação por RT-PCR de partes
do genoma do CLRDV usando dois diferentes
pares de oligos. A - Amplificação com os oligos
PLF e PLR
Os fragmentos amplificados, correspondendo a parte da polimerase e capsídeo e a região
intergênica inteira foram separados em gel de agarose (painel superior), transferidos para membranas
de nylon e hibridados com fragmento correspondente a CP viral (painel inferior).
Após amplificação e seqüênciamento, a análise do capsídeo completo mostrou que o vírus
associado à Doença azul do algodoeiro apresenta 92% de identidade com o capsídeo do Chickpea
stunt disease associated virus (CpSDaV), um membro da família Luteoviridae ainda não classificado
(Tab.1). Como esperado, alta identidade foi obtida também com capsídeo de diferentes membros do
gênero Polerovirus.
A
B
Figura 4. Análise filogenética das seqüências amplificadas. A – Filogenia com a CP do CLRDV e outros
Luteoviridae. B – Filogenia da parte C-terminal da polimerase do CLRDV e outros Luteoviridae.
Figura 5. Microscopia eletrônica do CLRDV em
plantas com Doença azul do algodoeiro.
Partículas virais forma coradas com contraste
negativo. A – Aumento de 160.000 x, PTA, pH
6,2; B – 120.000 x, 2% uranila; C – Leaf-tip,
76.000x, 2% uranila; D – 240.000 x, 2% uranila.
Barras correspondem a 30 nm.
Além disso, a seqüência da CP obtida foi utilizada como sonda para a detecção viral através de
Northern blot.
Partindo de aproximadamente 70g de folhas infectadas em casa de vegetação realizamos a
purificação do vírus. Através de microscopia eletrônica foi possível a identificação de partículas virais
isométricas com tamanho aproximado de 30 nm (Fig. 5).
CONCLUSÕES
1. A partir das análises pudemos identificar o agente causal da Doença azul do algodoeiro,
comprovando tratar-se de um vírus da família Luteoviridae. Foi possível também o isolamento e o
seqüenciamento de parte de seu genoma correspondente a CP, a região intergênica, parte da replicase
e a proteína do movimento do vírus;
2. A partir destes resultados pretende-se obter a expressão in vitro da região amplificada que poderá
ser utilizada para o desenvolvimento de anticorpos contra o vírus. Assim, métodos de diagnóstico em
larga escala e de baixo custo, como ELISA, poderão ser desenvolvidos e aplicados, o que facilitará
muito a identificação e o controle da doença no campo.
REFERÊNCIAS BIBLIOGRÁFICAS
CAUQUIL, J. Etudes sur une maladie d’ origine virale du cotonnier: la maladie bleue. Cot. Fib. Trop., v.
32, p. 259-278. 1977.
CAUQUIL, J.; VAISSAYRE, M. La "maladie bleue" du cotonnier en afrique : transmission de cotonnier à
cotonnier par aphis gossypii glover. Cot. Fib. Trop., v. 26, p. 463-466.1971.
COSTA, A. S.; FORSTER, R. Nota preliminar sobre uma nova moléstia de vírus do algodoeiro mosaico das nervuras. Campinas: Instituto Agronômico de Campinas, 1938. p. 51: 11 (Boletim
Técnico).
MAYO, M. A.; ZIEGLER-GRAFF, V. Molecular biology of luteoviruses. Adv. Virus Res., v. 46, p. 413–
460. 1996.
RAGUEH, F.; FESCURE, N.; ROBY, D.; MARCO, Y. Gene expression in Nicotiana tabacum in
response to compatible and incompatible isolates of Pseudomonas solonaciarum. Physiol. Mol. Plant
Pathol., n. 35, p. 23–33. 1989.