1
© 2016 Dr. Walter F. de Azevedo Jr.
>
Download do pyzo
www.pyzo.org
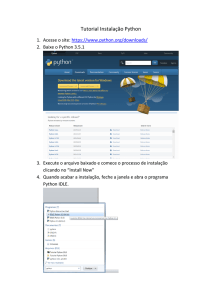
A partir do pyzo (disponível em http://www.pyzo.org/), o processo de instalação do
Python fica facilitado. A instalação integra, além do Python, um conjunto de bibliotecas,
necessárias para computação científica e bioinformática. Clique em “Quickstart”.
2
Download do Pyzo IDE
www.pyzo.org
Clique em “Pyzo for Windows”. Nesta etapa instalaremos o ambiente integrado de
desenvolvimento do Pyzo, chamado Pyzo IDE (Integrated Development Environment).
3
Download do Pyzo IDE
www.pyzo.org
Clique em “Download”.
4
Instalação do Pyzo IDE
www.pyzo.org
Para instalação do Pyzo IDE, abra a pasta de “Downloads” e clique em “Abrir pasta”.
5
Instalação do Pyzo IDE
www.pyzo.org
Clique duas vezes no arquivo de instalação baixado.
6
Instalação do Pyzo IDE
www.pyzo.org
Clique “Next” no instalador.
7
Instalação do Pyzo IDE
www.pyzo.org
Deixe a pasta “default” indicada para instalação e clique “Next”.
8
Instalação do Pyzo IDE
www.pyzo.org
Deixe as configurações indicadas e clique “Next”.
9
Instalação do Pyzo IDE
www.pyzo.org
Clique “Install” para iniciar o processo de instalação.
10
Instalação do Pyzo IDE
www.pyzo.org
A barra verde indica o progresso da instalação.
11
Instalação do Pyzo IDE
www.pyzo.org
Instalação finalizada com sucesso, clique em “”Finish”.
12
Download do Ambiente Python
www.pyzo.org
Para download do ambiente Python, clique em “Miniconda for Windows (64 bits)”.
13
Download do Ambiente Python
www.pyzo.org
Clique em “Download”.
14
Instalação do Ambiente Python
www.pyzo.org
Para instalar o ambiente Python, temos que abrir a pasta de “Downloads”.
15
Instalação do Ambiente Python
www.pyzo.org
Na pasta “Downloads”, clique duas vezes no arquivo de instalação baixado.
16
Instalação do Ambiente Python
www.pyzo.org
No instalador, clique “Next”.
17
Instalação do Ambiente Python
www.pyzo.org
Se concordar com os termos, clique em “I Agree”.
18
Instalação do Ambiente Python
www.pyzo.org
Selecione “Just Me” e clique “Next”.
19
Instalação do Ambiente Python
www.pyzo.org
Deixe a pasta indicada e clique “Next”.
20
Instalação do Ambiente Python
www.pyzo.org
Para instalar, clique em “Install”.
21
Instalação do Ambiente Python
www.pyzo.org
A evolução do processo de instalação é indicado pela barra verde.
22
Instalação do Ambiente Python
www.pyzo.org
Uma vez finalizada a instalação, você tem a mensagem abaixo. Clique em “Next”.
23
Instalação do Ambiente Python
www.pyzo.org
Clique em “Finish”. A instalação do ambiente Python está concluída.
24
Instalação das Bibliotecas Científicas
www.pyzo.org
Para instalação das bibliotecas científicas, clique no ícone do pyzo, como mostrado
abaixo.
25
Instalação das Bibliotecas Científicas
www.pyzo.org
Clique em “Shell>Create shell 1: (Pyhton)”.
26
Instalação das Bibliotecas Científicas
www.pyzo.org
Você terá a janela de comandos (shell) aberta, como mostrado abaixo.
27
Instalação das Bibliotecas Científicas
www.pyzo.org
Na janela abaixo (shell), digite “conda install scipy”, como mostrado abaixo. Depois
pressione <Enter>.
28
Instalação das Bibliotecas Científicas
www.pyzo.org
Quando aparecer a pergunta abaixo, digite y e pressione a tecla <Enter>.
29
Instalação das Bibliotecas Científicas
www.pyzo.org
É mostrada a evolução da instalação das bibliotecas científicas.
30
Instalação das Bibliotecas Científicas
www.pyzo.org
O processo pode levar vários minutos.
31
Instalação das Bibliotecas Científicas
www.pyzo.org
Ao finalizar, você terá a mensagem mostrada abaixo.
32
Instalação das Bibliotecas Científicas
www.pyzo.org
Para continuar a instalação, digite “conda install pyqt matplotlib pandas sympy” e
depois pressione a tecla <Enter>.
33
Instalação das Bibliotecas Científicas
www.pyzo.org
Ao aparecer a mensagem abaixo, digite y e pressione a tecla <Enter>.
34
Instalação das Bibliotecas Científicas
www.pyzo.org
Como sempre, você pode acompanhar a evolução do processo de instalação.
35
Instalação das Bibliotecas Científicas
www.pyzo.org
Quando você tiver a mensagem abaixo, a instalação estará completa.
Instale também o scikit-learn com “conda install scikit-learn”.
36
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
Podemos usar o Integrated Editor for Python (IEP) para desenvolver códigos. Vamos
ilustrar com um código que faz uso de duas bibliotecas científicas. Clique em
“File>New”, como mostrado abaixo.
37
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
Temos a janela de edição. Nela podemos digitar nosso código.
38
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
Abaixo temos um pequeno programa que gera um gráfico, a partir do uso da biblioteca
Matplotlib. O gráfico é da função quadrática, que usa um array da biblioteca NumPy.
39
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
Para salvar o código digitado, clique em “File>Save as...”, como mostrado abaixo.
40
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
Escolha uma pasta para salvar o arquivo fonte, e coloque como nome “test01.py”,
como indicado. Depois clique em “Salvar”.
41
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
Para executar o código, clique em “Run>Run file as script”, como mostrado abaixo.
42
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
O gráfico é gerado e mostrado na tela.
43
Usando IEP para Desenvolver Códigos em Python
www.pyzo.org
Para finalizar, clique em “File>Quit Pyzo” como indicado abaixo.
44
Referências
-BRESSERT, Eli. SciPy and NumPy. Sebastopol: O’Reilly Media, Inc., 2013. 56 p.
-DAWSON, Michael. Python Programming, for the absolute beginner. 3ed. Boston: Course Technology, 2010. 455 p.
-HETLAND, Magnus Lie. Python Algorithms. Mastering Basic Algorithms in the Python Language. Nova York: Springer
Science+Business Media LLC, 2010. 316 p.
-IDRIS, Ivan. NumPy 1.5. An action-packed guide dor the easy-to-use, high performance, Python based free open source
NumPy mathematical library using real-world examples. Beginner’s Guide. Birmingham: Packt Publishing Ltd., 2011. 212 p.
-KIUSALAAS, Jaan. Numerical Methods in Engineering with Python. 2ed. Nova York: Cambridge University Press, 2010. 422
p.
-LANDAU, Rubin H. A First Course in Scientific Computing: Symbolic, Graphic, and Numeric Modeling Using Maple, Java,
Mathematica, and Fortran90. Princeton: Princeton University Press, 2005. 481p.
-LANDAU, Rubin H., PÁEZ, Manuel José, BORDEIANU, Cristian C. A Survey of Computational Physics. Introductory
Computational Physics. Princeton: Princeton University Press, 2008. 658 p.
-LUTZ, Mark. Programming Python. 4ed. Sebastopol: O’Reilly Media, Inc., 2010. 1584 p.
-MODEL, Mitchell L. Bioinformatics Programming Using Python. Sebastopol: O’Reilly Media, Inc., 2011. 1584 p.
-TOSI, Sandro. Matplotlib for Python Developers. Birmingham: Packt Publishing Ltd., 2009. 293 p.
Última atualização: 23 de novembro de 2016.
45
Colophon
This text was produced in a DELL Inspiron notebook with 6GB of memory, a 750 GB
hard disk, and an Intel® Core® i5-3337U CPU @ 1.80 GHz running Windows 8.1. Text
and layout were generated using PowerPoint 2013. This tutorial uses Arial font.
54
Author
I graduated in Physics (BSc in Physics) at University of Sao Paulo (USP) in 1990. I
completed a Master Degree in Applied Physics also at USP (1992), working under
supervision of Prof. Yvonne P. Mascarenhas, the founder of crystallography in Brazil.
My dissertation was about X-ray crystallography applied to organometallics compounds
(De Azevedo Jr. et al.,1995).
During my PhD I worked under supervision of Prof. Sung-Hou Kim (University of
California, Berkeley. Department of Chemistry), on a split PhD program with a
fellowship from Brazilian Research Council (CNPq)(1993-1996). My PhD was about the
crystallographic structure of CDK2 (Cyclin-Dependent Kinase 2) (De Azevedo Jr. et al.,
1996). In 1996, I returned to Brazil. In April 1997, I finished my PhD and moved to Sao Jose do Rio Preto (SP,
Brazil) (UNESP) and worked there from 1997 to 2005. In 1997, I started the Laboratory of Biomolecular SystemsDepartment of Physics-UNESP - São Paulo State University. In 2005, I moved to Porto Alegre/RS (Brazil), where I
am now. My current position is coordinator of the Laboratory of Computational Systems Biology at Pontifical
Catholic University of Rio Grande do Sul (PUCRS). My research interests are focused on application of computer
simulations to analyze protein-ligand interactions. I'm also interested in the development of biological inspired
computing and machine learning algorithms. We apply these algorithms to molecular docking simulations, proteinligand interactions and other scientific and technological problems. I published over 160 scientific papers about
protein structures and computer simulation methods applied to the study of biological systems (H-index: 36).
These publications have over 4000 citations. I am editor for the following journals:
47