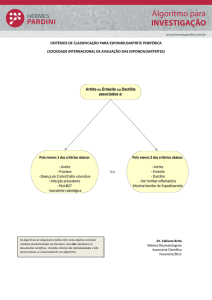
X Congresso Brasileiro de Informática em Saúde
BIRAM: Sistema para Recuperação
de Imagens por Conteúdo
1
1
Ramon A. Moreno , Sérgio S. Furuie
1
Divisão de Informática, Instituto do Coração (InCor),
Hospital das Clínicas - USP, Brasil
Resumo – As imagens digitais estão se tornando cada vez mais importantes, no campo da Medicina, para o
diagnóstico e tratamento do paciente. Ao mesmo tempo, há um aumento no volume de imagens produzidas
nos hospitais, causado pelo desenvolvimento de novas tecnologias e maior adoção de equipamentos
digitais. Este crescimento torna necessário o desenvolvimento de novas técnicas para a recuperação das
imagens que não sejam limitados a campos textuais, como nos si stemas PACS tradicionais, entre as quais
a recuperação baseada em conteúdo. Neste artigo é apresentado o sistema BIRAM, protótipo desenvolvido
em Java, que permite a utilização de diferentes algoritmos para a busca e recuperação de imagens por
conteúdo. O BIRAM também oferece a possibilidade de codificação do laudo de acordo com um dicionário
médico e a busca pelo laudo ou por palavras-chave do cabeçalho DICOM.
Palavras-chave: CBIR, imagens médicas, recuperação de informação.
Abstract – In the Medical field, digital images are becoming increasingly important for the diagnosis and
treatment of the patient. At the same time, the development of new technologies and its adoption caused the
increase in the volume of digital images produced in the hospitals. This growth made necessary to develop
new techniques for the retrieval of the information that are not limited to textual fields as traditional PACS. In
this article, it is presented the BIRAM system, which allows the use of several algorithms for the search and
retrieval of images using their content. The system also allows the search by textual information by means of
a report associated with the study or by keywords from the DICOM header.
Key-words: CBIR, medical images, information retrieval.
CBIR são interessantes para utilização em
hospitais-escola, como o Instituto do Coração do
Hospital das Clínicas (HCFMUSP).
A pesquisa em CBIR pode ser classificada
em três tipos:
x Recuperação por desenho (sketch): neste
tipo de pesquisa, o usuário esboça uma
imagem aproximada daquilo que ele deseja
recuperar;
x Recuperação por exemplos (QBE – Query By
Example): é a forma mais comum nos
sistemas atuais. Nela, o usuário fornece uma
imagem de exemplo, similar à qual ele deseja
recuperar;
x Busca Parametrizada: O usuário fornece
parâmetros de busca que descrevem a
imagem. Por exemplo, ‘buscar imagens com
60% de vermelho e 40% de verde’.
Os si stemas de recuperação baseada em
conteúdo são sempre compostos dos seguintes
módulos [8]: interface gráfica de consulta, sistema
de armazenamento e indexação de dados,
medida
de
similaridade,
extração
de
características e sistema de recuperação
(retrieval engine).
Parte da dificuldade em desenvolver
sistemas CBIR é determinar algoritmos que
caracterizem o conteúdo da imagem, diminuindo
Introdução
As imagens médicas têm se tornado cada
vez mais importantes para o diagnóstico e terapia
dos pacientes [1,2]. Porém, a sua manipulação
computacional tem se tornado cada vez mais
difícil devido ao aumento do volume de imagens
produzidas nos hospitais [3] (a produção anual de
um departamento radiológico de médio porte
representa dezenas Terabytes [4]) e devido às
crescentes necessidades dos médicos no uso
clínico das imagens [5]. Os si stemas PACS
(Picture, Archiving and Communication Systems)
existentes atualmente permitem somente a
pesquisa por palavras chave, como o identificador
do paciente, número do exame, data e
modalidade, os quais são parâmetros relevantes
para o médico, porém não em todos os caso s
[6,7]. Muitas vezes a recuperação de imagens
baseado em características intrínsecas da
imagem é benéfica para o médico, auxiliando-o
na tomada de decisões, ensino e pesquisa [8,9].
Os sistemas de recuperação de imagens baseado
em conteúdo (CBIR – Content Based Image
Retrieval) permitem a recuperação de imagens
utilizando características como cor, textura e
forma [10]. Devido a sua aplicação em
treinamento e pesquisa em Medicina, os sistemas
554
X Congresso Brasileiro de Informática em Saúde
o chamado “gap semântico” [8]. Dessa maneira, é
necessário ter a possibilidade de desenvolver a
testar algoritmos de uma maneira simples.
Neste artigo é apresentado o BIRAM (Base
de Imagens Relacional de Algoritmos e Métricas),
o qual é um protótipo de um sistema CBIR capaz
de suportar diversos algoritmos de extração de
características.
As imagens são processadas invocando-se
o método processar() da classe Algorithm (Figura
3), o qual retorna um objeto do tipo Descriptor.
O descritor é armazenado no banco de
dados (Figura 2, tabela Alg_x_Img) na forma
serializada. A partir deste momento a base pode
ser pesquisada utilizando-se este algoritmo.
A busca por imagens semelhantes é
realizada da seguinte forma: a imagem fornecida
como exemplo é processada por um dos
algoritmos armazenados, gerando um descritor.
Em seguida, o descritor é comparado com
aqueles armazenados no banco de dados. A
comparação é feita percorrendo-se os descritores
um a um, deserializando-os e invocando o
método calculateDistance(). Com o resultado das
distâncias é possível ordenar as imagens por
similaridade, usando o critério de medida de
distância do algoritmo.
O sistema ainda permite a associação de
um laudo ao estudo (tabela Report, Figura 2). O
laudo é composto de cinco itens: tipo de exame,
histórico, procedimento, resultados encontrados e
conclusões (tabela Conclusion, Figura 2). As
conclusões são codificadas segundo um
dicionário médico (tabela Dictionary, Figura 2).
Atualmente, é suportado o CID-10 (Classificação
Internacional de Doenças – versão 10).
Também foi prevista a possibilidade de
associação de uma região de interesse (ROI) ao
algoritmo (tabela ROI_Algorithm, Figura 2),
apesar de a mesma ainda não ter sido
implementada.
As tabelas armazenam as imagens
segundo o padrão DICOM (Digital Imaging and
Communications in Medicine), no qual o paciente
possui estudos, os quais possuem séries que
possuem imagens (tabelas Patient, Study, Series
e Image, Figura 2).
Metodologia
O BIRAM foi implementado utilizando-se a
linguagem Java e banco de dados MS Access 97.
O BIRAM atualmente utiliza características
especificas da linguagem Java como serialização,
invocação de métodos remotos (RMI) e
instanciação
dinâmica.
Suas
principais
funcionalidades são (Figura 1):
x Armazenamento de imagens;
x Armazenamento de algoritmos;
x Associação de laudos com imagens;
x Recuperação de algoritmos;
x Recuperação de imagens por semelhança;
x Associação de um laudo com as imagens.
O armazenamento de algoritmos é feito
através do envio de um (ou mais) arquivo(s) jar
(Java ™ Archive) para o servidor. Uma vez salvos
os arquivos, é instanciada a classe principal do
algoritmo (aquela que implementa a interface
Algorithm, Figura 3). A partir dessa instância, são
inseridas meta-informações sobre o algoritmo no
banco de dados (Figura 2, tabela Algorithm).
O armazenamento em banco permite a
realização de consultas SQL que tragam somente
as imagens apropriadas para um determinado
algoritmo (no caso, imagens com modalidade,
parte do corpo e número de bits iguais aos
suportados pelo algoritmo). Dessa maneira, é
possível limitar o processamento somente às
imagens selecionadas.
Buscar Imagem Laudo
Armazenar Laudo
Medi co
Usuario
Buscar Imagem Similari dade
Buscar Algoritmo
Armazenar Imagem
Armazenar Algoritmo
Figura 1 – Casos de uso do BIRAM
555
Desenvolvedor
X Congresso Brasileiro de Informática em Saúde
Figura 2 – Diagrama de tabelas do BIRAM
<<Interface>>
java.rmi .Remote
<<Interface>>
java.io.Serializable
<<Interface>>
A lgorithm
<<Interface>>
Descriptor
+produces
calculateDistance()
getName()
getDescription()
+contains
getAuthor()
getVersion()
1
0..*
getBodyPart()
getModality()
getNumberOfBits()
process()
<<Interface>>
Server
storeDICOMImage()
searchImage()
li stAlgorithms()
storeAlgorithm()
applyAlgorithm()
saveReport()
li stDictionaries()
li stDictionaryCodes()
Figura 3 – Diagrama das classes principais do BIRAM
algoritmos armazenados. Atualmente, foram
implementados
algoritmos
simples
como
momento, histograma, histograma métrico e
centro de massa.
Na Figura 4 é mostrada a tela de Laudo,
que permite associação do laudo (composto de
histórico,
procedimentos,
resultados
e
conclusões) com o estudo. O estudo é
selecionado de uma lista de pacientes.
Atualmente, o BIRAM torna anônimos os nomes
dos pacientes inseridos no sistema.
Resultados
O BIRAM foi implementado juntamente
com um cliente standalone. O cliente permite a
associação do laudo com um estudo (Figura 4).
Também permite a atribuição da conclusão
utilizando um dicionário médico (Figura 5).
Atualmente é suportado o CID-10.
O servidor é capaz de armazenar novos
algoritmos e imagens no padrão DICOM. Permite
a pesquisa por similaridade, utilizando os
556
X Congresso Brasileiro de Informática em Saúde
seleciona o algoritmo que deseja utilizar para
fazer a busca por similaridade (canto inferior
esquerdo). Pressionando o botão “Buscar
imagens Semelhantes” é recuperada uma lista de
imagens, como mostrado na Figura 7. Os valores
mostrados abaixo das imagens são o valor de
distância
retornado
pelo
método
calculaDistancia() conforme explicado na seção
anterior.
Na Figura 5 pode ser vista a atribuição de
uma conclusão ao laudo, utilizando para isso um
dicionário médico selecionado em uma list box.
Figura 6 – Tela de busca de imagens
semelhantes
Figura 4 – Visualização de laudos
A tela da Figura 6 também permite que
sejam enviadas imagens para a base, as quais
são indexadas automaticamente para pesquisa.
Figura 5 – Atribuição de uma conclusão ao laudo
Figura 7 – Resultado de pesquisa utilizando
histograma métrico
Na Figura 6 é mostrada a tela de
pesquisa de imagens por similaridade. Nela, o
usuário abre uma imagem local, a qual é exibida
(canto superior esquerdo). Em seguida, o usuário
557
X Congresso Brasileiro de Informática em Saúde
Discussão e Conclusões
A implementação atual do BIRAM é
promissora, porém, ainda tem alguns problemas a
serem resolvidos:
x Embora o protocolo DICOM forneça dados
textuais sobre a imagem e o paciente, existe
uma imprecisão no preenchimento dos
mesmos (por exemplo, os campos ‘body part’
e ‘study description’ [11]). Isso torna não
confiável a utilização de tal informação para
divisão dos algoritmos por tipo de imagem;
x A utilização de um sistema CBIR na prática
clínica depende da existência de uma
interface que permita pesquisas mais
complexas, embutindo informações como
localização relativa e aspecto morfológico
[11].
x A implementação atual não realiza a
indexação das imagens armazenadas. Assim,
é necessário percorrer todas as imagens do
banco para encontrar aquelas mais similares.
Isto pode ser evitado com o uso de árvores
de indexação, como a slim-tree [12, 13] e
métricas que satisfaçam as três regras do
espaço métrico (simetria, não-negatividade e
desigualdade triangular).
O BIRAM apresenta as seguintes
vantagens: (i) Pode receber diversos tipos de
algoritmos para pesquisa por similaridade; (ii)
Permite a codificação do laudo de acordo com um
dicionário médico, melhorando a indexação das
imagens e, consequentemente, sua recuperação;
(iii) Os algoritmos podem ser seletivamente
aplicados nas imagens utilizando metainformações sobre o mesmo (por exemplo, aplicar
o algoritmo somente para imagens de
ressonância magnética).
O BIRAM ainda pode ser melhorado com o
uso de regiões de interesse (ROI), indexação com
métodos de acesso métricos (metric access
methods - MAM) e integração com PACS (usando
protocolo DICOM).
2.
3.
4.
5.
6.
7.
8.
Agradecimentos
9.
Este projeto foi suportado parcialmente
pela FAPESP processo s 1997/14206-5 e
2001/05987-0, Fundação Zerbini e Escola
Politécnica (EPUSP).
Agradecemos também à Natalia Rosa e
Agma Traina por cederem gentilmente o algoritmo
de histograma métrico.
10.
Referências
11.
1. Bruce I. Reiner, Eliot L. Siegel, Frank J.
Hooper, Stephen Pomerantz, Andrew Dahlke,
David Rallis (2001), “Radiologists’ Productivity
in the Interpretation of CT Scans: A
Comparison of PACS with Conventional Film”,
12.
558
American
Journal
of
Roentgenology,
176(4):861-4.
B. Kaplan, H.P. Lundsgaarde, (1996), Toward
an evaluation of an integrated clinical imaging
sy stem: Identifying clinical benefits, Methods
Inform. Med. 35, 221—229.
J. Brender; C. Nohr; P. McNair (2000).
“Researchs and
priorities in
health
informatics”, International Journal of Medical
Informatics, vol.58-59, pp. 257-289.
Tristan Glatard, Johan Montagnat, Isabelle E.
Magnin (2004), “Texture based medical image
indexing and retrieval: application to cardiac
imaging”, International Multimedia Conference
archive, Proceedings of the 6th ACM SIGMM
international
workshop
on
Multimedia
information retrieval, New York, NY, USA, pp.
135-142.
K. P. Andrioli, “Addressing the Coming
Radiology Crisis: The Society for Computer
Applications” in Radiology Transforming the
Radiological Interpretation Process (TRIP™)
Initiative, November 2003. Disponível em:
http://www.scarnet.org/pdf/TRIPwhitepaper11
03.pdf.
Wesley W. Chu, Chih-Cheng Hsu, Alfonso F.
Cárdenas, Rick K. Taira (1998), “KnowledgeBased Image Retrieval with Spatial and
Temporal Constructs”, IEEE Transactions on
Knowledge and Data Engineering, Vol. 10,
No. 6.
Thomas M. Lehmann, Mark O. Guld, Thomas
Deselaers, Daniel Keysers,Henning Schubert,
Klaus Spitzer, Hermann Ney, Berthold B.
Wein (2005), "Automatic categorization of
medical images for content-based retrieval
and data mining", Computerized Medical
Imaging and Graphics, 29, 143–155.
Henning Müller, Nicolas Michoux, David
Bandon, Antoine Geissbuhler (2004), “A
review of content-based image retrieval
systems in medical applications–—clinical
benefits and future directions”, International
Journal of Medical Informatics 73, 1—23.
Mojsilovic, and J. Gomes, (2002), “Semantic
based categorization, browsing and retrieval
in medical image databases”, Proc. Int. Conf.
Image Processing, ICIP 2002, Rochester,
New York, Sept.
Arnold W.M. Smeulders, Marcel Worring,
Simone Santini, Amarnath Gupta, and
Ramesh Jain (2000). “Content-Based Image
Retrieval at the End of the Early Years”, IEEE
Transactions on Pattern Analysis and
Machine Intelligence, vol. 22, no. 12.
Daniel Keysers, Jörg Dahmen, Hermann Ney
(2003), “Statistical framework for modelbased
image
retrieval
in
medical
applications”, Journal of Electronic Imaging
12(1), 59–68.
Caetano Traina Jr, Agma Traina, Christos
Faloutsos, Bernhard Seeger, (2002), “Fast
X Congresso Brasileiro de Informática em Saúde
indexing and Visualization of Metric Data Sets
Using Slim-Trees”, IEEE Transactions on
Knowledge and Data Engineering, vol 14(2).
13. Caetano Traina Jr, Agma Traina, Bernhard
Seeger, Christos Faloutsos, (2000) “SlimTrees: High Performance Metric Trees
Minimizing
Overlap
Between
Nodes”,
International Conference on Extending
Database Technology, 2000.
Contato
Ramon A. Moreno. Endereço: Instituto do
Coração – Hospital das Clinicas da Faculdade de
Medicina da USP (InCor - HCFMUSP), Av. Dr.
Enéas de Carvalho Aguiar, 44, 2º. Andar,
Unidade
de
Informática.
E-mail:
[email protected]. Telefone: (0xx11)
3069-5547.
559