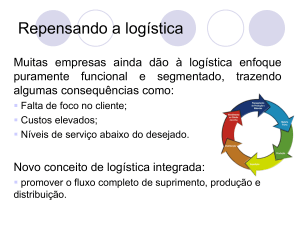
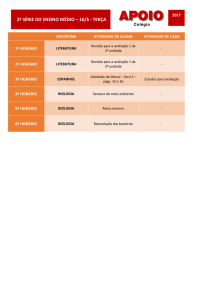
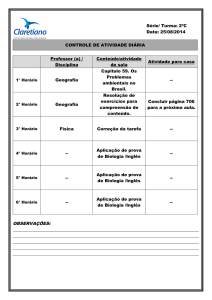
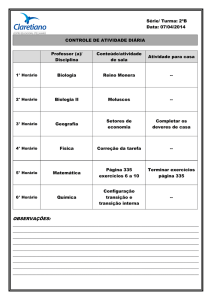
Apesar da grande quantidade de seqüências determinadas (260.175
seqüências e 3.874.166 transcrições no UniProtKB/TrEMBL em
06/03/2007) a quantidade de estruturas tridimensionais
determinadas é significativamente menor (38.750 no PDB em
15/03/2007).
O problema de determinar a estrutura tridimensional partindo de uma
seqüência conhecida (folding problem) é portanto a chave do
desenvolvimento da biologia pos-genômica.
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Homologia : similaridade de estrutura, fisiologia, desenvolvimento e
evolução de organismos baseados em fatores genéticos comuns:
Evolução de um ancestral comum.
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
homologia
1164 – BIOLOGIA ESTRUTURAL
Analogia (evolução convergente)
Aula 4
Prof. Dr. Valmir Fadel
•Enterotoxin (1lts-d)
é estruturalmente
similar à todas as
outras proteínas
mostrada na figura.
•A similaridade estrutural com a toxina cholera (1chp-d) é homologamente próxima uma vez que a
similaridade estrutura/função é acompanhada por uma alta identidade sequencial (80%).
•A segunda similaridade estrutural com a toxina toxic shock syndrome (TSS; 1tss) funcional, mas
com insignificante similaridade sequencial (8.8%) e são então classificada como homologas
remotas.
•A terceira similaridade estrutural com um domínio da aminoacyl tRNA synthetase (1krs) não é
acompanhada nem por significante similaridade seqüencial (4.4%) nem por similaridade funcional
sendo então classificadas como analogas.
Recognition of analogous and homologous protein folds: Analysis of sequence and structure conservation. RB Russell, MAS Saqi, RA Sayle,PA Bates & MJE Sternberg J. Mol. Biol. 269, 423-439, 1997.
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Homologia pode ser identificada através de similaridades
estatisticamente significantes e a similaridade seqüencial é a
mais evidente em proteínas.
Usualmente homologia implica em similaridade funcional em
diferentes organismos (ortologos) mas existem exceções.
Pela evolução uma proteína pode adquirir novas funções
como, por exemplo uma proteína que tem funções estrutural
na cristalino que é homologamente relacionada com a enzima
lactase dhidrogenase (parologos).
Duas proteínas são passíveis de serem homólogas de se
compartilham uma alta identidade seqüencial, ou seja, resíduos de
aminoácidos idênticos em posições seqüenciais (>30%) embora a
homologia possa ser identificada por similaridade estrutural mesmo
quando a identidade seqüencial é baixa.
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Proteínas homologas compartilham um núcleo estruturalmente
conservado, mesmo em proteínas com homologia distante,
composto geralmente por estruturas secundárias.
Regiões variáveis que conectam estas estruturas
secundárias variam consideravelmente em tamanho e
forma.
Hemoglobina canina superposta à hemoglobina
humana (branca). Vê-se a ajuste das estruturas
secundárias para compensar diferenças na
seqüência.
1164 – BIOLOGIA ESTRUTURAL
Fosfolipase A2 suina e bobina superpostas onde se vê a
conservação do núcleo a pequenos ajustes nas alças.
Aula 4
Prof. Dr. Valmir Fadel
O problema de determinar uma estrutura por homologia
consiste basicamente em determinar a forma destes loops e
a posição das cadeias laterais após ajustar o núcleo.
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Loops não apresentam estruturas completamente aleatórias
sendo dependente do número de resíduos que o compõe e
quais elementos ele conecta.
Sítios de ligação de antígenos (alças) em imunoglobinas. 1ª linha: modelados. 2ª linhas: experimental
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Cadeias laterais tem suas possíveis orientações determinadas
por minimização energética enquanto que análise de
estruturas conhecidas mostram que poucas conformações
ocorrem freqüentemente (rotâmeros).
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
O conhecimento das regras de empacotamento das
estruturas secundárias gerando um limitado número de
possíveis empacotamentos fazem com que a predição
das estruturas secundárias seja atualmente a chave da
predição da estrutura terciária.
O problema da predição das estruturas secundárias é que a
sua formação e interligada com a estrutura terciária. Por
outro lado co conhecimento do tipo de domínios existentes
pode facilitar a predição das estruturas secundarias.
Diferentes métodos de predição de estruturas
secundarias, baseados em critérios empíricos ou em
critérios estereoquimicos, podem apresentar precisão
de ate 75%.
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Alinhamento de seqüências
Objetivos:
•Medir a similaridade
•Determinar a relação resíduo-resíduo
•Observar padrões de conservação e variabilidade
•Inferir relações evolucionárias
•Pesquisar bancos de dados
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Um alinhamento (resultado):
Fosfolipase porcina e bovina
CLUSTAL W (1.83) multiple sequence alignment
1P2P__|PDBID|CHAIN|SEQUENCE -------ALWQFRSMIKCAIPGSHPLMDFNNYGCYCGLGGSGTPVDELDR 43
2BP2__|PDBID|CHAIN|SEQUENCE EAGLNSRALWQFNGMIKCKIPSSEPLLDFNNYGCYCGLGGSGTPVDDLDR 50
*****..**** **.*.**:*******************:***
1P2P__|PDBID|CHAIN|SEQUENCE CCETHDNCYRDAKNLDSCKFLVDNPYTESYSYSCSNTEITCNSKNNACEA 93
2BP2__|PDBID|CHAIN|SEQUENCE CCQTHDNCYKQAKKLDSCKVLVDNPYTNNYSYSCSNNEITCSSENNACEA 100
**:******::**:*****.*******:.*******.****.*:******
1P2P__|PDBID|CHAIN|SEQUENCE FICNCDRNAAICFSKAPYNKEHKNLDTKKYC 124
2BP2__|PDBID|CHAIN|SEQUENCE FICNCDRNAAICFSKVPYNKEHKNLD-KKNC 130
***************.********** ** *
* Resíduos idênticos - : resíduos conservados - . Resíduos semi-conservados
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel
Outro alinhamento (resultado):
Subtisilina e chimotripsina
CLUSTAL W (1.83) multiple sequence alignment
1GNS_A|PDBID|CHAIN|SEQUENCE GPYGVSQIKAPALHSQGYTGSNVKVAVIDSGIDSSHPALKVAGGASFVPS 50
2CHA_A|PDBID|CHAIN|SEQUENCE --CGVPAIQP------VLSGLXXIVNGEEAVPGSWPWQVSLQDKTGFHFC 42
**. *:.
:*
*
:: .*
:.: . :.* .
1GNS_A|PDBID|CHAIN|SEQUENCE ETNPFQDNNSHGTHVAGTVLAVAPSASLYAVKVLGADGSGQYSWIINGIE 100
2CHA_A|PDBID|CHAIN|SEQUENCE GGSLINEN--------WVVTAAHCGVTTSDVVVAGEFDQGSSSEKIQKLK 84
. :::*
.* *. ..:
* * * ..*. * *: ::
1GNS_A|PDBID|CHAIN|SEQUENCE WAIAN-NMDVINMSLGGPSGSAALKAAVDKAVASGVVVVAAAGNEGTSGS 149
2CHA_A|PDBID|CHAIN|SEQUENCE IAKVFKNSKYNSLTINNDITLLKLSTAASFSQTVSAVCLPSASDDFAAGT 134
* . * . .:::..
*.:*.. : : ..* :.:*.:: ::*:
1GNS_A|PDBID|CHAIN|SEQUENCE SSTVGYPGKYPSVIAVGAVDSSNQRASFSSVGPELDVMAPGVSIWSTLPG 199
2CHA_A|PDBID|CHAIN|SEQUENCE TCVT-------TGWGLTRYXXANTPDRLQQASLPLLSNTNCKKYWGTKIK 177
:...
: .:
:*
:.... *
: . *.*
1GNS_A|PDBID|CHAIN|SEQUENCE NKYGAKSGTCMASPHVAGAAALILSKHPNWTNTQVRSSLENTTTKLGDSF 249
2CHA_A|PDBID|CHAIN|SEQUENCE DAMICAGASGVSSCMGDSGGPLVCKKNGAWTLVGIVSWGSSTCSTSTPGV 227
:
. ..: ::*
....*: .*: ** . : * ..* :.
..
1GNS_A|PDBID|CHAIN|SEQUENCE YYG-KGLINVEAAAQ--- 263
2CHA_A|PDBID|CHAIN|SEQUENCE YARVTALVNWVQQTLAAN 245
*
..*:*
:
1164 – BIOLOGIA ESTRUTURAL
Aula 4
Prof. Dr. Valmir Fadel