Ação Gênica
Estágio Docência
Vanessa Veltrini Abril – Doutoranda em
Genética e Melhoramento Animal
Março de 2007
Qual é a função do DNA?
Como a informação genética é
transportada?
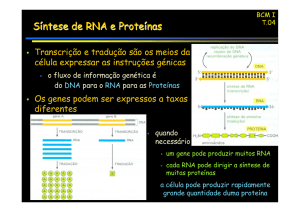
Genes
TRANSFERÊNCIA DE INFORMAÇÃO
DNA
DNA
Replicação
Proteínas
RNA
Transcrição
Tradução
Universalidade: Dogma Central
Como a informação genética está representada em
cada processo?
• Replicação – seqüências de desoxiribonucleotídeos
(A, T, C, G) = fita dupla DNA
• Transcrição – seqüência de ribonucleotídeos (A, U,
C, G) = fita simples RNA
• Tradução – seqüência de aminoácidos (leucina,
valina, alanina, metionina...) =
polipeptídios/proteínas
LOCALIZAÇÃO
REPLICAÇÃO
TRANSCRIÇÃO
DNA
RNA
= pequena proporção do DNA total >RNA
Fita molde DNA 3’-5’ (anti-senso)
Fita não molde DNA 5’-3’ (senso)
Fita RNA cresce 5’-3’ (similar à fita não molde)
RNA polimerase dependente de DNA (RNA pol)
=Vídeo Transcrição
TRANSCRIÇÃO – Procariotos
1. Iniciação
2. Alongamento
3. Término
α2ββ’σ - Holoenzima
α ββ’ - Cerne da enzima
RNA polimerase
- Regiões promotoras:
-35
5’ TTGACA 3’
-10
5’ TATAAT 3’
- Subunidade σ
- Complexo promotor fechado
- Complexo promotor aberto
- Bolhas de transcrição
-RNApol migra no sentido 3’5’do DNA
- Ribonucleotídeos adicionados
na extremidade 3’ da fita RNA
crescente;
Término
- Dissociação da RNApol e do
DNA = liberação do RNA
transcrito
- Finalizadores
Terminação:
- Palíndromos complementares
- Finalizadores dependentes de rô
TRANSCRIÇÃO - Eucariotos
- 3 tipos RNApol – promotores próprios
RNApol I – genes rRNA
RNApol II – genes de proteínas (mRNA)
RNApol III - tRNA, rRNA 5S
RNA polimerase II
Complexo pré-iniciação: polimerase + fatores de transcrição
promotor: TATA box: 5’ -TATAAAT- 3’
TBP – proteína de ligação ao TATA
2. Vídeo Transcrição eucariotos
PRODUTOS DA TRANSCRIÇÃO
mRNA
Intermediário entre gene e produto final
da expressão – passa pelo 2° estágio da
expressão gênica: TRADUÇÃO
rRNA
RNA estáveis : Produtos finais da
expressão gênica
tRNA
mRNA
-maioria das moléculas são instáveis
-sofrem modificações e processamento antes de
irem para o citoplasma
5’: Trifosfato de 7-metilguanosina - capeamento
3’: resíduos de adenilato (AMP) - poliadenilação
GENE: ÉXONS + ÍNTRONS
-Remoção de íntrons (splicing)
-Edição do RNA
Splicing do gene da Ovoalbumina
Edição do mRNA da APOLIPROTEÍNA-B humana
Apoliproteína-B100: sintetizada nas células hepáticas
Apoliproteína-B48: sintetizada pelas células intestinais
1 gene especificando 2 proteínas diferentes
mRNA das células intestinais: uma CITOSINA é
desaminada (perda de um NH2), convertendo-se em
URACILA
CAA
UAA
(Códon finalizador)
Conseqüência: proteína do intestino é menor que a do fígado.
rRNA
-Componentes dos ribossomos (“fábricas” para síntese proteíca);
- Ribossomos procariontes: 3 moléculas rRNA
eucariontes: 4 moléculas rRNA
tRNA
-Papel adaptador: leitura da seqüência de nucleotídeos
do mRNA, os convertendo em aminoácidos.
-Estrutura:
a. Trevo de tRNA
TRADUÇÃO
mRNA + rRNA + tRNA
tRNA: elo físico e informacional entre a seqüência de
nucleotídeos do mRNA e a seqüência de aminoácidos
do polipeptídeo.
tRNA – ligação covalente com o aminoácido no braço
aceptor do trevo
Aminoacil--tRNAAminoacil
tRNA-sintetase
Síntese Proteíca em Procariotos:
1. INICIAÇÃO:
- 30S + mRNA (sítio de ligação do ribossomo) – até
encontrar códon iniciação AUG
- Complexo de iniciação: pareamento do tRNAfmet
com códon AUG
(Formilmetionina)
- Fatores de iniciação
2. ALONGAMENTO
- Ligação da subunidade 50S;
- Sítio peptidil (P’) e sitío aminoacil (A);
- Fatores de alongamento;
- Peptidiltransferase: ligação entre aminoácidos
3. FINALIZAÇÃO
- Códon finalizador (UAA, UAG, UGA) no sítio A;
- Fatores de liberação: se ligam ao códon
finalizador.
Síntese Proteíca em Eucariotos:
-Subunidade 40S reconhece a extremidade 5’cap
(como sítio de ligação);
-Aminoácido iniciador é uma METIONINA não
modificada;
-Vários fatores de iniciação;
-Hidrólise de ATP para iniciar;
-Hidrólise de GTP para finalizar.
3. Vídeo Tradução
Para que saber tudo isso?
Aplicações práticas?
4. Vídeo Vaga-lume
INFORMAÇÃO GENÉTICA
Regras
Código Genético
Código Genético
Marshall Nirenberg -1961
Nobel 1968
Regras que determinam
qual seqüência de
nucleotídeo especifica
qual seqüência de
aminoácido.
2 suposições para decifrar o código genético:
A. COLINEARIDADE ENTRE GENE E
PROTEÍNA;
B. CADA CÓDON DO CÓDIGO É
UMA TRINCA DE NUCLEOTÍDEOS.
Características do Código:
-É redundante:
64 trincas
20 aminoácidos
(Códons sinônimos)
Ex. metionina e triptofano: 01 códon
Todos os outros: +01 códon
-Códons de Pontuação:
-Códon de iniciação: AUG (metionina)
-Códons de terminação: UAA, UGA, UAG
-Código não é UNIVERSAL:
-Mitocôndria
-Drosophila, protozoários, bactérias, etc
MUTAÇÕES NOS CÓDONS
1ª
2ª
3ª
- Alterações na 1ª posição gera substituição de aa.
com propriedades fisíco-químicas semelhantes;
- Alterações na 2ª posição gera substituição de aa.
não relacionados;
- Alterações na 3ª posição não gera substituição.
(DEGENERAÇÃO)
SUBSTITUIÇÕES DE AMINOÁCIDOS:
– mis-sense ou de sentido trocado: mudança de 1 aa
por outro
– non-sense ou sem sentido: terminação precoce
(Códon finalizador)
- perda de fase, frameshift ou mudança de matriz de
leitura: deleções ou inserções de bases
ex: normal AUG GCU UCU GCG CAG AUU AGG CAC ...
AC UGC GGC AGA UUA GGC AC ...
mutante AUG GCU UA
inserção