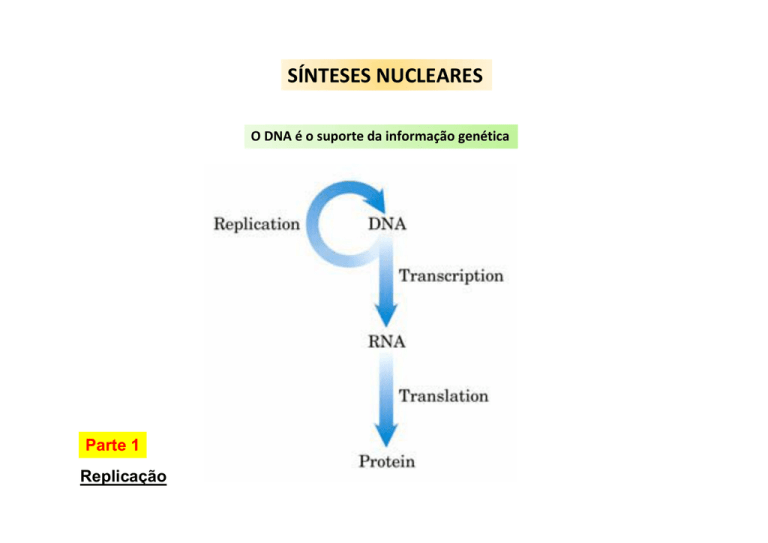
SÍNTESES NUCLEARES
O DNA é o suporte da informação genética
Parte 1
Replicação
Replicação do DNA
Estrutura do DNA
Nucleótidos
A informação genética das células é armazenada sob a forma de 2 moléculas similares: DNA (ácido desoxirribonucléico) e RNA (ácido ribonucléico).
Na sua estrutura primária, ambas as moléculas são polímeros de unidades químicas: os nucleotídeos.
Um nucleotídeo tem 3 partes:
‐ um grupo fosfato: responsável pela acidez de DNA e RNA, uma vez que fica carregado negativamente em pH intracelular
‐ um pentose: é um resíduo de açúcar com 5 átomos de carbono. No RNA o pentose é sempre um ribose, no DNA é sempre um desoxirribose;
‐ uma base orgânica azotada: A, T, G ou C para o DNA; A, U, G ou C para o RNA.
Replicação do DNA
Estrutura do DNA
Nucleótidos
As sequências nucleotídicas derivam de 4 bases heterocíclicas azotadas:
‐ bases púricas: Adenina (A) e Guanina (G)
‐ bases pirimídicas: Timina (T) ou Uracilo
(U) e Citosina (C). Replicação do DNA
Estrutura do DNA
Polimerização de nucleótidos
Quando os nucleotídeos polimerizam para formar ácidos nucléicos, o grupo OH ligado ao carbono 3’ do açúcar de um nucleotídeo, forma um ponte éster com o fosfato ligado ao carbono 5’ do açúcar do nucleótido seguinte.
Esta ligação entre os nucleótidos é chamada: ligação fosfodiéster.
A cadeia do ácido nucléico tem uma orientação:
‐ extremidade 3’: com um grupo hidroxilo (OH) livre no carbono 3’ do açúcar;
‐ extremidade 5’: com um grupo fosfato livre no carbono 5’ do açúcar.
A orientação da cadeia de um ácido nucléico é uma característica extremamente importante da molécula.
Polimerização de
nucleótidos
Replicação do DNA
Estrutura do DNA
A era da biologia molecular moderna começou em 1953 quando James Watson e Francis Crick descreveram, por difracção aos raios X, a organização em dupla hélice do DNA.
O DNA consiste na associação entre 2 cadeias polinucleotídicas com enrolamento helicoidal no espaço descrito como dupla hélice.
Os resíduos de açúcar + fosfato encontram‐se no exterior da hélice, as bases no interior. A orientação das 2 cadeias é
antiparalela e a sua associação faz‐se através de ligações de hidrogénio entre as bases, bem como através de interacções
hidrofóbicas. Para manter a geometria da dupla hélice, as purinas (A ou G) devem emparelhar com as pirimidinas (T ou C): i.e. as bases grandes emparelham com as bases pequenas.
Modelo de Watson‐Crick
Replicação do DNA
Estrutura do DNA
Desnaturação‐Renaturação do DNA
Durante a replicação do DNA ou a síntese de RNA, as 2 cadeias separam‐se provisoriamente. É possível induzir a separação das 2 cadeias no laboratório. Por exemplo, aquecendo uma solução de DNA, a energia térmica aumenta os movimentos moleculares quebrando os pontes de hidrogénio e destabilizando a dupla hélice, separando assim as 2 cadeias: fala‐se de desnaturação do DNA. Replicação do DNA
A replicação do DNA é semi‐conservativa
Cada cadeia de DNA é duplicada,
formando uma cadeia híbrida: após
a replicação, cada cadeia parental
emparelha‐se com uma cadeia nova, formando um novo DNA.
Replicação do DNA
A replicação do DNA é bi‐direccional
Forquilha de replicação
John Cairns evidenciou por autoradiografia a presença de "olhos" ou "bolhas de replicação" nos cromossomas circulares de bactérias crescidas em meio de cultura contendo [3H]‐timidina.
A análise indica que o DNA dupla hélice se replica por um processo progressivo de separação das duas cadeias parentais, acompanhada pela síntese de uma nova cadeia complementar a este DNA (cadeia‐
filha).
As 2 extremidade da "bolha" representam o local de replicação do DNA é são chamadas forquilhas de replicação (setas na figura).
Replicação do DNA
A replicação do DNA é bi‐direccional
Forquilha de replicação
Em procariotas e bacteriófagos, o DNA tem apenas uma "bolha“ de replicação, enquanto em eucariotas há formação de várias unidades de replicação (replicões) distribuídas ao longo do DNA.
Replicação do DNA
Origem de replicação
Nos procariotas e eucariotas, a origem das forquilhas de replicação situa‐se nos “olhos de replicação” (regiões onde as 2 cadeias se separaram) que se formam ao nível de sequências específicas do DNA chamadas: origens de replicação. Estas sequências servem de ligação à proteínas que iniciam o processo de replicação: iniciadores.
Enquanto que o genoma das bactérias e dos vírus tem uma única origem de replicação, o genoma dos eucariotas possui múltiplos origens de replicação (~30000 no homem).
Replicação do DNA
A maquinaria de replicação do DNA
‐ DNA polimerases
‐ Primase
‐ DNA helicases
‐ DNA topoisomerases (DNA girase em procariotas)
‐ DNA ligase
‐ Telomerase
‐ Proteínas de ligação às cadeias simples de DNA (ssDBP)
‐ Proteínas acessórias das DNA polimerases
Replicação do DNA
DNA polimerases
3 DNA polimerases em procariotas: I, II e III
‐ DNA polimerases I e III: são essenciais ao processo de replicação
‐ DNA polimerase II: está mais ligada aos processos de reparação de erros no DNA
5 DNA polimerases em eucariotas: α, β, γ, δ, e ε
‐DNA polimerases α, δ e ε : são essenciais ao processo de replicação do DNA nas células em divisão
‐ DNA polimerase β : está mais ligada aos processos de reparação de erros no DNA
‐ DNA polimerase γ: localiza‐se na mitocôndria e é responsável pela replicação do DNA mitocondrial
Replicação do DNA
DNA polimerases
Todas as DNA polimerases partilham 2 características fundamentais:
1. Todas as DNA polimerases só sintetizam o DNA na direcção de 5’ Æ 3’, adicionando os dNTPs ao 3’‐hidroxilo da cadeia nova em crescimento e utilizando a cadeia molde na direcção de 3’ Æ 5’.
2. São incapazes de sintetizar o DNA de novo: precisam de uma pequena sequência de RNA (4 a 10 nucleótidos) pré‐formada emparelhada à sequência complementar na cadeia molde. Esta sequência é designada por RNA iniciador ou primer.
Replicação do DNA
Iniciação da replicação do DNA
Utilizando uma cadeia simples de DNA como molde e um pequeno fragmento de RNA iniciador sintetizado pela primase, a DNA polimerase adiciona o grupo fosfato de um novo nucleótido ao grupo hidroxilo da extremidade 3’ da cadeia sintetizada de novo, que se alonga na direcção 5´para 3´.
Replicação do DNA
DNA polimerases
A DNA polimerase adiciona o grupo fosfato de um novo nucleótido ao grupo hidroxilo da extremidade 3’ da cadeia sintetizada de novo, que se alonga na direcção 5´para 3´.
A escolha do nucleótido a adicionar à cadeia nova é
ditado pela sequência da cadeia molde.
Replicação do DNA
Papel das proteínas iniciadoras
Nos procariotas e eucariotas, o local de iniciação da replicação do DNA é determinado por sequências particulares do DNA chamadas: origens de replicação. Estas sequências servem de ligação à proteínas que iniciam o processo de replicação: proteínas
iniciadoras.
Replicação do DNA
A replicação do DNA é semi‐contínua
As cadeias do DNA são anti‐paralelas (uma cadeia tem direcção de 5’ para 3’ enquanto que a outra tem a direcção de 3’ para 5’). Como a DNA polimerase realiza a replicação da cadeia que está na direcção de 5’para 3’?
Resposta: As duas cadeias do DNA parental são replicadas por caminhos diferentes.
‐ Uma das cadeia novas é sintetizada de maneira contínua (é cadeia avançada ou cadeia leading) na direcção de 5’para 3’, pela acção da DNA polimerase, na mesma direcção de movimento da forquilha.
‐ A outra nova cadeia é sintetizada de maneira descontínua (é a cadeia atrasada ou cadeia lagging) na direcção de 5’para 3’ pela DNA polimerase, mas em sentido contrário à direcção da forquilha de replicação . A síntese desta cadeia é feita aos poucos sob a forma de fragmentos de Okazaki.
DNA polimerase
Estrutura da
forquilha de replicação
Replicação do DNA
Iniciação da replicação do DNA
A cadeia leading é sintetizada pela DNA polimerase III nos procariotas e pelas polimerases
δ/ε nos eucariotas.
A síntese da cadeia lagging é inicada pela primase e extendida pela polimerase III nos procariotas, e é iniciada pelo complexo primase/polimerase α e extendida pelas polimerases
δ/ε em eucariotas. δ/ε
α
δ/ε
Replicação do DNA
Remoção do RNA iniciador (primer do RNA)
Nos procariotas, a DNA polimerase I utiliza a sua actividade exonucleásica para remover os primers e substituir os ribonucleótidos
pelos desoxirribonucleótidos. Nos eucariotas, esta actividade é
desempenhada pela polimerase δ.
Os fragmentos de DNA são depois unidos pela DNA ligase, que cataliza uma ligação fosfodiéster entre o último nucleótido em 3’ de um fragmento de Okazaki e o primerio
nucleótido em 5’ do fragmento seguinte.
Replicação do DNA
Acção da DNA helicase e das proteinas
de ligação às cadeias simples de DNA
As DNA helicases separam as duas cadeias parentais do DNA a frente da forquilha de replicação, rompendo os pontes hidrogénio entre as bases, um esforço que necessita ATP.
Proteínas de ligação ao DNA cadeia simples
cooperam para estabilizar e estender o DNA cadeia simples, facilitando a acção de DNA Polimerases.
Replicação do DNA
Acção das topoisomerases durante
a replicação do DNA
As topoisomerases actuam a montante da forquilha da replicação para aliviar a tensão de torção das cadeias parentais durante o seu desenrolar pela helicase.
Replicação do DNA
As enzimas de replicação do DNA na forquilha da replicação
As enzimas envolvidas na replicação do DNA actuam de maneira coordenada para sintetizar simultaneamente
as duas cadeias leading e lagging ao nível da forquilha de replicação.
O molde da cadeia lagging
dobra‐se de modo a permitir
à DNA polimerase actuar na direcção da forquilha de
replicação ajudando assim a contornar o problema da
orientação antiparalela das cadeias parentais.
Replicação do DNA
A fidelidade na replicação do DNA
A DNA polimerase pode corrigir erros de replicação
Durante a replicação ocorre cerca de 1 erro em cada 9 ou 10 milhões de nucleótidos. Os estudos apontam para uma frequência de erros muito mais elevada se estes derivarem exclusivamente do emparelhamento das bases. Assim, pensou‐se que deveriam existir outros mecanismos que explicassem a elevada fidelidade do processo de replicação.
A resposta a esta dúvida foi esclarecida através da observação da existência de uma acção das enzimas DNA polimerases I e III, na sua acção exonucleásica de 3’para 5’, retirando nucleótidos em direcção oposta àquela em que funciona a polimerase. Se um nucleótido errado é
inserido na cadeia, a enzima polimerase reconhece‐o, (uma vez que os nucleótidos não irão formar pontes de hidrogénio) e retorna ao ponto onde ocorreu o erro, hidrolisando o nucleótido errado a partir da extremidade 3’. Depois de removido o nucleótido a enzima polimerase continua agindo, agora com actividade polimerásica.
Esta revisão e a capacidade de correcção (proofreading) é muito importante pelo facto de que erros na replicação comprometem toda a descendência (enquanto que erros na transcrição ou na tradução comprometem apenas uma proteína de determinada célula).
Replicação do DNA
Replicação dos telómeros do DNA
As DNA polimerases não são capazes de copiar as extremidades dos cromossomas lineares dos eucariotas. As extremidades dos cromossomas lineares consistem em sequências repetitivas ricas em “G” chamadas telómeros.
Os telómeros são replicados por uma enzima especial: telomerase.
A telomerase é uma transcriptase reversa, i.e. sintetiza DNA a partir de RNA. A cadeia de RNA molde faz parte da telomerase e é complementar ao DNA telomérico.
A telomerase é extremamente importante, pois evita que o DNA seja encurtado nas suas extremidades após cada ciclo de replicação.
Replicação do DNA
Replicação dos telómeros
Modo de acção da telomerase
1. Ligação do RNA da telomerase à sequência complementar do telómero
na cadeia leading;
2. Extensão da cadeia leading
(formação de sequências repetitivas) através da actividade de transcrição reversa;
3. Extensão da cadeia lagging
por primase e DNA polimerase;
4. Remoção do primer de RNA.
1
2
3
4
A cadeia de RNA molde é complementar ao DNA do telómero da cadeia leading.
Replicação do DNA
Aplicação: PCR (Polymerase Chain
Reactions)
A reacção de PCR necessita de:
. DNA molde (template)
. Primers específicos
. dNTPs
. Polimerase
. Tampão de reacção
Esta reacção realiza‐se num termociclador








