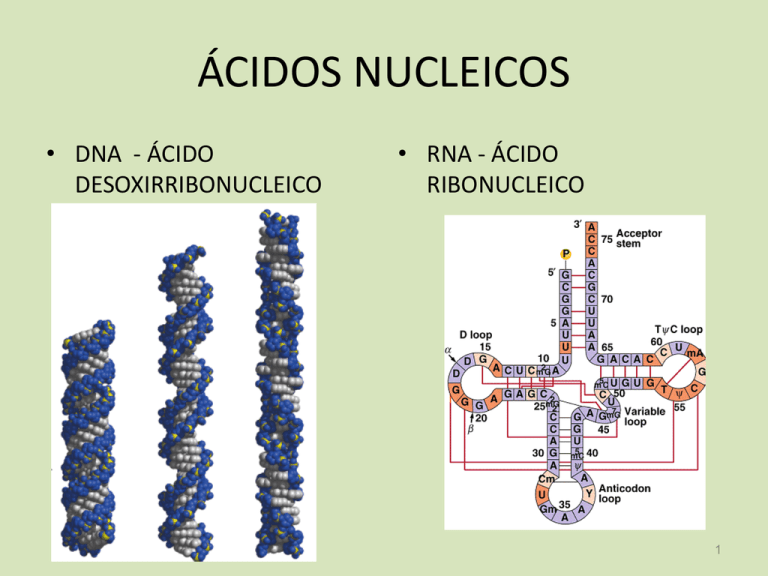
ÁCIDOS NUCLEICOS
• DNA - ÁCIDO
DESOXIRRIBONUCLEICO
• RNA - ÁCIDO
RIBONUCLEICO
1
Funções dos ácidos nucleicos
• Armazenar e expressar a informação genética
Replicação
– Cópia da mensagem contida
no DNA, que será
transmitida para as célulasfilhas
• Processo preciso
Transcrição
– primeiro estágio na
expressão da informação
genética
– Processo seletivo
Tradução
– Fase final do processo de
expressão gênica
2
DNA
• Funções:
– Armazenamento e expressão da informação genética
• Replicar de forma precisa cada que vez a célula se divide
• Expressar a informação que contém de forma seletiva
• Onde é encontrado?
– Em eucariotos:
• Cromossomos
• Mitocôndrias
• Cloroplastos
– Em procariotos:
• Cromossomo
• Plasmídeos
Cromossomo enovelado
de E. Coli
3
Molécula do DNA
5’3’
5’ACGT3’
Ligação fosfodiéster
4
Unidades que formam o DNA
Nucleotídeo
Base
nitrogenada
fosfato
pentose
Ligações
fosfodiéster
Esqueleto covalente do DNA
5
Bases Nitrogenadas
Adenina
Guanina
Purinas
Citosina
Timina
Pirimidinas
6
DUPLA HÉLICE DO DNA
Características:
Polaridade
Pareamento
Fitas antiparalelas
Fenda
menor
3’
5’
Fenda
maior
7
Principais formas da dupla hélice do DNA
A
B
Z
8
Principais formas estruturais da dupla hélice
B DNA
– Dupla hélice clássica
– Forma + abundante na
célula
• Umidade relativa 92%
• Baixa força iônica
– Giro para a direita
– 10-10,4 pb por volta
A DNA
• obtida por desidratação do B DNA
•
•
•
•
Cavidade > + estreita e profunda
Cavidade < + larga e rasa
11 pb por volta
Inclinação dos planos de pares de
bases
• Híbridos
DNA-RNA
9
Z DNA
• Conformações ANTI e SYN
alternadas
– Alteração promovida por
elevadas concentrações
de cátions no meio
– Comum em seqüências
nas quais G e C se
alternam
• Bases púricas na
forma Syn e
pirimidínicas na
forma Anti
• Giro para a esquerda
• 12 pb por volta
10
Outras estruturas do
DNA
Estrutura Cruciforme
Estrutura
em grampo
11
Moléculas de DNA circulares
• Eucariotos
– Mitocôndrias
– Cloroplastos
• Procariotos
– Cromossomos
• Associado a proteínas e
RNA
– Plasmídeos
– Alguns vírus
12
Replicação
Processo semiconservativo
13
Início da replicação
• Procariotos
– Uma única origem de replicação
– Bidirecional
• Eucariotos
– Bidirecional
– Múltiplos sítios de replicação
• distando de 30.000 a
300.000 pb
• Sítios com muita A e T
– Seqüências de
consenso
» Ordem dos
nucleotídeos é a
mesma em cada
sítio
14
Sentido da replicação: 5’ 3’
Fita líder
Direção do movimento
da zona de replicação
Fragmentos
de Okazaki
Fita atrasada
15
Replicação
16
Etapas da Replicação
1)
Separação das duas fitas:
realizada pelas helicases
2)
Alívio do estresse topológico
catalisado pelas
topoisomerases
3)
Manutenção das duas fitas
separadas envolve as
proteínas de ligação do DNA de
fita simples SSB
DNA
polimerase
SSB
helicase
17
Etapas da Replicação
4) Síntese do RNA iniciador (primer)
– primase
5)
Adição ao primer de
desoxirribonucleotídeos pela
DNA polimerase III
6)
Retirada do primer pela DNA
polimerase I
substituição dos
ribonucleotídeos
pelos
desoxirribonucleotídeos
18
Etapas da Replicação
7)
Ligação do DNA recém sintetizado - DNA ligase
8)
Síntese da fita atrasada – fragmentos de Okazaki
19
Síntese simultânea das fitas
20
Correção do
DNA recém
sintetizado
Atividade de
verificação
Exonuclease
1 erro a cada 106-108
nucleotídeos adicionados
DNA polimerase III
Atividade de correção
3’ 5’ exonuclease
21
TIPOS DE DNA POLIMERASE
Escherichia coli: I, II e III
I – reparo, recombinação e replicação
II – reparo
III - replicação
Eucariotos: , , , e
- síntese da fita retardatária (primase)
- reparo
- síntese da fita líder
- reparo e preenchimento dos espaços entre
os fragmentos de Okazaki
- síntese do DNA mitocondrial
22
CAUSAS DAS MUTAÇÕES
1 – Erro da DNA polimerase – 1 a cada 108 nucleotídeos
2 – Desaminação espontânea de bases
célula: G-C G-U
adenina hipoxantina
3– Quebra da ligação entre a base e a pentose
1/105 purinas/dia (=10.000/dia em 1 célula)
4 – Radiações ionizantes: raios cósmicos, radioatividade
(rádio, plutônio, urânio, radônio, C14, H3), raios-X
(radiografias, radioterapia), radiação
23
5 – Exposição a agentes químicos
a – desaminantes – bissulfito e ácido nitroso
b – alquilantes – dimetilsulfato
c – radicais livres
d- compostos semelhantes às bases do DNA
6 – Intercalação de compostos com anel planaracridinas, brometo de etídio deslocamento da
leitura
24
REPARO DO DNA
25
Reparo de lesão causada por luz UV
Dímero de timina
• Endonuclease UV
específica reconhece o
dímero de timinas
• Clivagem Exonuclease de
excisão reconhece a
incisão feita pela
endonuclease
• Reposição das bases
(polimerase)
26
Correção da alterações ou perdas de bases
1) Remoção das bases
anormais
– glicosilases específicas
Uracil DNA
glicosidase
27
Correção da alterações ou perdas de bases
2) Reconhecimento e
reparo do sítio AP
– Endonucleases AP
– Excisão e
preenchimento da
lacuna
AP endonuclease
DNA polimerase e
DNA ligase
28
Referências Bibliográficas
• Champe, P.C.; Harvey, R.A. Bioquímica Ilustrada, traduzido por
Ane Rose Bolner. 2ª Ed., ARTMED, 2002.
• Campbell, M. K. Bioquímica; traduzido por Henrique B. Ferreira
et al. 3ª ed. Porto Alegre: Artes Médicas, 2001
• Devlin, T.M. Manual de Bioquímica Química Clínica com
Correlações Clínicas; traduzido por Yara M. Michelacci et
al.
4ª ed, São Paulo: Edgard Blücher Ltda, 1997.
• Lehninger, A; Nelson, D.; Cox, M. Princípios de Bioquímica.
Traduzido por Arnaldo A Simões, Wilson R. N. Lopes. 2ª ed.,
São Paulo: SARVIER, 2000
29








