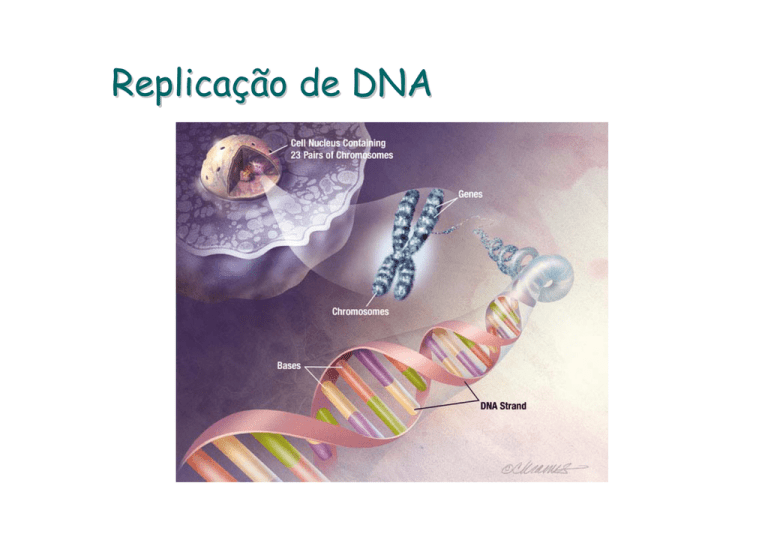
Replicação de DNA
DNA
Dupla-hélice composta
de nucleotídeos ligados
entre si e cujas bases
nitrogenadas de uma
hélice fazem pontes de
hidrogênio com bases
nitrogenadas de outra
hélice, numa direção
anti-paralela.
Replicação do DNA
Processo que precede a divisão celular e através do qual são
geradas cópias idênticas das moléculas de DNA presentes
na célula-mãe, a seguir herdadas pelas duas células-filhas
Duplicação dos
cromossomos
Separação dos
cromossomos
Divisão celular
Cada vez que uma
célula divide a
informação
genética precisa
ser transmitida às
células filhas
formação de
gametas para
transmissão da
informação
para a próxima
geração
Ciclo Celular
Antes de iniciar a
condensação dos
cromossomos o DNA
necessita ser duplicado :
é a fase S
Experimento de Meselson & Stahl, 1958
A pergunta
Experimento de Meselson & Stahl, 1958
Molécula parental
A resposta
Primeira geração de
moléculas “filhas”
Segunda geração de
moléculas “filhas”
Cultivo de bactérias
em meio contendo
15NH Cl,
4
Isótopo pesado de N
Transferência para
meio contendo 14NH4Cl
Extração e purificação do DNA
seguida de centrifugação em
gradiente de CsCl
Replicação do DNA - características
Fita 1 parental
Fita 2 parental
Fita 1
Fita 2 Fita 1 Fita 2
parental “filha” “filha” parental
• Síntese de DNA é
semi-conservativa
Replicação do cromossomo bacteriano
Cultivo de bactérias
em meio contendo
Trítio (3H), isótopo
radioativo de H
Isolamento do DNA,
espalhamento em
emulsão fotográfica
Experimento de J. Cairns, 1963
Observação em
microscópio eletrônico
Replicação do DNA - características
•Começa numa
origem de
replicação (Ori) e é
bidirecional
O genoma bacteriano circular constitui um único replicon
• A velocidade da forquilha de replicação
bacteriana é 50.000 pb/min
• Um única origem de replicação em E.coli
(OriC, 245 pb)
Forquilha de replicação
Região do DNA onde ocorre a transição do DNA parental fita dupla para as
novas fitas filhas duplas
O genoma eucariótico possui vários replicons
• A velocidade da forquilha de replicação eucariótica é 2.000 pb/min
• Os replicons eucarióticos tem 40-100 kb e são iniciados em tempos diferentes
• Fase S demora ~ 6hrs em uma célula somática
Replicon
Unidade do DNA onde está ocorrendo um evento de replicação
•
Origem + Término
•
Ativados apenas uma única vez em cada ciclo celular
•
O genoma de uma célula procariótica possui um único replicon
•
Cada cromossomo eucariótico possui vários replicons e todos são
ativados uma única vez no ciclo celular ainda que não
simultaneamente
Primeiro passo: abrir o DNA (forquilha de replicação) e
manter as fitas separadas
DNA ou
iniciador RNA
DNA polimerase
Fita de DNA
molde
O que dirige a escolha do nucleotídeo a ser adicionado à fita é a
especificidade A-T/C-G e o alongamento ocorre somente no
sentido 5’ – 3’, devido à necessidade de um grupamento 3’OH
livre para formação da ligação fosfodiéster
Síntese da cadeia de DNA envolve a formação
das ligações fosfodiéster
Desoxiribonucleosídeo
5´trifosfato
(substrato)
Fita sendo
polimerizada
Fita molde
iniciador
trifosfato
de guanosina
Fita de DNA molde
A ligação fosfodiéster se dá entre o grupamento 3’OH de um
nucleotídeo já existentes na fita e o grupamento 5’P do
nucleotídeo livre
iniciador
Fita de DNA molde
Liberação de pirofosfato
iniciador
Fita de DNA molde
Na formação da ligação fosfodiéster é liberado um grupo
pirofosfato rico em energia
DNA é sintetizado por
DNA
polimerases
• DNA polimerase I foi isolada a
partir de E.coli em 1955 por A.
Kornberg
• As DNA polimerases necessitam
sempre de um DNA molde e uma
sequência iniciadora.
• O substrato da síntese é o
desoxiribonucleosídeo 5´-trifosfato.
• A síntese de DNA ocorre pela
adição de nucleotídeos a
extremidade 3´OH da cadeia em
crescimento.
•Sentido da síntese sempre é
5’ → 3’.
Replicação do DNA - características
• Síntese de DNA procede
na direção 5´-3´
DNA parental
Fita contínua
Fita descontínua
A replicação se dá nas duas fitas ao mesmo tempo, porém
em sentidos opostos
Fita contínua
Fita descontínua
Iniciador
de RNA
No caso da síntese da fita descontínua, iniciadores de RNA
são produzidos ao longo de todo o processo pela primase
Fita contínua
Fita descontínua
Observe o alongamento das duas fitas em sentidos opostos
Fita contínua
Fita
desc
ontín
ua
Fragmento de
Okazaki
Os fragmentos sintetizados na fita descontínua são
chamados de fragmentos de Okazaki e são ligados
enzimaticamente para gerar uma fita contínua
Síntese de DNA ocorre sempre na direção
5’ → 3’ e é semi-descontínua
Fita com síntese contínua
Fita com síntese descontínua
Fragmentos de Okazaki:
Bactéria: 1.000 a 2.000 pb
Eucariotos: 150 a 200 pb
As sub-unidades Beta da DNA-polimerase envolvem a
dupla hélice formada durante a replicação
Sub-unidades beta da Pol
Dupla - hélice
nascente
PRINCIPAIS COMPONENTES ENVOLVIDOS NA REPLICAÇÃO
proteína ligante
de DNA
DNA polimerase
DNA polimerase
Proteínas presentes na origem de Replicação de E.coli
DnaA
Reconhece a origem oriC e abre a dupla fita em sítios
específicos
DnaB (helicase)
Desenrola o DNA
DnaC
Auxilia a ligação de DnaB na origem
HU
Proteína do tipo histona que estimula a iniciação
Primase (DnaG)
Sintetiza os iniciadores de RNA
Single strand binding
(SSB)
Liga a fita simples de DNA
RNA polimerase
Facilita a ação da DnaA
DNA girase
(topoisomerase)
Alivia a tensão torsional gerada pela abertura da dupla-fita
Dam Metilase
Metila as sequências GATC na OriC
Propriedades das DNA-polimerases Bacterianas
Polimerização 5’ → 3’
Exonuclease 3’ → 5’
Exonuclease 5’ → 3’
Número de subunidades
Velocidade de
Polimerização(nt/seg)
Pol I
+
+
+
1
Pol II
+
+
≥4
Pol III
+
+
≥ 10
16-20
40
250-1.000
1.500
≥ 500.000
Processividade
3-200
(nt adicionados antes
da dissociação da fita-molde)
Reparo (Pol IV e V)
Principal enzima de
replicação em E.coli
Atividade exonuclease 5’ → 3’,
responsável pelo processo de remoção dos iniciadores de RNA
A DNA-polimerase III é multimérica, formada por mais
de 10 cadeias, com uma simetria dimérica
Fita na qual ocorre
a síntese contínua
6 sub-unidades
As sub-unidades τ mantém a
estrutura dimérica
Fita na qual ocorre a
síntese descontínua
6 sub-unidades
Iniciação - 1º estágio da replicação em E.coli
Estrutura da origem de replicação bacteriana oriC
• A origem de replicação OriC é extremamente conservada.
• sequências repetidas com 9 e 13 bases, ricas em A-T são
reconhecidas por > 9 enzimas diferentes.
• enriquecida na sequência palindrômica GATC, alvo de metilação
enzimática na adenina .
Origem de Replicação em bactérias
Somente origens completamente metiladas podem iniciar a replicação
Elongação - 2º estágio da replicação
Replicação do DNA - Resumo
•• Replicação
-conservativa
Replicação do
do DNA
DNA éé semi
semi-conservativa
•• Replicação
-direcional.
Replicação éé bi
bi-direcional.
•• Cromossomos
Cromossomos bacterianos
bacterianos possuem
possuem apenas
apenas 11 replicon
replicon,,
cromossomos
cromossomos eucariotos
eucariotos possuem
possuem vários
vários amplicons
amplicons..
•• O
O DNA
DNA éé sintetizado
sintetizado por
por DNA
DNA polimerase
polimerase,, uma
uma enzima
enzima multimérica
multimérica,,
•• A
A síntese
síntese ocorre
ocorre sempre
sempre no
no sentido
sentido 5´
5´ -- 3’,
3’, com
com atividade
atividade revisora
revisora
3´
3´-- 5´.
5´.
•• A
osta
A síntese
síntese éé contínua
contínua em
em uma
uma das
das fitas
fitas ee descontínua
descontínua na
na fita
fita op
oposta
•• Um
Um complexo
complexo de
de mais
mais 99 enzimas
enzimas está
está envolvido
envolvido na
na iniciação,
iniciação,
elongação
elongação ee terminação
terminação da
da replicação.
replicação.
Geometria do pareamento das
bases contribui para a fidelidade
da replicação do DNA
Pareamento incorreto
A
Pareamento correto
T
G
A
C
A
C
G
T
G
Atividade revisora (atividade exonuclease 3’ → 5’)
garante a fidelidade da replicação
Reparo do DNA
- “Mismatch repair”
- Reparo por Excisão de Bases
- Reparo por Excisão de Nucleotídeos
- Reparo Direto
- Reparo por Recombinação
Reparo do DNA
“Mismatch repair” :
Corrige erros de replicação
Reparo do DNA
Reparo por Excisão
de Bases:
Remove a Uracila
(citosina alterada)
Citosina
Uracila
Reparo do DNA
Reparo por
Excisão de
Nucleotídeos :
Remove o
nucleotídeo
Reparo do DNA
Reparo Direto :
Não há remoção de nucleotídeos
Reparo do DNA
Reparo por
Recombinação :
Usa-se uma outra
molécula de DNA
Célula
Normal
Câncer
estima-se que 6-7 eventos devem ocorrer (em ~2040 anos) para indução do câncer
em certos casos a propensão ao câncer é hereditária
Recombinação
9constitui
na troca ou
incorporação de uma
seqüência de DNA com
(à) outra
9na
meiose ocorre a
recombinação homóloga
(troca de trechos)
9a
recombinação
somática é responsável
pelo rearranjo de BCR e
TCR
9DNA
de bactérias e
virus também sofrem
recombinação
recombinação homóloga
recombinase RecA e Rec BCD
Recombinação Somática
cadeia leve λ
cadeia leve κ
cadeia pesada
recombinase RAG1 e RAG2
RSS: a regra de recombinação
há a formação
de alças durante
o rearranjo
e parte do
DNA
genômico é
perdido
Repertório Imunológico
DNA germinativo
Eventos Imunoglobulinas
κ+λ
H
DNA rearranjado
mRNA maduro
proteína
V
51
D
30
J
5
inserções 2
TOTAL
69
0
5
(1)
1016
A hipermutação somática ocorre após a
ativação do linfócito B no centro
germinativo e leva a maturação por
afinidade.
taxa de mutação neste processo : 1/1000
bp
(normal : 1/ 106 ou menos)
a cada divisão 1/2 linfócitos modificam sua
seqüência
mutação ocorre nas regiões V rearranjadas
conseqüência da grande proliferação e hipermutação
Ciclo de vida de um bacteriófago lambda
na fase após infecção de uma bactéria pode ocorrer a
recombinação (integração) do DNA do fago no DNA da bactéria
na fase após infecção de uma bactéria ocorre a recombinação
(integração) sítio-específica (att) do DNA do fago no DNA da
bactéria
Há replicação do DNA do fago, formação de novas
partículas e o ciclo de fecha
os fagos permanecem dentro das bactérias e assim
podem ser usados para formar bibliotecas de DNA








