Exercícios (aula de duplicação)
1. De que material básico dispunha Watson e Crik para desenvolver um modelo de DNA?
R: Eles dispunham das Regras de Chargaff da composição de bases e dos de Chargaff da composição
de bases
2. Quais os tipos de ligações químicas na molécula de DNA?
R: Pontes de hidrogênio entre bases nitrogenadas e ligações fosfodiéster entre o grupo fosfato do
carbono 5’ de um nucleotídeo e o cabono 3’ do nucleotídeo seguinte.
3. Uma alga vermelha estoca sua informação genética na dupla hélice de DNA. quando o DNA foi
extraído de células de alga e analisado, observou-se que 32% das bases eram guaninas. Por esta
informação, é possível determinar que porcentagem das bases neste DNA eram timinas? Caso sim,
qual a porcentagem?
R: Sim, é possível determinar a quantidade de timinas. A quantidade de timinas é de 18%.
4. Se um filamento de DNA na dupla hélice tem uma sequencia de bases de 5’GTCATGACTTGCAAATGC-3’, qual a sequencia de bases do filamento complementar?
R: A sequencia complementar é 3’-CAGTACTGAACGTTTACG-5’.
5. Quais as diferenças entre DNA e RNA?
R: A molécula de DNA difere da molécula de RNA nos seguintes aspectos: bases nitrogenadas – o
DNA possui Timina, Adenina, Guanina e Citosina, enquanto que o RNA possui, no lugar da Timina, uma
Uracila; o açúcar pentose que compõem o nucleotídeo – no DNA o açúcar é uma desoxirribose e no RNA o
açúcar é uma ribose; e diferem também com relação ao número de filamentos – o DNA possui dois
filamentos unidos por pontes de hidrogênio, enquanto que o RNA possui um único filamento.
6. DNA foi extraído de células de Staphylococcus afermentans e analisado quanto a composição de
bases. Foi observado que 14% das bases são citosinas. Com base nesta informação é possível prever
qual a porcentagem de adeninas? Caso sim, qual a porcentagem?
R: Sim é possível. A quantidade de adeninas presente no DNA de Staphylococcus afermentans é de
36%.
7. O DNA extraído das células de cebola apresentou 40% de bases púricas. Qual a porcentagem de
bases pirimídicas? Quais são as bases púricas e as pirimidínicas? (desconsiderar a questão)
R: as bases púricas são adenina e guanina e as pirimídicas são citosina e timina. A quantidade de
bases pirimídicas seria de 40% também.
8. Assinale verdadeiro ou falso:
(F)A+T=G+C
( F ) A = G; C = T
( V ) A/T = C/G
(V)A+G=C+T
( V ) G/C = 1
( F ) A = T dentro de cada filamento
( V ) uma vez conhecida a sequencia de bases de um filamento da dupla hélice de DNA, a sequencia
de bases do segundo filamento pode ser deduzida.
Exercícios (aula de replicação)
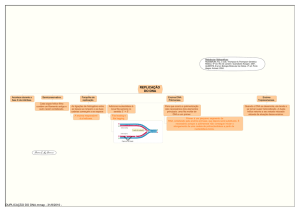
1. Defina replicação conservativa, semiconservativa e dispersiva. Qual delas é aceita como forma de
replicação da molécula de DNA?
R: NA replicação semiconservativa a dupla hélice de cada molécula filha de DNA contém um
filamento da molécula original de DNA e um filamento recém-sintetizado; na replicação conservativa
a molécula parental de DNA é conservada, e uma única dupla hélice filha é produzida, consistindo em
dois filamentos recém-sintetizados; e na replicação dispersiva: as moléculas filhas consistem em
filamentos cada um contendo segmentos, tanto do DNA parental quanto do DNA recém-sintetizado. O
DNA é replicado de forma semiconservativa.
2. O que é um primer, e porque são necessários para a replicação de DNA?
R: O primer é um fragmento de RNA (formado de nucleotídeos de RNA) e é necessário para iniciar
a duplicação da molécula do DNA porque a DNA polimerase necessita de uma extremidade 3’OH livre para
adicionar nucleotídeos de DNA. Essa extremidade não é fornecida apenas pelo rompimento da dupla hélice,
por isso é necessário a formação do primer.
3. Quais os requisitos necessários para a replicação do DNA?
R: Para que ocorra a duplicação do DNA são necessários um molde de DNA unifilamentar (fornece a
sequencia de nucleotídeos que especifica a sequencia complementar da cadeia nascente de DNA);
Enzimas e outras proteínas que leiam o molde e montem a nova molécula de DNA; Trifosfatos de
desoxiribonucleotídeos (dNTPs); e primers (que fornece um término com uma ponta 3’OH livre onde
são adicionados nucleotídeos pela DNA polimerase durante a síntese de DNA).
4. Quais as funções de:
a) Primer: fornecer uma extremidade 3’OH livre para que a DNA polimerase adicione nucleotídeos de
DNA durante a duplicação do DNA.
b) Primase: sintetizar o primer.
c) Proteína SSB: manter a dupla hélice na forma unifilamentar (dupla hélice separada)
d) Topoisomerase: diminuir a tração formada após as forquilhas de replicação.
e) Helicase: quebram as pontes de hidrogênio entre as bases nitrogenadas abrindo os dois filamentos da
dupla hélice.
f) DNA ligase: promove a ligação entre os fragmentos de DNA do fragmento sintetizado
descontinuamente.
g) DNA polimerase III: adicionar nucleotídeos de DNA na extremidade 3’OH livre.
h) DNA polimerase I: possui atividade exonuclease 5’→3’, retirando os nucleotídeos do primer e
atividade polimerase 5’→3’, adicionando nucleotídeos de DNA ao filamento.
5. Porque a síntese do DNA é contínua em um filamento e descontínua no outro?
R: Porque a DNA polimerase só adiciona nucleotídeos na extremidade 3’OH livre, sendo assim um
filamento proporciona essa extremidade e o outro não.
6. Desenhe os filamentos novos a partir do filamento abaixo:
3’AGCTTTAGGGCAAATCGATGCA5’
5’TCGAAATCCCGTTTAGCTACGT3’
3’AGCTTTAGGGCAAATCGATGCA5’
5’TCGAAATCCCGTTTAGCTACGT3’
7. Quais as atividades da polimerase I, II e III na E. coli.
R: DNA polimerase I que tem atividade de: Exonuclease 5’ → 3’; Exonuclease 3’ → 5’; Polimerase 5’
→ 3’
DNA polimerase II que tem atividade de: Polimerase 5’ → 3’; Exonuclease 3’ → 5’;
DNA polimerase III que tem atividade de: Polimerase 5’ → 3’; Exonuclease 3’ → 5’; Exonuclease 5’
→ 3’
Exercícios (aula de transcrição)
1. Que bases no mRNA transcrito representaria a seguinte sequencia molde de DNA: 3’TGCAGACATTAACGAGT-5’.
R: 3’-ACGUCAGUAAUUGCUCA-5’
2. Que bases no filamento transcrito de DNA dariam origem à seguinte sequencia de bases do mRNA:
5’-CUGAUCGCAUUAUCG-3’.
R: 3’-GACTAGCGTAATAGC-5’
3. Onde ocorre a transcrição em eucariontes e em procariontes?
R: A transcrição em eucariontes ocorre no núcleo e em procariontes ocorre no citoplasma.
4. Quais as RNA polimerases presentes em eucariontes e que grupos de RNA cada uma delas
transcreve?
R: RNA polimerase I – transcreve genes de rRNA (exceto 5S rRNA); RNA polimerase II –
transcreve genes de mRNA; RNA polimerase III – transcreve genes de tRNA, snRNA e 5S rRNA.
5. Descreva como ocorre o processo de iniciação da transcrição em procariontes e em eucariontes.
R: Em procariontes a RNA polimerase se liga ao DNA nos promotores -35 e -10 e inicia a
duplicação. Após iniciada a subunidade σ se desliga e a transcrição continua até uma sinalização de término.
Em eucariontes a iniciação necessita de fatores de transcrição que se ligam primeiro no DNA em regiões
conhecidas como CAAT e TATA Box (promotores) e depois a RNA polimerase se liga para iniciar a
transcrição.
6. Como ocorre o término da transcrição em eucariontes? Qual a importância do cap-7MG e da cauda
poli(A) adicionados ao RNA transcrito?
R: Ocorre uma clivagem que produz uma ponta 3’ de um transcrito geralmente ocorre no sítio de 11
a 30 nucleotídeos posteriores a sequência conservada de consenso AAUAA e antecede uma região rica em
GU. Após a clivagem a enzima poli(A)-polimerase adiciona uma cauda poli(A) com cerca de 200 adeninas
(poliadenilação). Essa cauda aumenta a estabilidade e tem um papel importante em seu transporte do núcleo
para o citoplasma. Além disso, é adicionada uma 7-MG na extremidade 5’ do transcrito que forma uma
ligação incomum 5’-5’-trifosfato e dois ou mais grupos metila. Esse cap 7-MG protege as cadeias de RNA
da degradação de nucleases.
7. Como e porque ocorre a retirada dos íntrons do RNA transcrito? Qual a importância dos éxons?
R: O processo de retirada dos íntrons ocorre em duas etapas e necessitam de 5 snRNA conhecidos
como U1, U2, U4, U5 e U6, que juntos formam o spliceossomo (um complexo de snRNA e
proteínas). NA primeira etapa ocorre a clivagem no sítio de clivagem do íntron 5’ (GU-íntron) e a
formação de uma ligação fosfodiéster intramolecular entre o C5’ da G do sítio de clivagem com o
C2’ de uma A conservada presente no íntron. Evidências indicam que a U1 deve se ligar ao sítio de
corte 5’antes da reação inicial de clivagem. Depois ocorre a adição da U2 ao complexo, onde contém
uma A conservada formando um ponto de ramificação no íntron. U5 e U4/U6 se ligam na
extremidade 3’ do íntron. Quando a extremidade 5’ do íntron é cortada, a U4 é liberada do
spliceossomo. O sítio 3’ do íntron é cortado e os dois éxons são unidos por uma ligação fosfodiéster.
Os éxons são importantes porque carregam a informação codificante do DNA.
Exercícios (ultima aula)
1. Uma alga vermelha estoca sua informação genética na dupla hélice de DNA. Quando o DNA foi
extraído de células de alga e analisado, observou-se que 23% das bases eram guaninas. Por esta
informação, é possível determinar que porcentagem das bases neste DNA eram timinas? Caso
sim, qual a porcentagem?
R: 27% de timinas
2. Qual a função do RNA ribossômico (rRNA), RNA mensageiro (mRNA) e o RNA transportador
(tRNA) durante a tradução?
R: rRNA: ler a mensagem do mRNA; mRNA carregar a mensagem do DNA; tRNA levar os
aminoácidos correspondentes aos códons do mRNA para formar a proteína.
3. Onde ocorre a duplicação, transcrição e duplicação em procariontes e em eucariontes?
R: Eucariontes: duplicação e transcrição nu núcleo e tradução no citoplasma; procariontes: todas
as fases ocorrem no citoplasma.
4. Porque o código genético é considerado degenerado?
R: Porque um aminoácido pode ser transcrito por mais de um códon.