Genética Molecular
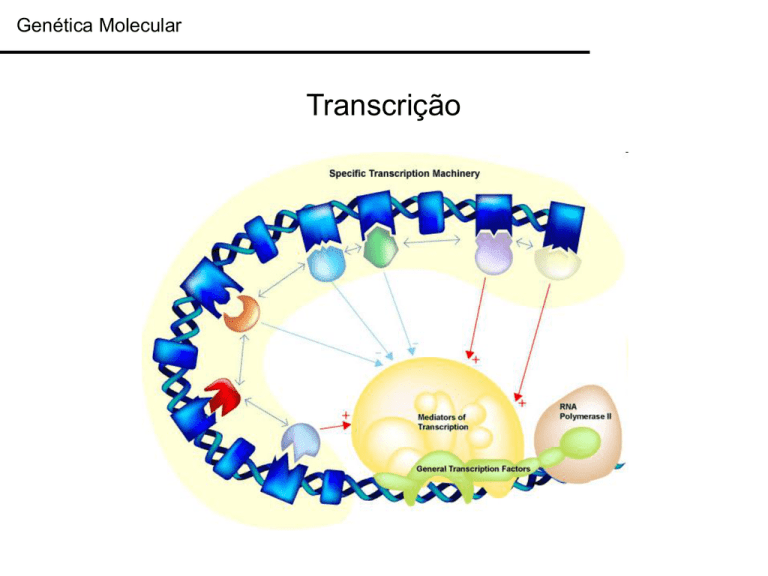
Transcrição
Genética Molecular
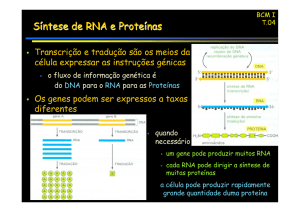
Dogma central da biologia molecular: a informação genética segue
um fluxo unidirecional.
transcrição reversa
replicação
DNA
RNA
transcrição
polipeptídeo
tradução
Transcrição
Transcrição: ato ou efeito de reproduzir por meio de cópia.
Processo a partir do qual a informação genética de certos segmentos
de DNA (os genes) especifica a síntese de RNA.
Somente pequena parcela do DNA total das células é transcrito.
E deste, apenas uma pequena porção é traduzido (eucariotos):
• Algumas unidades de transcrição expressam-se produzindo outras
moléculas de RNA que não especificam polipeptídeos.
• O transcrito primário das unidades de transcrição que codificam
polipeptídeos está sujeito a eventos de processamento de RNA
• Apenas uma parte do mRNA maduro é traduzida
Transcrição
Genoma = DNA codificante + DNA não-codificante (“DNA lixo”)
Transcrição
Genes: tradicionalmente é a unidade fundamental da hereditariedade.
Na genética moderna, é uma seqüência de nucleotídeos do DNA que
pode ser transcrita em uma versão de RNA.
Também pode ser visto como um segmento de um cromossomo a que
corresponde um código distinto, uma informação para produzir uma
determinada proteína ou controlar uma característica.
Estrutura de um gene – inclui regiões que antecedem e que seguem a
região codificadora, bem como sequências que não são traduzidas
(íntrons) que se intercalam aos segmentos codificadores individuais
(éxons), que são traduzidos.
Transcrição
Estrutura do gene
Procariotos
Eucariotos
Transcrição
Principio: complementaridade de bases.
Filamento de DNA transcrito – fita molde.
Filamento de DNA complementar – fita não-molde
mRNA possui igual seqüência que a fita não-molde (trocando T por U).
Transcrição
Transcrição em Procariotos
Ocorre diretamente no citoplasma (procariotos não possuem núcleo).
Um tipo de RNA polimerase.
Início
Alongamento
Término
RNA polimerase
Síntese 5’ -> 3’
Transcrição em Procariotos
Início
• RNA polimerase (fator sigma) reconhece e se liga especificamente
à região promotora (região -35 e -10). DNA ainda em dupla hélice –
complexo fechado.
• DNA é aberto em fita simples na região próxima da iniciação da
transcrição (região +1) – complexo aberto – formação da bolha de
transcrição.
• A RNA polimerase transcreve o DNA, mas produz cerca de 10
transcritos abortivos, que não conseguem sair da enzima uma vez
que o canal de saída está bloqueado pelo fator sigma.
• Eventualmente o fator sigma se dissocia da enzima, e a transcrição
se procede.
NTP’s são substrato da enzima, e fornecem a energia a partir da quebra do
trifosfato em monofosfato
Transcrição em Procariotos
Transcrição em Procariotos
Término
a. Direto / Intrínseco / Rô-independente: envolve seqüência de terminação
(sequência palindrômica com ca. 40pb com região rica em GC seguido por
repetições de A). Formação de alça em grampo – dissociação da RNA pol
da fita molde.
b. Rô-dependente: usa um fator de término (rô) para parar a síntese num ponto
específico. mRNA’s com sinais de término dependentes de rô não possuem
seqüência palindrômica.
–
Rô (hexâmero) – ligação à região rut – separação do RNA da RNA pol
– Translocação de rô ao longo do mRNA.
Sequência palindrômica: Sequência de bases no sítio de reconhecimento,
lida de 5´para 3´, é a mesma em ambos os filamentos (e.g., 5´GAATTC-3´; 5´-CTTAAG-3´; 5´-AAGCTT-3´; 5´TTCGAA-3´).
Trancrição
Transcrição em Eucariotos
Envolve mais fatores de transcrição que procariotos.
Transcrição ocorre no núcleo.
Três tipos de RNA polimerase:
I - trancreve o rRNA.
II - trancreve o mRNA.
III - transcreve o tRNA, snRNA e outros RNA’s pequenos.
Regulação: Dependende de fatores cis- e trans-atuantes.
Processamento do mRNA - Regulação pós-transcripcional.
Transcrição em Eucariotos
Padrão de sequêncial de ligação dos
Fatores de transcrição.
1) TFIID se liga ao TATA box;
2) Ligação de TFIIA e TFIIB.
3) Complexo resultante se liga à RNA polimerase II, a
qual já se ligou ao TFIIF.
4) Complexo de iniciação é completado com a adição
de TFIIE, TFIIJ, e TFIIH.
5) Depois de um passo de ativação (fosforilação
dependente de ATP) da RNA polimerase II, esta
pode iniciar a transcrição no ponto de início (+1).
Genética Molecular
Tipos de RNA: estrutura e função
Tipos de RNA: estrutura e função
Tipos de RNA: estrutura e função
Diferentes elementos
estruturais na estrutura
secundária do RNA.
A - hairpin loop
B - stacking
C - bulge loop
D - interior loop
E - multi loop
F - exterior loop
G - dangling nucleotide
H - par que não G-C
I - Coaxial stacking
Tipos de RNA: estrutura e função
RNA’s informacionais
• mRNA – carrega informação genética pros ribossomos (expressão
gênica).
– Procariotos: transcrito prim’ario = mRNA
– Eucariotos: transcrito prim’ario = pré-mRNA
RNA’s funcionais
•
•
•
•
rRNA – constitui, junto com proteínas, os ribossomos.
tRNA – transporta aminoácidos para os ribossomos.
snRNA – junto com proteínas constituem as snRNP (recombinação)
Outros – snoRNA, siRNA, gRNA, scRNA, tmRNA
Tipos de RNA: estrutura e função
Conteúdo de RNA de uma célula
RNA total
~4%
96%
RNA codificante
RNA não-codificante (ncRNA)
Pré-RNA codificante
ou hnRNA
mRNA
snRNA
Pré-rRNA
scRNA
srpRNA
tRNA
snoRNA
rRNA
Pré-tRNA
tmRNA e
outros tipos
gRNA
RNAi
Tipos de RNA: estrutura e função
mRNA (messenger RNA)
Molécula mensageira que converte
a informação genética do DNA
para o ribossomo.
Transcriptoma: todo o conteúdo de
mRNA de um organismo.
mRNA em eucariotos – sintetizado
como uma forma precursora,
chamada pré-mRNA ou hnRNA
(heterogeneous nuclear RNA).
Processamento do pré-mRNA.
Tipos de RNA: estrutura e função
rRNA (ribosomal RNA)
Juntamente com as proteínas (+50), formam os ribossomos.
Ribossomos: grandes complexos para a síntese protéica.
tamanho: grande, estimado pelo coeficiente de sedimentação
(unidade S, Svedberg)
Formado por 2 subunidades: maior e menor
Procariotos – 70S
Eucaritos – 80S
Subunidade maior – 50S
Subunidade menor – 30S
Subunidade maior – 60S
Subunidade menor – 40S
23S e 5S
16S
28S, 5.8S e
5S
18S
rRNA
Estrutura 3D das subunidades do ribossomo procarioto
30S
50S
rRNA
genes rRNA nucleares
Repetição in tandem
Variação intragenômica
Evolução em concerto
rRNA
tRNA (transfer RNA)
20 ou mais tipos de pequenas moléculas de RNA atuando
na tradução.
Cada tipo carrega um aminoácido específico para um sítio
especificado por um códon de RNA.
tRNA
Grupo 5’-fosfo terminal.
Braço aceptor – tronco com 7
pb, que contém grupo
CCA 3’- terminal –
aminoacetilado
Alça D – tronco com 4 pb
terminando numa alça que
contém diidrouridina
(DHU)
Alça do anticódon – tronco
com 5 pb que contém o
anticódon.
Alça TΨC– tronco com 5 pb
contendo a sequência
TΨC (onde Ψ é uma
pseudouridina)
tRNA
Aminoacilação – processo de adição de um grupo aminoacil a um
composto. Na molécula de tRNA, resulta na ligação covalente de
um aminoácido no terminal CCA-3`.
Cada tRNA é aminoacilado (ou carregado) com um aminoácido
específico por uma aminoacil tRNA sintetase.
Há uma aminoacil tRNA sintetase para cada aminoácido, a despeito de
poder haver mais de um tRNA e mais de um anticódon para cada
aminoácido.
Reconhecimento do tRNA apropriado pela sintetase não é mediado só
pelo anticódon, mas também pela alça aceptora.
Reação:
amino ácido + ATP → aminoacil-AMP + PPi
aminoacil-AMP + tRNA → aminoacil-tRNA + AMP
tRNA
Tipos de RNA: estrutura e função
snRNA (small nuclear RNA)
Classe de moléculas encontrada no núcleo.
Associadas à proteínas específicas, formas as
snRNP (ribonucleoproteínas nucleares pequenas).
Atuam:
• Nas reações de recombinação (splicing)
(remoção de introns do hnRNA);
• Na regulação de fatores de transcrição ou da
RNA polimerase;
• E, na manutenção dos telômeros.
Um grande grupo de snRNA é conhecido como snoRNAs (small
nucleolar RNAs). Importantes na biogênese de rRNAs e outros genes
de RNA (tRNA e snRNA). Localizados nos nucléolos (lugar de maior
síntese de RNA)
Tipos de RNA: estrutura e função
RNAi (RNA interference)
Sistema das células que ajuda a controlar a expressão de genes.
Mecanismo de defesa da célula contra invasões (sobretudo virais).
Aplicações biotecnológicas.
Dois tipos de RNAs participam deste processo: miRNA (microRNA) e
siRNA (small interfering RNA).
dsRNA exógenos são inicialmente clivado pela enzima Dicer.
Fragmentos pequenos de 20-25 pb de dsRNA (siRNA) são separados
em fitas simples e integrados à RISC (RNA-induced silencing
complex).
siRNA pareia com mRNA complementar, induzindo clivagem deste
mRNA.
dsRNA exógeno é detectado e ligado por uma proteína efetora, que
atua como a Dicer.
Este caminho inicial pode ser amplificado pela célula através da
síntese de siRNAs secindários, usando siRNAs primários como
molde. Estes são distintos dos siRNAs produzidos pela Dicer, e são
produzidos por uma RNA polimerase dependente de RNA (RdRP).
RNAi
RNAi
miRNAs são produzidos a partir de Dna
não-codificante.
Ajudam no controle da expressão gênica,
principalmente durante o desenvolvimento.
miRNAs são similares aos siRNAs, após sua maturação.
Transcrito primário de miRNA (pri-riRNA) é processado em um premiRNA (70 pb, haste-alça) pelo complexo microprocessador (MPC).
MCP consiste de uma RNase III e uma proteína ligadora à dsRNA.
A porção dsRNA do pre-miRNA é ligada e clivada pela Dicer, para
produzir o miRNA maduro, para ser incorporada ao complexo RISC.
A partir daí miRNA e siRNA compartilham a mesma maquinaria celular.
RNAi