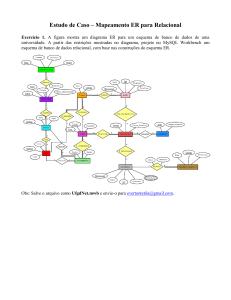
Bancos de Dados Biológicos
Luis Artur Ribeiro
Rafael Justino
Poline Lottin
Debora Comochina
Conceitos
Bioinformática: especialização da Informática aplicada à informação
originada pela/para Biologia; ou estudo da aplicação de técnicas computacionais
e matemáticas à geração e gerenciamento de (bio)informação.
A Bioinformática combina conhecimentos de química, física, biologia, ciências da
computação, informática e matemática/estatística para processar dados
biológicos ou biomédicos.
Exemplos em Softwares: identificar genes, prever a configuração
tridimensional de proteínas, identificar inibidores de enzimas, organizar e
relacionar informação biológica, simular células, agrupar proteínas homólogas,
montar árvores filogenéticas, analisar experimentos de expressão gênica entre
outras inúmeras aplicações.
Conceitos
“BD biológicos se tornaram uma importante ferramenta no
entendimento da vasta quantidade de fenômenos
biológicos existentes, desde a estrutura das biomoléculas
e sua interação ao metabolismo como um todo e a
evolução das espécies.
Este entendimento contribui para facilitar a luta contra
doenças, auxilia no desenvolvimento de novos fármacos e
na descoberta de relações entre espécies.”
(Wikipedia, 2006)
Conceitos
•São tanto bancos públicos (ex: Genbank) como privados
•Mais de 1000 bancos de dados biológicos comerciais e
públicos disponíveis atualmente
•Acesso a esses bancos de dados através de padrões abertos
(open standards) como a web é importante dadas as
características dos usuários destes bancos – Servidores UNIX
x Macintosh
•A revista NucleicAcidsResearch é um importante recurso com
informações sobre estes BD
(http://www3.oup.co.uk/nar/database/c/)
Motivação
• Disponibilizar dados biológicos para os cientistas.
• O máximo possível de um tipo particular de informação
deve estar disponível em um único lugar.
• Dados publicados podem ser difíceis de encontrar ou
acessar.
• Coletá-los da literatura consume muito tempo.
• Disponibilizar dados em formato que possa ser lido por
um computador.
Conteúdo
Dados de genômica, transcriptômica, proteômica, taxonomia,
ecologia, doenças, fármacos...
Informações:
• Seqüências de nucleotídeos, aminoácidos
• Função, estrutura, localização no cromossomo
• Mapas metabólicos
• Efeitos clínicos de mutações
• Características genéticas de populações específicas
• Catálogo de espécies ou recursos naturais
Conteúdo
Bancos de Dados bibliográficos.
• Resumem a literatura cientifica de uma forma legível para a máquina.
Bancos de Dados Taxonômicos.
• Bancos de dados de
classificação de espécies. São extremamente dependentes da classificaç
ão feita por um especialista.
Bancos de Dados de Seqüências de Nucleotídeos.
• Enfocam entidades biológicas como genes e ácidos nucléicos. Visam o
armazenamento e divulgação dedados de seqüências de nucleotídeos de
uma comunidade de pesquisa. As seqüências de DNA e RNA são
normalmente apresentadas juntamente com outras informações como o
organismo a qual a seqüência pertence ou ainda com as funções fisiológicas
relacionadas à seqüência.
Conteúdo
Bancos de Dados Genômicos.
• Disponibilizam dados genéticos de um organismo especial, variando muito no
conteúdo. As informações armazenadas em bancos de
dados genômicos incluem informações sobre genótipos, nome de
genes, propriedades de genes, mutações específicas, assim como mapas
genômicos e informações referentes a raças.
Bancos de Dados Proteômicos.
• Em geral, podem ser vistos como uma mistura de banco de dados de
seqüências de nucleotídeos, seqüências de proteínas e outros.
Bancos de Dados de Vias Metabólicas.
• Armazenam informações sobre o metabolismo de um organismo ou de vários
organismos diferentes. As enzimas participantes de reações são
freqüentemente relacionadas com bancos de dados de seqüências.
Conteúdo
Bancos de Dados de Seqüências de Proteínas.
• Proporcionam informações sobre proteínas. Bancos de dados universais que
armazenam informações sobre proteínas de todos organismos devem ser
diferenciados de bancos de dados especializados que armazenam
informações sobre famílias especificas ou grupo de proteínas ou sobre
as proteínas em espécies específicas.
Bancos de Dados de Estrutura Protéica.
• Estes bancos mantêm dados relativos à estrutura de proteínas. A estrutura
3D completa de proteínas é representada pelo armazenamento de
coordenadas no espaço 3D.
Bancos de Dados Híbridos.
• Trata-se de bancos de dados que armazenam diferentes conteúdos,
pertencendo a mais de um dos grupos citados.Com o crescente número de
dados biológicos que vem sendo
gerados,vários bancos de dados têm surgido.
Exemplos
Alguns tipos de bancos de dados biológicos:
•Bancos de dados primários de seqüência (nucleotídeos e
aminoácidos) –GenBank, UniProt
•Bancos de genomas – Mouse GenomeDatabase, NCBI
Genomic Biology
•Bancos de dados especializados - Flybase, Wormbase, CGAP
•Bancos de dados de estrutura de proteínas – PDB, SCOP
•Bancos de dados de interações proteína-proteína – STRING,
BioGRID
•Bancos de Cadastro de recursos naturais – AmazonLink,
ENDS, National Whale and Dolphins Stranding Database
Relembrando
Nucleotídeos ou nucleótidos são compostos ricos em energia e
que auxiliam os processos metabólicos, principalmente as
biossínteses, na maioria das células.
Funcionam ainda como sinais químicos, respondendo assim a
hormônios e outros estímulos extracelulares; eles são também
componentes estruturais de cofactores enzimáticos,
intermediários metabólicos e ácidos nucleicos. Os nucleóticos
podem ser considerados os monómeros da DNA/RNA, sendo o
polimero, o próprio DNA/RNA.
Relembrando
Genótipos, o conjunto dos cromossomos que se situam no
núcleo das células. Os cromossomos são interpretados como
uma sequência de genes. São os genes os portadores das
informações que condicionam o fenótipo. Ao conjunto dos
genes de um indivíduo damos o nome de genótipo.
• É o conjunto dos genes, condiciona os fenótipos totais.
• São as informações hereditárias de um organismo contidas
em seu genoma.
• gene é uma seqüência de nucleotídeos do DNA que pode
ser transcrita em uma versão de RNA.
Relembrando
Fenótipo são as características observáveis ou caracteres de um
organismo como, por exemplo: morfologia, desenvolvimento,
propriedades bioquímicas ou fisiológicas e comportamento.
Resulta da expressão dos genes do organismo, da influência de
fatores ambientais e da possível interação entre os dois.
Nem todos os organismos com um mesmo fenótipo parecem ou agem
da mesma forma, porque a aparência e o comportamento, assim como
os demais componentes do fenótipo, são modificados por condições
ambientais e de desenvolvimento. Do mesmo modo, nem todos os
organismos cujas aparências se assemelham possuem
necessariamente o mesmo genótipo.
O DNA é uma hélice dupla onde os pares seguem algumas regras.
Características BDB
• Tratam de dados biológicos altamente heterogêneos
mas relacionados
• Informações dinâmicas, modificações e atualizações
constantes
• Grande volume e variedade de dados biológicos
Geração de Dados
Fontes de dados:
• sequenciamento e anotação de genomas
• experimentos em larga escala: identificação de proteínas,
estrutura 3D de proteínas, microarranjos de DNA
Anotação:
• anexar conhecimento biológico relevante aos dados de
sequenciamento do genoma de um organismo.
Geração de Dados
• Visão direcionada para a biologia molecular
• Base da biologia molecular: DNA
o Nucleotídeos {A, C, G, T}
• Além do DNA, existem outros tipos de dados na
linha de evolução (RNA e proteínas)
• Uma seqüência de DNA pode possuir milhares de
pares de nucleotídeos.
• Cada seqüência possui uma identificação, funções
biológicas e podem pertencer a vários organismos.
Requisitos
•Qualidade dos dados (obtenção).
•Anotações consistentes.
•Integração entre ferramentas.
•Fácil acesso às informações disponíveis.
•Mecanismos para extrair do conjunto de dados apenas
aqueles de interesse do pesquisador.
Fatos
Mais de 1000 bases de dados diferentes
• 968 databases em The Molecular Biology Database
Collection: 2007 update by Galperin, Nucleic Acids
Research, 2007, Vol. 35, Database issue D3-D4
Tamanho dos bancos: de 100kB até 100GB
• DNA: >100GB
• Proteina: 1GB
• 3D structure: 5GB
Fatos
Organismos, Número de genes:
Plantas, <50.000
Mamíferos, 100.000
Vermes, 14.000
Bactérias, 2-4.000
dsDNAVírus
Vacina, <300
ssRNAVírus
INfluenza 12
Modelagem
Atualmente, os modelos de dados “tradicionais” mais referenciados
para
desenvolvimento de bancos de dados de biologia molecular são:
o modelo relacional, o modelo orientado a objetos e o modelo semiestruturado (bancos de dados XML).
Cada vez mais bancos de dados com diferentes conteúdos
do domínio abordado estão utilizando um SGBD relacional.
Por exemplo:
• Genome Sequence Database (GSDB), um banco de dados de seqüências de
nucleotídeos, é implementado usando o SGBD Relacional Sybase.
• CyanoBase, um banco de dados genético, também está implementado
usando o Sybase
• O banco de dados proteômico YPD é gerenciado por um SGBD relacional
Oracle.
Modelagem - Modelo Relacional
Dados biológicos são complexos. Um típico tipo de dado tem uma estrutura
aninhada de difícil representação no modelo relacional. Sistemas gerenciadores
de bancos de dados relacionais freqüentemente proporcionam um projeto
fragmentado e não intuitivo.
São melhores quando o esquema é simples, o dado é regular e consultas
sucessivas são independentes. Apesar disso, SGBDs relacionais são utilizados
para gerenciar todo tipo de dado biológico.
O esquema do modelo relacional é pré-definido. Os dados são abstraídos em
entidades e relacionamentos, sendo armazenados em tabelas. Além disso, o
modelo relacional é muito restritivo quanto à habilidade de ser estendido
O fato de decisões serem tomadas nos estágios iniciais, como a definição das
entidades e dos atributos, caracteriza uma desvantagem do modelo
relacional. Em dados biológicos, não há como predizer qual fator se provará
importante ou sujeito à modificação, isto se tornar um problema, pois se mostra
difícil e trabalhoso, senão impossível, alterar o esquema.
Modelagem - Modelo Relacional
A força do modelo relacional é sua total provisão por SQL. Mas sua
principal fraqueza é a ineficiência para modelagem de objetos
genômicos complexos.
O modelo relacional é orientado em direção a um eficiente
armazenamento e gerenciamento de dados, mas não provê
construtores para uma boa captura da semântica dos dados: a
representação de um objeto conceitual complexo em um banco de
dados relacional
pode se estender por muitos registros em várias tabelas distintas,
fazendo do esquema relacional um pobre veículo para comunicação
semântica de um banco de dados.
Com relação ao desempenho pode–se destacar que SGBDs
relacionais apresentam melhor desempenho em operações de
pesquisa para bases de dados de grande dimensão.
Modelagem - Modelo Orientado a
Objeto
Assim como o modelo relacional, o modelo orientado a objeto está
sendo bastante empregado para o tratamento de dados biológicos.
O INTERACT, por exemplo, um banco de dados sobre interações de
proteína, utiliza o SGBD orientado a objetos Poet.
Outros bancos de dados como o PSD/PIR, um banco de seqüências
de proteínas também foram implementados usando um SGBD
orientado a objetos.
Modelagem - Modelo Orientado a
Objeto
No modelo orientado a objeto os dados são abstraídos e armazenados como
objetos, possuindo estruturas com tipos pré-definidos. Sistemas orientados a
objeto são melhores quando o esquema é complexo, o dado irregular e as
consulta correlatas, sendo mais fácil pesquisar nas vizinhanças.
Uma vantagem de armazenar dados em um SGBD orientado a objeto é que ele é
capaz de proporcionar uma melhor performance para dados complexos (ex:
dados biológicos) como gráficos complicados, de 10-1000% comparado com o
padrão de banco de dados relacional.
Diferentemente do modelo relacional, modelos orientados a objeto são
extensíveis, contudo enfatizam o comportamento de objetos e insistem que cada
objeto tem sua própria identidade.
Modelagem - Modelo Orientado a
Objeto
A extensibilidade de sistemas de bancos de dados baseados em orientação a
objeto também nos permite incorporar operações sobre os dados diretamente
nas descrições de classe do objeto no banco de dados, deste modo escondendo
os detalhes de implementação do usuário e permitindo ser usado diretamente
com a linguagem de consulta do banco de dados.
A principal força do modelo orientado a objeto é seu poder de modelagem de
dados altamente flexível, oferecendo uma elegante maneira de representação de
objetos genômicos complexos. A principal fraqueza inclui a falta de uma forma
genérica de acesso aos objetos complexos.
A orientação a objeto permite ainda mais concisão e facilidade para entender
definições do banco de dados quando comparados com construtores de bancos
de dados relacionais.
A experiência tem mostrado que este tipo de
modelagem permite criar esquemas que são mais intuitivos para biólogos
moleculares compreender se comparados com os esquemas relacionais.
Modelagem - Modelo Semi-Estruturado (XML)
Dados biológicos nem sempre são bem estruturados, muitas vezes se mostram
incompletos, irregulares, redundantes ou contem erros. A maioria deles são
implicitamente estruturados. Portanto, dados da biologia molecular são bons
candidatos para um modelo de dados semi-estruturado.
No modelo semi-estruturado, o esquema é definido dinamicamente através dos
dados (auto-descritivo), apresentando uma descrição flexível de dados com
relacionamentos complexos.
A natureza auto-descritiva de XML a torna uma forma promissora para definição
de dados semi-estruturados.
XML é altamente flexível. Atualizar um modelo de dados é, portanto, simples.
XML é orientado a Internet e tem grande capacidade para vincular dados,
podendo ser utilizado para interconectar bancos de dados.
Modelagem - Modelo Semi-Estruturado (XML)
XML proporciona uma área aberta para definir especificações padronizadas.
Ponto importante, pois claramente há falta de padronização na bioinformática.
O custo de um formato baseado em texto na analise de dados, armazenamento e
transmissão precisam ser avaliados antes de adotar XML como uma solução
geral. Contudo, um formato texto significa que o código fonte pode ser lido e
editado com um editor de texto.
A expressividade de um modelo de dados XML provavelmente não será
suficiente para a biologia molecular. A semântica de dados biológicos é bastante
rica e requer um modelo de dados bastante expressiva.
XML não tem mecanismos de herança e nem métodos em objetos. O conceito de
relacionamento pode ser imitado através de referências lentas, mas não existe
como tal.
Apenas restrições de unicidade, cardinalidade e não nulo podem ser
especificadas: não há simetria, nem restrições elaboradas e nem ‘triggers’;
XML não tem suporte para valores numéricos, tabelas e matrizes.
Desafios
Armazenar dados de sequenciamento, experimentos e
resultado das anotações (conhecimento biológico).
Projetar banco de forma eficiente para facilitar recuperação de
informações.
Disponibilizar acesso às informações (ferramentas)
• Interface com usuário: intuitiva, fácil manipulação
• Flexibilidade de acesso: tipos de informação que podem ser
recuperadas (consultas)
• Flexibilidade de visualização: formato de apresentação dos
dados (texto, gráfico, links)
Desafios
Integrar dados heterogêneos
• Integração física (bancos distribuídos geograficamente)
o
Tecnologias de integração de dados (web services,
DAS)
• Integração conceitual (semântica)
o
Ontologias (bases de conhecimento)
Desafios
Diferentes bases de dados para armazenamento de dados
biológicos:
• arquivos em formato de texto
• arquivos estruturados
• bancos de dados relacionais
• bancos de dados objeto-relacionais
• bancos de dados orientado a objetos
Ontologias
Desafios
Muitas bases de dados foram construídas por biólogos.
• Não padronização da taxonomia
• Dificuldade na adoção de um vocabulário comum entre
os grupos de pesquisa
• Termos diferentes para conceitos iguais
• Conceitos diferentes para termos iguais
Desafios
Qualidade dos dados disponíveis na Web
• Grupos de pesquisa submetem suas descobertas
• Algumas bases aceitam de qualquer entrada
• Muitas bases não verificam a qualidade dos dados
• Outras bases preocupam-se com a qualidade dos
dados, onde um comitê valida-os. Estas bases ganham
destaque da comunidade científica.
Buscas
Muitos tipos de seqüências de entrada
• Pode ser uma seqüência de aminoácido ou de nucleotídeo
• Genômica, cDNA/mRNA, proteína
• Completa ou fragmentada
Matches exatos são raros
• Em geral, o objetivo é recuperar um conjunto de seqüências
similares
Buscas
O que queremos saber sobre a seqüência?
• Ela é similar ao algum gene conhecido? Quão próximo é o
melhor match? Significância?
• O que sabemos sobre este gene?
o Genômica(localização no cromossomo, regiões
reguladoras, ...)
o Estrutural (estrutura conhecida? ...)
o Funcional (molecular, celular e doença)
•Informação evolutiva
o Este gene é encontrado em outros organismos?
o Qual é sua árvore taxonômica?
NCBI - National Center for Biotechnology
Information - GenBank
NCBI disponibiliza o banco de sequência de DNA GenBank desde
1992. O GenBank tem acesso aberto e possui uma coleção de todas
as sequências de nucleotídios disponíveis publicamente e suas
proteínas traduzidas.
• Recebe sequências prodozidas em laboratórios de todo o mundo
de mais de 100,000 organismos.
• Cresce a ritmo exponencial, duplicando sua base a cada 18 meses.
• Em agosto de 2006 possuía mais de 65 milhões de bases de
nucleotídios em mais de 61 milhões de sequências.
Se coordena com laboratórios individuais e outros bancos de
sequências como European Molecular Biology Laboratory (EMBL) e
DNA Database of Japan (DDBJ).
Basic Local Alignment Search Tool (BLAST)
Algoritmo de comparação de sequências biológicas primárias
(aminoácidos e nucleotídios)
Permite ao pesquisador comparar uma sequência com outras
disponíveis na base de dados e idientificar as parecidas com
um determinado grau de confiabilidade.
•
•
•
•
Algorítmo de busca de sequências genéticas
Cria lista de partes da sequência de busca
Pesquisa a base de dados
Calcula semelhanças
Protein Data Bank (PDB) & Fasta
PDB:
• Banco de dados que compõe o projeto NCBI.
• É um repositório de informações estruturais 3-D de grandes
moléculas biológicas como preoteínas e ácidos nucléicos.
• Trabalho com o MMDB (Molecular Modeling DB) .
Fasta
• Formato de texto que representa sequências de
nucleotídios ou peptídios, em que pares base ou
aminoácidos são representados usando letras.
Exemplo
Influenzavirus A
• gripe aviária
• gripe suína
o depende da espécie do hospedeiro
Neuraminidase
• enzima presente na superfície dos vírus influenza que
permite ao vírus ser liberado da célula hospedeira
Exemplo
Links:
http://en.wikipedia.org/wiki/Influenzavirus_A
http://en.wikipedia.org/wiki/Viral_neuraminidase
Exemplo
Influenza Research Database
http://www.biohealthbase.org/GSearch/fluSegmentDetails.do?ncbiGenomicAcces
sion=FJ998214&decorator=influenza
NCBI - Nucleotide Database
http://www.ncbi.nlm.nih.gov/nuccore/229484043?report=genbank
NCBI - Taxonomy Browser
http://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Info&id=6437
80
NCBI - Blast
*http://blast.ncbi.nlm.nih.gov/Blast.cgi*
NCBI - PDB - MMDB
http://www.ncbi.nlm.nih.gov/Structure/mmdb/mmdbsrv.cgi?uid=67153
Influenza Research Database
Através de palavras-chave pesquisa-se:
• Segmentos de Nucleotídios
• Proteínas
• Tipos de Influenzas
o tipos de hospedeiro
o país
o data de adição
o e outros.
Pesquisando Neuraminidase em tipo s de influenza, obtêm-se mais de
10 mil resultados, e arbitrariamente seleciona-se um caso mexicano de
2009 com um hospedeiro humano.
Atributos (taxonomia, segmento...) possuem IDs que "linkam"
diretamente outras bases usadas na consulta. (NCBI, UniProtKB,
GBrowse, TARGET, IEDB)
NCBI - Nucleotide - Taxonomy
Através do ID de segmento (FJ998214) se acessa página do
mesmo na base de Nucleotídios do NCBI.
• autores, referências, sequência do segmento, tradução
Fasta.
Através do ID de taxonomia (643780) se acessa a página da
espécie (Influenza A virus (A/Mexico/InDRE4487/2009(H1N1)))
na base de Taxonomia do NCBI.
Na página de segmento já há um link para usar o Blast e fazer
comparação do segmento genético em outras espécies sem
precisar copiar e colar a sequência Fasta.
NCBI - Blast - PDB - MMDB
Através de múltiplas consultas como resultado são
apresentadas dezenas de variações de vírus que possuem a
mesma sequência exata (100%) e aproximadas (99%); toas
com seus IDs linkados para continuar a pesquisa no NCBI.
No mesmo portal há ferramentas de visualização 3D das
estruturas dos vírus, o formato pode ser baixado e visualizado
com o uso de um software (Cn3D) na máquina do usuário.
Toda a navegação é intuitiva feita a partir de links e IDs e as
bases funcionam de forma interligada.
Bibliografia
http://www.icb.ufmg.br/biq/maira/biodbfiles/aula1-introducaobioinfo.pdf
http://biotec.icb.ufmg.br/cabi/aulas/aula21.pdf
http://www.inf.ufrgs.br/~clesio/cmp151/cmp15120031/BDsBiologicos.pdf
http://en.wikipedia.org/wiki/National_Center_for_Biotechnology_Information
http://en.wikipedia.org/wiki/BLAST
http://en.wikipedia.org/wiki/Protein_data_bank#Viewing_the_data
http://en.wikipedia.org/wiki/FASTA_format
http://pt.wikipedia.org/wiki/Gene
Bibliografia
http://pt.wikipedia.org/wiki/Gen%C3%B3tipo
http://pt.wikipedia.org/wiki/Fen%C3%B3tipo
http://www2.dc.uel.br/nourau/document/?view=127