7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
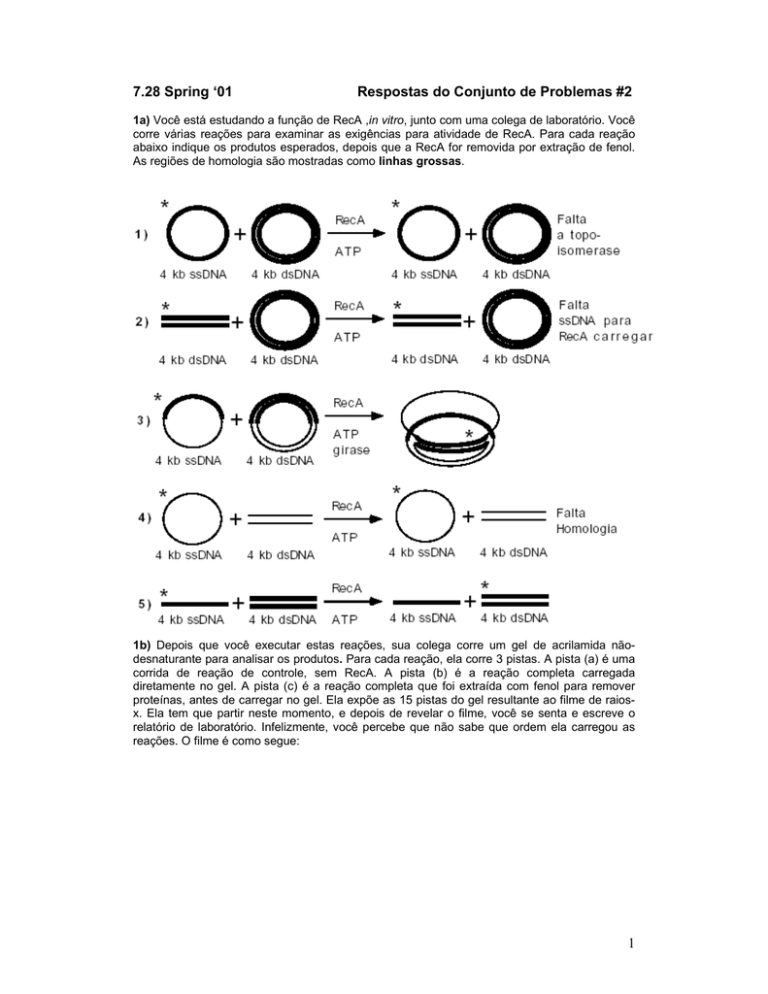
1a) Você está estudando a função de RecA ,in vitro, junto com uma colega de laboratório. Você
corre várias reações para examinar as exigências para atividade de RecA. Para cada reação
abaixo indique os produtos esperados, depois que a RecA for removida por extração de fenol.
As regiões de homologia são mostradas como linhas grossas.
1b) Depois que você executar estas reações, sua colega corre um gel de acrilamida nãodesnaturante para analisar os produtos. Para cada reação, ela corre 3 pistas. A pista (a) é uma
corrida de reação de controle, sem RecA. A pista (b) é a reação completa carregada
diretamente no gel. A pista (c) é a reação completa que foi extraída com fenol para remover
proteínas, antes de carregar no gel. Ela expõe as 15 pistas do gel resultante ao filme de raiosx. Ela tem que partir neste momento, e depois de revelar o filme, você se senta e escreve o
relatório de laboratório. Infelizmente, você percebe que não sabe que ordem ela carregou as
reações. O filme é como segue:
1
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
Quais números das reações (1-5) correspondem às reações no filme (U-Z)? Explique as
diferenças entre as pistas a, b, e c para cada reação.
Reação U = 3, W = 5, X = 2, e Y ou Z = 1 ou 4 (mesmas bandas no gel)
1 & 4 a: Estas têm o mesmo tamanho do ssDNA circular, aparentemente corre a 3 kb.
b: A recA liga ambas as moléculas de DNA, forma uma banda para 2 DNAs + troca
de proteína (8kb).
c: Os componentes faltando indicados em (a), assim nenhum produto de DNA
estável novo é formado (3 kb)
2
a: dsDNA linear radioativo de 4 kb.
b e c: Nenhum ssDNA, a recA não pode se ligar, mesmo tamanho observado (4 kb).
3
a: mesma molécula de DNA radioativo como 1 e 4 (3 kb).
b: recA liga e forma produto de DNA novo que combina os 2 DNAs, permanece
ligada (8 kb)
c: Produto final estável sem recA combina dois substratos de DNA (6 kb)
5
a: DNA quente inicial é ssDNA linear, aparentemente, corre mais rápido que o
circular (2kb)
b: recA liga e forma produto de DNA novo que combina os 2 DNAs, permanece
ligada (8 kb)
c: produto radioativo final é justamente dsDNA de 4 kb.
2
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
2a) Alguns elementos móveis (transposable) em E. coli, por exemplo, Tn10, transpõem a uma
frequência alta, imediatamente, depois que a forquilha de replicação passa. Esta indução de
transposição, através da replicação, é um grande efeito, de forma que a maioria dos eventos de
transposição acontece em um período curto de tempo, logo depois da replicação. Que
mecanismo o Tn10 poderia usar para acoplar a transposição à passagem da forquilha de
replicação? Proponha um experimento simples, in vivo, para testar se este mecanismo é
verdadeiro.
A transposição poderia ser acoplada à replicação pelo mesmo sistema dam - dependente
de metilação de DNA, que acopla o reparo de pareamento incorreto à replicação. Neste
caso, a transposição seria induzida em transposons que contém sequências metiladas
pela metade. [De fato, a transposase de Tn10 não se liga ao DNA metilado
completamente, mas se liga ao DNA metilado parcialmente, ou não metilado, desta forma
ativando a transposição depois da passagem da forquilha de replicação.] Um
experimento in vivo poderia envolver a comparação da frequência de transposição em
tipo selvagem, dam - e cepas dam superexpressando metilase. O Tn10, frequentemente,
deveria transpor mais que normal em células dam - (onde os sítios sempre deveriam ser
não metilados) e menos frequentemente que normal em superexpressadores dam (onde
os sítios são metilados mais rapidamente após a replicação). A frequência de
transposição poderia ser medida através de Southern blots com uma sonda de Tn10,
como descrito na parte b).
2b) Você tem uma cepa de E. coli (cepa 708A) que tem uma única cópia de Tn10 no genoma,
integrada no gene de hisB. Você isola um derivado desta cepa (o qual você nomeou 708A+) na
qual o Tn10 transpôs e causou uma mutação no gene requerido para crescimento na ausência
de leucina. Abaixo se encontra o Southern blot de DNA de uma cepa 708, que é relacionada a
708A, mas faltando Tn10, 708A e 708A+. A membrana foi sondada com 3 sondas radioativas
diferentes: (1) para o gene de hisB, (2) para o gene de leu e (3) para sequências de Tn10.
Dado o padrão mostrado para as outras cepas, esquematize as bandas você esperaria ver
para a cepa 708A+ em cada dos filmes (abaixo):
3
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
2c) Você declara, em reunião de grupo, que Tn10 é um transposon replicativo. Porém, seu
professor o informa isso baseado apenas na experiência executada na parte b), você não pode
dizer se Tn10 é um transposon replicativo ou não replicativo. Por que não? Explique
brevemente a consideração do seu professor.
Depois que a forquilha de replicação atravessa a sequência de Tn10, há duas cópias de
Tn10, temporariamente, na célula, no cromossomo “pai” e “filho” no mesmo local (o
gene His).
(1) se uma cópia transpõe por um mecanismo replicativo, a célula de progênie
resultante, leva dois transposons de Tn10 em dois sítios diferentes.
(2) se uma cópia do transposon excisa por um “corte e cola”, um processo não
replicativo, deixará para trás uma falha em dupla fita. Esta falha será reparada usando a
região homóloga no cromossomo oposto que contém a cópia, recentemente, replicada
da sequência de Tn10. Assim, os transposons serão achados também em ambos locais
velho e no novo sítio alvo na célula de progênie.
3a) Você está estudando o problema de resistência a antibióticos, que está se expandindo na
população bacteriana hospitalar. Você tem razão para suspeitar que alguns fagos,
previamente, não caracterizados estão causando o problema. Estes fagos levam genes de
resistência a antibióticos, levando, desta forma, as células infetadas a ficarem resistentes,
quando o fago se integra no DNA cromossomal da célula. Como um primeiro passo, tentando
prevenir a expansão adicional deste fago, você caracteriza o mecanismo deles de integração.
Para tentar adquirir uma compreensão melhor dos mecanismos que eles usam, você infecta
dois dos fagos em várias cepas de E. coli diferentes, com mutações em genes que efetuam
processo de DNA. Abaixo, uma tabela dos resultados.
Cepa de E. coli
Tipo selvagem
Fago A
Integra
Fago B
Integra
polA- (gene de DNA
polimerase I)
Não Integra
Integra
lig- (gene de DNA ligase)
Não Integra
Integra
recB- (gene de RecB)
Integra
Integra
Baseado nestes dados, que tipo de recombinação: homóloga, transposição ou sítio específica,
é, mais provável, ser usada para integração por um fago A ? Explique sua resposta.
A transposição. O fago A não requer a recB, assim não é recombinação homóloga.
Porém, ele requer polimerase e ligase que são necessárias para a transposição, mas não
para recombinação sítio específica.
4
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
3b) Qual tipo de recombinação: homóloga, transposição ou sítio específica, é mais provável
ser usada para integração por um fago B ? Explique sua resposta.
A recombinação sítio específica. O fago B não requer recB também, e assim não usa
recombinação homóloga. A recombinação sítio específica não requer qualquer síntese
de DNA novo, e assim não requer polimerase. A recombinase une as fitas partidas, de
forma que a ligase não é necessária também.
3c) Você decide que este método de determinar um mecanismo de integração é bastante
trabalho intensivo e indireto. Sugira um ensaio mais direto que deverá lhe permitir determinar
se o fago que você isolou usa o processo de integração usado pelo fago A ou aquele usado
pelo fago B. Você tem uma sonda de DNA, à sua disposição, para os genes de resistência a
antibióticos, levados por ambos os tipos de fago. Explique o ensaio, e o que você esperaria ver
com fago de cada classe.
Um ensaio simples seria fazer um Southern blot genômico nas cepas E. de coli
infectadas por cada fago. Isole o DNA de E.coli infectado com cada fago, digira com uma
enzima de restrição, e corra um Southern blot (corra em um gel de não desnaturante,
desnature o DNA, transfira para uma membrana, incube com uma sonda, e exponha ao
filme). A E. coli infectada pelo fago B sempre deverá produzir uma banda do mesmo
tamanho, quando sondada com o DNA do fago B, uma vez que a recombinação sítioespecífica sempre causa integração no mesmo lugar. A E. coli infectada com fago A
testada produz muitos bandas diferentes, quando sondada com o DNA do fago A, já que
os transposons transpõem em pontos randômicos do genoma.
3d) Para parar a expansão deste fago de resistência antibiótico, você quer isolar um inibidor do
processo de integração. Baseado em estudos com outros inibidores de enzima, você
racionaliza que o melhor objetivo para o inibidor seria o sítio ativo da enzima de recombinação,
responsável pela integração. Para identificar o sítio ativo na recombinase para fago B, você usa
uma abordagem de sequências consensos. Primeiro, você identifica uma região do genoma do
fago B que é fracamente homóloga ao gene da integrase do fago λ. Então você sequencia
esta região de cinco fagos que são todos os parentes próximos do fago B. Abaixo estão as
regiões de sequência de aminoácidos (código de 1 letra) destes seis fagos semelhantes a B.
Fago λ
B-001
B-002
B-003
B-004
B-005
IFGYARVSYSQE
LFGYARVSYSQE
LFGYARVSTSQQ
IAGWIRVSTFDQ
IAGYIRVSTFDQ
IAGYIRVSTFDQ
Baseado nestes dados de alinhamento, que aminoácido nesta região é provável formar parte
do sítio ativo? Dê duas razões para justificar sua resposta.
A serina conservada é provável parte do sítio ativo. Está presente em todo fago parente
de B, e logo deve ser importante. Também, a serina contém um grupo OH que é
necessário para atacar o fosfato do DNA e formar um intermediário de DNA-proteína
covalente.
5
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
4a) Um colega está estudando uma doença dominantemente herdada e encontra um paciente
sem história da doença na família dele. Depois de averiguar que o paciente realmente estava
correto sobre a identidade dos pais dele, seu colega contatou você, o perito em transposons.
Você sequencia o gene da doença do paciente, e acha uma inserção grande no gene. Que
características de sequência você poderia esperar esta inserção ter, para ser um transposon?
Repetições diretas curtas (duplicação de sítio alvo), repetições invertidas, genes com
homologia para transposases.
4b) Você decide testar se você achou um transposon novo realmente, conferindo se o
transposon pode se transferir entre DNAs diferentes em uma célula bacteriana. (Assuma que
os elementos humanos podem transpor em bactérias.) Sua UROP insere um gene AmpR na
sequência suspeito de transposon e a clona em um plasmídeo bacteriano, que contém um
gene TetR. A UROP transforma este plasmídeo em uma variante bacteriana que leva um
plasmídeo F' com um gene KanR. Então ela cresce estas células doadoras, as junta com uma
variante recipiente AmpS KanS TetS, mata as células doadoras, e plaqueia as bactérias
sobreviventes sobre várias placas com antibiótico, para testar o fenótipo das células
recipientes. 0,05% das células KanR que ela recupera são KanR AmpR TetS, enquanto as
restantes 99,5% são KanR AmpS TetS. O que pode concluir você destes dados?
Transposon “corte e cola”, transpondo a uma baixa taxa.
4c) Você acusa sua UROP de invalidar o ensaio, como você esperou uma porcentagem mais
alta de células AmpR. Ofendida, sua UROP repete a experiência em duplicata, desta vez
transformando diversas variantes bacterianas com plasmídeos F’ diferentes, todos contendo
genes KanR . Desta vez, ela acha que uma das variantes resultou em células recipiente nas
quais são 40% dos genes KanR são KanR AmpR. Você diz para ela sequenciar um plasmídeo F'
que ganhou uma inserção de transposon e descobre que ele tem um conteúdo de GC muito
alto. Dado que regiões do genoma humano que contém sequências codificantes têm um
conteúdo de GC muito mais alto que regiões que não fazem, por que é estranho que este
transposon pareça preferir integrar em DNA rico em GC? (Por que a maioria do transposons
pode integrar em regiões não codificantes?)
A maioria do transposons tem uma preferência para o DNA não codificante,
presumivelmente, de forma a poderem ser retidos no genoma sem, acidentalmente,
matar o hospedeiro.
4d) Qual é a explicação pela qual este transposon poderia preferir integrar em regiões
ativamente transcritas com estruturas de cromatina abertas? (Que tipo de transposon poderia
ser?)
Talvez seja um retro-transposon, e precisa ser transcrito em RNA antes de transpor. Os
retro-transposons, normalmente, têm os próprios promotores deles, mas poderiam
transpor, presumivelmente, com mais frequência se eles estiverem em regiões ativas do
genoma com estruturas de cromatina abertas.
6
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
4e) Como esta hipótese muda sua conclusão sobre os resultados na parte b?
Os retro-transposons transpõem replicativamente (eles deixam para trás uma cópia no
genoma do qual o RNA foi transcrito), mas eles não fazem uma co-integração. Então, no
ensaio bacteriano, eles não trariam o plasmídeo inteiro que contém o gene TetR com eles
para a célula recipiente. O que parecia uma transposição “corta e cola” no seu ensaio foi
de fato transposição replicativa.
4f) Como este transposon poderia alcançar esta preferência para DNA rico em GC?
A transposase poderia especificamente reconhecer uma sequência curta rica em GC no
DNA alvo.
5) Você está interessado em estudar a regulação de genes em camundongos, importante
durante desenvolvimento precoce. Seu professor lhe pede que continue o trabalho de um
estudante graduado anterior. Este estudante estava estudando dois genes novos que ele
nomeou Hip e Hop. Hip e Hop são necessários, precocemente em embriões, mas não depois.
O estudante graduado pôde gerar o mapa de restrição seguinte para Hip e Hop:
Você decide executar um Southern blot em Hip e Hop. Sabendo que estes genes são
importantes durante desenvolvimento precoce, você isola o DNA genômico de embriões e
também de adultos jovens. Você digere com HindIII e acha os resultados seguintes:
5a) Você está surpreso pelos resultados. Como você pode explicar o resultado visto quando
sondando seu Southern blot com Hop? Dada sua explicação, o que você pode concluir sobre a
regulação de Hop durante desenvolvimento de camundongos? Hop é necessário em
camundongos adultos, ou só no desenvolvimento embrionário?
O gene HOP só é importante, durante desenvolvimento embrionário. A falta de uma
banda ao sondar o DNA adulto indica que o gene de HOP não está mais presente em
camundongos adultos. É provável que uma recombinação sítio específica seja usada
para excisar este gene, depois que ele não for mais necessário para desenvolvimento.
Este tipo de regulação só é possível em células que são terminalmente diferenciadas.
7
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
5b) Você gostaria de investigar mais a regulação de Hip. Em uma tentativa para fazê-lo, você
digere o DNA que você isolou acima com duas enzimas, HindIII e EcoRI. Desta vez, você vê os
seguintes resultados:
Dados estes novos dados, como você pensa que Hip é regulado durante desenvolvimento do
camundongo? Forneça uma explicação a partir dos dados acima.
Neste caso, a recombinação sítio específica também está sendo usada para regular o
gene HIP. Porém, em lugar de um evento de excisão, uma inversão aconteceu. Isto pode
ser visto pela mudança nos tamanhos de fragmentos de restrição no Southern blot. O
local assimétrico do sítio de EcoRI produz dois padrões distintos antes e depois do
evento de inversão.
5c) Usando as mesmas cepas de camundongos que você está estudando, uma colega do
laboratório criou camundongos mutantes que não desenvolvem corretamente. Um dos
mutantes dela mostra múltiplas características embrionárias, até mesmo quando o
camundongo alcançou a fase adulta. Ela lhe pede que execute sua caracterização de Southern
blot nos camundongos mutante dela ver se HIP ou HOP são mutantes na cepa dela. Você está
contente de fazer isto e digere o DNA genômico dos camundongos dela com HindIII e EcoRI.
Você acha os seguintes resultados:
8
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
A partir dos dados acima, qual gene você pensa é modificado nos camundongos da sua
colega? Como você pensa que a regulação deste gene é modificada para dar as
características de mutante descritas acima?
O gene HIP parece ser modificado nas cepas da sua colega. O gene normalmente é
regulado por um evento de inversão. O padrão de restrição no gel acima sugere que
algumas células não sofreram este evento de inversão. As duas bandas medianas são
geradas a partir de células onde HIP é regulado corretamente, considerando que as duas
bandas externas são geradas a partir de células que não invertem o gene de HIP.
6) Você desenvolveu um ensaio de transcrição que recapitula a regulação in vivo do promotor
da RNA Pol II transcrita do gene Ratchet. Através do fracionamento deste extrato, você
purificou e clonou dois fatores de transcrição específicos que, em adição aos fatores de
transcrição comuns da RNA Pol II, são requeridos para regulação correta do promotor. Como
um primeiro passo para entender a função destes fatores, você faz uma série de mutantes de
deleção do promotor de Ratchet e testa o efeito deles na transcrição, na presença e ausência
de fatores Click e Clack. Você acha os resultados seguintes.
6a) Baseado nestes dados, o que você preveria como sendo a função de elementos 3, 6, e 7?
O elemento 3 é mais provavelmente um sítio ativador de ligação. Considerando que este
elemento é requerido para ativação de transcripcional por Click e Clack, provavelmente é
o sítio de ligação para uma destas proteínas.
Os elementos 6 e 7 são, provavelmente, parte do elemento central do promotor,
possivelmente o TATA box, e a sequência de INR. Isto pode ser determinado a partir dos
locais deles relativos ao início da transcrição e pelo fato de que a eliminação destas
sequências dianteiras conduz à perda de níveis basais de transcrição.
9
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
Para caracterização adicional dos fatores que você identificou, você executa um ensaio de
alteração em gel, para ver a habilidade dos diferentes fatores para ligar ao promotor Ratchet.
Você obtém os seguintes resultados.
6b) Baseado nestes dados, o que pode concluir você relativo à função de Click e Clack? Como
este achado é consistente com os dados obteve na parte3a?
Isto mostra que Clack é uma proteína de ligação com o DNA, e que Click depende de
Clack para associação com o DNA. Isto é consistente com o fato de que só há um sítio
ativador de ligação, como determinado por sua análise na parte 3a. Considerando que
Click depende de Clack para associação com o DNA, o único sítio de ligação requerido é
o para o que Clack se liga. Se ambas as proteínas ligassem ao DNA, uma pessoa
observaria um sítio de ligação, provavelmente, para cada proteína.
6c) Como você determinaria a região do fator Clack requerida para ativação do promotor de
Ratchet?
Para mapear o domínio de ativação de Clack:
1.Fusione Clack ao domínio de ligação com DNA de uma proteína de ligação com DNA
conhecida (i.e. LexA). Isto lhe permite mapear domínios de ativação, independente de
efeitos na ligação com o DNA.
2.Faça uma série de deleções no fim de Clack, e fusione cada uma ao domínio de ligação
do LexA no DNA.
3.Teste a habilidade de cada fusão para ativar transcrição a partir um promotor teste.
Este promotor tem que conter sítios de ligação de LexA, acima na região do sítio de
iniciação da transcrição, e um gene repórter (como o LacZ) para medir a transcrição.
Esta técnica lhe permite determinar quais regiões da proteína de Clack são necessárias e
suficientes para ativação transcripcional.
* * IMPORTANTE: Se você respondeu a pergunta, explicando como mapear o domínio de
ligação de Clack no DNA, isto poderia levar a perda pontos em um exame. Muitas
proteínas têm *ambos * os domínios de ligação com DNA *e* domínios de ativação
transcripcional. Esteja seguro que você leu e respondeu a pergunta que é feita.
10
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
Ao longo de sua análise do promotor Ratchet você foi frustrado pela falta de efeito de um
terceiro fator de transcrição, Clock , que é conhecido por ativar este promotor in vivo. Todos
seus ensaios prévios para transcrição foram executados, usando análise com S1, e uma sonda
de ssDNA de 300 bases de comprimento, que sobrepõe o sítio iniciação de Ratchet por 100
bases.
Pensando que você pode ter perdido algo, você analisa os produtos de um conjunto de
reações de transcrição executados na presença de UTP, marcado radioativamente, em um gel
de agarose desnaturante. Para causar a terminação da RNA Pol II artificialmente, você cortou
o template de DNA 2 kb abaixo do promotor, com uma enzima de restrição. Você obtém os
resultados seguintes (sugestão: liberação do promotor é completada após 100 bases de
síntese de RNA).
6d) Que aspecto da função da RNA Pol II é efetivada pela adição de Clock? Por que isto efeito
foi perdido pela análise com S1 e detectado pelo ensaio de “escape” da transcrição?
O Clock serve como um fator de processabilidade para a RNA Pol II. Note que quando
Clock é adicionado, os transcritos aumentam em comprimento de ~300 bp para 2000 bp.
Este não é um efeito na liberação do promotor, uma vez que a liberação de promotor (e
portanto a iniciação da transcrição) está completo em 100 bp.
Este é um efeito no alongamento do novo transcrito. Outra resposta plausível é que
Clock atos atua como um regulador negativo de terminação de transcrição. Assim,
quando Clock estiver presente,os transcrito são mais longos que na sua ausência.
11
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
No ensaio S1, uma sonda de DNA de 200 bp marcada que é hibridizada com o transcrito
de RNA é a sua referência de leitura para transcrição. Uma banda que corresponde a
esta sonda 200 de bp será observada no auto-radiograma quando o RNA que é
hibridizado tem 200 bp ou 2000 bp de comprimento. Assim, em um ensaio de proteção
S1, nenhuma informação do comprimento do RNA é provida, e os resultados com e sem
proteína CLOCK seria o mesmo.
No ensaio de escape da transcrição, alguém mede incorporação de rNTPs. Os RNTPs
serão incorporados do sítio de iniciação até que a terminação aconteça (que dependerá
da processabilidade intrínseca da RNA Pol II sem CLOCK, e do RNA Pol II “escapando”
do DNA com CLOCK). Este ensaio lhe permite determinar a informação do comprimento
do RNA o, e assim lhe permitirá ver produtos diferentes com e sem a proteína CLOCK.
7) Seu laboratório estuda uma cepa especializada de E. coli que contém genes incomuns que
ajudam em seu crescimento sob de condições de baixos nutrientes. Você está examinando
melhor um gene que é induzido sob de condições de baixos nutrientes (starvation), o gene
funE. Você usou a análise de proteção de nuclease S1 para determinar o sítio de iniciação da
transcrição para o mRNA de funE. O sequência inclui regiões -10 e –35, que diferem
substancialmente das sequências consensos padrão para E . coli acima do sítio de iniciação.
Usando um sítio de restrição conveniente, você fusiona o promotor de funE com um gene
repórter de β-galactosidase em um plasmídeo para permitir o estudo detalhado da expressão
deste promotor.
7a) Você induz mutações nesta cepa e a transforma com seu plasmídeo repórter para achar
mutantes que afetam expressão da β-galactosidase. Um mutante, o mutante de craZ, tem uma
perda completa de indução de β atividade, sob de condições de baixos nutrientes. Você
examina a expressão de outros genes que seu laboratório estuda, que são induzidos em
condições de baixos nutrientes, e acha que estes genes também não são mais induzidos no
mutante de craZ. Dado que estes genes têm uma região -35 semelhante ao gene de funE,
proponha uma explicação para a natureza do mutante de craZ.
Relembre que a região -35 é primariamente envolvida na especificidade da ligação
holoenzima de RNA Polimerase. É provável que a mutação de craZ esteja em um fator
sigma especializado (σ) que reconhece as regiões -35 incomuns do gene de funE e
genes relacionados. A expressão ou atividade desta subunidade σ deve ser induzida sob
condições de baixos nutrientes.
12
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
7b) Você testa uma variedade de combinações para ver se elas aumentam a expressão da
fusão funE/β-galactosidase em células de tipo selvagem. Você acha que a adição de cálcio,
significativamente, aumenta o sinal de - β-gal. Para examinar este efeito no nível
transcripcional, você executa ensaios de incorporação primeiro em extratos celulares tipo
selvagem, adicionando o plasmídeo repórter e [H3]UTP. Você faz o ensaio na presença e
ausência de cálcio, então corre a reação em gel e expõe o gel ao filme. O filme resultante é
mostrado abaixo:
Este resultado sugere uma diferença em uma etapa limitante da taxa da iniciação
transcripcional, na presença e ausência de cálcio. O que é a etapa limitante mudada?
Para o ensaio de incorporação na ausência de cálcio, observa você que a maioria dos
produtos é de fragmentos curtos (~5 bp). Isto indica que os complexos ternários
formaram, mas a liberação de promotor não aconteceu. Para o ensaio de incorporação
na presença de cálcio, observa você que a maioria de produtos é grande (>300 bp),
presumivelmente transcritos completos. Então na ausência de cálcio, a liberação de
promotor deve ser a etapa limitante da taxa, mas na presença de cálcio este passo é feito
mais eficientemente e a transcrição não mais limitada.
7c) Usando fracionamento bioquímico e um ensaio de gel shift, seu colega isola uma proteína
que se liga acima da região –35 de funE. Ele determina a sequência do gene e nota que a
proteína tem dois domínios, um domínio de ligação com DNA e um domínio de ligação com
cálcio. Vocês dois concluem que é provável que esta proteína esteja mediando os efeitos que
você estudou em (b). Sugira DOIS possíveis mecanismos para como este fator poderia resultar
na transcrição dependente de Ca++. Que experiência genética você poderia executar para
distinguí-los?
A proteína ou poderia estar agindo como uma repressora da transcrição que é inativada
pela ligação com cálcio, ou como um ativador da transcrição que é ativado através da
ligação de cálcio.
++
Ca --| o Repressor--| FunE
OU
++
Ca →Ativator→FunE
Para distinguir entre estas possibilidades, você pode inativar o gene. Se a proteína for
uma repressora, a inativação será insensível ao cálcio e FunE será expresso
constitutivamente a um alto nível (o nível causado por cálcio no tipo selvagem). Se a
proteína for um ativador, a inativação insensível ao cálcio e FunE será expresso a um
nível mais baixo (o nível sem cálcio no tipo selvagem).
13
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
7d) Para o estudo adicional da transcrição a partir deste promotor, você induz mutações na
região promotora do seu plasmídeo repórter e procura efeitos na expressão de β-gal, na
presença de cálcio. Você obtém uma mutação na região -10, chamada silE, que aumenta a
expressão de β-gal, na presença de cálcio. Porém, esta mutação não tem nenhum efeito na
expressão de β-gal, na ausência de cálcio.
Você usa KMnO4 para examinar o desenrolamento do DNA nas reações de transcrição
executadas in vitro com fragmento de DNA dupla fita linear, marcado na extremidade, que leva
a fusão promotor de funE/ β-gal. O DNA substrato e o filme resultante são mostrados abaixo.
O que indicam estes resultados sobre a natureza da mutação silE? Por que a mutação silE
afeta só a expressão de β-gal, na presença de cálcio (Sugestão: considere também os dados
da parte (b))?
A mutação silE aumenta a extensão do DNA que desenrola e abre a formação do
complexo, talvez trazendo a sequência incomum de funE -10 para mais perto da região
consenso –10 de E. coli.
A expressão de β-gal do gene de fusão é determinada por uma etapa limitante
da taxa da iniciação. Na ausência de cálcio, liberação de promotor é a limitante da taxa.
Logo, o aumentar o desenrolamento do DNA e abrir a formação do complexo, na
ausência de cálcio não têm nenhum efeito na expressão de β-gal. Já na presença de
cálcio, a liberação do promotor não mais é limitante da taxa. A formação do complexo
aberto parece ser limitante da taxa sob estas condições, de forma que a mutação silE
que aumenta a extensão do DNA desenrolado (e então formação complexa aberta),
também aumenta a expressão de β-gal.
14
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
8) você está estudando um par de fatores de transcrição que regulam o promotor Pumped
(envolvido na produção de uma proteína músculo-específica). Você purificou ambos fatores e
testou a habilidade deles para ou regular a transcrição do promotor Pumped, sem ou com
mutações em vários elementos fundamentais do promotor.
8a) Baseado nos dados da transcrição acima, proponha papéis para os elementos A e B do
promotor Pumped. Seja breve.
O elemento A só é requerido para transcrição ativada. É provável que contenha uma
sequência (UAS) reconhecida por um ativador transcripcional de ligação com DNA,
sequência-específico. O elemento B é requerido para a transcrição basal e ativada. É
provável conter o TATA-box e/ou os elementos iniciadores reconhecidos pelos fatores
gerais da transcrição.
8b) Você esta intrigado pelos resultados da análise da transcrição, na qual ambos PTF1 e
PTF2 foram adicionados, ao mesmo tempo. Você hipotetisa que PTF2 poderia inibir a função
de PTF1, alterando suas propriedades de ligação com DNA. Para esclarecer isto, você executa
um experimento de gel shift, usando o promotor Pumped.
As experiências prévias argumentaram que PTF1 e PTF2 são dímeros, quando ligados ao
DNA. Com isso em mente, como você pode explicar os resultados deste ensaio de gel shift?
A PTF2 é uma proteína maior que PTF1, e o complexo DNA-proteína, que contém um
dímero de PTF2, corre mais lentamente que o que contém um dímero de PTF1. Quando
PTF1 e PTF2 estiverem presentes, um heterodímero que contém uma molécula de PTF1 e
uma molécula de PTF2 é formado. Este complexo DNA-proteína corre em um tamanho
intermediário entre os dois homodímeros.
15
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
8c) Tendo mapeado o domínio de ativação de PTF1, você agora pergunta que parte de PTF2 é
necessária para inibir função de PTF1. Você identifica três regiões como importantes. Duas
destas regiões são envolvidas na ligação com DNA e na função de dimerização de PTF2. A
terceira é um domínio separado, que não é requerido para ligação com DNA ou dimerização.
Proponha uma hipótese para a função deste terceiro domínio e descreva um modelo para
resultados de transcrição na parte (a).
O terceiro domínio é provavelmente responsável pela inibição da transcrição basal em
conjunção com o domínio de ativação de PTF1 (talvez por uma interação física de
proteína- proteína). (Note que o homodímero de PTF2 não inibe a transcrição basal)
MODELO: Normalmente, ambos PTF1 e PTF2 estão presentes em concentrações iguais
na célula. Eles formam, predominantemente, heterodímeros a estas concentrações, e
estes heterodímeros se ligam ao A-box no promotor. Os domínios inibição de PTF2 e de
ativação de PTF1 interagem para inibir a transcrição basal, até mesmo no promotor
Pumped.
Sob condições de ativação, a relação de PTF1 para PTF2 na célula muda de forma que
PTF1 predomina (por exemplo, quanto mais PTF1 é produzido, menos PTF2 é produzido,
ou PTF2 é degradado). Isto força a formação de homodímeros de PTF1 (em lugar de
heterodímeros), que se ligam ao A-box e ativam a transcrição no promotor Pumped,
através dos domínios de ativação.
[PTF1= [PTF2]
[PTF1] >> [PTF2]
OFF
ON
16
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
9) A sketch of the lac promoter region is shown below. Please fill in the information missing on
the chart below.
Condições
de cresc.
Atividade
de LacZ
Explicação
(se menos que atividade máxima)
Proteínas ligadas ao promotor
e a posição de cada uma.
Glicose +
IPTG–
-
reprimido por LacI
LacI ligada ao operador
Glicose +
IPTG +
-
não ativado por CAP
~ nada
Glicose–
IPTG–
-
CAP- cAMP promove a
formação do complexo
fechado e LacI promove a
formação do complexo
fechada, mas bloqueia a
formação do complexo
fechado.
CAP- cAMP se liga ao sítio
de CAP, a sigma70 -RNAP
se ligou a -35 e -10, e LacI
se ligou ao operador.
Glicose–
IPTG +
+
vai!
CAP- cAMP se ligando ao
sítio de CAP ativa o
promotor, sigma 70-RNAP
se ligará e rapidamente
libera a região do promotor.
cya IPTG +
-
Nenhum cAMP para ligar ao
CAP, logo CAP não se ligará
ao sítio de CAP e o promotor
não é ativado.
~ nada
cya - = um mutante no gene de adenilato ciclase.
A ativação CA-cAMP do promotor de Lac aumenta a transcrição em ~50 vezes, logo
assuma que a atividade de LacZ se refere a esta atividade altamente ativada (ativada
através de CAP e não reprimida por LacI).
17
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
10) Você está estudando operon bacteriano recentemente identificado, YGO. Parece ser
transcripcionalmente regulado por YGP e um produto de molécula pequena do operon, YGO.
Este é transcrito pela RNAP mais sigma 70.
a) Os resultados de análise genética da regulação do promotor que usa uma fusão de promotor
ao lacZ são mostrados abaixo. Interprete cada conjunto de resultados, preenchendo a coluna
de defeito molecular, e se a mutação é cis ou trans. Assuma em todos os casos que o repórter
lacZ não sofre mutação.
YGP Selv. + Pygo Selv. +
defeito molecular
tipo de mutação
+YGO+YGO+++
na
na
nenhum
Constitutiva
+++
+++/+++
+++/+++ defeito de operador (não pode
ligar o repressor YGP) (O segundo (cis)
operon que você adicionou para
checar cis x trans está reprimido,
mas também está fundido ao LacZ,
que já está sendo feito. Teria sido
esclarecido se fundido a um gene
diferente como luciferase, como
discutido em classe.)
genótipo YGO + YGO Tipo selv.
mutante 1
+++
mutante 2
-
-
/-
-/+++
mutante 3
+++
+++
-/+++
mutante 4
+++
+++
mutante 5
-
-
Defeito de promotor
Não indutível
(cis)
+++/+++
repressor YGP não se liga
ao +/- operador indutor
de YGO
Constitutiva
(trans
recessiva)
+++/+++
+++/+++
repressor mutante YGP
não reprime e bloqueia a
repressão do tipo selv.
Constitutiva
Não indutível,
dominante
negativa (trans)
-/-
-/-
repressor YGP se liga ao
operador independente
de indutor YGO e
não se libera.
Não indutível
(trans
dominante)
Note que os mutantes 1 e 4 têm os mesmos fenótipos, mas há duas possíveis mutações
que poderiam causar isto.
18
7.28 Spring ‘01
Respostas do Conjunto de Problemas #2
b) Você isola uma sexta mutação, que tem um fenótipo como o mutante 3 a não ser que se
você superexpressar a proteína mutante 6 (é uma mutação trans ) a repressão é observada
agora na presença de YGO. Como você pode explicar este achado? Que ensaio poderia fazer
você para testar sua hipótese e que resultado você esperaria?
YGP tipo selv. quando ligado por YGO poderia se ligar ao operador de uma maneira
cooperativa e a mutação 6 abole esta ligação cooperativa. A superexpressão poderia
suprimir o efeito da perda de cooperatividade e então poderia restabelecer a repressão.
Para testar esta hipótese, use um de ensaio gel shift do promotor de operon de YGO,
com concentrações crescentes de YGP selv. ou do mutante 6, na presença de YGO. O
resultado que você espera se YGP selv. se ligar ao operador cooperativamente, quando
ativado por YGO, e o mutante 6 não, é mostrado abaixo.
19