MECANISMOS DE REPARO
Eduardo Montagner Dias
Apesar de mutações genéticas serem de extrema importância para a
evolução de uma espécie, a sobrevivência do indivíduo depende da estabilidade
do seu genoma. A estabilidade resulta não só de um acurado mecanismo de
replicação, mas também de mecanismos que reparem os danos que ocorrem
continuadamente no DNA.
Entende-se por reparo a capacidade da maquinaria celular de corrigir os
erros causados por mutações. Muitos danos sofridos pelo DNA podem ser
reparados porque a informação genética é preservada em ambas as fitas da
dupla-hélice, de tal forma que a informação perdida em uma fita pode ser
recuperada a partir da fita complementar. Os mecanismos existentes e conhecidos
de reparação do DNA lesado são provavelmente universais e uma célula pode ter
vários sistemas capazes de atuar ao mesmo tempo no DNA lesado.
Como os sistemas de reparação são mais bem compreendidos e estudados
na Escherichia coli, muitos mecanismos discutidos farão referência a esta
bactéria.
Reparo por fotorreativação enzimática
Um dos mecanismos de reparo melhor estudados é a remoção dos dímeros
de pirimidina, formados pela exposição do DNA à luz ultravioleta. Este dímero não
se encaixa bem na estrutura de dupla-hélice e, assim, a replicação e a expressão
gênica são bloqueadas até que a lesão seja removida. Esse tipo de lesão pode ser
reparado de diferentes formas. A forma mais direta envolve enzimas que
simplesmente revertem a modificação química que originou o dano. Dímeros de
pirimidina são o alvo universal da enzima fotoliase, que se liga ao dímero e
catalisa uma segunda reação fotoquímica, desfazendo o anel formado pela luz UV
e refazendo as bases pirimídicas individuais. Esse processo é chamado
fotorreativação e envolve as seguintes etapas: inicialmente, a enzima reconhece e
liga-se ao dímero (mesmo na ausência de luz visível); depois, a absorção de luz
fornece energia para converter o dímero em monômero de pirimidina; por fim, a
enzima dissocia-se do DNA. A reação depende do gene que codifica a fotoliase e
ocorre especificamente em procariotos.
Reparo de bases alquiladas
A base nitrogenada primariamente afetada pelos agentes alquilantes é a
guanina. Na E. coli, esta lesão é removida pela enzima O6-metilguaninametiltransferase, que reconhece a alteração no DNA e remove o grupamento
metila causador da mutação. A mesma enzima também pode remover
grupamentos metila dos fosfatos que possivelmente interrompem a cadeia de
DNA. Uma característica importante dessa reação é que cada grupamento metila
removido consome uma enzima O6-metilguanina-metiltransferase, a qual não pode
ser recuperada para nova utilização.
Reparo por excisão de bases
Ocorre quando a remoção da base defeituosa é feita pela clivagem da
ligação base nitrogenada – desoxirribose, seguida pelo preenchimento da região
com a base correta por ação da DNA-polimerase. Um exemplo comum de reparo
por excisão de base é o que acontece na correção da desaminação da citosina à
uracila. O sistema de reparação reconhece a uracila como uma base estranha ao
DNA. Primeiramente, a uracila-DNA-glicosilase hidrolisa a ligação entre a uracila e
a molécula de desoxirribose. Nesse estágio, a cadeia de DNA está intacta, mas a
base é perdida. A enzima AP endonuclease reconhece, então, essa fenda e cliva
a cadeia em regiões adjacentes à base perdida. A DNA-polimerase insere
novamente uma citosina, de acordo com a orientação fornecida pela fita
complementar não-danificada, que contém uma guanina. Finalmente, a
integridade da fita corrigida é restaurada pela DNA-ligase. Existem outras
glicosilases que reconhecem e removem hipoxantina, 3-metil-adenina e purinas
com anel imidazol aberto por radiação ionizante ou pH elevado.
Reparo por excisão de nucleotídeos (REN)
Ocorre quando a remoção da base defeituosa é feita pela incisão
endonucleolítica nos dois lados da lesão, com liberação dos nucleotídeos, seguida
pelo preenchimento da região por ação da DNA-polimerase. Em todos os
organismos onde já foi estudado, o processo de REN consiste em cinco etapas:
1. Reconhecimento da lesão por um complexo multienzimático;
2. Incisão da fita anormal em ambos os lados da lesão a alguma distância
desta;
3. Excisão do segmento contendo a lesão;
4. Síntese de um novo segmento de DNA utilizando a fita não-danificada
como molde, por ação da DNA-polimerase;
5. Ligação/restauração da molécula de DNA por ação da enzima DNA-ligase.
A extensão média do fragmento de DNA removido é de 12 nucleotídeos,
razão pela qual esse modelo é chamado de reparação de regiões curtas. Em caso
de grandes quantidades de lesões, em que a substituição envolve de 1500 a 9000
nucleotídeos, o modelo é conhecido como reparação de regiões longas. A
diferença entre esses dois modelos de reparação é que o primeiro é uma função
constitutiva da célula, enquanto o segundo deve ser induzido por lesões no DNA –
pelo menos no que diz respeito à célula bacteriana.
O modelo proposto para o sistema de REN em mamíferos é o seguinte:
1. As proteínas XPB, XPD e XPA estão envolvidas na detecção da lesão,
sendo que as duas primeiras percorrem a hélice e a segunda reconhece a
lesão;
2. Após encontrar a lesão, o DNA do local do dano é desnaturado e as
endonucleases XPF e XPG fazem a dupla incisão na cadeia;
3. O oligonucleotídeo contendo a lesão é removido pelas DNAs-polimerases δ
e ε e pelos seus cofatores PCNA e RF-C, que também são responsáveis
pelo passo seguinte;
4. Ressíntese do DNA utilizando a fita não danificada como molde;
5. Ligação da extremidade 5’ da região recém-sintetizada à seqüência original,
pela DNA-ligase.
Outro exemplo muito interessante de REN é o sistema de reparação global
X genes ativos. Foi demonstrado em E. coli que, embora sítios distintos
contenham o mesmo tipo de lesão, o sistema pode diferenciar um gene ativo de
uma região não-codificadora; assim, os sítios críticos do genoma são os primeiros
a ser reparados. Em mamíferos, onde a quantidade de DNA presente no núcleo é
muito maior do que na E. coli, a reparação preferencial de seqüências transcritas
também ocorre.
Reparo de bases malpareadas
Algumas bases incorretamente pareadas conseguem escapar da revisão
realizada pela DNA-polimerase durante o processo de replicação. Por isso, na E.
coli, a etapa final que confere precisão ao processo de replicação é de
responsabilidade do sistema de correção de erro, que consiste em várias
proteínas codificadas pelos genes mut. Esse sistema percorre o DNA
recentemente sintetizado à procura de pares de base malpareadas e remove os
segmentos de fita simples contendo nucleotídeos incorretos. Isso permite que a
DNA-polimerase insira a base correta na lacuna formada. A dificuldade que surge,
aparentemente, é a de distinguir qual das bases de um par erroneamente formado
é a incorreta, porque ambas são componentes naturais do DNA. Se a remoção de
uma das bases fosse ao acaso, haveria a probabilidade de 50% de a base correta
ser removida e a mutação poderia ser perpetuada, ao invés de ser corrigida.
Existe, porém, um sinal específico que direciona o sistema de excisão do erro
exclusivamente para a fita recém-sintetizada: o reconhecimento de seqüências
GATC próximas ao erro. GATC metiladas indicam a fita parental, enquanto que
ausência de metilação nestas seqüências caracteriza a fita recém-sintetizada.
O sistema de correção detecta não somente pares únicos de bases
malpareadas, mas também adições e deleções, o que reduz a incidência de
mutações por modificação no módulo de leitura, além daquelas envolvendo
substituição de bases.
Reparo por recombinação
No processo de reparação por recombinação, a fita complementar nãodanificada é utilizada como molde na substituição do fragmento lesado. Algumas
vezes, porém, esse molde não está disponível, por motivos diversos. A DNApolimerase, então, é forçada a passar pela lesão sem replicar aquela região. Uma
lacuna com um tamanho considerável é deixada na fita recém-sintetizada. Toda a
informação do sítio danificado é perdida e somente pode ser recuperada pela
utilização de uma outra molécula idêntica de DNA.
Como as células de organismos superiores são geralmente diplóides, elas
possuem a mesma seqüência, ou praticamente a mesma, em cada um dos
cromossomos homólogos. Uma fonte apropriada de DNA, portanto, pode ser
encontrada na célula.
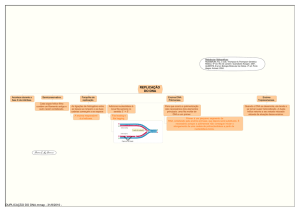
Assim, como esquematizado na figura abaixo, após uma mutação, a fita 1
do homólogo A (1A) não está disponível; a fita 2 do homólogo A (2A) possui uma
lacuna. As fitas 1 e 2 do homólogo A’ (1A’ e 2A’, respectivamente) estão íntegras.
No reparo por recombinação, a fita 1A’ é permutada pela fita 2A, de modo que 1A’
sirva de molde para reconstrução de 1A; e 2A’ sirva de molde para 2A.
Na E. coli, é de fundamental importância a proteína RecA, pois só ela é
capaz de parear duas moléculas de DNA homólogas e catalisar as reações de
trocas de fitas, permitindo reparação e recuperação de lesões.
Reparo através do sistema SOS
Uma vez que a célula pode regular a expressão gênica de acordo com sua
necessidade, muitas enzimas envolvidas no reparo do DNA são induzidas pelo
próprio defeito da molécula de DNA. As enzimas mais importantes desse processo
são derivadas dos genes SOS, cuja expressão é induzida por um defeito severo o
bastante a ponto de impedir a síntese de DNA. Estes genes dão origem a
proteínas de reparo como, por exemplo, endonucleases de excisão, helicases,
entre outras.
Os dímeros de pirimidinas constituem um exemplo típico desse tipo de
dano. Quando a forquilha de replicação encontra um dímero, a síntese pára e só é
reiniciada a alguma distância além do dímero, ficando uma lacuna no DNA na qual
a enzima de recombinação RecA se liga. Essa ligação ativa em RecA uma
atividade enzimática completamente independente da recombinação: ela destrói o
repressor dos genes SOS (repressor LexA). Dessa maneira, a proteína RecA
promove reparação do material genético de duas formas: por recombinação, com
troca das fitas, e por indução dos genes SOS.
Reparo sujeito a erro
Existem ocasiões em que o dano no DNA é tão extremo que não há como
os mecanismos celulares de reparo corrigirem de forma precisa a molécula.
Nesses casos, a célula utiliza como último recurso o sistema de reparo sujeito a
erro, no qual qualquer uma das quatro bases é inserida no local lesado, a fim de
garantir a continuidade do processo de replicação. Devido ao fato de não haver a
informação precisa (molde), o próprio mecanismo de reparo acaba sendo o
causador de uma mutação, pois a chance de introduzir uma base incorreta na
cadeia é grande. De qualquer forma, para a célula é preferível incorporar uma
base incorreta e permitir que a replicação continue, do que não replicar mais.
Na E. coli, esse sistema de reparação envolve dois genes principais: umuC
e umuD. Esses genes são também genes SOS e, portanto, seus produtos estão
em abundância somente após a célula ter o sistema SOS ativado. Devido a isso, a
proteína RecA também é necessária no sistema de reparo sujeito a erro, a fim de
comandar a indução dos genes umuC e umuD.
Reparo por união de extremidades não-homólogas
Quebras nas duas fitas de uma mesma molécula de DNA ocorrem nas
células em diversas circunstâncias e constituem uma das principais ameaças à
integridade do genoma. Se não forem reparadas, podem causar a morte celular e,
em organismos multicelulares, podem causar o câncer. Uma grande variedade de
agentes exógenos, incluindo a radiação ionizante e um número considerável de
quimioterápicos (como a bleomicina), causam essas quebras, bem como agentes
endógenos, a exemplo dos radicais livres. O principal mecanismo de reparação
dessas quebras duplas, que ocorre comumente em mamíferos, é chamado de
união de extremidades não-homólogas. Neste mecanismo, as extremidades de
uma molécula de DNA, apesar de terem perdido alguns nucleotídeos por
degradação espontânea, são justapostas para recombinar. O mesmo complexo
enzimático realiza o processo de ligação entre as duas extremidades. Desta
forma, a seqüência original de DNA acaba sendo alterada.
Reparo por união de extremidades homólogas
O segundo mecanismo reparador de quebras na dupla-fita é a união de
extremidades homólogas. O processo é bem mais comum em bactérias e
leveduras, e também mais preciso. Por este mecanismo, o cromossomo homólogo
àquele lesado faz uma cópia da seqüência de nucleotídeos perdida com quebra
da dupla-fita e transfere esta seqüência para o sítio de quebra no cromossomo
lesado; este cromossomo lesado, então, tem sua seqüência original restaurada.
Doenças Causadas por Deficiência de Reparo no Homem
DOENÇA
SENSIBILIDADE SUSCETIBILIDADE SINTOMAS
AO CÂNCER
Xeroderma
pigmentoso
UV, purina
alquilada
Ataxia
Radiação gama
teleangiectasia
Carcinoma de pele
Fotossensibilidade
de pele e olhos
Linfoma
Ataxia; dilatação
de vasos
sangüíneos na pele
e olhos; aberrações
cromossômicas.
Pancitopenia
hipoblástica;
anomalias
congênitas
Fotossensibilidade;
mudanças faciais
Anemia de
Fanconi
Agentes que fazem Leucemias
ligação cruzada
Síndrome de
Bloom
UV
Leucemias
ENZIMA
OU
PROCESSO
AFETADO
Reparo por
excisão de
nucleotídeos
Proteína
cinase ATM,
ativada por
quebra nas
duas fitas
Reparo de
DNA
cruzado
interfitas
DNAhelicase
acessória
para
replicação
Bibliografia:
• Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P. Molecular Biology of
the Cell. 4th edition, New York: Garland Science, 2002
• Zaha A, Ferreira HB, Passaglia LMP. Biologia Molecular Básica. 3ª edição, Porto
Alegre: Mercado Aberto, 2003.