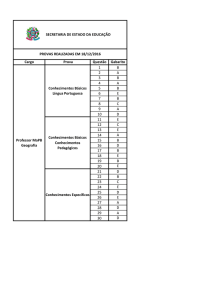
Boletim Técnico
ANO 4 | NÚMERO 29 | JUNHO DE 2016
Análise do sequenciamento do Exoma humano
Denomina-se Exoma o conjunto de éxons que compõem o
Estudos estimam um rendimento diagnóstico entre 16 e
material genético de um indivíduo, constituindo a porção
50% com a análise do Exoma (30% em média). Contudo, o
do genoma que efetivamente codifica proteínas. Em
restante dos dados está repleto de variantes de significado
humanos corresponde a 1% de todo o DNA, ou seja,
incerto ou desconhecido (Variant of Unclear Significance –
aproximadamente 22 mil genes do total da sequência
VUS) sendo, portanto, prudente a reanálise periódica.
genômica haploide (cerca de 30 megabases). Apesar de
Diferentemente da análise do genoma, o Exoma é capaz de
98% do DNA corresponder a genes não codificantes, as
detectar diminutas variações de cópia (p. ex. Indels), sendo
variações do Exoma estão relacionadas à maior parte dos
considerado atualmente a ferramenta com melhor
fenótipos
custo-benefício em casos em que testes genéticos
associados
a
doenças
Mendelianas
(ou
hereditárias).
padronizados falham ao estabelecer o diagnóstico de uma
doença potencialmente Mendeliana.
Um genoma diploide humano possui cerca de 3,5 milhões
de variações nucleotídicas (SNPs) e 1000 variações de
No entanto, é importante salientar que a análise das
números de cópias (Copy Number Variation – CNVs; >500 pb)
variações de número de cópias (por CGH-array ou
em relação a um genoma de referência. Aproximadamente
SNP-array)
20 a 25 mil destas variações estão envolvidas na
especialmente quando o fenótipo não é específico de uma
codificação de proteínas, sendo que em 10 mil há alteração
condição genética conhecida e quando a história familiar
de um aminoácido e destes, entre 50-100, formam
não indica um padrão de herança ligada ao X ou recessiva
proteínas truncadas ou perdas de variantes funcionais. Tais
(p. ex. pais consanguíneos). Além disto, diante da análise
estimativas são baseadas apenas em cerca de 5 a 10% dos
de um painel atualizado com resultado negativo, não é
genes reconhecidamente causadores de doenças.
mandatório a realização do Exoma, a menos que o clínico
deve
ser
considerada
antes
do WES,
tenha forte suspeita do seu diagnóstico genético ou tenha
Uma revisão manual deste grande número de variações
ocorrido alterações no fenótipo do paciente.
torna-se impraticável, sendo fundamental o auxílio da
informática e estatística para a seleção das variações de maior
Para a solicitação do Exoma, além do conhecimento clínico
impacto sobre o indivíduo. Diante disso, o sequenciamento
e genético, é necessário reconhecer características como a
completo do Exoma (Whole Exome Sequencing – WES) foi
adequação, acessibilidade, utilidade e aceitabilidade do
escolhido como técnica a ser utilizada para decodificar este
paciente e dos familiares com relação ao exame. Sugere-se
emaranhado químico e converter em informação linear,
que o clínico possua os conhecimentos básicos na
possibilitando a interpretação destas sequências de forma a
avaliação genética tanto do paciente quanto do teste,
estabelecer quais podem ser patogênicas, benignas ou
saber interpretar cada alteração independente da
fatores de proteção.
pré-interpretação laboratorial ou análises automatizadas e
prestar o aconselhamento genético adequado para tais
validação biológica ou estatística da alteração encontrada.
alterações. Portanto, é fundamental o aconselhamento
Este é o caso em que ocorre segregação de uma variante
genético pré-teste com assinatura do termo de
em uma família ou em três ou mais indivíduos com as
consentimento esclarecido por um médico geneticista ou
mesmas características.
clínico reconhecidamente habilitado.
A constante atualização do clínico sobre o registro de
Atualmente, a utilização clínica do Exoma tem se
variações nas bases de dados doença-específica e o
restringido por divulgação de resultados de variações
conhecimento dos catálogos de ontologia clínica e gênica,
patogênicas já conhecidas ou em genes essencialmente
auxiliam na busca ativa de informações. Quando
constituídos por uma condição genética. A utilização desta
compartilhadas, corroboram com outros profissionais e
ferramenta como uma prática de pesquisa para novos
pacientes na sua jornada através da busca de um
genes ou variações de significado incerto não tem sido
diagnóstico fidedigno, especialmente quando se trata de
amplamente aplicada na clínica por causa da escassez de
doenças ou variações raras.
OLIVEIRA, Guilherme de Macêdo
Residência em Genética Médica - Instituto Fernandes Figueira - Fiocruz
Mestre em Medicina - Instituto Oswaldo Cruz - Fiocruz
Graduado em Medicina - Faculdade de Medicina de Petrópolis
Experiência nas áreas de Genética Clínica e Genômica
Atua como consultor em Genética Médica e Sequenciamento de Nova Geração
Referências Bibliográficas:
1. Gonzaga-Jauregui C, Lupski JR, Gibbs RA. Human genome sequencing in health and disease.Annu Rev Med. 2012;63:35-61. doi:
10.1146/annurev-med-051010-162644.
2. Volk A, Conboy E, Wical B, Patterson M, Kirmani S. Whole-Exome Sequencing in the Clinic: Lessons from Six Consecutive Cases from the Clinician's
Perspective. MolSyndromol. 2015 Feb;6(1):23-31. doi: 10.1159/000371598. Epub 2015 Feb 3.
3. Smith LA, Douglas J, Braxton AA, Kramer K. Reporting Incidental Findings in Clinical Whole Exome Sequencing: Incorporation of the 2013 ACMG
Recommendations into Current Practices of Genetic Counseling. J Genet Couns. 2015 Aug;24(4):654-62.
4. Delaney SK, Hultner ML, Jacob HJ, Ledbetter DH, McCarthy JJ, Ball M, Beckman KB, Belmont JW, Bloss CS, Christman MF, Cosgrove A, Damiani SA, Danis T,
Delledonne M, Dougherty MJ, Dudley JT, Faucett WA, Friedman JR, Haase DH, Hays TS, Heilsberg S, Huber J, Kaminsky L, Ledbetter N, Lee WH, Levin E, Libiger
O, Linderman M, Love RL, Magnus DC, Martland A, McClure SL, Megill SE, Messier H, Nussbaum RL, Palaniappan L, Patay BA, Popovich BW, Quackenbush J,
Savant MJ, Su MM, Terry SF, Tucker S, Wong WT, Green RC. Toward clinical genomics in everyday medicine: perspectives and recommendations. Expert Rev
MolDiagn. 2016 May;16(5):521-32. doi: 10.1586/14737159.2016.1146593. Epub 2016 Feb 24.
5. O'Donnell-Luria AH, Miller DT.A Clinician's perspective on clinical exome sequencing. Hum Genet. 2016 Jun;135(6):643-54. doi: 10.1007/s00439-016-1662-x.
Epub 2016 Apr 28.
6. Goldfeder RL, Priest JR, Zook JM, Grove ME, Waggott D, Wheeler MT, Salit M, Ashley EA. Medical implications of technical accuracy in genome sequencing.
Genome Med. 2016 Mar 2;8(1):24.
7. Bowdin S, Gilbert A, Bedoukian E, Carew C, Adam MP, Belmont J, Bernhardt B, Biesecker L, Bjornsson HT, Blitzer M, D'Alessandro LC, Deardorff MA, Demmer
L, Elliott A, Feldman GL, Glass IA, Herman G, Hindorff L, Hisama F, Hudgins L, Innes AM, Jackson L, Jarvik G, Kim R, Korf B, Ledbetter DH, Li M, Liston E, Marshall
C, Medne L, Meyn MS, Monfared N, Morton C, Mulvihill JJ, Plon SE, Rehm H, Roberts A, Shuman C, Spinner NB, Stavropoulos DJ, Valverde K, Waggoner DJ,
Wilkens A, Cohn RD, Krantz ID. Recommendations for the integration of genomics into clinical practice. Genet Med. 2016 May 12.
Grupo Hermes Pardini
Rua Aimorés, 66, Funcionários, Belo Horizonte/MG. Cep: 30140-070 · Tel.: (31) 3228-6200.
Acesse www.portalmedicopardini.com.br para baixar este e os demais Boletins Técnicos.
Responsável Técnico: Ariovaldo Mendonça - CRMMG 33477 / Inscrição CRM 356 - MG